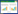扩展功能
文章信息
- 李晓亮, 蔡明明, 李阳, 张培玉, 林学政
- LI Xiao-Liang, CAI Ming-Ming, LI Yang, ZHANG Pei-Yu, LIN Xue-Zheng
- 第六次北极科学考察海洋沉积物可培养细菌的多样性分析
- Biodiversity analysis of cultivable bacteria isolated from marine sediments collected during 6th Arctic Expedition
- 微生物学通报, 2016, 43(5): 974-983
- Microbiology China, 2016, 43(5): 974-983
- DOI: 10.13344/j.microbiol.china.150875
-
文章历史
- 收稿日期: 2015-11-04
- 接受日期: 2015-12-23
- 优先数字出版日期(www.cnki.net): 2016-01-04
2. 国家海洋局第一海洋研究所国家海洋局海洋生物活性物质重点实验室 山东 青岛 266061
2. Key Laboratory of Marine Bioactive Substances SOA, the First Institute of Oceanography, SOA, Qingdao, Shandong 266061, China
由于地理位置相对封闭、受人类活动的影响较小、以及对环境变化敏感等特性,使极地成为研究地球环境变化和认识探索生命起源和生命机理的最佳场所[1]。极地气候条件恶劣、光照辐射变化极大、常年有6级以上的大风,为低温高盐环境,如此酷寒、强辐射的自然环境造就了极地微生物特殊的生物学特征,使其在环境适应性机制研究及生物活性物质的研发方面具有广阔的前景,被认为是一个潜在、重要的微生物资源库[2]。其中,由于长时间的沉淀和积累,使得极地海洋沉积物成为一个成分复杂且巨大的微生物栖息地。海洋沉积物中有机质含量是海水的104−105倍,充足的营养物质蕴藏了大量的微生物,并且这些微生物往往具有适应其特殊环境的形态学、生理学上的特异性[3]。以主要温室气体之一的甲烷为例,约90%的海洋甲烷循环过程发生在深海沉积物中,海底沉积物产生的甲烷含碳总量比地球表面的生物及陆地泥土产生的甲烷含碳量的总和要高4−8倍,而微生物在深海甲烷的生成与消耗过程中起着非常重要的作用[4]。研究海洋沉积物的微生物多样性不仅可以为开发利用微生物资源提供重要的菌种资源,还可以为人类了解生命起源和进化提供重要的线索[5]。我国对于极地微生物的研究起步较晚,但近年来发展迅速。2014年7−9月我国进行了第六次北极科学考察,对北极海域(白令海和楚科奇海)海洋沉积物进行了大面积调查和样品的采集工作。本文通过对海洋沉积物进行涂布培养、分离纯化及分子鉴定与系统发育分析,分离获得并发现了大量新的细菌菌种资源,这对于了解北极海域海洋沉积物微生物的多样性、发现新的微生物菌种资源、开发利用其特殊的功能基因和生物活性物质具有重要意义。
1 材料与方法 1.1 样品北极海域海洋沉积物样品为2014年7−9月中国第六次北极科学考察采集,对箱式采样器和重力采样器采取的海洋沉积物表层样品,用无菌药匙采集后置于无菌封口袋中,于4 °C低温保存,回实验室后进行可培养细菌的分离纯化与鉴定。
采样区域包括白令海和阿留申群岛周边海域在内的北太平洋北极边缘海,沿我国以往历次北极科考白令海西侧航线、阿姆奇卡特岛-努尼瓦克岛之间连线设置一条南北横跨白令海西部及中部海域的B断面,沿白令海峡南部常规测线设置调查断面BS,在圣劳伦斯岛两次分别与俄罗斯和阿拉斯加之间设置NB断面;在本海域内,共对13个站位的沉积物样品进行了可培养细菌的分离纯化与多样性分析。白令海峡以北的楚科奇海,以横断北极主要流系为原则,设置7个常规作业断面;其中楚科奇海陆架区设置3个常规作业断面,分别是R0断面、CC断面和C0断面;楚科奇海台和加拿大海盆区设置4个作业断面,分别为R1断面、S0断面、C1断面和C2断面;在本海域内,共对 27个站位的沉积物样品进行了可培养细菌的分离纯化与多样性分析。
1.2 分离培养基与可培养细菌的分离纯化改良的海水Zobell 2216E培养基(g/L):蛋白胨1,酵母粉0.2,琼脂粉15,过滤陈海水:自来水(体积比为2:1) 。用无菌药匙移取适量沉积物样品于含5 mL无菌海水的试管中,经充分混匀和适当系列稀释后,取50 μL涂布2216E培养基平板,于4 °C倒置培养15 d。根据菌落形态学特征,挑取不同的单菌落进行进一步的分离纯化与分子鉴定。
1.3 分子鉴定与系统发育分析将处于指数生长期的100 μL细菌培养液加入1.5 mL EP管中,沸水煮沸10 min后再冰浴5 min,以此作为PCR扩增的DNA模板。采用细菌16S rRNA基因扩增通用引物27F (5′-AGAGTTTGATCC TGGCTCAG-3′)和1492R (5′-GGTTACCTTGT-TAC GACTT-3′)。细菌16S rRNA基因的扩增采用50 μL反应体系:1 μL引物27F (10 μmol/L),1 μL引物1492R (10 μmol/L),25 μL Master mix (KT201,北京Tiangen),21 μL无菌去离子水,2 μL DNA模板。反应条件:95 °C 5 min;95 °C 1 min,55 °C 30 s,72 °C 1.5 min,30个循环;72 °C 7 min。PCR产物经琼脂糖凝胶电泳检验合格后送南京金斯瑞生物股份有限公司进行纯化和测序;获得的16S rRNA基因序列有效长度为1 300−1 400 bp,测序结果在NCBI进行BLAST分析。序列比对采用BioEdit的多序列比对排列(Clustalw multiple alignment),系统发育分析采用Mega 5.0的邻接法(Neighbor-joining method)。与模式菌株的 16S rRNA基因相似性比较利用EzTaxon-Database进行(http://eztaxon-e.ezbiocloud.net)[6]。
2 结果与分析 2.1 菌株的分离纯化及鉴定采用营养物浓度稀释5倍的Zobell 2216E培养基4 °C培养15 d,从40个站位的北极海洋沉积物样品中共分离并获得16S rRNA基因有效序列的细菌达445株。采用EzTaxon在线比对分离菌株与模式菌株之间的16S rRNA基因序列相似性,结果显示,绝大多数菌株的16S rRNA基因序列与模式菌株的相似性高,达97%以上的菌株占总数的97.3%。系统发育分析结果表明,分离获得的细菌分别属于4个门,分别为变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、厚壁菌门(Firmicutes)和拟杆菌门(Bacteroidetes),6个纲,其中γ-变形菌纲(γ-Proteobacteria)的菌株数量最多,共283株,占总分离菌株数的63.6%,其次为黄杆菌纲(Flavobacteriia)、放线菌纲(Actinobacteria)、α-变形菌纲(α-Proteobacteria)、芽孢杆菌纲(Bacilli)和鞘脂杆菌纲(Sphingobacteria),菌株数分别为99、23、21、18和1株,分别占总数的22.2%、5.2%、4.7%、4.0%和0.2% (图 1);其中有12株菌株与已有模式菌株的16S rRNA基因序列相似性低于97%,可能代表了6个不同的细菌新物种。

|
| 图 1 北极海洋沉积物可培养细菌的组成分析 Figure 1 Composition analysis of culturable bacteria isolated from Arctic Ocean sediments |
|
|
分离获得的北极海洋沉积物细菌的种属类型、菌株数以及站位等情况详见表 1。从40个站位的北极海洋沉积物样品中共分离获得细菌445株,分属于49个属和91个种;其中,在属水平上,γ-变形菌纲细菌的多样性最为丰富,共14个属,黄杆菌纲和放线菌纲次之,各10个属,α-变形菌纲和芽孢杆菌纲各7个属,鞘脂杆菌纲仅含1个属。在菌株数量上,以希瓦氏菌属(Shewanella)所含数量最多,共有83株,分属于8个种,占总菌株数的18.7%,且其分布较广,从29个站位的沉积物中分离到该属菌株。其次为科尔韦尔氏菌属(Colwellia),共有81株,分属于5个种,占总菌株数的18.2%,其分布也较广,从33个站位的沉积物样品中分离到该属菌株。海洋杆菌属(Maribacter)和嗜冷单胞菌属(Psychromonas)菌株的出现频率及多样性也较高,分别为2个种49株和5个种35株。
| 纲 Class | 属 Genus | 种/菌株数量 Species/Number | 出现站位数/频率 Stance/Frequency (%) |
| α-proteobacteria | Sphingopyxis | 1/1 | 1/2.5 |
| Sulfitobacter | 2/5 | 5/12.5 | |
| Paracoccus | 1/4 | 2/5.0 | |
| Loktanella | 2/4 | 4/10.0 | |
| Halocynthiibacter | 1/2 | 2/5.0 | |
| Rhizobium | 1/4 | 3/7.5 | |
| Aliihoeflea | 1/1 | 1/2.5 | |
| γ-proteobacteria | Colwellia | 5/81 | 33/82.5 |
| Thalassomonas | 2/2 | 2/5.0 | |
| Glaciecola | 1/1 | 1/2.5 | |
| Marinobacter | 1/12 | 7/17.5 | |
| Moritella | 4/16 | 9/22.5 | |
| Pseudoalteromonas | 3/22 | 11/27.5 | |
| Pseudomonas | 1/1 | 1/2.5 | |
| Psychromonas | 5/35 | 16/40.0 | |
| Shewanella | 8/83 | 29/72.5 | |
| Marinomonas | 1/6 | 5/12.5 | |
| Neptunomonas | 1/8 | 6/15.0 | |
| Halomonas | 1/4 | 1/2.5 | |
| Photobacterium | 2/11 | 9/22.5 | |
| Stenotrophomonas | 1/1 | 1/2.5 | |
| Actinobacteria | Janibacter | 1/1 | 1/2.5 |
| Serinicoccus | 1/1 | 1/2.5 | |
| Arthrobacter | 1/2 | 1/2.5 | |
| Microbacterium | 6/9 | 7/17.5 | |
| Chryseoglobus | 1/1 | 1/2.5 | |
| Agrococcus | 2/2 | 2/5.0 | |
| Cryobacterium | 1/1 | 1/2.5 | |
| Rhodococcus | 2/3 | 2/5.0 | |
| Dietzia | 1/2 | 1/2.5 | |
| Nocardioides | 1/1 | 1/2.5 | |
| Bacilli | Bacillus | 7/9 | 6/15.0 |
| Planococcus | 1/2 | 1/2.5 | |
| Paenisporosarcina | 1/1 | 1/2.5 | |
| Lysinibacillus | 1/1 | 1/2.5 | |
| Chryseomicrobium | 1/1 | 1/2.5 | |
| Exiguobacterium | 1/2 | 2/5.0 | |
| Staphylococcus | 1/2 | 2/5.0 | |
| Flavobacteriia | Bizionia | 1/1 | 1/2.5 |
| Flavobacterium | 2/6 | 5/5.0 | |
| Gillisia | 1/1 | 1/2.5 | |
| Maribacter | 2/49 | 18/45.0 | |
| Psychroserpens | 1/2 | 2/5.0 | |
| Sediminicola | 1/6 | 5/12.5 | |
| Winogradskyella | 2/6 | 4/10.0 | |
| Nonlabens | 1/8 | 5/12.5 | |
| Algibacter | 2/18 | 11/27.5 | |
| Lutibacter | 2/2 | 2/5.0 | |
| Sphingobacteria | Sphingobacterium | 1/1 | 1/2.5 |
此外,一些属的分离菌株数量少,如鞘氨醇单胞菌属(Sphingomonas)、假单胞菌属(Pseudomonas)和盐单胞菌属(Halomonas)等仅分离获得1株菌,但可反映出该海域沉积物中存在此类微生物。
一般认为,16S rRNA基因序列相似性小于97%,可认为属于不同的种;相似性小于93%−95%,可以认为属于不同的属[7]。经16S rRNA基因的序列测定与对比分析,本文发现有 12个菌株序列与相似性最高的模式菌株的16S rRNA基因序列相似性<97% (表 2),对其进行系统发育分析发现,这12株菌株可能为6个潜在的新物种(图 2)。
| 分离站位 Sampling site | 经纬度 Longitude and latitude | 编号 Number | 相似性最高模式菌株(注册号) The highest-similarity species (accession No.) | 相似度 Similarity (%) |
| B14 | 61°56′03″W 176°21′01″E | 478 | Thalassomonas haliotis A5K-61T (AB369381) | 96.40 |
| LIC | 81°04′40″W 157°11′55″E | 332 | Bacillus persicus B48T (HQ433471) | 96.47 |
| NB04 | 61°12′02″W 171°33′19″E | 383-1 | Colwellia piezophila Y223GT (AB094412) | 96.22 |
| 383-2 | Thalassomonas actiniarum A5K-106T (AB369380) | 96.26 | ||
| 394 | Algibacter_agarivorans KYW560T (JN864025) | 96.98 | ||
| R02 | 67°40′52″W 169°01′18″E | 138 | Winogradskyella ulvae KMM 6390T (HQ456127) | 96.86 |
| R03 | 68°37′23″W 169°03′12″E | 441 | Winogradskyella ulvae KMM 6390T (HQ456127) | 96.85 |
| 445 | Winogradskyella ulvae KMM 6390T (HQ456127) | 96.88 | ||
| 453 | Winogradskyella ulvae KMM 6390T (HQ456127) | 96.90 | ||
| 145 | Winogradskyella ulvae KMM 6390T (HQ456127) | 96.88 | ||
| R09 | 74°36′42″W 168°59′09″E | 287 | Lutibacter litoralis CL-TF09T (AY962293) | 96.99 |
| R11 | 76°08′26″W 166°20′13″E | 271 | Colwellia aestuarii SMK-10T (DQ055844) | 96.83 |

|
| 图 2 北极海洋沉积物中可培养细菌疑似新种的系统发育树 Figure 2 Neighbor-Joining tree showing the phylogenetic relationships of potential novel species sequences of culturable bacteria isolated from Arctic Ocean sediments |
|
|
基于16S rRNA基因序列的系统发育分析结果,进一步证实,分离株分属6个纲、49个属和91个种(图 3和图 4)。从物种水平上,γ-变形菌纲数量最多(36个种,占39.6%);其次为放线菌纲 (17个种,占18.7%);黄杆菌纲和芽孢杆菌纲的数量也较多(分别为15个和13个,占16.5%和14.3%);鞘脂杆菌纲只有1个种,仅占0.2%。

|
| 图 3 北极海洋沉积物中可培养细菌中变形菌门的系统发育树 Figure 3 Neighbor-joining tree showing the phylogenetic relationship of Proteobacteria sequences of culturable bacteria isolated from Arctic Ocean sediments |
|
|

|
| 图 4 北极海洋沉积物中可培养细菌中黄杆菌纲、放线菌纲、芽孢杆菌纲和鞘脂杆菌纲的系统发育树 Figure 4 Neighbor-joining tree showing the phylogenetic relationship of Flavobacteriia,Actinobacteria,Bacilli and Sphingobacteria sequences of culturable bacteria isolated from Arctic Ocean sediments |
|
|
系统发育结果显示(图 2),疑似新种分布于多个不同分支,反映了北极菌种资源的丰富性和新颖性。例如,在γ-变形菌纲中发现了多个疑似新种,菌株271与模式菌株Colwellia aestuarii SMK-10T的16S rRNA基因序列相似性仅96.80%;菌株478与模式菌株Thalassomonashaliotis A5K-61T的相似性为96.40%;菌株383-2与模式菌株Thalassomonas actiniarum A5K-106T的相似性为96.26%等。
黄杆菌纲中也发现了疑似新种,菌株138、145、441、445和453与模式菌株Winogradskyella ulvae KMM6390T的16S rRNA基因序列相似性分别为96.86%、96.88%、96.85%、96.88%和96.90%;菌株287与模式菌株Lutibacter litoralis CL-TF09T的相似性为96.98%。
α-变形菌纲中也发现了一个疑似新种,菌株394与模式菌株Algibacter agarivorans KYW560T的16S rRNA基因序列相似性为96.98%。此外,芽孢杆菌纲中也发现了一个疑似新种,菌株332与模式菌株Bacillus persicus B48T的16S rRNA基因序列相似性为96.47%。
3 讨论通过对第六次北极科学考察获取的40个站位的海洋沉积物中的细菌分离纯化与培养,获得了较丰富的极地微生物菌种资源。在所获取的445株细菌中,变形菌门细菌占可培养细菌总数的绝大多数,其中以γ-变形菌纲的细菌数量最多,占总分离菌株的63.6%,这与以往国内外的研究结果相似。γ-变形菌为深海沉积环境中的优势类群,不乏一些具有特殊生理功能的菌株[8]。如张荣秋等[9]在白令海和楚科奇海表层沉积物中分离培养出以萘、菲和芘为唯一碳源和能源的菌株51株,其中优势菌为海杆菌属、假交替单胞菌属(Pseudoalteromonas)和假单胞菌属,可以有效降解不同有机污染物。李会荣等[10]采用PCR结合变性梯度凝胶电泳技术(DGGE)对北极太平洋扇区3个不同深度的海洋沉积物样品的细菌系统发育分析发现,其优势细菌类群为γ-Proteobacteria。
本文分离获得假交替单胞菌属、希瓦氏菌属和黄杆菌属(Flavobacterium)的菌株较多,占总分离菌株的25.6%。已有的研究表明,这3个属的菌株均能分泌胞外多糖物质和适冷蛋白酶,其研究与应用将对传统的食品、医药、化工、能源和环保的发展产生深远影响[11]。
本次分离获得放线菌23株,占总数的5.2%;目前广泛应用的抗生素约70%是由各种放线菌所产生。如Bredholdt等[12]从挪威最大的峡湾——特隆赫姆峡湾(Trondheim Fjord)的海底沉积物中分离出3 200株放线菌,并对所分离出的放线菌进行抗细菌和抗真菌筛选,结果发现这些放线菌具有强大的抗生素生产潜力。
本次分离获得黄杆菌99株,占总数的22.2%,Mojib等[13]在南极湖泊中的一株Flavobacterium sp| Ant342中分离出黄色色素F-YOP,抑菌实验结果表明该物质对结核分枝杆菌等结核病菌具有很高的抗性,或将成为结核病化学治疗药的先导化合物。
通过从第五次和第六次北极(简称五北和六北)科考采集的海洋沉积物中分离获得的细菌在纲水平上的组成比较分析表明,两次科学考察获得的可培养细菌在纲水平上的组成基本相似,均以γ-变形菌纲和黄杆菌纲为主;但相比于第五次北极考 察[14],第六次北极考察所分离获得的菌株新增加了鞘脂杆菌纲,尽管其仅占总分离株数的0.2%;黄杆菌纲菌株的比例明显上升,达到了22.2%;而γ-变形菌纲的比例略微下降但其仍为最多(图 5)。
|
| 图 5 在纲水平上五北与六北可培养细菌组成的比较分析 Figure 5 Comparable analysis of culturable bacterial composition between the isolates from 5th and 6th Arctic Expedition at the class level |
|
|
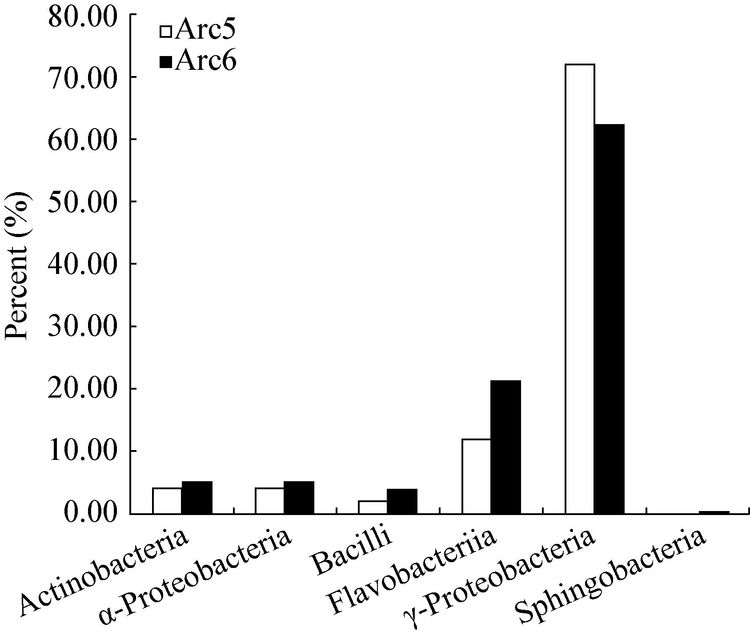
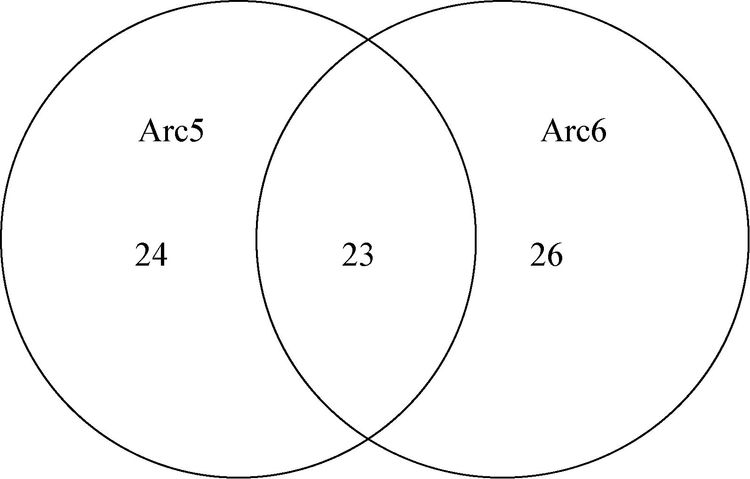
两次科学考察分离获得的细菌组成在属水平上有着较为明显的差异,从五北中分离获得的菌株分布于47个属,从六北中分离获得的菌株分布于49个属,二者仅有23个属相同,表现了较大的差异性(图 6),这可能与两次分离菌株所用的培养基组成成分有所差异造成的,与五北采用的Zobell 2216E相比,六北采用的分离培养基将营养物浓度稀释了5倍。进一步分析两次北极科考数量前十位的属的可培养细菌的组成情况(图 7),结果发现,希瓦氏菌属菌株数量均最多,比例分布26.8%和18.7%;在六北中属于科尔韦尔氏菌属的菌株数量为第二,占总分离菌株的18.2%,但其在五北中只有4.4%,为数量第4的属;海洋杆菌属在六北中占总分离菌的11.0%,含量第三,而在五北中只有3.0%;藻杆菌属(Algibacter)菌株在六北中占总分离菌的4.0%,而在五北中未分离到。然而,在五北中占总数3.3%的嗜冷杆菌属(Psychrobacter)在六北中没有分离到;在五北中占总分离菌株数6.3%的亚硫酸杆菌属(Sulfitobacter)在六北中只有1.1%。从属水平上看,五北与六北两次考察培养出的细菌具有较大的差异性,体现出了北极微生物的多样性;同时也表明,为获得尽可能多的微生物菌种资源,同时采用多种培养条件进行分离纯化的必要性。
|
| 图 6 属水平上五北与六北可培养细菌的组成分析 Figure 6 Comparison analysis of culturable bacteria of marine sediments between Arc5 and Arc6 at the genus level |
|
|
|
| 图 7 属水平上五北与六北可培养细菌组成分析图 Figure 7 Composition analysis of culturable bacteria of marine sediments between Arc6 and Arc5 at the genus level |
|
|
极地特殊的环境造就了极地微生物特殊的生物学特征,为适应极地的特殊条件,极地微生物必需采用独特的生存策略来获得生存优势[15],这使其具备了特殊的生理生化特征,对其开发利用可以在医药、食品、日化及环保等诸多领域给人们的生活带来巨大的方便。本文通过分离纯化得到了丰富的细菌菌种资源,并与第五次采集得到的菌株进行了对比分析,发现北极微生物的多样性远不止现阶段所了解的情况,今后仍需对其进行不断地探索和认识。
| [1] | Chen HW, Gao AG. Progress on investigation and research of polar microbiology in China[J]. Chinese Journal of Polar Research, 2005, 17 (4): 299–307. (in chinese) 陈皓文, 高爱国. 中国极地微生物学调查研究进展[J]. 极地研究, 2005, 17 (4):299–307. |
| [2] | Chen JG, Yang JF. The industrial applications of polar microorganism and connectiong with astrobiology research[J]. Chemistry of Life, 2008, 28 (1): 97–100. (in chinese) 陈吉刚, 杨季芳. 极地微生物的工业应用及其与天体生物学研究的联系[J]. 生命的化学, 2008, 28 (1):97–100. |
| [3] | Zeng YX, Zou Y, Chen B, et al. Phylogenetic diversity of sediment bacteria in the northern Bering Sea[J]. Polar Biology, 2011, 34 (6): 907–919. |
| [4] | Su YH. Molecular analysis of microbial communities in Pacific Arictic surficial sediment[D]. Qingdao: Master’s Thesis of Ocean University of China, 2006 (in Chinese) 苏玉环. 北极太平洋扇区表层沉积物中微生物群落的分子生物学分析[D]. 青岛: 中国海洋大学硕士学位论文, 2006 |
| [5] | Liu P, Wang HM. The investigation of microbial diversity in marine sediments[J]. Journal of Anhui Agricultural Sciences, 2009, 37 (10): 4569–4571. (in chinese) 刘璞, 王红梅. 海洋沉积物微生物多样性研究[J]. 安徽农业科学, 2009, 37 (10):4569–4571. |
| [6] | Kim OS, Cho YJ, Lee K, et al. Introducing EzTaxon-e: aprokaryotic 16S rRNA gene sequence database with phylotypes that represent uncultured species[J]. International Journal of Systematic and Evolutionary Microbiology, 2012, 62 (Pt 3): 716–721. |
| [7] | Xu XW, Wu M, Huang WD. Isolation and 16S rDNA sequence analysis of extremely halophilic archaea from saline in Altun region[J]. Journal of Zhejiang University (Engineering Science), 2004, 38 (4): 508–512. (in chinese) 许学伟, 吴敏, 黄伟达. 阿尔金山盐湖极端嗜盐古生菌的分离及16S rDNA序列分析[J]. 浙江大学学报: 工学版, 2004, 38 (4):508–512. |
| [8] | Head IM, Jones DM, Röling WFM. Marine microorganisms make a meal of oil[J]. Nature Reviews Microbiology, 2006, 4 (3): 173–182. |
| [9] | Zhang RQ, Dong CM, Sheng HF, et al. Biodiversity of polycyclic aromatic hydrocarbon-degrading bacteria from surficial sediments of the Bering Sea and Chukchi Sea in the Arctic[J]. Acta Oceanologica Sinica, 2014, 36 (4): 52–61. (in chinese) 张荣秋, 董纯明, 盛华芳, 等. 白令海和楚科奇海表层沉积物中多环芳烃降解微生物多样性[J]. 海洋学报, 2014, 36 (4):52–61. |
| [10] | Li HR, Yu Y, Zen YX. Phylogenetic analysis of bacterial diversity in Pacific Arctic sediments[J]. Acta Microbiologica Sinica, 2006, 46 (2): 177–183. (in chinese) 李会荣, 俞勇, 曾胤新. 北极太平洋扇区海洋沉积物细菌多样性的系统发育分析[J]. 微生物学报, 2006, 46 (2):177–183. |
| [11] | Nichols CAM, Garon S, Bowman JP, et al. Production of exopolysaccharides by Antarctic marine bacterial isolates[J]. Journal of Applied Microbiology, 2004, 96 (5): 1057–1066. |
| [12] | Bredholdt H, Galatenko OA, Engelhardt K, et al. Rare actinomycete bacteria from the shallow water sediments of the Trondheim fjord, Norway: isolation, diversity and biological activity[J]. Environmental Microbiology, 2007, 9 (11): 2756–2764. |
| [13] | Mojib N, Philpott R, Huang JP, et al. Antimycobacterial activity in vitro of pigments isolated from Antarctic bacteria[J]. Antonie van Leeuwenhoek, 2010, 98 (4): 531–540. |
| [14] | Wang Z, Li Y, Che S, et al. Diversity analysis of culturable bacteria isolated from marine sediments of Arctic[J]. Acta Oceanologica Sinica, 2014, 36 (10): 116–123. (in chinese) 王桢, 李阳, 车帅, 等. 北极海洋沉积物中可培养细菌及其多样性分析[J]. 海洋学报, 2014, 36 (10):116–123. |
| [15] | Giudice AL, Bruni V, Michaud L. Characterization of Antarctic psychrotrophic bacteria with antibacterial activities against terrestrial microorganisms[J]. Journal of Basic Microbiology, 2007, 47 (6): 496–505. |
 2016, Vol. 43
2016, Vol. 43