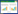扩展功能
文章信息
- 黄兴如, 张彩文, 张瑞杰, 章俊, 张晓霞
- HUANG Xing-Ru, ZHANG Cai-Wen, ZHANG Rui-Jie, ZHANG Jun, ZHANG Xiao-Xia
- 多环芳烃降解菌的筛选、鉴定及降解特性
- Isolation, identification and degrading properties of PAHs-degrading bacteria
- 微生物学通报, 2016, 43(5): 965-973
- Microbiology China, 2016, 43(5): 965-973
- DOI: 10.13344/j.microbiol.china.150949
-
文章历史
- 收稿日期: 2015-11-19
- 接受日期: 2016-01-27
- 优先数字出版日期(www.cnki.net): 2016-02-03
2. 首都师范大学资源环境与旅游学院 北京 100048
2. College of Resources Environment and Tourism, Capital Normal University, Beijing 100048, China
多环芳烃(Polycyclic aromatic hydrocarbons,PAHs)是指由两个或两个以上的苯环稠合在一起的一类持久性有机污染物,主要源于石油、煤等化石燃料以及木材、天然气、秸秆等有机物的不完全燃烧或者在还原条件下经热分解而生成,其在环境中普遍存在[1]。PAHs具有高毒性、热稳定性、生物富集性、疏水性、不易降解等特点[2-3],极易在环境中累积,并可通过食物链传递而在生物体内富集,对人类健康和生态环境具有很大的危害性[4];因此美国环保局于1976年将16种多环芳烃列为优先控制污染物名单[5]。PAHs依据苯环数量可分为低环和高环(苯环≥4) PAHs两类,且研究发现,随着环数的增加,其毒性越高,生物可利用性越差,越不易被降解[6]。
释放到自然环境中的PAHs能够通过多种途径移除,包括挥发、光氧化、化学氧化、生物富集、微生物降解和土壤吸附等,大量的研究证明微生物降解是去除环境中PAHs的最主要途径[7]。PAHs的微生物修复因其经济、高效且无二次污染等特点,已成为去除环境中PAHs的理想方法和重要手段[8]。目前,研究人员已从环境中分离出多种PAHs降解菌,主要包括假单胞菌属(Pseudomonas)[9]、红球菌属(Rhodococcus)[10]、气单胞菌属(Aeromonas)[11]、芽孢杆菌属(Bacillus)[12]、伯克氏菌属(Burkholderia)[13]、分支杆菌属(Mycobacterium)[14]和鞘氨醇单胞菌属(Sphingomonas)[15]等,但其中所涵盖的高效降解菌株并不多[16],特别是高环PAHs降解菌更为稀少。因此筛选PAHs高效降解菌或混合菌系,具有重要的现实意义。本研究从中国海洋微生物菌种保藏中心和中国农业微生物菌种保藏中心获得的25株细菌中筛选高分子量PAHs的高效降解菌,分析其在液体培养基中的降解特性,为PAHs污染环境的微生物修复提供资源保障和科学依据。
1 材料与方法 1.1 实验材料1.1.1 供试菌株: 从中国海洋微生物菌种保藏中心和中国农业微生物菌种保藏中心获取25株具有耐受或降解PAHs功能的细菌,相应的编号分别为:01990、02172、02173、02588、02830、02755、10542、10543、11238、11239、11240、11241、11242、11243、11244、11245、11246、14971、K13(H25) 、S23(1) 、MCT5、F44-8、RC99-3、SL-1、F11,用于筛选多环芳烃高效降解菌。
1.1.2 多环芳烃: 萘、菲、芴、芘、苯并[a]芘(浓度>97%)均购于中国国家标准物质研究中心。
1.1.3 培养基: 无机盐基础培养基(g/L)[17]:NH4Cl 1.1,K2HPO4 1.0,NaCl 0.5,KCl 0.2,MgSO4·7H2O 0.2,FeSO4 0.001,CaCl2 0.01,微量元素混合液1 ml,pH 7.0,固体培养基则再加20 g琼脂。TSB合成培养基(g/L):胰蛋白胨17,大豆胨3,葡萄糖2.5,氯化钠5,磷酸氢二钾2.5,蒸馏水1 000 mL,pH 7.3±0.2。微量元素参照参考文献[18]配制。
1.2 多环芳烃降解菌的筛选将获得的25株菌依次点接到无机盐培养基平板上,以菲为底物,采用平板升华法[19]将PAHs镀于平板上,而后置于30 °C恒温培养箱中培养,4−5 d后观察降解结果。对出现明显降解圈的菌株再次进行平板复筛,通过牛津杯使加入的菌苔的直径定量为6 mm,以芘为底物,培养5 d后定量测定降解圈直径,筛选高分子量PAHs的高效降解菌株。
1.3 菌种鉴定采用热裂解法提取菌体总DNA[20],并利用通用引物27f (5′-AGAGTTTGATCCTGGCTCAG-3′)和1492r (5′-GGTTACCTTGTTACGACTT-3′)对细菌16S rRNA基因进行PCR扩增。25 µL扩增体系为:DNA模板2 µL,2×Taq PCR Master Mix 12.5 µL,27f (10 μmol/L)和1492r (10 μmol/L)各 1 µL,ddH2O 8.5 µL。PCR扩增程序为:95 °C 5 min;95 °C 50 s,55 °C 50 s,72 °C 1 min,30个循环;72 °C 7 min。获得的PCR产物采用0.8%的琼脂糖凝胶电泳检测后,送往北京美吉生物科技有限公司完成测序工作。降解菌16S rRNA基因序列通过Blast程序与GenBank中核酸数据进行比对分析。然后利用MEGA 6.0软件构建系统发育树,采用Neighbor-Joining法进行系统发育分析。
1.4 降解菌株生长曲线的测定将筛选出的降解菌株接种于TSB液体培养基中,摇床振荡(30 °C、180 r/min)培养活化20 h,取出2 ml菌液转入盛有100 ml TSB的300 mL三角瓶中,充分摇匀后依次吸取10 mL菌液于无菌试管中,将试管30 °C、180 r/min振荡培养。每隔4 h取出试管,以未接种TSB液体培养基作空白对照,利用UV-1800 紫外分光光度仪测定不同培养时间的细菌菌液的OD600值。
1.5 降解菌生理学特征1.5.1 NaCl生长浓度范围及最适值: 在含有不同NaCl浓度(0、1%、2%、3%、4%、5%和6%)的TSB液体培养基中接种相同浓度的菌液(1%接种量,体积比,并设立不接菌液为对照组,每个浓度设3个平行),30 °C振荡培养24 h,用UV-1800 紫外分光光度仪测量600 nm处的吸光值。
1.5.2 生长pH范围及最适值: 在已确定的最适NaCl生长浓度培养液中用HCl或KOH调pH值至5.0、6.0、7.0、8.0、9.0、10.0和11.0,接菌后置于30 °C振荡培养24 h,用UV-1800紫外分光光度仪测量600 nm处的吸光值。
1.5.3 生长温度范围及最适值: 在已确定的最适NaCl生长浓度和生长pH的培养液中接入菌液,分别在15、20、25、30、35、40 °C条件下振荡培养,24 h后利用UV-1800 紫外分光光度仪测定细菌菌液的OD600值。
1.6 菌株的降解特性1.6.1 菌悬液的制备: 在无菌条件下将菌株接种于添加菲(100 mg/L)的TSB液体培养基中,30 °C、180 r/min振荡培养3 d,离心(10 000 r/min,6 min)收集菌体,并用无菌水反复洗涤3次,再用无机盐培养基将菌悬液调至所需浓度备用。
1.6.2 菌株对混合PAHs的降解: 向盛有50 mL无机盐培养基的150 mL三角瓶中加入适量的PAHs母液,使每个三角瓶内的萘、菲、芴、芘的浓度为200、50、100、50 mg/L,每株菌设3个平行,每个平行设3个重复,并设一不加菌空白对照组,30 °C、170 r/min条件下避光振荡培养,分别于24、48和72 h后取样,对样品中的全部PAHs进行萃取。
1.6.3 菌株对单一PAH 的降解: 向无机盐培养基中分别加入适量的菲和芘,使其终浓度达到100 mg/L和50 mg/L,其中菲设3个平行,每个平行设3个重复,并设一不加菌空白对照;芘设3个平行,每个平行设5个重复,并设一不加菌空白对照,30 °C、170 r/min条件下避光振荡培养,分别于24、48和72 h后取样,对样品中的全部PAHs菲和芘进行萃取。
1.6.4 溶液中多环芳烃的提取与分析: 培养基中的PAHs利用50 mL乙酸乙酯进行两次中性和两次酸性萃取(pH 2.0) ,合并萃取液后用无水Na2SO4去除水分。而后将萃取液用高纯氮吹干,残留物用色谱级甲醇重新溶解,用高效液相色谱(HPLC)测定。HPLC使用色谱柱为C18反相柱(Agilent,USA,250 mm×4.6 mm,5 μm),流动相为甲醇:水=80:20,流速为1 mL/min进行线性洗脱,进样量20 μL,检测波长为254 nm,柱温为40 °C。
1.6.5 共代谢底物对菌株降解多环芳烃的影响: 取2 ml菌悬液接入100 mL无机盐培养基中(芘浓度为100 mg/L),向三角瓶中添加邻苯二酚母液,使液体中邻苯二酚的终浓度分别为10、20、50和100 mg/L,30 °C、170 r/min振荡培养7 d,采用1.6.4节的方法萃取和测定培养基中残留的芘,以未加邻苯二酚的培养基为对照。每个浓度均设置 3个平行。
2 结果与分析 2.1 多环芳烃降解菌的筛选以菲为底物进行平板初筛,从供试菌株中筛选出10株能以PAH菲为唯一碳源和能源生长的降解菌株。而后以芘为底物进行平板复筛,最终获得3株降解圈直径大于13 mm的菌株,分别为02830、02173和SL-1 (图 1)。
|
| 图 1 降解菌株在固体平板上的降解圈直径 Figure 1 Diameter of the transparent circle created by the degradative strains in the solid plate |
|
|
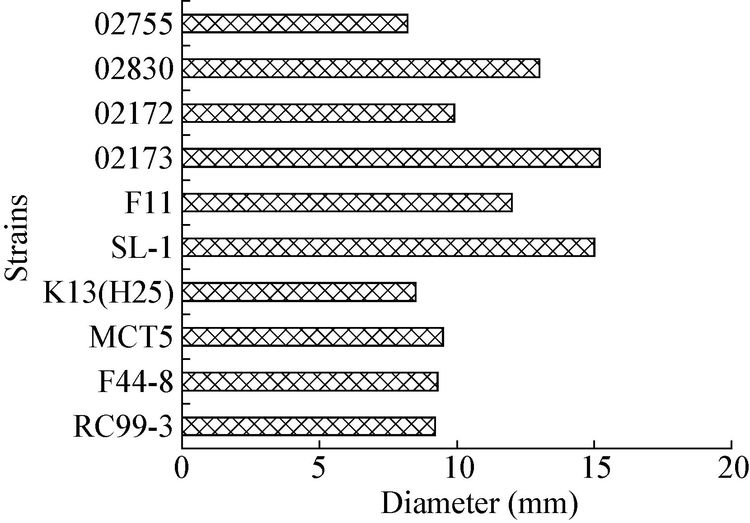
对以菲和芘作为惟一碳源的PAHs高效降解菌株进行基因组DNA的提取,并完成16S rRNA基因测序,将得到的序列与GenBank中已知序列进行比对,并对两种未知的降解菌02173和02830分别构建系统发育树,结果如图 2所示。根据16S rRNA序列同源性比对表明,菌株02173与假单胞菌属(Pseudomonas)中的Pseudomonas alcaliphila同源性最近,序列相似性高达99%;02830则与假单胞菌属(Pseudomonas)中的Pseudomonas corrugata同源性最近,其序列相似性达96.75%;而菌株SL-1为本课题组发表新类群Rhizobium petrolearium的模式菌株。菌株02173和02830所测序列提交到Genbank数据库,获得序列登录号分别为KU306076和KU306077。
|
| 图 2 基于菌株02173 和02830 16S rRNA基因序列构建的系统发育树 Figure 2 Neighbour-joining phylogenetictree based on nearly complete 16S rRNA gene sequences ofstrains 02173 and 02830 The numbers at the nodes indicate the levels of bootstrap support (%) based on 1 000 resampled data sets; only values above 50% are given. GenBank accession numbers are given in brackets. The scale bar represents 0.005 (A)/0.002 (B) substitutions per nucleotide position. |
|
|
由图 3可知,3株菌均适宜在TSB培养基中生长,延滞期都较短,菌株02173和02830于4 h后进入对数期,随着培养基中营养物质的消耗,24 h后开始逐渐进入稳定期;SL-1的延滞期稍长,8 h后开始进入对数期。3株菌的生长趋势大致保持一致,其中02173与02830的长势最好,略高于SL-1的生物量。

|
| 图 3 降解菌株生长曲线 Figure 3 Growth curve of degradative strains |
|
|
如图 4a所示,降解菌02173和02830在各个盐浓度下长势良好,盐度在0−5%之间时OD600值均高于1,其中菌株02173最适宜盐度为3%−4%,菌株02830最适宜盐度为1%−2%。菌株SL-1对盐度的变化较为敏感,当盐浓度大于4%时,其生长受到抑制,最适宜生长盐浓度为1%。3株菌最适宜的pH值均为7.0,且发现菌株SL-1在pH 11.0时生长受到抑制(图 4b)。由图 4c可知,所有菌株最适宜生长温度为30 °C,其中菌株02173适宜生长温度范围较广,在20−40 °C下生长良好;菌株02830适宜生长温度为20−35 °C,不耐受高温;而菌株SL-1生长温度范围较为受限(25−35 °C),低温和高温均不适宜其生长。总之,菌株02173和02830对逆境的耐受能力较SL-1略强。

|
| 图 4 不同盐浓度(A)、pH (B)、温度(C)条件下菌株的生物量 Figure 4 Biomass of each strain in the condition of different salinity (A),pH (B) and temperature (C) |
|
|
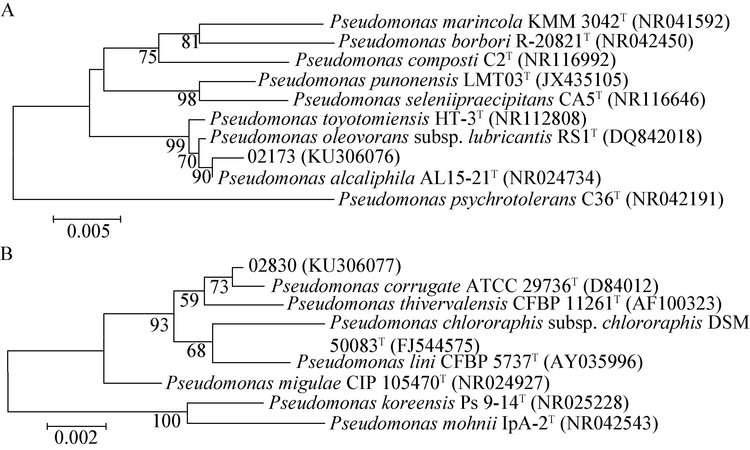
2.5.1 菌株对混合多环芳烃的降解:污染环境中的多环芳烃均以混合形式存在,通过测定菌株对混合PAHs中高低环PAHs的降解率,相对于单一PAHs更加实际。如图 5a所示,3株菌对混合PAHs中萘(200 mg/L)的降解主要发生在1 d内,降解率均达到了87%以上,而菲(100 mg/L)、芴 (50 mg/L)和芘(50 mg/L)的降解主要发生在2 d内(图 5b,c,d),降解速率明显弱于萘。研究发现,3 d后3株菌对萘的降解率均达到或接近100%;与此同时,菌株02173和02830对菲、芴和芘的降解率均达到97%以上,而菌株SL-1对PAHs (菲、芴和芘)的降解要明显弱于菌株02173和02830,3 d后对菲、芴和芘的降解率分别达到75.89%、81.04%和81.98%。
|
| 图 5 供试菌株对混合PAHs萘(a)、菲(B)、芴(C)和芘(D)的降解特性 Figure 5 Degradation of Nap (A),Phe (B),Flu (C) and Pyr (D) in the mixed PAHs by functional strains |
|
|
2.5.2 菌株对单一多环芳烃的降解: 为分析多环芳烃降解特征,以菌株SL-1为代表,分析其对单一PAH菲和芘的降解率。如图 6所示,3 d后菌株SL-1对菲(100 mg/L)和芘(50 mg/L)的降解率分别达到100%和48%,而培养5 d后能够降解74%的芘,且发现接种后1 d内菲和芘的降解率最高。

|
| 图 6 菌株SL-1对单一PAHs菲(A)和芘(B)的降解特性 Figure 6 Degradation of Phe (A) and Pyr (B) by strain SL-1 |
|
|
选取PAHs降解途径中最普遍的中间产物邻苯二酚作为共代谢底物,分析其对高分子量PAHs降解的影响。如图 7所示,添加外加碳源邻苯二酚对菌株SL-1降解芘产生了一定的影响。实验结果表明,第7天的时候,添加了邻苯二酚浓度为10、20、50、100 mg/L的培养基中芘的降解率分别为69.79%、78.86%、86.93%、61.44%,而未添加外源碳源的空白对照的降解率为60.83%。由此可见,共代谢底物邻苯二酚的添加,能促进高分子量PAHs芘的降解,当邻苯二酚达到一定浓度时,芘的降解反而被抑制。

|
| 图 7 不同邻苯二酚浓度对SL-1 降解芘的影响 Figure 7 Different concentration of catechol affecting the Pyr degradation by strain SL-1 |
|
|
利用微生物处理法解决自然环境中的PAHs污染问题已越来越受大家青睐[21],其优点在于费用低、二次污染少、修复彻底等,是一类低耗高效和环境安全的生物修复技术[22],而筛选分离PAHs高效降解菌是该项技术的关键环节[23]。本研究从25株供试菌株中分离到3株可高效降解低环和高环PAHs的细菌SL-1、02173和02830,通过16S rRNA基因序列比对确定02173和02830分别与假单胞菌属中的Pseudomonas alcaliphila和Pseudomonas corrugate同源性最近,SL-1属于模式菌株Rhizobium petrolearium SL-1。已有研究表明,根瘤菌具有较好的有机污染物降解能力,如多氯联苯[24]、三硝基甲苯[25]、二苯并噻吩[26]、2,4-二硝基甲苯[27]等,而利用其降解PAHs的研究较少。本研究所筛选的根瘤菌SL-1能够以菲和芘为唯一碳源和能源,3 d内对菲(100 mg/L)和芘(50 mg/L)的降解率分别达到100%和48%,虽然菌株SL-1在 50 mg/L的芘液体培养基中生长比较缓慢,但是培养5 d后能够降解74%的芘。陈芳艳等[28]从某焦化厂生物处理装置的活性污泥中分离筛选出一株游离茄镰孢菌,经过108 h对菲的降解效率达到49.25%;毛健等[29]筛选出一株可降解芘的副球菌,该菌株在芘浓度为50 mg/L的培养基中7 d内可降解47.2%的芘;相较而言,菌株SL-1具有较高的降解能力,且对高分子量PAHs具有很好的降解潜力。与此同时,菌株02173和02830对PAHs也同样表现出高效的降解能力,3 d内对混合多环芳烃中萘(200 mg/L)、芴(50 mg/L)、菲(100 mg/L)和芘(50 mg/L)的降解均分别超过97%,虽然多有研究表明,假单胞菌对PAHs具有较好的降解能力,但主要针对四环以下的PAHs[30-32]。由此可见,该研究筛选出的3株菌不但对低分子量PAHs具有较好的降解能力,还可以高效降解高分子量PAHs,为多环芳烃污染土壤的生物修复技术提供宝贵的微生物资源。
污染环境中的多环芳烃均以混合形式存在,通过测定菌株对混合PAHs中高低环PAHs的降解率,相对于单一PAHs更具有现实意义。本研究中3株降解菌对混合多环芳烃均表现出高效的降解能力,尤其是菌株02173和02830;且研究发现,混合多环芳烃中萘的降解主要发生在1 d内,降解率均达到了87%以上,而芴、菲和芘的降解主要发生在2 d内,且降解速率明显弱于萘;说明多种PAHs存在的情况下,降解菌优先选择低环PAHs作为碳源和能源。菌株SL-1 3 d内对混合PAHs中菲和芘的降解分别为75.89%和81.98%,与单一PAHs菲和芘的降解相较而言,小分子量PAHs菲的降解率有所下降,而大分子量PAHs芘的降解率明显上升,这说明小分子量PAHs可促进大分子量PAHs的降解,而与此同时,小分子量PAHs的降解会受到抑制。这与目前已报道的多环芳烃降解方式相符,低环PAHs可以作为菌株生长的唯一碳源和能源而被直接降解,但高环PAHs需与其它有机质共代谢(或共氧化)而被降解[33-34]。该研究也同时发现,适当的共代谢底物邻苯二酚能促进高分子量PAHs芘的降解,当随着共代谢底物浓度的不断提高,芘的降解反而被抑制。由此可见,共代谢作用因改变了微生物碳源与能源的底物结构,增大微生物对碳源和能源的选择范围,从而使得难降解PAHs最终被微生物利用降解;而随着共代谢底物的不断提高,引起了竞争抑制,从而使得PAHs降解率下降。
| [1] | Wilcke W. Synopsis polycyclic aromatic hydrocarbons (PAHs) in soil—a review[J]. Journal of Plant Nutrition and Soil Science, 2000, 163 (3): 229–248. |
| [2] | Arulazhagan P, Vasudevan N. Biodegradation of polycyclic aromatic hydrocarbons by a halotolerant bacterial strain Ochrobactrum sp. VA1[J]. Marine Pollution Bulletin, 2011, 62 (2): 388–394. |
| [3] | Feng TC, Cui CZ, Dong F, et al. Phenanthrene biodegradation by halophilic Martelella sp. AD-3[J]. Journal of Applied Microbiology, 2012, 113 (4): 779–789. |
| [4] | Haritash AK, Kaushik CP. Biodegradation aspects of polycyclic aromatic hydrocarbons (PAHs): a review[J]. Journal of Hazardous Materials, 2009, 169 (1/3): 1–15. |
| [5] | Keith L, Telliard W. ES & T special report: priority pollutants: I-a perspective view[J]. Environmental Science & Technology, 1979, 13 (4): 416–423. |
| [6] | Seo JS, Keum YS, Li QX. Bacterial degradation of aromatic compounds[J]. International Journal of Environmental Research and Public Health, 2009, 6 (1): 278–309. |
| [7] | Patel V, Patel J, Madamwar D. Biodegradation of phenanthrene in bioaugmented microcosm by consortium ASP developed from coastal sediment of Alang-Sosiya ship breaking yard[J]. Marine Pollution Bulletin, 2013, 74 (1): 199–207. |
| [8] | Rockne KJ, Strand SE. Anaerobic biodegradation of naphthalene, phenanthrene, and biphenyl by a denitrifying enrichment culture[J]. Water Research, 2001, 35 (1): 291–299. |
| [9] | Lin M, Hu XK, Chen WW, et al. Biodegradation of phenanthrene by Pseudomonas sp. BZ-3, isolated from crude oil contaminated soil[J]. International Biodeterioration & Biodegradation, 2014, 94 : 176–181. |
| [10] | Finkelstein ZI, Baskunov BP, Golovlev EL, et al. Fluorene transformation by bacteria of the genus Rhodococcus[J]. Microbiology, 2003, 72 (6): 660–665. |
| [11] | Sàágua MC, Baeta-Hall L, Anselmo AM. Microbiological characterization of a coke oven contaminated site and evaluation of its potential for bioremediation[J]. World Journal of Microbiology and Biotechnology, 2002, 18 (9): 841–845. |
| [12] | Feitkenhauer H, Müller R, Märkl H. Degradation of polycyclic aromatic hydrocarbons and long chain alkanes at 6 070 °C by Thermus and Bacillus spp.[J]. Biodegradation, 2003, 14 (6): 367–372. |
| [13] | Weissenfels WD, Beyer M, Klein J. Degradation of phenanthrene, fluorene and fluoranthene by pure bacterial cultures[J]. Applied Microbiology and Biotechnology, 1990, 32 (4): 479–484. |
| [14] | Li QX, Fan BQ, Gong MB, et al. Isolation, identification of a pyrene-degrading strain Mycobacterium sp. M11 and its degrading characteristics[J]. Environmental Science, 2008, 29 (3): 763–768. (in chinese) 李全霞, 范丙全, 龚明波, 等. 降解芘的分枝杆菌M11的分离鉴定和降解特性[J]. 环境科学, 2008, 29 (3):763–768. |
| [15] | Roy M, Khara P, Dutta TK. meta-Cleavage of hydroxynaphthoic acids in the degradation of phenanthrene by Sphingobium sp. strain PNB[J]. Microbiology, 2012, 158 (3): 685–695. |
| [16] | Jia YH, Qu YY, Zhou JT, et al. Characteristics and enzyme purification of Burkholderia sp. AFF for phenanthrene biodegradation[J]. Environmental Science & Technology, 2009, 32 (5): 21–25. (in chinese) 贾玉红, 曲媛媛, 周集体, 等. 菲降解菌的特性及其降解酶纯化研究[J]. 环境科学与技术, 2009, 32 (5):21–25. |
| [17] | Yan QX, Wang YX, Li SP, et al. Sphingobium qiguonii sp. nov., a carbaryl-degrading bacterium isolated from a wastewater treatment system[J]. International Journal of Systematic and Evolutionary Microbiology, 2010, 60 (12): 2724–2728. |
| [18] | Zhang XF, Hiorshi O. Study on isolation, identification and characteristics of polycyclic aromatic hydrocarbons degrading bacteria[J]. Shanghai Environmental Science, 2003, 22 (8): 544–547. (in chinese) 张小凡, 小柳津广志. 多环芳烃化合物菲分解菌的分离鉴定及分解特性研究[J]. 上海环境科学, 2003, 22 (8):544–547. |
| [19] | Alley JF, Brown LR. Use of sublimation to prepare solid microbial media with water-insoluble substrates[J]. Applied and Environmental Microbiology, 2000, 66 (1): 439–442. |
| [20] | Zhang XX, Sun L, Qiu FB, et al. Rheinheimera tangshanensis sp. nov., a rice root-associated bacterium[J]. International Journal of Systematic and Evolutionary Microbiology, 2008, 58 (10): 2420–2424. |
| [21] | Gibson DT, Mahadevan V, Jerina DM, et al. Oxidation of the carcinogens benzo[a] pyrene and benzo[a] anthracene to dihydrodiols by a bacterium[J]. Science, 1975, 189 (4199): 295–297. |
| [22] | Yang H, Wang HX, Li XJ, et al. Research progress on the bioremediation technology of polycyclic aromatic hydrocarbons contaminated soil[J]. Journal of Anhui Agricultural Sciences, 2011, 39 (3): 1427–1429. (in chinese) 杨辉, 王海霞, 李晓军, 等. 多环芳烃污染土壤生物修复技术研究进展[J]. 安徽农业科学, 2011, 39 (3):1427–1429. |
| [23] | Tao XQ, Lu GN, Yi XY, et al. Isolation of phenanthrene-degrading microorganisms and analysis of metabolites of phenanthrene[J]. Journal of Agro-Environment Science, 2006, 25 (1): 190–195. (in chinese) 陶雪琴, 卢桂宁, 易筱筠, 等. 菲高效降解菌的筛选及其降解中间产物分析[J]. 农业环境科学学报, 2006, 25 (1):190–195. |
| [24] | Damaj M, Ahmad D. Biodegradation of polychlorinated biphenyls by rhizobia: a novel finding[J]. Biochemical and Biophysical Research Communications, 1996, 218 (3): 908–915. |
| [25] | Labidi M, Ahmad D, Halasz A, et al. Biotransformation and partial mineralization of the explosive 2,4,6-trinitrotoluene (TNT) by rhizobia[J]. Canadian Journal of Microbiology, 2001, 47 (6): 559–566. |
| [26] | Frassinetti S, Setti L, Corti A, et al. Biodegradation of dibenzothiophene by a nodulating isolate of Rhizobium meliloti[J]. Canadian Journal of Microbiology, 1998, 44 (3): 289–297. |
| [27] | Dutta SK, Hollowell GP, Hashem FM, et al. Enhanced bioremediation of soil containing 2,4-dinitrotoluene by a genetically modified Sinorhizobium meliloti[J]. Soil Biology and Biochemistry, 2003, 35 (5): 667–675. |
| [28] | Chen FY, Liang LL, Tang YB, et al. Isolation and identification of a fungous strain for degrading phenanthrene and its immobilized conditions[J]. Environmental Science & Technology, 2007, 30 (12): 19–21. (in chinese) 陈芳艳, 梁林林, 唐玉斌, 等. 一株菲降解菌的分离鉴定及其固定化条件研究[J]. 环境科学与技术, 2007, 30 (12):19–21. |
| [29] | Mao J, Luo YM, Teng Y, et al. Isolation and characterization of a high-molecular-weight (HMW) PAHs degrading bacterial strain[J]. Microbiology China, 2008, 35 (7): 1011–1015. (in chinese) 毛健, 骆永明, 滕应, 等. 一株高分子量多环芳烃降解菌的筛选、鉴定及降解特性研究[J]. 微生物学通报, 2008, 35 (7):1011–1015. |
| [30] | Masakorala K, Yao J, Cai MM, et al. Isolation and characterization of a novel phenanthrene (PHE) degrading strain Psuedomonas sp. USTB-RU from petroleum contaminated soil[J]. Journal of Hazardous Materials, 2013, 263 : 493–500. |
| [31] | Cai ZQ, Shi S, Li SS, et al. Isolation and characterization of Pseudomonas sp. nai8 capable of naphthalene degradation[J]. Asian Journal of Chemistry, 2014, 26 (1): 164–168. |
| [32] | Zhou W, He D, Li X, et al. Isolation and characterization of naphthalene-degrading strains, Pseudomonas sp. CZ2 and CZ5[J]. African Journal of Microbiology Research, 2013, 7 (1): 13–19. |
| [33] | Johnsen AR, Karlson U. PAH degradation capacity of soil microbial communities—does it depend on PAH exposure?[J]. Microbial Ecology, 2005, 50 (4): 488–495. |
| [34] | Zhong Y, Zou SC, Lin L, et al. Effects of pyrene and fluoranthene on the degradation characteristics of phenanthrene in the cometabolism process by Sphingomonas sp. strain PheB4 isolated from mangrove sediments[J]. Marine Pollution Bulletin, 2010, 60 (11): 2043–2049. |
 2016, Vol. 43
2016, Vol. 43