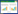扩展功能
文章信息
- 郭棵棵, 杨雨卉, 沈伟, 费文斌, 乐率, 胡福泉
- GUO Ke-Ke, YANG Yu-Hui, SHEN Wei, FEI Wen-Bin, LE Shuai, HU Fu-Quan
- 一株感染铜绿假单胞菌的双链RNA噬菌体PaP6的分离和鉴定
- Isolation and characterization of PaP6, a newly isolated Pseudomonas aeruginosa bacteriophage containing three dsRNA genomic segments
- 微生物学通报, 2016, 43(5): 948-954
- Microbiology China, 2016, 43(5): 948-954
- DOI: 10.13344/j.microbiol.china.151019
-
文章历史
- 收稿日期: 2015-12-09
- 接受日期: 2016-03-02
- 优先数字出版日期(www.cnki.net): 2016-03-04
2. 菲吉乐科生物科技有限公司 江苏 南京 210000
2. Phagelux (Nanjing) Bio-tech Company Limited, Nanjing, Jiangsu 210000, China
铜绿假单胞菌是一种常见的机会致病菌,常导致囊性纤维化病人的肺部感染以及免疫力低下人群的感染,如烧伤和艾滋病人[1]。随着抗生素的广泛应用甚至滥用,铜绿假单胞菌的耐抗生素现象越来越普遍,多重耐药的铜绿假单胞菌越来越常见于临床,给临床治疗带来巨大挑战。因此,噬菌体治疗再次受到人们的关注[2-3]。
噬菌体是专门感染细菌的一类病毒,可以杀死细菌。目前,越来越多的实验证明了铜绿假单胞菌噬菌体治疗的可行性,如Morello等用裂解性噬菌体成功治疗铜绿假单胞菌肺部感染的小鼠[4]。Torres-Barceló等用噬菌体LUZ7和链霉素联合使用,可以更好地杀灭铜绿假单胞菌[5]。2014年欧盟发布了一项噬菌体临床试验“Phagoburn”,在法国、比利时和荷兰招募220名伤口受到铜绿假单胞菌或大肠杆菌感染的烧伤患者,用Pherecydes制药公司生产的噬菌体鸡尾酒制剂进行治疗的临床试验正在进行中[6]。铜绿假单胞菌感染的噬菌体治疗前景被人们看好。
目前,NCBI中可查询到的假单胞菌属噬菌体全基因组序列共有139条(2015年12月1日)。其中,92.8%的噬菌体都属于有尾噬菌体目(Caudovirales),具有双链DNA,根据其噬菌体尾丝的特点可以分为:短尾病毒科(Podoviridae)、肌尾病毒科(Myoviridae)和长尾病毒科(Siphoviridae)。目前已知的感染假单胞菌属的无尾噬菌体仅10株:2株ssDNA噬菌体,属于丝状噬菌体科(Inoviridae),2株ssRNA 噬菌体属于光滑病毒科(Leviviridae family),属于分节段dsRNA囊病毒科(Cystoviridae)的噬菌体有6株,分别是phi6、phi8、phi12、phi13、phiNN和phi2954[7-12]。
囊病毒科噬菌体是一类结构特殊的噬菌体,具有3段双链RNA,且核衣壳外有一层外膜包裹。以研究最为深入的phi6为例,其含有的3节双链RNA分别长6 374、4 063和3 948 bp,共编码14个基因,绝大多数主要为结构蛋白。phi6的完整病毒颗粒具有三层衣壳结构。最内层是由120个P1蛋白组成的核心衣壳,核心衣壳外面由一层P8蛋白包裹。这两层一起称为核衣壳。核衣壳外面是一层脂质包膜,包膜表面有P9、P3和P6三种蛋白,其中P3和P6蛋白是噬菌体的受体吸附蛋白,决定了其宿主的特异性[13]。目前,NCBI中报道的6株囊病毒科噬菌体都以丁香假单胞菌(Pseudomonas syringae)为宿主,从植物表面分离得到。本文首次报道以铜绿假单胞菌为宿主,从西南医院污水中分离到一株具有3节段dsRNA基因组的囊病毒科噬菌体,命名为PaP6 (Pseudomonas aerugingnosa phage 6)。本课题组曾经报道了多株铜绿假单胞菌噬菌体的相关研究,如PaP1[14]、PaP2[15]、PaP3[16],而PaP6是一株国际上首次报道的感染铜绿假单胞菌的囊病毒科噬菌体。本课题对PaP6进行生物学特性的研究,丰富了对噬菌体、尤其是囊病毒科噬菌体多样性的认识,也为今后噬菌体治疗潜力的研究奠定了基础。
1 材料与方法 1.1 材料1.1.1 菌株:112株铜绿假单胞菌临床分离株分别来自重庆西南医院、新桥医院和大坪医院临床科室。标准株PAO1为科室保存。噬菌体PaP6是以临床分离株PA038为宿主菌从西南医院污水中分离得到。
1.1.2 主要试剂:液体LB培养基(1% NaCl,1%胰蛋白胨,0.5%酵母提取物)、固体培养基(含1.5%琼脂粉的液体培养基)、半固体培养基(含0.75%琼脂粉的液体培养基)、RNase A、DNaseⅠ、TE缓冲液(1 mol/L pH 8.0的EDTA,10 mmol/L TrisCl)、1%琼脂糖凝胶、CsCsCl2溶液(密度分别为1.15 g/mL、 1.35 g/mL、1.50 g/mL、1.70 g/mL)。
1.1.3 主要仪器:JM-250核酸电泳仪,大连捷迈科技发展有限公司;透射式电镜,Philips;高速低温离心机,Sigma;核酸凝胶扫描仪、分光光度计、蛋白凝胶扫描仪,Bio-Rad。
1.2 方法1.2.1 细菌和噬菌体的分离:从西南医院污水处理站取得的污水,10 000×g离心10 min,取上清液,用0.22 μm无菌滤膜过滤除菌。向过滤后的上清液中加入200 mL液体LB[16]和3 mL培养至对数早期的临床菌株PA038菌液。置于37 °C、 220 r/min培养过夜。次日,将混合液10 000×g离心10 min,取上清,用0.22 μm无菌滤膜过滤除菌。将滤液进行10倍的梯度稀释后,从各个梯度中分别取100 μL与100 μL宿主菌PA038混合,室温静置15 min。加入大约50 °C、0.75%琼脂的半固体LB 3 mL,充分混匀后,倒于普通的LB平板上,半固体胶凝固后,将双层琼脂平板置于37 °C培养过夜。次日观察是否有噬斑长出,并观察噬斑特点。待噬斑形成后,挑取单个噬斑,将其接种于培养至对数早期的宿主菌中,进一步纯化和扩增分离到的噬菌体,低温保存备用。
1.2.2 噬菌体基因组的提取和鉴定:采用天根生化科技(北京)有限公司的病毒基因组DNA/RNA提取试剂盒(Lot:N2603)进行抽提。抽提得到基因组后,分别进行DNase和RNase酶切,并进行琼脂糖电泳观察结果。
1.2.3 噬菌体的透射电镜观察:CsCl2梯度离心纯化噬菌体颗粒,80 000×g离心1.5 h,然后取20 μL纯化后的噬菌体,滴加在铜网上,自然沉淀15 min,用滤纸从侧面吸取多余液体,加1滴2%磷钨酸于铜网上,染色30 s,同样将多余的染液吸干,干燥后电镜观察。
1.2.4 噬菌体对氯仿的敏感性:取PaP6噬菌体 200 μL,加入等体积的氯仿,充分振荡混合1 min。10 000×g离心10 min。取上层液体,即氯仿处理后的噬菌体。将氯仿处理前后的噬菌体液各1 μL分别滴加在含有宿主菌的双层琼脂平板上,观察氯仿处理后噬菌体是否形成噬斑,以确定其是否具有繁殖活性。
1.2.5 最佳感染复数测定:感染复数(Multiplicity of infection,MOI)是指初始感染时加入噬菌体的总数与宿主菌总数的比值。方法是挑取宿主菌单菌落接种于3 mL液体LB中,37 °C、220 r/min振荡培养至OD600=0.2,相当于1×108 CFU/mL。按照1:10 000、1:1 000、1:100、1:10、1:1、10:1六种不同的比例加入噬菌体和宿主菌,37 °C、220 r/min振荡培养至宿主菌被裂解澄清,再10 000×g离心10 min,收集上清,并用噬斑实验测定噬菌体滴度。产生最高噬菌体滴度的比例即为最佳感染复数。
1.2.6 噬菌体的一步生长曲线:一步生长曲线是指从噬菌体吸附到宿主菌被裂解而释放子代噬菌体这样一个完整的增殖周期,它反映的是噬菌体在一个复制周期中的数量增殖变化规律。方法是将噬菌体比宿主菌为10:1充分混合,置37 °C、220 r/min振荡培养15 min后,13 000×g离心30 s,弃上清,用1 mL液体LB洗涤3次后,再用预热的LB液混悬沉淀(总体积为5 mL)并将其充分混匀,然后迅速置于37 °C摇床中160 r/min振荡培养,同时开始计时,分别在0时刻和每隔10 min各取样50 μL,共取样2 h。取样后,立即13 000×g离心30 s,吸取上清后用双层琼脂平板法测定噬菌体滴度。实验生物学重复3次。最后将实验所得数据进行统计绘图,得出PaP6的潜伏期、暴发期和暴发量3个参量。
1.2.7 噬菌体结构蛋白的分析:参考萨姆布鲁克《分子克隆实验指南》第3版的提取方法,略有改动。取PaP6噬菌体原液600 mL,加入固体PEG8000 (聚乙二醇)至终浓度10% (质量体积比),振荡溶解后冰浴1 h。然后,4 °C、12 000×g离心10 min,尽量弃掉上清;用30 mL TM液 (0.05 mol/L Tris-HCI,pH 7.5,0.2% MgSO4·7H2O)混悬沉淀。然后对噬菌体粗制颗粒做CsCsCl2密度梯度离心,对样本进行浓缩,再进行SDS-PAGE。SDS凝胶,分离胶浓度为12%,浓缩胶浓度为4%,上样量为50 μL,电泳采用横流20 mA、100 min。
1.2.8 PaP6的宿主谱研究:利用噬斑实验检验PaP6对112株临床菌株和PAO1的裂解能力。
2 结果与分析 2.1 噬菌体的噬斑特征从西南医院污水中分离得到噬菌体PaP6。在平板上可观察到直径为2 mm−4 mm的圆形噬斑,呈透明样,边界清晰,表现出裂解性噬菌体的噬斑特征(图 1A)。噬菌体在液体培养过程中,可以完全将宿主菌裂解澄清,因此,初步断定PaP6为裂解性噬菌体。
|
| 图 1 噬菌体PaP6噬斑、电镜形态和氯仿敏感实验 Figure 1 PaP6 plaques,electron microscopy photograph of phage and chloroform sensitive experiment A:噬菌体PaP6 噬斑;B:PaP6电镜形态;C:氯仿敏感实验. A: PaP6 plaques; B: Electron microscopy photograph of PaP6; C: Chloroform sensitive experiment. |
|
|
CsCsCl2密度梯度离心纯化噬菌体PaP6后,用透射电镜进行观察。电镜照片可见,噬菌体PaP6呈二十面体结构,直径大小约45 nm (图 1B),未见尾部结构。
2.3 噬菌体对氯仿敏感将噬菌体PaP6与等体积氯仿混合振荡1 min后,噬菌体完全失去感染活性,不能形成噬斑(图 1C),表明噬菌体PaP6具有脂质囊膜结构,对氯仿敏感。但在电镜放大12万倍的条件下,未能看清楚噬菌体的囊膜结构。
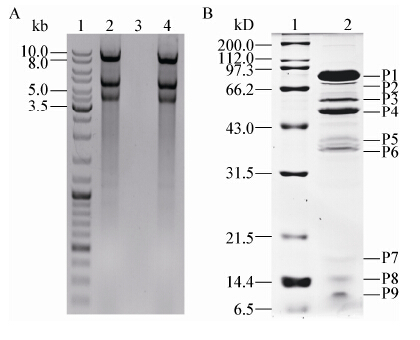
2.4 噬菌体的核酸类型鉴定图 2A为噬菌体基因组的酶切鉴定,泳道2显示未用核酸酶切的PaP6的基因组有3条带,说明天然的PaP6基因组由3个节段组成。图 2A中的3、4泳道显示,3个节段的基因组均可被RNA酶降解,而不能被DNA酶降解,说明PaP6的基因组为3节段的双链RNA。3节段大小分别约为:9.0、4.5、3.5 kb (图 2A)。
|
| 图 2 噬菌体PaP6全基因组电泳结果和结构蛋白电泳图谱 Figure 2 PaP6 genome and SDS-PAGE analysis of PaP6 structure proteins A:PaP6基因组电泳图. 1:蛋白分子量标准;2:未酶切的PaP6基因组;3:用RNA酶酶切后的PaP6基因组;4:用DNA酶酶切后的PaP6基因组. B:结构蛋白SDS-PAGE电泳图谱. 1:蛋白分子量标准;2:PaP6噬菌体结构蛋白. A: Agarose gel electrophoresis of PaP6 genome. 1: Protein ladder; 2: PaP6 genome; 3: RNase digestion of the PaP6 genome; 4: DNase digestion of the PaP6 genome. B: SDS-PAGE analysis of PaP6 structure proteins. 1: Protein ladder; 2: Structural protein of PaP6. |
|
|
SDS-PAGE主要是对噬菌体PaP6的结构蛋白进行分析。由电泳结果可知,凝胶上至少出现了9条蛋白条带,说明PaP6至少有9种结构蛋白(图 2B)。
2.6 最佳感染复数的测定按照不同比例加入噬菌体和宿主菌,培养 3.5 h后,测定各管中的噬菌体滴度。结果如表 1所示,当噬菌体与细菌的比例为0.1,产生的子代噬菌体滴度最高(8.5×109 PFU/mL)。因此,PaP6的最佳感染复数为0.1。
| 管号 Tube No. | 细菌数 Number of bacteria | 噬菌体数 Number of bacteriophages | 感染复数 MOI | 3.5 h 后滴度Titer at 3.5 h (PFU/mL) |
| 1 | 1×108 | 1×109 | 1×101 | 2.5×107 |
| 2 | 1×108 | 1×108 | 1×100 | 3.3×107 |
| 3 | 1×108 | 1×107 | 1×10−1 | 8.5×109 |
| 4 | 1×108 | 1×106 | 1×10−2 | 3.4×109 |
| 5 | 1×108 | 1×105 | 1×10−3 | 4.5×107 |
| 6 | 1×108 | 1×104 | 1×10−4 | 2.8×107 |
噬菌体的繁殖经历3个时期,即潜伏期、裂解期和平台期。潜伏期是指从噬菌体开始吸附至细菌开始释放出新的噬菌体之间的一段时期;裂解期是被感染细菌开始裂解至全部菌细胞都被裂解之间的一段时期;平台期是指裂解期之后,噬菌体滴度保持稳定的时期。一步生长曲线显示,PaP6感染宿主菌的潜伏期约为10 min,裂解期约为50 min (图 3),50 min之后滴度变化不大,为平台期。根据裂解量计算公式:裂解量=暴发末期噬菌体滴 度÷感染初期宿主菌浓度,即得出PaP6感染宿主菌PA038的裂解量约为10。

|
| 图 3 噬菌体PaP6一步生长曲线 Figure 3 One-step growth curve of PaP6 |
|
|
通过对112株临床菌株和标准株PAO1进行噬斑实验,发现PaP6不能裂解标准株PAO1,但是能裂解40.1%的临床分离株,说明PaP6是一株相对广谱的噬菌体。
3 讨论噬菌体是最具生物多样性的一类生物。有报道称生物圈中的噬菌体超过1031种,目前已分离鉴定的噬菌体仅1万种左右。因此,分离鉴定尽可能多的新噬菌体,对于人们了解噬菌体的生物多样性及其基因多样性和生物学特性具有重要意义。
我们首次分离得到一株感染铜绿假单胞菌的dsRNA噬菌体。其噬菌体颗粒形态为对称的二十面体结构,基因组为3节段的双链RNA,大小分别约为9.0、4.5和3.5 kb,总共约17 kb。氯仿敏感实验证实PaP6对氯仿敏感,说明PaP6是具有囊膜的噬菌体。PaP6的SDS-PAGE结果显示其具有9个明显的结构蛋白,其它几株囊病毒科噬菌体也具有8−9个结构蛋白,与PaP6的蛋白电泳图类 似[9, 11]。根据噬菌体的形态学特征、有无尾部结构、有无囊膜、基因组结构特征、对氯仿的敏感性以及结构蛋白特征,我们初步将PaP6确定为囊病毒科噬菌体。目前,囊病毒科噬菌体的代表株是噬菌体phi6,已报道属于囊病毒科的噬菌体还包括phi8、phi12、phi13、phiNN和phi2954,但之前报道的这6株噬菌体均是以丁香假单胞菌(植物寄生菌)为宿主分离得到的。本研究分离鉴定的PaP6,是首次报道的一株能感染人类病原体铜绿假单胞菌的dsRNA噬菌体,并将其分类到囊病毒科。这说明囊病毒科噬菌体不仅仅限于感染丁香假单胞菌,也有感染铜绿假单胞菌的噬菌体。推测囊病毒科噬菌体在自然界的分布可能更为广泛。
铜绿假单胞菌是一种临床常见的机会致病菌,耐药现象常常困扰着临床治疗。因此,铜绿假单胞菌的噬菌体治疗已经从动物实验走向临床应用阶段了。噬菌体治疗相对抗生素治疗有一个缺点,即噬菌体具有高度的菌株特异性,可以杀死某些菌株,往往不能杀死绝大多数其他菌株[17]。因此,噬菌体治疗大多需要制备含多种噬菌体的鸡尾酒混合制剂,以提高其裂菌谱的覆盖率。在这种情况下,广谱噬菌体的分离和应用则能更好地用于临床治疗。本课题分离的PaP6噬菌体宿主谱相对较广,能感染约40.1%的临床菌株。因此,PaP6有用于噬菌体鸡尾酒治疗的更大潜力。噬菌体鸡尾酒制剂常常需要根据细菌的变异情况而进行调整,补充或替换一些噬菌体。因此,根据细菌变异情况而分离新的不同噬菌体来重新组合噬菌体鸡尾酒具有重要意义。
另外,用一株噬菌体通过试管内人工进化、突变,构建具有巨大库容量的噬菌体突变库,从中筛选能裂解目标病原体的策略,也是具有潜力的研究方向之一。细菌对噬菌体的不敏感性也可以理解为细菌对噬菌体的耐受,而噬菌体又可通过突变来再次感染耐受了的细菌,这种噬菌体-宿主菌之间的寄生与反寄生历程是在相互斗争中的共进化现象[18]。如,Ferris等利用phi6快速筛选到了40株可感染新宿主菌的突变噬菌体[19],而Paterson等用荧光假单胞菌及其噬菌体phi2进行共进化实验,发现细菌-宿主共进化4周后,新的噬菌体的宿主谱得到扩 展[20]。dsRNA的丁香假单胞菌噬菌体phi6出现感染新宿主的突变概率是1.156×10−6[21],而dsDNA的铜绿假单胞菌噬菌体PaP1出现感染新宿主的突变的概率约1×10−8[17],也就是说RNA噬菌体的突变率相对DNA噬菌体高出约100倍。这是因为dsRNA噬菌体复制时需要RNA依赖的RNA聚合酶,RNA聚合酶没有DNA聚合酶的保真性高,因此RNA噬菌体(或者RNA病毒)常常具有较高的突变率[19],利用dsRNA噬菌体进行共进化实验更容易筛选到宿主谱突变或扩展宿主谱的噬菌体。这可能也是应对细菌突变而快速筛选新的突变噬菌体的方法之一。因此,本课题组下一步将围绕PaP6的基因组特点及其突变和进化规律进行深入研究。
| [1] | Berrazeg M, Jeannot K, Enguéné VYN, et al. Mutations in β-Lactamase AmpC increase resistance of Pseudomonas aeruginosa isolates to antipseudomonal cephalosporins[J]. Antimicrobial Agents and Chemotherapy, 2015, 59 (10): 6248–6255. |
| [2] | Beeton ML, Alves DR, Enright MC, et al. Assessing phage therapy against Pseudomonas aeruginosa using a Galleria mellonella infection model[J]. International Journal of Antimicrobial Agents, 2015, 46 (2): 196–200. |
| [3] | Pires DP, Boas DV, Sillankorva S, et al. Phage therapy: a step forward in the treatment of Pseudomonas aeruginosa infections[J]. Journal of Virology, 2015, 89 (15): 7449–7456. |
| [4] | Morello E, Saussereau E, Maura D, et al. Pulmonary bacteriophage therapy on Pseudomonas aeruginosa cystic fibrosis strains: first steps towards treatment and prevention[J]. PLoS One, 2011, 6 (2): e16963. |
| [5] | Torres-Barceló C, Arias-Sánchez FI, Vasse M, et al. A window of opportunity to control the bacterial pathogen Pseudomonas aeruginosa combining antibiotics and phages[J]. PLoS One, 2014, 9 (9): e106628. |
| [6] | Resch G, Que YA, Gabard J, et al. Update on PHAGOBURN, the First European clinical trial of phage therapy[A]//Abstracts of the General Meeting of the American Society for Microbiology[M]. Boston, Massachusetts: American Society for Microbiology, 2014, 114: 1277 |
| [7] | Gottlieb P, Wei H, Potgieter C, et al. Characterization of variant φ 12, a bacteriophage related to variant φ 6: nucleotide sequence of the small and middle double-stranded RNA[J]. Virology, 2002, 293 (1): 118–124. |
| [8] | Hoogstraten D, Qiao XY, Sun Y, et al. Characterization of Φ 8, a bacteriophage containing three double-stranded RNA genomic segments and distantly related to Φ 6[J]. Virology, 2000, 272 (1): 218–224. |
| [9] | Mäntynen S, Laanto E, Kohvakka A, et al. New enveloped dsRNA phage from freshwater habitat[J]. Journal of General Virology, 2015, 96 (5): 1180–1189. |
| [10] | Qiao XY, Qiao J, Onodera S, et al. Characterization of Φ 13, a bacteriophage related to Φ 6 and containing three dsRNA genomic segments[J]. Virology, 2000, 275 (1): 218–224. |
| [11] | Qiao XY, Sun Y, Qiao J, et al. Characterization of Φ 2954, a newly isolated bacteriophage containing three dsRNA genomic segments[J]. BMC Microbiology, 2010, 10 : 55. |
| [12] | Vidaver AK, Koski RK, van Etten JL. Bacteriophage φ 6: a lipid-containing virus of Pseudomonas phaseolicola[J]. Journal of Virology, 1973, 11 (5): 799–805. |
| [13] | Katz A, Alimova A, Futerman E, et al. Bacteriophage φ6-structure investigated by fluorescence stokes shift spectroscopy[J]. Photochemistry and Photobiology, 2012, 88 (2): 304–310. |
| [14] | Lu SG, Le S, Tan YL, et al. Genomic and proteomic analyses of the terminally redundant genome of the Pseudomonas aeruginosa Phage PaP1: establishment of genus PaP1-Like phages[J]. PLoS One, 2013, 8 (5): . |
| [15] | Huang JJ, Hu XM, Rao XC, et al. Biological characteristics of Pseudomonas aeruginosa phage PaP2[J]. Acta Academiae Medicinae Militaris Tertiae, 2004, 26 (13): 1133–1136. (in chinese) 黄建军, 胡晓梅, 饶贤才, 等. 铜绿假单胞菌噬菌体PaP2生物学特性的研究[J]. 第三军医大学学报, 2004, 26 (13):1133–1136. |
| [16] | Zhou YB, Shen XD, Li M, et al. The biological characterization of Pseudomonas aeruginosa phage PaP3[J]. Medical Journal of Chinese People's Liberation Army, 2006, 31 (10): 999–1001. (in chinese) 周莹冰, 申晓冬, 李明, 等. 铜绿假单胞菌噬菌体PaP3生物学特性的研究[J]. 解放军医学杂志, 2006, 31 (10):999–1001. |
| [17] | Le S, He XS, Tan YL, et al. Mapping the tail fiber as the receptor binding protein responsible for differential host specificity of Pseudomonas aeruginosa bacteriophages PaP1 and JG004[J]. PLoS One, 2013, 8 (7): e68562. |
| [18] | Labrie SJ, Samson JE, Moineau S. Bacteriophage resistance mechanisms[J]. Nature Reviews Microbiology, 2010, 8 (5): 317–327. |
| [19] | Ferris MT, Joyce P, Burch CL. High frequency of mutations that expand the host range of an RNA virus[J]. Genetics, 2007, 176 (2): 1013–1022. |
| [20] | Paterson S, Vogwill T, Buckling A, et al. Antagonistic coevolution accelerates molecular evolution[J]. Nature, 2010, 464 (7286): 275–278. |
| [21] | Ford BE, Sun B, Carpino J, et al. Frequency and fitness consequences of bacteriophage Φ 6 host range mutations[J]. PLoS One, 2014, 9 (11): e113078. |
 2016, Vol. 43
2016, Vol. 43