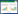扩展功能
文章信息
- 吴清平, 李玉冬, 张菊梅
- WU Qing-Ping, LI Yu-Dong, ZHANG Ju-Mei
- 常见食源性致病菌代谢组学研究进展
- Advances in metabolomics of common food-borne pathogen
- 微生物学通报, 2016, 43(3): 609-618
- Microbiology China, 2016, 43(3): 609-618
- 10.13344/j.microbiol.china.150478
-
文章历史
- 收稿日期: 2015-06-23
- 接受日期: 2015-10-10
- 优先数字出版日期(www.cnki.net): 2015-12-16
2. 中国科学院广州化学研究所 广东 广州 510650;
3. 中国科学院大学 北京 100049
2. Guangzhou Institute of Chemistry,Chinese Academy of Sciences,Guangzhou,Guangdong 510650,China;
3. University of Chinese Academy of Sciences,Beijing 100049,China
随着国内外对微生物食品安全问题的更加重视,世界各国针对食源性病原微生物的监控和溯源工作不断加强。据中国疾病预防控制中心(CDC)统计,我国最近十年由食源性致病微生物引起的食品安全事件比例高达60%以上,因此针对食源性致病微生物监测和控制的研究亟待加强。近年来,本实验室通过进行全国主要食品中致病微生物风险调查和识别研究,分离了约16 000株包括沙门氏菌(Salmonella)、副溶血性弧菌(Vibrio parahaemolyticus)、单核细胞增生李斯特菌(Listeria monocytogenes)、小肠结肠炎耶尔森氏菌(Yersinia enterocolitica)、大肠杆菌O157 (Escherichia coli O157)、阪崎肠杆菌(Enterobacter sakazakii)、金黄色葡萄球菌(Staphylococcus aureus)、空肠弯曲杆菌(Campylobacter jejuni)和蜡样芽胞杆菌(Bacillus cereus)等食源性致病微生物,为深入开展食源性致病微生物鉴别和控制研究打下了较好的基础[1, 2, 3]。迄今为止,随着分子测序技术的进步,部分食源性致病微生物的基因组被国内外成功测序,根据基因组等信息发掘,已探索出各种检测新靶标。此外,由于不同种属致病微生物、相同种属不同来源的菌株代谢产物均可能存在较大差异。因此寻找各种微生物的特征性代谢产物及代谢产物谱已成为目前病原微生物快速检测和鉴定研究的一个重要方向,国际上已经初步建立了微生物挥发性代谢产物数据库[Microbial Volatile Metabolites (MVOCs) Database]。
由于代谢组学主要研究某一时刻细胞内外所有代谢产物的集合,因此可以通过代谢组学技术快速获得细胞内外整体代谢产物信息,分析细胞间差异和用于样品分类。如图 1所示,目前代谢组学技术主要的分析方法有靶标代谢组学分析、非靶标代谢组学分析(代谢轮廓分析、代谢指纹分析和代谢足迹分析)。靶标代谢组学分析主要研究一些特定的代谢产物及其代谢途径。非靶标代谢组学主要定性或半定量分析细胞内外整体代谢产物,是获得细胞内外全面代谢产物信息的主要技术手段。例如凭借食品组学[4]来控制食品安全和质量的出发点就是通过非靶标代谢组学技术对食品进行全面的表征,其中包括代谢相关基因对食物影响[5](转基因前后代谢产物差异),食品原材料生产、采集和加工过程中在不同胁迫因子下代谢产物变化(例如食源性致病菌污染[6]),食物的营养状况,质量等级(样品组成成分和质量),食物外源性物质(农药、兽药和添加剂等)和环境因素影响下的食物代谢产物变化(例如生长在不同地区食物代谢产物差异[7])。随着代谢组学技术的逐步成熟,目前不断用于监控食品安全质量,尤其在借助代谢组学技术研究食源性致病微生物代谢组特征,并用以鉴别和监控食源性致病微生物上运用更为成熟[8]。
基于代谢组学食源性致病菌代谢产物研究主要集中在胞内代谢轮廓分析[9, 10, 11, 12, 13]和胞外挥发性代谢轮廓分析[14, 15],并发现通过代谢指纹图谱可以将不同菌种明显区分开来,且极可能发现生物标志分子。因此,以代谢组学技术为基础的常见食源性致病菌代谢轮廓研究是食源性致病菌代谢产物数据库建立和特征性代谢产物研究的基础。本文针对近年来食源性致病菌代谢组学研究方法和应用状况进行归纳,并重点阐述食源性致病菌代谢产物数据库建立和潜在生物标志物在食源性致病菌快速检测技术研发等方面的应用前景,以期为建立食源性致病微生物代谢组学快速检测技术提供参考。
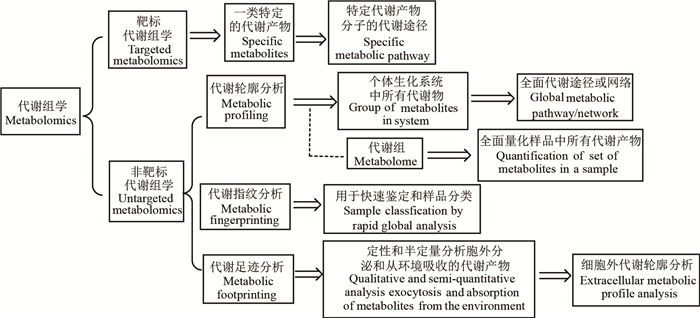
1 常见食源性致病菌代谢组学研究方法通过非靶标代谢组学技术研究食源性致病菌代谢产物轮廓是建立食源性代谢产物数据库基础,同时,研究不同食源性致病菌间代谢轮廓相似性和差异性是寻找不同菌种之间潜在生物标志物分子必要手段。食源性致病菌代谢组学研究方法主要如图 2所示。
由于细胞受环境变化影响,其生理机能会发生较大改变,产生一些其他的代谢产物干扰检测结果,其次不同的提取方法得到的代谢产物不同,因此样品前处理在特征性代谢产物研究中尤为关键。目前常采用大肠埃希氏杆菌(E. coli)作为食源性致病菌前处理优化模式菌,常见食源性致病菌前处理方法见表 1。除主要采用冷甲醇和液氮淬灭外,还常使用酸碱淬灭,但在代谢轮廓分析中不常用,主要由于其容易造成代谢产物发生理化反应而整体失真。
| 细菌 Bacteria | 淬灭 Quenching | 提取 Extractions | 仪器分析 Analyses | 相关文献报道 References |
| E. coli | (60%,-48 °C)甲醇 | 冷冻-解冻甲醇提取 MeOH (-48 °C) | GC/TOF-MS | [17] |
| E. coli,B. subtilis | -45 °C纯甲醇 | CHCl3 (-45 °C) | GC/MS和LC/MS | [18] |
| P. aeruginosa,S. typhimurium, S. aureus,E. coli | None | 冷甲醇/H2O (9:1) 溶液 (-80 °C) | GC-MS/TOF | [19] |
| Vibrio parahaemolyticus | 液氮 液氮 | 75%甲醇(-80 °C) 75%甲醇(-80 °C) | GC-MS UPLC/Q-TOF-MS | [20] [21] |
| B. subtilis | 液氮 | 60%甲醇/纯水再提取 | LC/MS GC-MS | [22] |
| E. coli,E. coli O157:H7,P. aeruginosa,Salmonella,S. aureus | None | 冷甲醇提取 | GC-MS | [23] |
| E. coli,B. subtilis,B. megaterium | None | 50/50 MeOH/H2O/超声辅助提取 | LC-MS | [24] |
细胞内代谢产物提取时,主要的提取溶剂有冷甲醇液、甲醇/氯仿等,同时配合超声波破碎、玻璃球磨、循环冻融和微波等辅助处理等。本文作者以E. coli为模式菌株,通过高压细胞破碎、超声破碎和微波辅助等提取方法进行优化前处理方法,发现超声辅助6:3:1的CH3OH/CH2Cl2/H2O提取效果最优,并用于其他常见食源性致病菌代谢轮廓分析。Schaefer等[25]使用自动制样装置,以4.5 Hz采样频率对大肠杆菌胞内代谢动力学进行监控,为后续自动化高通量检测提供参考。
此外,由于细胞内酶活性较强和淬灭产生的胞内代谢产物泄漏等因素造成重现性较差,同时难以寻找普适的淬灭及提取方法,而胞外代谢产物半衰期较长,较为稳定,操作简单,利于高通量分析,因此是寻找食源性特征性代谢产物较为理想途径。目前针对微生物代谢产生的一些小分子挥发性物质(如酸类、酮类、醛类、醇类、芳香类及杂环化合物类等)报道较多。Lemfack等[26]特别针对微生物挥发性代谢产物初步建立了一个在线数据库http://bioinformatics.charite.de/mvoc。相对于挥发性代谢产物,胞外代谢足迹分析报道较少,虽然只能显示部分代谢组学信息,但它能提供重要的功能基因信息和菌株之间的差异信息,对环境变化进行应答时细胞代谢释放一些特异性代谢产物信息,同时有助于了解细胞之间的交流机制、代谢工程和生物技术产业过程等[27],对完善食源性代谢产物数据库和寻找特征性代谢产物具有重要意义。
食源性致病菌挥发性代谢产物研究方法与胞内代谢产物分析比较而言方法较为简单,并且已有专门针对于挥发性物质实时监控仪器出现,例如选择性离子流管质谱(SIFT-MS)及电子鼻设备等。目前主要的挥发性代谢产物提取方法有顶空固相微萃取、顶空直接进样、吹扫捕集和动态顶空提取[28]等。Tait等[29]针对不同培养基、不同的固相微萃取柱头及极性和非极性毛细管色谱柱对挥发性代谢产物产生、提取及检测等影响进行研究,发现在EF培养基中,E. coli的发酵液中并没有检测到吲哚。不同的固相微萃取柱头对不同菌的萃取效果不同,例如DVB-CAR-PDMS固相微萃取头对于金黄色葡萄球菌的萃取效果最佳,而对于克雷伯氏肺炎菌3种萃取柱头萃取效果类似,说明金黄色葡萄球菌含有较多的痕量MVOCs,而其他菌种的挥发性代谢产物含量较高。因此,虽然挥发性代谢产物研究方法较为简单,操作方便,但仍存在挥发性物质难以完全富集、分离和检测等问题。
1.2 分析技术平台对于分析细胞产生的复杂微量代谢产物,需要采用高效、高分辨、灵敏的分离检测技术。近年来,国内外对食源性致病菌特征性代谢产物的分析手段主要是通过GC/HPLC/CE-MS、MS/MS和NMR等[30, 31],并不断出现专门针对常见食源性致病菌挥发性代谢产物检测仪器。Marcinowska等[19]通过GC-MS分析了假结核耶尔森菌25%的代谢产物,同时通过GC-MS/TOF分析了铜绿假单胞菌(P. aeruginosa)、鼠伤寒耶尔森杆菌(Y. pestis)、E. coli和金黄色葡萄球菌胞内代谢产物,发现各菌株间代谢产物存在显著差异。Wu等[24, 32]通过LC-MS同位素标记定量分析大肠埃希氏杆菌和芽孢杆菌时,结合多元分析发现每种细菌均含有2 500余种代谢产物,同时他们利用LC检测出样品中由丹磺酰标记后的代谢产物总量。随着代谢组学发展,不断出现高效分离色谱如GC×GC、LC×LC及新型高灵敏高分辨MS检测器来辅助色谱分离鉴定生物分子,例如三重四级杆质谱(QQQ-MS)、基质辅助激光解析电离质谱(MALDI-MS)、四级杆串联飞行时间质谱(Q-TOF-MS)、离子阱串联飞行时间质谱(Trap-TOF-MS-MS)、傅里叶变换离子回旋共振质谱仪(FT-ICR-MS)及基质辅助激光解析电离飞行时间质谱(MALDI-TOF-MS)等MS检测器,并且陆续出现相应的LC-MS代谢产物数据库。
1.3 数据分析平台食源性致病菌代谢产物经色谱分离后,产生的每个信号峰均包含代谢产物中各种物质定性和定量信息,特别是分析使用不同样品前处理手段分析大量样品时,生成含有大量多变量数据,常规数据分析方法已不适用,不能直接用于食源性致病菌代谢组学研究,需转换为代谢组学研究格式。目前主要的代谢产物数据分析方法[33]为聚类分析(CA)、PCA、线性判别分析(LDA)、偏最小二乘法(PLS)、正交-偏最小二乘投影判别分析(OPLS-DA)和ANN等。例如Cevallos-Cevallos等[23]通过PCA方法分析肠杆菌科中沙门氏菌和大肠杆菌时发现,通过简单PCA很难将上述两种菌分开,但通过PLS模型预测分析,在鸡肉和牛肉中沙门氏菌响应标准差(MSE)和预测MSE值均98%以上,而E. coli O157:H7的预测值达到100%。Marcinowska等[19]使用PCA分析铜绿假单胞菌、鼠伤寒沙门氏菌、大肠埃希氏菌和金黄色葡萄球菌时发现,大肠埃希氏菌和鼠伤寒沙门氏菌代谢产物较类似,但能明显区分,与革兰氏阳性菌相比具有较大差异,肠杆菌与假单胞菌属之间差异也较大。此外,本文作者通过对13种常见食源性致病菌进行胞内外代谢轮廓分析,结合PCA主成分分析和聚类分析,发现各菌株代谢产物之间存在较大差异,且蜡样芽孢杆菌及其类似菌株(苏云金芽孢杆菌和蕈状芽孢杆菌)可以通过胞内代谢轮廓进行聚类区分,并发现蜡样芽孢杆菌、苏云金芽孢杆菌和蕈状芽孢杆菌在L-脯氨酸代谢途径中的差异,同时发现部分潜在的生物标志分子。
1.4 食源性致病菌代谢产物数据库建立微生物代谢产物受环境变化而呈现多样性变化,使得特征性代谢产物分离鉴定成为食源性致病菌代谢组学研究的重点和难点,同时单纯的质谱图比对较化合物鉴定意义不大,但建立微生物代谢组学数据库将加速化合物的鉴定和菌种识别[34]。现阶段主要的微生物代谢产物数据库如表 2所示。目前从基因组水平上结合基因组学和代谢轮廓完成重建代谢网络只有大肠杆菌[34]、蜡样芽孢杆菌[35]和酵母菌[36],尚缺乏标准食源性致病菌代谢产物数据库。本实验室在系统调查全国食源性致病菌污染状况和风险识别的基础上,分离和保藏了大量菌株,并建立起致病菌菌种资源库,其中部分食源性致病微生物的基因组被成功测序。为探索各种检测靶标,拟从上述常见食源性致病菌出发,通过上述代谢组学研究方法对分离菌株和标准菌株进行代谢轮廓分析,建立常见食源性致病菌代谢产物数据库。此外,结合蛋白质组学进行代谢途径分析,了解代谢产物之间关系及其相关基因表达情况,从而建立较为完善的食源性致病菌代谢网络。此外,通过靶标代谢组学技术研究特征性代谢产物代谢途径,可获得各菌株间的特异性酶及酶底物,对寻找食源性致病菌新型检测靶点具有重要意义[37, 38]。
| 序号 serial number | 数据库 Datebase | 序号 serial number | 数据库 Datebase |
| 1 | HMP: The Human Microbiome Project | 9 | HMDB:Human Metabolome Database |
| 2 | EcoCyc: Encyclopedia of Escherichia coli K-12 Genes and Metabolism | 10 | BiGG Database |
| 3 | ECMDB: The Escherichia coli Metabolome Database | 11 | SYSTOMONAS genome Database |
| 4 | YMDB: The Yeast Metabolome Database | 12 | LIPID MAPS Database |
| 5 | NMD: National Microbiological Database | 13 | KEGGPATHWAY Database |
| 6 | MNPD: Microbial Natural Products Database | 14 | NCBI -PubChem Compound |
| 7 | UMBBD: University of Minnesota Biocatalysis/Biodegradation Database | 15 | MetaCyc Encyclopedia of Metabolic Pathways |
| 8 | BioCyc Pathway/Genome Databases and Pathway Tools Software | 16 | Microbial Volatile Metabolites Database |
食源性致病菌代谢组学研究是建立食源性代谢产物数据库的基础,同时,研究不同食源性致病菌间代谢产物相似性和差异性是寻找菌间潜在生物标志物分子的必要手段。目前,通过代谢组学研究发现不同致病微生物间的代谢产物差异性显著,菌间存在多种特征性代谢产物(表 3)。研究发现,细胞内最常见的生物标志物为脂类、多糖、脂多糖、分枝菌酸、胞壁酸和醌类等化合物及其他一些小分子化合物。此外,针对上述不同种类生物标志分子建立了多种商品化细菌鉴定系统,如细菌脂肪酸GC鉴定系统,细菌分枝菌酸HPLC鉴定系统等。采用常规鉴定方法难以区分蜡样芽孢杆菌、苏云金芽孢杆菌和炭疽芽孢杆菌,但其胞内碳水化合物轮廓可以将上述菌株明显区分,进一步研究还发现炭疽芽孢杆菌形成芽孢时产生鼠李糖和3-甲氧基鼠李糖,而蜡样芽孢杆菌形成芽孢时产生3-甲氧基鼠李糖和异鼠李糖,此外苏云金芽孢杆菌和蜡样芽孢杆菌中还含有海藻糖和2-甲氧基鼠李糖[39]。Hamid等[40]利用纸喷雾质谱对60多种菌种进行直接检测,质谱鉴别发现主要物质为磷脂质,且可以将细菌进行种间分类区分,同时革兰氏阳性菌(G+)预测模型预测成功率达到98%,革兰氏阴性菌(G-)可达到87%。
| 标志物类型Biomarker type | 生物标志分子Biomarker molecules | 微生物Microorganism | |
| 挥发性代谢产物Volatile metabolites | 长链脂肪醇 | Enterobacteriaceae family [15,16,41,42] | |
| 短链脂肪酸 | Anaerobic bacteria [43] | ||
| 氨 | S.aureus,P.aeruginosa [44,45] | ||
| 2-氨基苯乙酮,1-十一碳烯,2,4-二甲基庚烷,2-丁酮,4-甲基喹唑啉,HCN,甲基酮,硫氰酸甲酯 | P.aeruginosa [14,15,16,17,18,19,20,21,22,23,24,25,26,27,28,29,30,31,32,33,34,35,36,37,38,39,40,41,42,43,44,45,46] | ||
| 异戊酸,2-甲基丁醛 | S.aureus [15,47] | ||
| 甲醇,戊醇,乙酸乙酯,吲哚 | E.coli [14,48,49] | ||
| 乙酸,乙醇 | S.typhimurium [50] | ||
| 非挥发性代谢产物Non volatile metabolites | 细胞内Intracellular | 脂类为最常见,已建立相关脂肪酸GC鉴定系统(美国MIDI公司) | |
| 细菌分枝菌酸HPLC鉴定系统(美国MIDI公司的Sherlock分枝菌酸鉴定系统SMIS) | Mycobacterium | ||
| 多糖:多糖指纹图谱分析可区分不同致病菌,例如生成芽孢后的炭疽芽孢杆菌鼠李糖和3-O-甲基鼠李糖,蜡样芽孢杆菌和苏云金芽孢杆菌还含有海藻糖和2-O-甲基鼠李糖[39] | |||
| DPA(吡啶-2,6-二羧酸) | 区分细菌芽胞和繁殖体[51] | ||
| 脂寡糖和脂多糖(G -表面抗原决定簇) | G -进行区分(沙门氏菌等[52]) | ||
| 细胞外Extracellular | 铜绿假单胞菌特有群体感应信号分子(QS信号分子)喹诺酮(PQS)信号分子和G -细菌产生的乙酰基高丝氨酸内酯(AHLs) | P.aeruginosa和G -菌[53,54,55,56] | |
| 肌醇,D-松二糖 | L.monocytogenes [6] | ||
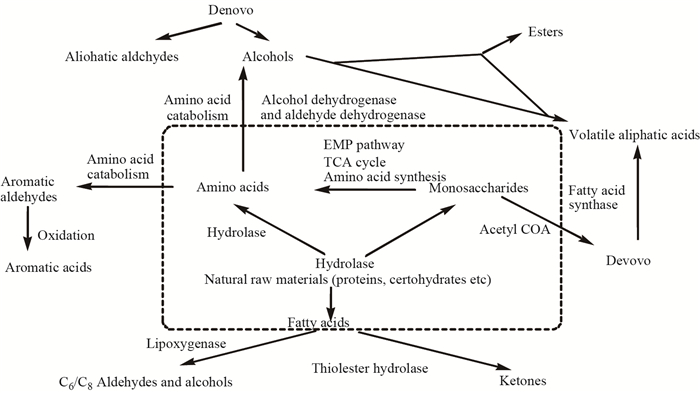
在胞外代谢轮廓分析中,Schulz等[15, 16]综述了微生物挥发性代谢产物和常见MVOCs的主要代谢过程,常见微生物挥发性代谢产物(MVOCs)的产生途径如图 3所示。长链脂肪醇通常作为肠杆菌科的标志性代谢产物,例如Hamilton-Kemp等[41]对于E. coli的培养液增补不同的脂肪酸分析可知,脂肪酸的减少伴随相应长链脂肪醇的增加。然而培养时间决定VOCs产生,Siripatrawan[42]在分析E. coli和鼠伤寒沙门氏菌时,培养10 h后发现两种肠杆科细菌均未检出长链醇。短链脂肪酸主要在厌氧菌中发现,主要来源于细菌对糖类及氨基酸类代谢产生,被认为是厌氧细菌的潜在标志物。对于铜绿假单胞杆菌及金黄色葡萄球菌产氨在早期就有报道[44, 45],特别在富含尿酸的培养基上生长时,发现有大量氨产生。Labows等[46]对11株铜绿假单胞杆菌和5株其他假单胞菌的VOCs分析时发现,所有菌株都产1-十一碳烯、甲基酮、2-壬酮和2-十一酮,但2-氨基苯乙酮被认为是铜绿假单胞杆菌的潜在特征性代谢产物。
随着食源性致病菌挥发性代谢产物研究技术不断成熟,已被建议作为临床和食品中致病菌鉴别的替代方法,并逐渐应用于临床和食品安全检测[48]。Bianchi等[28]发现利用β-月桂烯、邻甲基苯乙烯、甲基庚烯酮和辛醇可以明显区分污染大肠杆菌、金黄色葡萄球菌和炭黑曲霉菌的土豆和正常土豆。Bhattacharjee等[50]发现污染鼠伤寒沙门氏菌的牛肉产生大量的乙酸和乙醇,并认为乙酸和乙醇可作为污染鼠伤寒沙门氏菌牛肉的潜在生物标志物。此外,Zhu等[58]通过二次电喷雾质谱(SESI-MS)对E. coli、金黄葡萄球菌和鼠伤寒沙门氏菌在肉汁、蔬菜汁和苹果汁中生长产生的挥发性物质进行检测,发现污染4 h后,可以通过E. coli的特征质谱峰与上述其他菌株进行区分,同时也可以将E. coli O157:H7和E. coli O145与其他E. coli种进行区分。
此外,被致病微生物污染后的食品常含有大量代谢副产物和毒素,极容易造成食品安全事故。目前,可以初步借助代谢组学方法对食品中代谢副产物和毒素进行分析,从而进行快速评估食品污染情况。例如Beale等对比正常牛奶和被单增李斯特菌污染的牛奶,发现被污染的牛奶含有大量肌醇和D-松二糖等,通过PLS-DA分析可知,污染和无污染的牛奶可以明显区分[6]。
3 问题与展望随着分析技术的不断发展,食源性致病菌代谢组学研究取得重大进展,陆续建立了微生物挥发性代谢产物数据库和E. coli代谢产物数据库,此外,还基于代谢组学技术建立了相关食源性致病菌鉴定方法,但仍然存在较多问题有待解决。首先,胞内代谢组学研究中淬灭容易造成代谢产物泄露,且由于不同致病微生物间细胞壁和细胞膜结构存在差异,导致没有普适的淬灭和提取方法,因此全面分析代谢产物时需结合多种淬灭和提取方法。其次,胞内外代谢产物成份复杂,且胞外代产谢物难以与基质分离,各种代谢产物含量相差甚大,复杂的生物样本对分析检测技术中的色谱分离能力,质谱灵敏度、分辨率及高通量等需要更高的要求。最后,除个别致病菌代谢产物数据库建立外,目前尚缺乏标准化代谢产物数据库,同时现有的代谢途径数据库不能满足食品安全快速检测的需要,所以有必要建立我国食源性致病微生物系统代谢产物数据库和代谢网络等。
因此,在目前国内外研究基础上,进一步标准化食源性致病菌代谢组学前处理方法,系统研究食源性致病菌代谢组,建立标准化食源性致病菌代谢产物数据库,可以为寻找新型检测靶标和建立快速检测技术提供理论基础。本文作者多年来针对常见食源性致病菌代谢组学进行研究,业已发现部分常见食源性致病菌潜在生物标志物和蜡样芽孢杆菌及其类似菌株在L-脯氨酸代谢途径上的差异,后续将进一步针对本实验室在近几年全国污染调查中获得的分离株进行代谢轮廓分析,建立常见食源性致病菌代谢指纹谱图库和食源性致病菌代谢产物数据库,以期为建立我国食源性致病微生物代谢组学检测技术不断打下基础。
| [1] | Ye YW,Jiang QH,Wu QP,et al.The characterization and comparisonof Staphylococcus aureus by antibiotic susceptibility testing,enterobacterial repetitive intergenic consensus-polymerase chain reaction,and random amplified polymorphic DNA-polymerase chain reaction[J].Foodborne Pathogens and Disease,2012,9(2): 168-171 |
| [2] | Ye YW,Wu QP,Zhang JM,et al.Detection of viable Cronobacter spp.(Enterobacter sakazakii)by one-step RT-PCR in dry aquatic product[J].Journal of Food Science,2012,77(11): M616-M619 |
| [3] | Chen MT,Wu QP,Zhang JM,et al.Prevalence and characterization of Listeria monocytogenes isolated from retail-level ready-to-eat foods in South China[J].Food Control,2014,38: 1-7 |
| [4] | Inoue K,Toyo’oka T.Chapter 13-Foodomics[A]//Wilson CL,Wilson DW,Svehla G.Comprehensive Analytical Chemistry[M].Amsterdam: Elsevier,2015: 653-684 |
| [5] | Baker JM,Hawkins ND,Ward JL,et al.A metabolomic study of substantial equivalence of field-grown genetically modified wheat[J].Plant Biotechnology Journal,2006,4(4): 381-392 |
| [6] | Beale DJ,Morrison PD,Palombo EA.Detection of Listeria in milk using non-targeted metabolic profiling of Listeria monocytogenes: a proof-of-concept application[J].Food Control,2014,42: 343-346 |
| [7] | Rodríguez-Bencomo JJ,Andújar-Ortiz I,Moreno-Arribas MV,et al.Impact of glutathione-enriched inactive dry yeast preparations on the stability of terpenes during model wine aging[J].Journal of Agricultural and Food Chemistry,2014,62(6): 1373-1383 |
| [8] | Xu YJ,Wang CS,Ho WE,et al.Recent developments and applications of metabolomics in microbiological investigations[J].TrAC Trends in Analytical Chemistry,2014,56: 37-48 |
| [9] | Howlett RM,Davey MP,Quick WP,et al.Metabolomic analysis of the food-borne pathogen Campylobacter jejuni: application of direct injection mass spectrometry for mutant characterisation[J].Metabolomics,2014,10(5): 887-896 |
| [10] | Li H,Xia X,Li XW,et al.Untargeted metabolomic profiling of amphenicol-resistant Campylobacter jejuni by ultra-highperformance liquid chromatography-mass spectrometry[J].Journal of Proteome Research,2015,14(2): 1060-1068 |
| [11] | Singh AK,Ulanov AV,Li Z,et al.Metabolomes of the psychrotolerant bacterium Listeria monocytogenes 10403S grown at 37 ℃ and 8 ℃[J].International Journal of Food Microbiology,2011,148(2): 107-114 |
| [12] | Taymaz-Nikerel H,de Mey M,Ras C,et al.Development and application of a differential method for reliable metabolome analysis in Escherichia coli[J].Analytical Biochemistry,2009,386(1): 9-19 |
| [13] | Wu XH,Yu HL,Ba ZY,et al.Sampling methods for NMR-based metabolomics of Staphylococcus aureus[J].Biotechnology Journal,2010,5(1): 75-84 |
| [14] | Bos LDJ,Sterk PJ,Schultz MJ.Volatile metabolites of pathogens: a systematic review[J].PLoS Pathogens,2013,9(5): e1003311 |
| [15] | Schulz S,Dickschat JS.Bacterial volatiles: the smell of small organisms[J].Natural Product Reports,2007,24(4): 814-842 |
| [16] | Xu GW.Metabonomics-Methods and Application[M].Beijing: Science Press,2008: 1-421(in Chinese)许国旺.代谢组学——方法与应用[M].北京:科学出版社,2008: 1-421 |
| [17] | Winder CL,Dunn WB,Schuler S,et al.Global metabolic profiling of Escherichia coli cultures: an evaluation of methods for quenching and extraction of intracellular metabolites[J].Analytical Chemistry,2008,80(8): 2939-2948 |
| [18] | van der Werf MJ,Overkamp KM,Muilwijk B,et al.Microbial metabolomics: toward a platform with full metabolome coverage[J].Analytical Biochemistry,2007,370(1): 17-25 |
| [19] | Marcinowska R,Trygg J,Wolf-Watz H,et al.Optimization of a sample preparation method for the metabolomic analysis of clinically relevant bacteria[J].Journal of Microbiological Methods,2011,87(1): 24-31 |
| [20] | Zhao Q,Liu HQ,Lu Y,et al.Comparison of quenching and extraction methods for metabolome analysis of Vibrio parahaemolyticus[J].Chinese Journal of Analytical Chemistry,2011,39(11): 1689-1694(in Chinese)赵强,刘海泉,卢瑛,等.副溶血性弧菌细胞猝灭及代谢物组提取方法的优化[J].分析化学,2011,39(11): 1689-1694 |
| [21] | Guo ZR,Cui XW,Wang JJ,et al.Metabolome response to tdh expression of pathogenic Vibrio parahaemolyticus induced by different temperatures[J].Acta Microbiologica Sinica,2014,54(8): 882-888(in Chinese)郭卓然,崔晓文,王敬敬,等.不同温度条件下副溶血性弧菌tdh基因表达及其代谢组响应[J].微生物学报,2014,54(8): 882-888 |
| [22] | Meyer H,Weidmann H,Lalk M.Methodological approaches to help unravel the intracellular metabolome of Bacillus subtilis[J].Microbial Cell Factories,2013,12: 69 |
| [23] | Cevallos-Cevallos JM,Danyluk MD,Reyes-De-Corcuera JI.GC-MS based metabolomics for rapid simultaneous detection of Escherichia coli O157: H7,Salmonella typhimurium,Salmonella muenchen,and Salmonella hartford in ground beef and chicken[J].Journal of Food Science,2011,76(4): M238-M246 |
| [24] | Wu YM,Li L.Development of isotope labeling liquid chromatography-mass spectrometry for metabolic profiling of bacterial cells and its application for bacterial differentiation[J].Analytical Chemistry,2013,85(12): 5755-5763 |
| [25] | Schaefer U,Boos W,Takors R,et al.Automated sampling device for monitoring intracellular metabolite dynamics[J].Analytical Biochemistry,1999,270(1): 88-96 |
| [26] | Lemfack MC,Nickel J,Dunkel M,et al.mVOC: a database of microbial volatiles[J].Nucleic Acids Research,2014,42(D1): D744-D748 |
| [27] | Mapelli V,Olsson L,Nielsen J.Metabolic footprinting in microbiology: methods and applications in functional genomics and biotechnology[J].Trends in Biotechnology,2008,26(9): 490-497 |
| [28] | Bianchi F,Careri M,Mangia A,et al.Differentiation of the volatile profile of microbiologically contaminated canned tomatoes by dynamic headspace extraction followed by gas chromatography-mass spectrometry analysis[J].Talanta,2009,77(3): 962-970 |
| [29] | Tait E,Perry JD,Stanforth SP,et al.Identification of volatile organic compounds produced by bacteria using HS-SPME-GC-MS[J].Journal of Chromatographic Science,2014,52(4): 363-373 |
| [30] | Baidoo EEK,Benke PI,Keasling JD.Mass spectrometry-based microbial metabolomics[A]//Navid A.Microbial Systems Biology[M].Berlin: Springer,2012: 215-278 |
| [31] | Li XY,Legido-Quigley C.Advances in separation science applied to metabonomics[J].Electrophoresis,2008,29(18): 3724-3736 |
| [32] | Wu YM,Li L.Determination of total concentration of chemically labeled metabolites as a means of metabolome sample normalization and sample loading optimization in mass spectrometry-based metabolomics[J].Analytical Chemistry,2012,84(24): 10723-10731 |
| [33] | Aa JY.Analysis of metabolomic data: principal component analysis[J].Chinese Journal of Clinical Pharmacology and Therapeutics,2010,15(5): 481-489(in Chinese)阿基业.代谢组学数据处理方法——主成分分析[J].中国临床药理学与治疗学,2010,15(5): 481-489 |
| [34] | Guo AC,Jewison T,Wilson M,et al.ECMDB: the E.coli metabolome database[J].Nucleic Acids Research,2013,41(D1): D625-D630 |
| [35] | Oh YK,Palsson BO,Park SM,et al.Genome-scale reconstruction of metabolic network in Bacillus subtilis based on high-throughput phenotyping and gene essentiality data[J].Journal of Biological Chemistry,2007,282(39): 28791-28799 |
| [36] | Herrgård MJ,Swainston N,Dobson P,et al.A consensus yeastmetabolic network reconstruction obtained from a community approach to systems biology[J].Nature Biotechnology,2008,26(10): 1155-1160 |
| [37] | Lu MF,Wu QP,Liu YL,et al.The specificity of enzyme reaction in the application of the detection of foodborne pathogenic bacterial[J].Chinses Journal of Health Laboratory Technology,2005,15(3): 354-356(in Chinese)卢勉飞,吴清平,刘云林,等.特异性酶反应在食源性致病菌检测中的应用[J].中国卫生检验杂志,2005,15(3): 354-356 |
| [38] | Lu MF,Cai ZH,Chen LX,et al.Evaluation of the detecting effect for chromogenic Escherichia coli O157 medium[J].Microbiology China,2014,41(7): 1456-1462(in Chinese)卢勉飞,蔡芷荷,陈霖熙,等.大肠杆菌O157显色培养基检测效果的评价[J].微生物学通报,2014,41(7): 1456-1462 |
| [39] | Fox A,Stewart GC,Waller LN,et al.Carbohydrates and glycoproteins of Bacillus anthracis and related Bacilli: targets for biodetection[J].Journal of Microbiological Methods,2003,54(2): 143-152 |
| [40] | Hamid AM,Jarmusch AK,Pirro V,et al.Rapid discrimination of bacteria by paper spray mass spectrometry[J].Analytical Chemistry,2014,86(15): 7500-7507 |
| [41] | Hamilton-Kemp T,Newman M,Collins R,et al.Production of the long-chain alcohols octanol,decanol,and dodecanol by Escherichia coli[J].Current Microbiology,2005,51(2): 82-86 |
| [42] | Siripatrawan U.Rapid differentiation between E.coli and Salmonella typhimurium using metal oxide sensors integrated with pattern recognition[J].Sensors and Actuators B: Chemical,2008,133(2): 414-419 |
| [43] | Cummings JH,Macfarlane GT.Role of intestinal bacteria in nutrient metabolism[J].Clinical Nutrition,1997,16(1): 3-11 |
| [44] | Robacker DC,Lauzon CR,Patt J,et al.Attraction of Mexican fruit flies(Diptera: Tephritidae)to bacteria: effects of culturing medium on odour volatiles[J].Journal of Applied Entomology,2009,133(3): 155-163 |
| [45] | Allardyce RA,Langford VS,Hill AL,et al.Detection of volatile metabolites produced by bacterial growth in blood culture media by selected ion flow tube mass spectrometry(SIFT-MS)[J].Journal of Microbiological Methods,2006,65(2): 361-365 |
| [46] | Labows JN,Mcginley KJ,Webster GF,et al.Headspace analysis of volatile metabolites of Pseudomonas aeruginosa and related species by gas chromatography-mass spectrometry[J].Journal of Clinical Microbiology,1980,12(4): 521-526 |
| [47] | Filipiak W,Sponring A,Baur MM,et al.Molecular analysis of volatile metabolites released specifically by staphylococcus aureus and pseudomonas aeruginosa[J].BMC Microbiology,2012,12(23): 113 |
| [48] | Kubo I,Muroi H,Kubo A.Structural functions of antimicrobial long-chain alcohols and phenols[J].Bioorganic & Medicinal Chemistry,1995,3(7): 873-880 |
| [49] | Di Martino P,Fursy R,Bret L,et al.Indole can actas an extracellular signal to regulate biofilm formation of Escherichia coli and other indole-producing bacteria[J].Canadian Journal of Microbiology,2006,49(7): 443-449 |
| [50] | Bhattacharjee P,Panigrahi S,Lin DQ,et al.A comparative qualitative study of the profile of volatile organic compounds associated with Salmonella contamination of packaged aged and fresh beef by HS-SPME/GC-MS[J].Journal of Food Science and Technology,2011,48(1): 1-13 |
| [51] | Bhattacharjee P,Panigrahi S,Lin DQ,et al.A comparative qualitative study of the profile of volatile organic compounds associated with Salmonella contamination of packaged aged and fresh beef by HS-SPME/GC-MS[J].Journal of Food Science and Technology,2011,48(1): 1-13 |
| [52] | Gibson BW,Melaugh W,Phillips NJ,et al.Investigation of the structural heterogeneity of lipooligosaccharides from pathogenic Haemophilus and Neisseria species and of R-type lipopolysaccharides from Salmonella typhimurium by electrospray mass spectrometry[J].Journal of Bacteriology,1993,175(9): 2702-2712 |
| [53] | Dickschat JS.Quorum sensing and bacterial biofilms[J].Natural Product Reports,2010,27(3): 343-369 |
| [54] | Praneenararat T,Palmer AG,Blackwell HE.Chemical methods to interrogate bacterial quorum sensing pathways[J].Organic & Biomolecular Chemistry,2012,10(41): 8189-8199 |
| [55] | Borgos SEF,Skjåstad R,Tøndervik A,et al.Rapid metabolic profiling of developing Pseudomonas aeruginosa biofilms by high-resolution mass spectrometry fingerprinting[J].Annals of Microbiology,2015,65(2): 891-898 |
| [56] | Bukelman O,Amara N,Mashiach R,et al.Electrochemical analysis of quorum sensing inhibition[J].Chemical Communications,2009(20): 2836-2838 |
| [57] | Liang HZ,Zhang X,Rao J,et al.Microbial volatile organic compounds: generation pathways and mass spectrometric detection[J].China Biotechnology,2008,28(1): 124-133(in Chinese)梁华正,张燮,饶军,等.微生物挥发性代谢产物的产生途径及其质谱检测技术[J].中国生物工程杂志,2008,28(1): 124-133 |
| [58] | Zhu JJ,Hill JE.Detection of Escherichia coli via VOC profiling using secondary electrospray ionization-mass spectrometry(SESI-MS)[J].Food Microbiology,2013,34(2): 412-417 |
 2016, Vol. 43
2016, Vol. 43