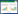扩展功能
文章信息
- 盛超兰, 王威, 胡洪波, 彭华松, 张雪洪
- SHENG Chao-lan, WANG Wei, HU Hong-bo, PENG Hua-song, ZHANG Xue-hong
- 一个甲基转移酶基因lomo3在洛蒙真菌素生物合成途径中的功能
- Function of a methyltransferase gene lomo3 involved in the biosynthesis of lomofungin
- 微生物学通报, 2016, 43(3): 575-582
- Microbiology China, 2016, 43(3): 575-582
- 10.13344/j.microbiol.china.150445
-
文章历史
- 收稿日期: 2015-06-09
- 接受日期: 2015-08-26
- 优先数字出版日期(www.cnki.net): 2015-10-30
吩嗪是一类含氮杂环化合物,由两个苯环和一个吡嗪环形成对称的稠环结构。吩嗪类化合物是含有吩嗪结构的一类化合物的总称,以吩嗪环为核心,经过羟基、羧基、甲基等不同的功能基团修饰,形成具有抗菌、抗肿瘤、抗疟疾以及抗寄生虫等不同生物活性的吩嗪衍生物[1]。
天然吩嗪类化合物主要由假单胞菌、链霉菌以及部分海洋菌属等合成。已报道的由假单胞菌产生的吩嗪类化合物主要有吩嗪-1-羧酸(phenazine-1-carboxylic acid,PCA)、1-羟基吩嗪(1-hydroxy-phenazine,1-OH-Phz)、吩嗪-1-甲酰胺(phenazine-1-carboxamide,PCN)、绿脓菌素(pyocyanin)等[2]。与假单胞菌相比,链霉菌不仅能够产生一些结构比较简单的吩嗪类化合物,如PCA、吩嗪-1,6-二羧酸(phenazine-1,6-dicarboxylic acid,PDC)等[3],还能合成一些结构更为复杂的,如吩嗪环上含有酯基、醛基、氨基、硫醚基等取代基的吩嗪类化合物[4]。
研究表明,假单胞菌中的吩嗪类化合物生物合成的前体是PCA,而链霉菌除了PCA,还可以PDC为前体生物合成不同的吩嗪类化合物[5]。PCA和PDC主要来源于莽草酸途径,其生物合成一般是由7个基因——phzABCDEFG组成的核心基因簇完成的[6],该核心基因簇在假单胞菌和链霉菌中都有发现。前体合成之后,通过不同的侧链修饰途径形成不同的吩嗪类化合物。假单胞菌中的修饰途径研究的比较多,如在铜绿色假单胞菌PAO1 (Pseudomonas aeruginosa PAO1)中,甲基转移酶PhzM催化PCA生成5-甲基吩嗪-1-羧酸甜菜碱(5-methylphenazine-1-carboxylic acid betaine),水杨酸羟化酶PhzS单独作用时可催化PCA转化为1-OH-Phz,天冬酰胺合成酶PhzH催化PCA转化为PCN[7]。关于链霉菌中吩嗪类化合物生物合成的修饰基因和修饰途径的研究还很少。
洛蒙真菌素是由洛蒙德链霉菌(Streptomyces lomondensis)合成的一种吩嗪类次级代谢产物,由美国Upjohn公司的Bergy博士等分离得到。洛蒙真菌素呈橄榄黄色,其化学结构为6-醛基-4,7,9-三羟基-吩嗪-1-甲酸酯,能够抑制多种革兰氏阳性和革兰氏阴性细菌生长,还具有抑制真菌的特性,是一种广谱抗生素[8]。
洛蒙德链霉菌S015 (S. lomondensis S015)是本实验室从土壤中分离得到的,我们在该菌株的发酵产物中纯化鉴定到了洛蒙真菌素[9]。通过对S015的全基因组测序分析,在其基因组中找到了由phzGFEDCB组成的吩嗪类化合物生物合成核心基因簇,并在此基因簇附近,发现了一个甲基转移酶基因——lomo3 (NCBI:KP721214.1),如图 1所示[10]。根据洛蒙真菌素的侧链结构,我们推测此基因可能参与了其C-1位的甲酯化反应。本文通过无痕敲除的方法,研究了该基因对洛蒙真菌素生物合成的影响。
1 材料与方法 1.1 实验材料 1.1.1 工具酶:DNA聚合酶rTaq (生化试剂),Xba I、Hind III、Bgl II、Nde I限制性内切酶(生化试剂),Solution I DNA连接酶试剂盒,均购自日本Takara公司;KOD-201高保真酶(生化试剂),购自上海硕盟有限公司。 1.1.2 培养基:LB培养基(g/L):胰蛋白胨10,酵母提取物5,NaCl 10,固体加琼脂15;MS培养基(g/L):黄豆饼粉30,加水煮沸后过滤,加甘露醇20,琼脂20;2×YT培养基(g/L):酵母提取物10,胰蛋白胨10,氯化钠5;1.0×105 Pa,20 min高温灭菌。YEME培养基(g/L):葡萄糖4,麦芽提取物10,酵母提取物4;0.68×105 Pa高温灭菌20 min。 1.1.3 菌株和质粒:所用菌株及质粒信息见表 1。| 菌株或质粒 Strains and plasmids | 特点 characteristics | 来源 Reference |
| Escherichia coli | ||
| DH5α | 克隆宿主 | TranGen Boitech |
| ET12567 (pUZ8002) | 接合转移供体菌,KmR,CmR | MacNeil等[11] |
| Streptomyces lomondensis | ||
| S015 | 野生型 | 本实验室保存 |
| S015Δlomo3 | lomo3基因敲除突变株 | 本实验室保存 |
| S015Δlomo3::lomo3 | lomo3基因回补菌株,AprR | 本实验室保存 |
| Plasmids | ||
| pKC1139 | 大肠杆菌-链霉菌穿梭质粒,温控型,AprR | Bierman等[12] |
| pIB139 | 链霉菌整合型质粒,包含强启动子PermE*,AprR | Wilkinson等[13] |
| pMD19-T vector | 克隆载体,AmR | Takara |
| pCC701 | lomo3基因同源左臂连接在pMD19-T载体上,AmR | 本实验室保存 |
| pCC702 | lomo3基因同源右臂连接在pMD19-T载体上,AmR | 本实验室保存 |
| pCC703 | lomo3基因左右同源臂拼接在pMD19-T载体上,AmR | 本实验室保存 |
| pKC1139-lomo3 | lomo3基因左右同源臂重组连接在pKC1139-T载体上,用于lomo3基因同框缺失,AprR | 本实验室保存 |
| pCC704 | lomo3完整基因片段连接在pMD19-T载体上,AmR | 本实验室保存 |
| pIB139-lomo3 | lomo3完整基因片段连接在pIB139载体上,回补载体,AprR | 本实验室保存 |
| 引物 Primers | 碱基序列 Base sequences (5′→3′) | 作用 Function |
| lomo3 left-F | AAATTTGAATTCTACAGATGTGCTGAAGGCTGGG (下划线为Hind III位点) | 克隆lomo3基因同源重组的左臂 |
| lomo3 left-R | AAAAAAAAAAAAATTTAGATCTCCCGACGTTCGCTTTCCAGTT (下划线为Bgl II位点) | |
| lomo3 right-F | AAATTTAGATCTAACGGGTCGAGGAACAGGGC (下划线为Bgl II位点) | 克隆lomo3基因同源重组的右臂 |
| lomo3 right-R | AAATTTTCTAGACAGTCGGGGAAGCACTTGAG (下划线为Xba I位点) | |
| lomo3-F | AAATTTCATATGGGGCAGACCGCGCTCTTCAT (下划线为Nde I位点) | 克隆携带有完整lomo3基因的片段 |
| lomo3-R | AAAATTTTTCTAGACCTGAGCGAAATACTCTCGG (下划线为Xba I位点) | |
| pIB-F | TTGCGCCCGATGCTAGTCG | pIB139载体通用引物,验证质粒整合入链霉菌中 |
| pIB-R | GCACGACAGGTTTCCCGACTG |
 |
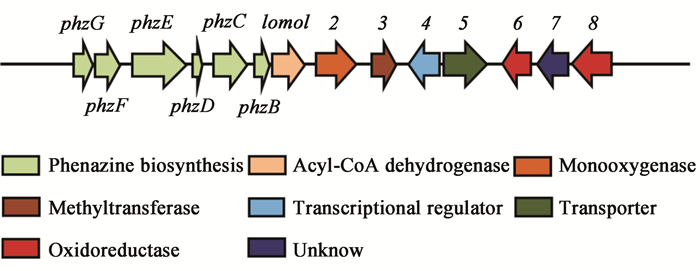
| 图 2 突变质粒pKC1139-lomo3构建及lomo3基因敲除突变株验证电泳图谱 Figure 2 Construction of pKC1139-lomo3 plasmid in vitro and validation of lomo3 gene knock-out strain
注: M:1 kb plus DNA ladder marker. A:1、2:以全基因组为模板,扩增lomo3基因左、右同源臂序列. B:1:以pKC1139-lomo3为模板,扩增同源臂序列. C:1:以S015基因组为模板;2:以S015Δlomo3基因组为模板. Note:M: 1 kb plus DNA ladder marker. A: 1,2: using the whole genome as a template,amplified homology left and right arm sequences. B: 1: using pKC1139-lomo3 as a template,amplified homologous recombination sequence. C: 1: using S015 genome as a template; 2: Using S015Δlomo3 genome as a template. |
| |
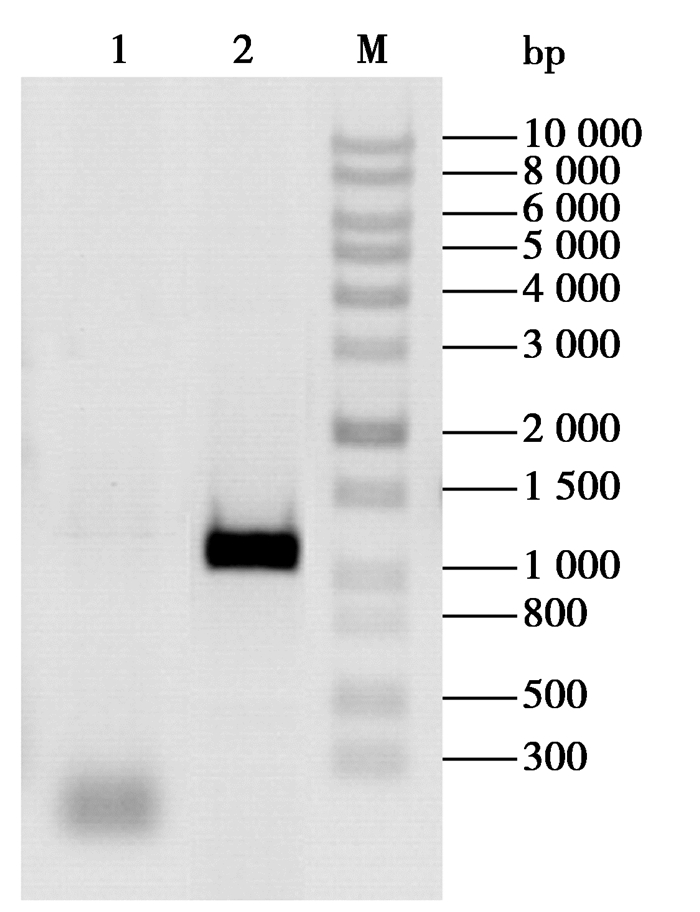 |
| 图 3 lomo3基因回补菌株的验证图谱 Figure 3 Validation of lomo3 gene complement strain
注:M:1 kb plus DNA ladder marker;1:以S015Δlomo3基因组为模板;2:以S015Δlomo3::lomo3基因组为模板. Note: M: 1 kb plus DNA ladder marker; 1: Using S015Δlomo3 genome as a template; 2: Using S015Δlomo3::lomo3 genome as a template. |
| |
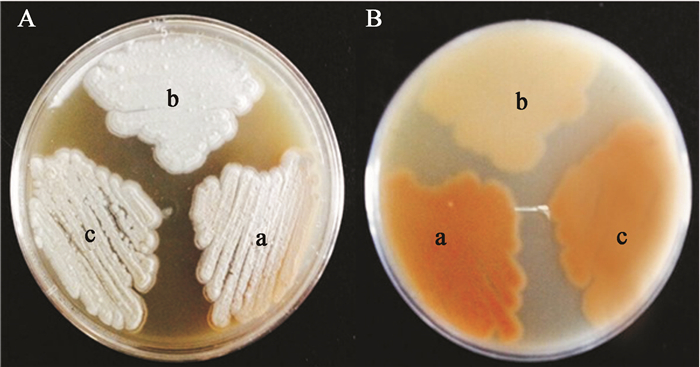 |
| 图 4 野生株S015 (a)、lomo3缺失株S015Δlomo3 (b)和lomo3回补株S015Δlomo3::lomo3 (c)在MS平板上生长6 d的形态 Figure 4 Phenotype of wild type S015 (a),lomo3 knock-out mutant S015Δlomo3 (b) and lomo3 complementary strain S015Δlomo3::lomo3 (c) grown on MS medium for 6 days
注:A:平板的正面;B:平板的背面. Note: A: Imagines from the right side of the culture dish; B: Imagines from the reverse side of the culture dish. |
| |
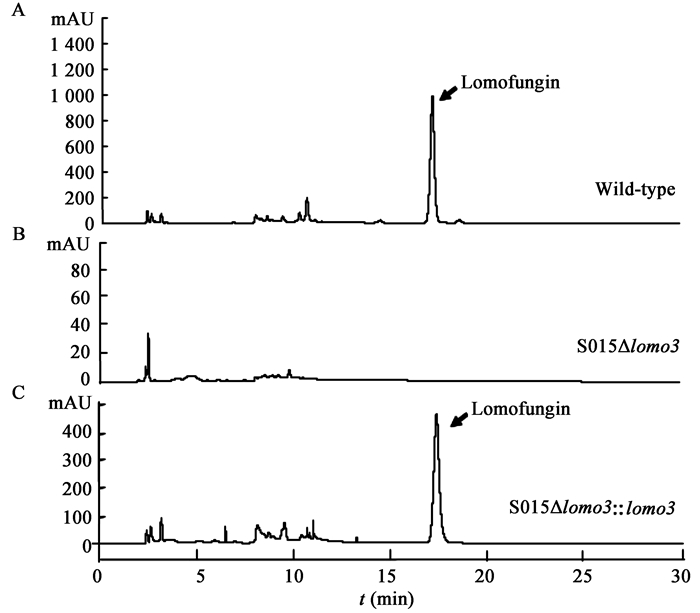 |
| 图 5 野生株S015 (a)、lomo3基因缺失株S015Δlomo3 (B)和lomo3基因回补株S015Δlomo3::lomo3 (C)发酵产物的HPLC图谱 Figure 5 HPLC profiles of fermentation products from wild type S015 (A),lomo3 knock-out mutant S015Δlomo3 (B) and complementary strain S015Δlomo3::lomo3 (C) |
| |
本文通过无痕敲除的方法,研究了洛蒙德链霉菌S015中的一个甲基转移酶基因lomo3对洛蒙真菌素生物合成的影响。
甲基转移酶在很多微生物的次级代谢产物合成中起着重要的作用[16]。在铜绿色假单胞菌PAO1 (Pseudomonas aeruginosa PAO1)中,甲基转移酶PhzM负责PCA吩嗪环上5位的甲基转移,生成5-甲基吩嗪-1-羧酸甜菜碱[7]。Streptomyces spheroides的新生霉素合成基因簇中,有一种S-腺苷甲硫氨酸依赖的氧位甲基转移酶NovP,负责诺维糖C-4位上羟基的甲基化[17]。Streptomyces iakyrus在合成放线菌素G的过程中,将编码一种甲基转移酶的基因acmG5′ 敲除后,会导致该菌株不再合成放线菌素G[18]。本研究中,甲基转移酶基因lomo3的敲除导致S015不能合成洛蒙真菌素;通过lomo3基因回补,菌株S015Δlomo3::lomo3恢复了洛蒙真菌素的合成。说明这个甲基转移酶基因lomo3在洛蒙真菌素的生物合成过程中起着重要的作用。
Buckland等[19]利用14C标记的方法,确定了PDC是洛蒙真菌素合成的前体;而PDC来源于莽草酸途径中产生的分支酸(chorismic acid)[5]。Zhang等的研究表明,在洛蒙真菌素的生物合成中,单加氧酶基因lomo2的功能是在其C-7位的羟基转移[10]。我们根据洛蒙真菌素的侧链结构,推测脱氢酶基因lomo1参与C-6位的羧基还原成醛基,lomo3参与了C-1位的甲酯化,lomo6可能作用于C-4和C-9位的羟基修饰,并据此推测出洛蒙真菌素的生物合成途径(图 6)。我们没有在lomo3基因缺失株S015Δlomo3中检测到其他吩嗪类产物,还不能确定该甲基转移酶作用的底物及相应的反应产物。这可能是因为Lomo3的底物是由一个或多个可逆反应合成的:当lomo3基因存在时,反应向着合成洛蒙真菌素的方向进行;而当该基因敲除以后,反应则逆向进行导致在基因缺失菌株中检测不到吩嗪类产物。目前还不能确定1位的甲酯反应、6位的脱氢反应,以及4、9位的两个羟基转移反应的先后顺序。我们正在进行lomo1、lomo6的基因敲除和功能的研究,同时还在进行Lomo3的体外酶学研究,希望通过这些研究,能够确定这些基因尤其是lomo3的功能,阐明洛蒙真菌素的生物合成途径。
| [1] | Schneemann I, Wiese J, Kunz AL, et al. Genetic approach for the fast discovery of phenazine producing bacteria[J]. Marine Drugs, 2011, 9(5): 772-789 |
| [2] | Mavrodi DV, Blankenfeldt W, Thomashow LS. Phenazine compounds in fluorescent Pseudomonas spp. biosynthesis and regulation[J]. Annual Review of Phytopathology, 2006, 44: 417-445 |
| [3] | Hui JY, Wang W, Hu HB, et al. Streptomyces griseoruber Y1B, a novel Streptomyces for 1-Hydroxyphenazine production[J]. Journal of Applied Biotechnology, 2014, 2(2): 13-31 |
| [4] | Laursen JB, Nielsen J. Phenazine natural products: biosynthesis, synthetic analogues, and biological activity[J]. Chemical Reviews, 2004, 104(3): 1663-1686 |
| [5] | Herbert RB, Holliman FG, Sheridan JB. Biosynthesis of microbial phenazines: incorporation of shikimic acid[J]. Tetrahedron Letters, 1976, 17(8): 639-642 |
| [6] | Mavrodi DV, Ksenzenko VN, Bonsall RF, et al. A seven-gene locus for synthesis of phenazine-1-carboxylic acid by Pseudomonas fluorescens 2-79[J]. Journal of Bacteriology, 1998, 180(9): 2541-2548 |
| [7] | Mavrodi DV, Bonsall RF, Delaney SM, et al. Functional analysis of genes for biosynthesis of pyocyanin and phenazine-1-carboxamide from Pseudomonas aeruginosa PAO1[J]. Journal of Bacteriology, 2001, 183(21): 6454-6465 |
| [8] | Johnson LE, Dietz A. Lomofungin, a new antibiotic produced by Streptomyces lomondensis sp. n.[J]. Applied Microbiology, 1969, 17(5): 755-759 |
| [9] | Wang HS, Bei XY, Hu HB, et al. Isolation, identification and medium optimization of modified phenazine derivative lomofungin from Streptomyces lomondensis S015[J]. Journal of Shanghai Jiaotong University (Agricultural Science), 2014, 32(2): 48-54 (in Chinese) 王华盛, 贝晓宇, 胡洪波, 等. 洛蒙德链霉菌S015中吩嗪类活性产物洛蒙真菌素的分离纯化、结构鉴定及发酵优化[J]. 上海交通大学学报: 农业科学版, 2014, 32(2): 48-54 |
| [10] | Zhang CX, Sheng CL, Wang W, et al. Identification of the lomofungin biosynthesis gene cluster and associated flavin-dependent monooxygenase gene in Streptomyces lomondensis S015[J]. PLoS One, 2015, 10(8): e0136228 |
| [11] | MacNeil DJ, Gewain KM, Ruby CL, et al. Analysis of Streptomyces avermitilis genes required for avermectin biosynthesis utilizing a novel integration vector[J]. Gene, 1992, 111(1): 61-68 |
| [12] | Bierman M, Logan R, O’Brien K, et al. Plasmid cloning vectors for the conjugal transfer of DNA from Escherichia coli to Streptomyces spp.[J]. Gene, 1992, 116(1): 43-49 |
| [13] | Wilkinson CJ, Hughes-Thomas ZA, Martin CJ, et al. Increasing the efficiency of heterologous promoters in actinomycetes[J]. Journal of Molecular Microbiology Biotechnology, 2002, 4(4): 417-426 |
| [14] | Bergy ME. Lomofungin, a new broad spectrum antibiotic isolation and characterization[J]. The Journal of Antibiotics, 1969, 22(3): 126-128 |
| [15] | Li S, Ye QM, Wang W, et al. High-performance liquid chromatography analysis of lomofungin in Streptomyces lomondensis[J]. Journal of Liquid Chromatography & Related Technologies, 2013, 36(15): 2059-2068 |
| [16] | Feitelson JS, Hopwood DA. Cloning of a Streptomyces gene for an O-methyltransferase involved in antibiotic biosynthesis[J]. Molecular and General Genetics MGG, 1983, 190(3): 394-398 |
| [17] | Freel Meyers CL, Oberthür M, Heide L, et al. Assembly of dimeric variants of coumermycins by tandem action of the four biosynthetic enzymes CouL, CouM, CouP, and NovN[J]. Biochemistry, 2004, 43(47): 15022-15036 |
| [18] | Qin ZW, Wang XL, Rateb ME, et al. Disruption of a methyltransferase gene in actinomycin G gene cluster in Streptomyces iakyrus increases the production of phenazinomycin[J]. FEMS Microbiology Letters, 2014, 352(1): 62-68 |
| [19] | Buckland P, Gulliford SP, Herbert RB, et al. The biosynthesis of phenazines-biosynthesis of lomofungin via phenazine-1, 6-dicarboxylic acid[J]. Journal of Chemical Research, 1981, 12: 362 |
 2016, Vol. 43
2016, Vol. 43