扩展功能
文章信息
- 杨娟, 郝志成, 张亚平
- YANG Juan, HAO Zhi-Cheng, ZHANG Ya-Ping
- 基于MiSeq测序分析新疆泥火山土壤细菌群落多样性
- Analysis the diversity of soil bacterial community from mud volcano in Xinjiang using MiSeq sequencing
- 微生物学通报, 2016, 43(12): 2609-2618
- Microbiology China, 2016, 43(12): 2609-2618
- DOI: 10.13344/j.microbiol.china.151073
-
文章历史
- 收稿日期: 2015-12-30
- 接受日期: 2016-03-08
- 优先数字出版日期(www.cnki.net): 2016-11-04
泥火山是由泥浆形成的,只因它的喷发形式和喷发后形成的堆积物在外形上与火山有些相似,因此称其泥火山[1]。新疆乌苏具有亚洲最大的泥火山群,其特殊的环境是研究微生物资源的最佳场所[2]。土壤微生物是土壤的重要组成部分,土壤微生物群落的变化在一定程度上可以反映土壤质量的变化趋势[3],而细菌是土壤中最丰富、分布最广泛的微生物类群[4],广泛参与土壤营养物质循环过程[5-6]。传统的土壤微生物培养方法存在着耗时、工作量大的缺点,并且自然界微生物多是未培养的,从而导致无法全面解释微生物群落结构。近年来,随着高通量测序技术的不断发展[7-9],人们对土壤中微生物的研究日渐深入[10],它被广泛应用于环境微生物菌群结构与演替规律研究,其中,Illumina公司开发的MiSeq方法避免了通量低、准确率低、操作复杂等问题[11],与454焦磷酸测序相比,通量更高,操作简便、成本更低,更适合分析环境中群落结构多样性[12]。本研究通过Illumina MiSeq测序技术分析新疆乌苏泥火山微生物的群落结构,这将为深入研究泥火山生态系统奠定基础,为合理利用和开发泥火山微生物资源提供指导。
1 材料与方法 1.1 生境概况乌苏泥火山位于新疆天山北麓乌苏市南约 40 km白杨沟镇东南约2 500 m处,属于天山北坡山前丘陵地带,四周群山环抱,属大陆干旱气候,空气干燥,降雨稀少,夏季炎热,光照充足,年均降水158 mm,平均气温7.3 ℃,其环境条件较为特殊。本课题以泥火山口为参照,选取具有代表的4种不同生境(命名为A、B、C、D)为研究对象,各个生境基本情况见表 1。
1.2 样品采集以4种不同生境(A、B、C、D)为研究对象,取样时间为2014年4、7、11月,以正方形5点取样法在样地内设置样方,草本采用1 m×1 m的样方,5次重复;灌木采用5 m×5 m的样方,3次重复。每个点取样量大体一致,去除土样里的植物根系和砾石,然后将重复的土样混合均匀,做好标记装入灭菌封口聚乙烯袋,低温保存带回实验室。将土样分成两份,一份在室内风干,将风干土分别过1 mm和0.15 mm筛子,用于理化因子的研究;一份放入-20 ℃,用于后续的分子方面的研究。最终,混合后的土壤样品共12个,根据采样季节不同,分别标记为A4、A7、A11,B4、B7、B11,C4、C7、C11,D4、D7、D11。
| 生境类型 Habitat types | 经度 Longitude (E) | 纬度 Latitude (N) | 海拔 Altitude (m) | 备注 Remarks column |
| A | 84°40′20″ | 44°18′00″ | 1 300 | 主要为一些零星灌木丛 |
| B | 84°23′00″ | 44°11′00″ | 1 275 | 泥火山口周围,几乎没有植被 |
| C | 84°22′10″ | 44°13′00″ | 1 100 | 一些矮小的禾本科、藜科植物等 |
| D | 84°29′20″ | 44°17′00″ | 762 | 一些多年生莎草科杂草,例如梭梭草等,地表较干旱,缺水 |
引物,上海生工合成;PCR和酶反应体系中DNA磁珠法回收纯化试剂盒,志昂生物科技有限公司。PCR仪,安捷伦科技有限公司;电泳仪和电泳凝胶成像系统,北京君意东方电泳设备有限公司。
1.4 研究方法1.4.1 土壤理化性质的测定:土壤含水量采用烘干测定法;pH和电导率采用pH电导率仪;根据水土比2.5:1和5:1进行测定;有机质采用重铬酸钾容量法-外加热法测定;土壤全磷采用HClO4-H2SO4法测定;土壤全氮采用半微量凯氏法测定;全钾含量测定采用NaOH熔融-火焰光度计法[13]。
1.4.2 土壤微生物基因组DNA的提取:DNA的提取采用美国(MOBIO PowerSoil DNA Isolation Kit)强力土壤DNA提取试剂盒。
1.4.3 土壤细菌16S rRNA基因PCR扩增:采用细菌16S rRNA基因V3-V4区的通用引物(336F:5′-GTACTCCTACGGGAGGCAGCA-3′;806R:5′-GTGGACTACHVGGGTWTCTAAT-3′)扩增泥火山土壤细菌16S rRNA基因片段。50 μL PCR反应体系:DNA模板1 μL,正反向引物(10 μmol/L)各 2 μL,dNTPs (2.5 μmol/L) 4 μL,10×Pyrobest Buffer 5 μL,Pyrobest DNA Polymerase (2.5 U/μL) 0.3 μL,ddH2O 35.7 μL。PCR程序为:95 ℃ 5 min;95 ℃ 30 s,56 ℃ 30 s,72 ℃ 40 s,共25个循环;72 ℃ 10 min。将PCR产物用1% (质量体积比)的琼脂糖凝胶进行检测,参照电泳初步定量结果,使用Qubit荧光定量系统对PCR产物进行检测定量,之后按照每个样本的测序量要求,根据精确定量结果进行相应比例的混合。随后进行MiSeq文库构建,Illumina上机测序。
1.5 数据处理通过COPE软件(Connecting Overlapped Pair-End,V1.2.3.3) ,对于Illumina测序的测序结果进行分析,去掉测序质量不好的序列,并对序列长度进行筛选,删掉长度小于200 bp的序列,根据Barcode将序列确定为本样品的序列,并去除Barcode和引物序列,最终得到有效的序列文件。在0.97相似度下利用Qiime (V1.8.0) 软件将其聚类为用于物种分类的OTU (Operational taxonomic units),统计各个样品每个OTU中的丰度信息,OTU的丰度初步说明了样品的物种丰富程度。
利用QIIME软件计算样品包括Chao1[14]指数、Shannon[15]指数和Simpson指数[16]的Alpha多样性值,其中Chao1指数表示群落丰富度,其值越高表示群落物种的丰富度越高;而Shannon指数和Simpson指数可以反映样品的多样性程度,其值越高表示群落物种的多样性越高;经过UniFrac算法利用系统进化的信息来比较样品间的物种群落差异,进行Beta多样性(Beta diversity)分析;通过SPSS 17.0[17]软件进行细菌多样性与环境因子的相关性分析。
2 结果与分析 2.1 土壤理化性质测定结果及分析由表 2可知,不同生境土壤理化性质存在显著性差异(P<0.05) ,pH、含水量、电导率、有机质、全磷、全氮和全钾的含量波动范围分别为7.41-10.20、2.72%-33.56%、0.12-7.97 ds/m、9.88-163.91 g/kg、0.33-1.06 g/kg、0.57-15.12 g/kg和3.24-6.27 g/kg。其中除了pH、电导率,在4种生境中,生境C的含水量、有机质、全磷、全氮和全钾含量最高;生境D的全氮、全磷、全钾含量相对较低;从时空动态变化方面看,pH、含水量、有机质(除了生境C)的变化趋势均是4月份最高;全磷的变化趋势均是11月份含量最高,7月份最低;全氮的变化趋势(除了生境A)是11月份含量最高;全钾的变化趋势均是4月份最高。
| 生境类型 Habitat types | 月份 Months | pH | 含水量 Moisture (%) | 电导率Electric Conductivity (ds/m) | 有机质 Organic matter (g/kg) | 全磷 Total P (g/kg) | 全氮 Total P (g/kg) | 全钾 Total K (g/kg) |
| A | 4 | 8.00±0.06b | 13.78±0.94a | 0.17±0.02a | 76.40±1.83b | 0.39±0.03a | 3.32±0.28b | 5.55±0.55a |
| 7 | 7.80±0.20b | 2.72±0.29a | 0.12±0.01a | 38.36±1.15b | 0.35±0.03a | 1.92±0.06b | 4.01±0.03a | |
| 11 | 7.71±0.17ab | 8.74±0.32a | 0.16±0.01a | 45.23±0.65b | 0.91±0.01ab | 3.31±0.09b | 3.24±0.12a | |
| B | 4 | 10.20±0.02d | 22.05±0.01b | 2.74±0.20c | 16.44±0.44a | 0.50±0.03b | 0.82±0.02a | 6.27±0.13b |
| 7 | 7.91±0.06b | 22.05±0.01b | 4.50±0.68b | 14.85±3.00ab | 0.43±0.01b | 0.74±0.15ab | 5.18±0.07c | |
| 11 | 7.91±0.06b | 18.92±0.27b | 7.97±0.05c | 11.50±0.42a | 0.85±0.03a | 1.08±0.06a | 3.26±0.17a | |
| C | 4 | 7.60±0.07a | 33.56±3.33c | 1.70±0.15b | 133.89±4.79c | 0.39±0.01a | 6.69±0.23c | 5.29±0.18a |
| 7 | 7.41±0.06a | 22.03±2.18c | 1.08±0.14a | 163.91±14.04c | 0.33±0.03a | 8.20±0.70c | 4.45±0.13b | |
| 11 | 7.41±0.06a | 24.06±3.56b | 0.56±0.03b | 112.65±2.32c | 1.06±0.03c | 15.12±0.39c | 5.06±0.04b | |
| D | 4 | 9.00±0.06c | 8.52±0.39a | 1.32±0.13b | 15.85±0.52a | 0.39±0.02a | 0.95±0.14a | 5.85±0.18b |
| 7 | 8.38±0.07c | 2.82±0.15a | 1.23±0.27a | 11.47±2.19a | 0.33±0.01a | 0.57±0.11a | 5.08±0.04c | |
| 11 | 8.38±0.06c | 7.72±0.17a | 4.33±0.21d | 9.88±0.51a | 0.97±0.02bc | 1.08±0.05a | 3.53±0.08a | |
| 注:表中数据为平均数±标准误. 同一指标中不同字母表示平均数的差异,在P<0.05时达到显著水平. Note: Data in the table are mean±SE. Different small letters in the same row mean significant difference at 0.05 level among treatments. | ||||||||
通过对泥火山土壤细菌V3-V4区测序,12个样品原始序列条数为302 439,过滤掉低质量的序列后,有效序列的总数为287 467。根据Barcode标签进行样品序列拆分,在对初始序列进行去冗余处理后,获得16S rRNA基因Unique Reads,并在97%相似度下将其聚类为用于物种分类的OTU (Operational taxonomic units),统计得到各个样品在不同OTU中的丰度信息,12个样品共产生29 005个OTU,平均测序读长在420-480 bp之间。每个样品的有效序列数量如表 3所示。
| 生境类型 Habitat types | Raw tags | Clean tags | Reads | OTUs |
| A4 | 14 410 | 10 073 | 8 899 | 1 717 |
| A7 | 20 882 | 20 282 | 15 470 | 3 102 |
| A11 | 19 181 | 17 061 | 13 708 | 2 642 |
| B4 | 21 236 | 19 794 | 18 436 | 988 |
| B7 | 23 430 | 23 305 | 21 834 | 1 708 |
| B11 | 26 153 | 23 605 | 21 850 | 1 754 |
| C4 | 15 973 | 15 973 | 11 284 | 1 600 |
| C7 | 25 466 | 24 650 | 16 786 | 3 024 |
| C11 | 56 415 | 56 261 | 55 074 | 2 265 |
| D4 | 28 157 | 27 512 | 23 494 | 4 643 |
| D7 | 25 635 | 24 989 | 20 188 | 2 941 |
| D11 | 25 501 | 23 962 | 20 643 | 2 621 |
稀释曲线(rarefaction)反映了样品的取样深度,可以用来评价测序量是否足以覆盖所有类群。从稀释曲线(图 1)中可知,12个样品稀释曲线有一些基本趋于平缓,例如样品C11,曲线已趋于平缓,即再增大数据量对OTU的发现也没有影响,可以说样本C11的OTU的覆盖度已达到饱和;而有一些仍未进入饱和状态,例如样品A4、C4,说明随着测序量的增加可能会发现新的细菌种系。

|
| 图 1 相似度为97%条件下各土壤样品的稀释曲线 Figure 1 Rarefaction curves of each soil sample at cutoff level of 3% |
|
|
2.3.1 Alpha多样性分析:Alpha多样性指数是对单个样品中物种多样性的分析。由表 4可知,从生境类型来看,生境D的平均Chao1指数3 666.25和平均Shannon指数9.65最高;生境A的平均Simpson指数0.99最高;最低的是生境C,Simpson值0.86。从季节变化方面看,OTU数、丰度指数Chao1、多样性Shannon和Simpson指数均是7月份最高,依次是2 694、3 240.40、88.94、0.98。
| 生境类型 Habitat types | OTUs | Chao1指数 Chao1 index | 香农指数 Shannon index | 辛普森指数 Simpson index |
| A4 | 1 717 | 3 011.260 | 8.689 | 0.989 5 |
| A7 | 3 102 | 4 182.282 | 9.789 | 0.997 1 |
| A11 | 2 642 | 3 394.613 | 9.709 | 0.997 1 |
| B4 | 988 | 1 118.684 | 6.326 | 0.956 7 |
| B7 | 1 708 | 1 978.086 | 6.503 | 0.947 3 |
| B11 | 1 754 | 2 052.854 | 7.622 | 0.985 5 |
| C4 | 1 600 | 2 133.440 | 8.929 | 0.993 1 |
| C7 | 3 024 | 3 380.447 | 9.879 | 0.996 9 |
| C11 | 2 265 | 1 987.890 | 3.885 | 0.587 1 |
| D4 | 4 643 | 4 956.987 | 10.239 | 0.993 1 |
| D7 | 2 941 | 3 420.772 | 9.592 | 0.995 6 |
| D11 | 2 621 | 2 621.000 | 9.114 | 0.993 0 |
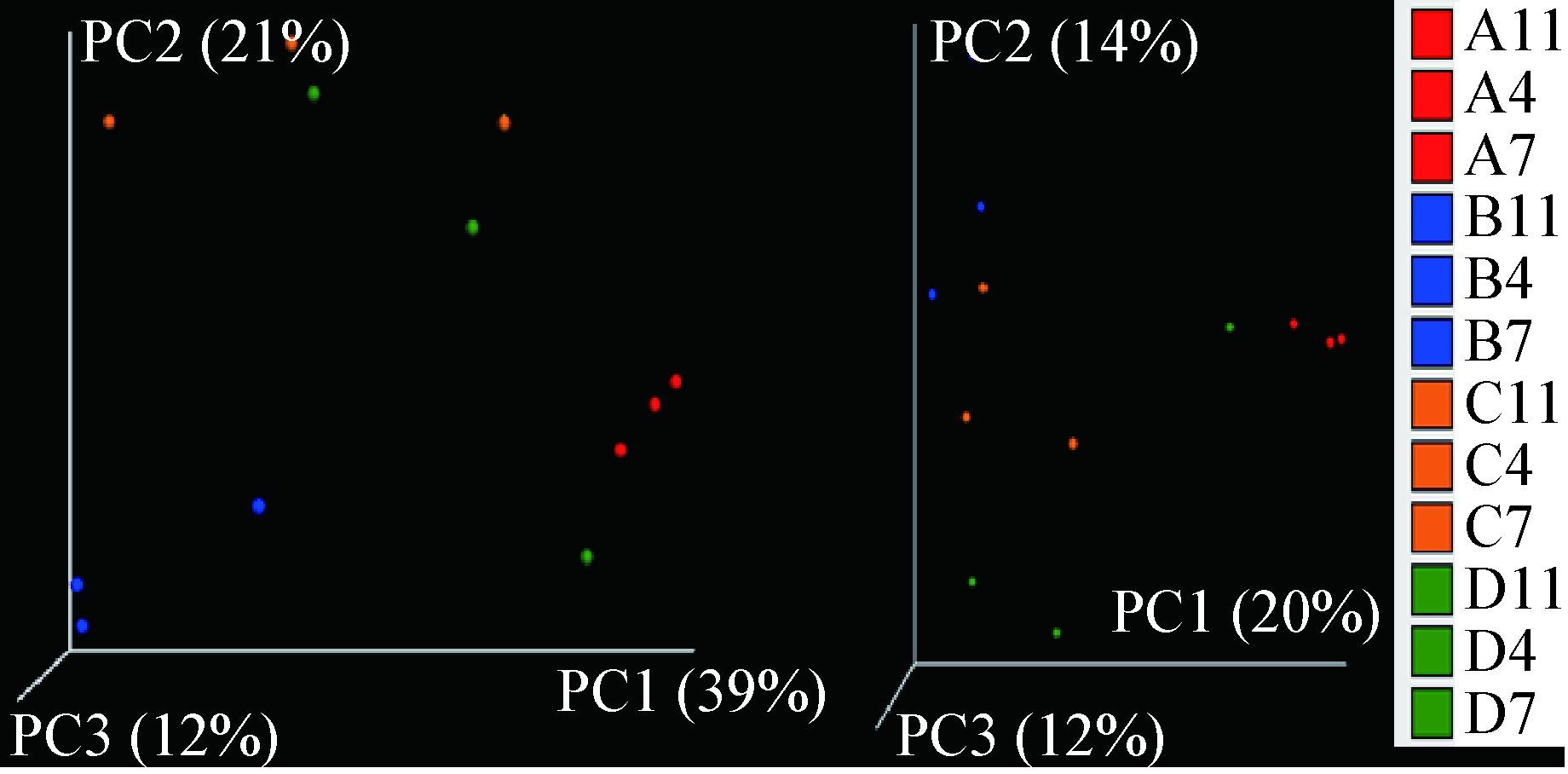
2.3.2 Beta多样性分析:Beta多样性(Beta diversity)分析是用来比较一对样品在物种多样性方面存在的差异大小。UniFrac是通过系统进化的信息来比较样品间的物种群落差异,其计算结果可以作为一种衡量Beta多样性的指数,它考虑了物种间的进化距离,该指数越大表示样品间的差异越大。由图 2可知,基于UniFrac的加权和非加权的主坐标分析发现其第一主成分、第二主成分和第三主成分的贡献率分别为39%、21%和12%,20%、14%和12%,生境A、生境B、生境C、生境D之间明显有分离的现象,但又存在一定的交叉。这种现象说明,有些分类操作单元(OTU)在不同生境之间均存在,但含量不同。其中明显可以看出生境A的4月、7月、11月份的3个样品聚集在一起,说明其组成较为相似,也就是说生境A的微生物群落在季节动态变化方面并无太大差异。
|
| 图 2 Beta多样性的主坐标分析(PCoA)图 Figure 2 Principal coordinate analysis (PCoA) scores plot of beta diversity |
|
|
2.3.3 细菌多样性与理化因子的相关性分析:从表 5可看出,Chao1指数、Shannon指数均与含水量呈极显著负相关关系,与全磷呈显著负相关关系;Simpson指数与含水量、有机质、全氮均呈极显著负相关关系,与全磷呈显著负相关关系。总体来看,土壤细菌多样性指数与理化因子之间呈负相关关系,且均与含水量、全磷相关性较高。
| 理化性质 Physicochemical properties | Chao1指数 Chao1 index | 香农指数 Shannon index | 辛普森指数 Simpson index |
| pH | -0.108 | -0.02 | 0.567 |
| 含水量 Water content | -0.821** | -0.889** | -0.891** |
| 电导率 Electrical conductivity | -0.464 | -0.373 | 0.227 |
| 有机质 Organic matter | -0.150 | -0.280 | -0.924** |
| 全磷 Total phosphorus | -0.609* | -0.642* | -0.612* |
| 全钾 Total potassium | -0.515 | -0.508 | -0.329 |
| 全氮 Total nitrogen | -0.177 | -0.307 | -0.938** |
| 注:*:显著相关(P<0.05) ;**:极显著相关(P<0.01) . Note: *: Significant level for P<0.05; **: Significant level for P<0.01. | |||
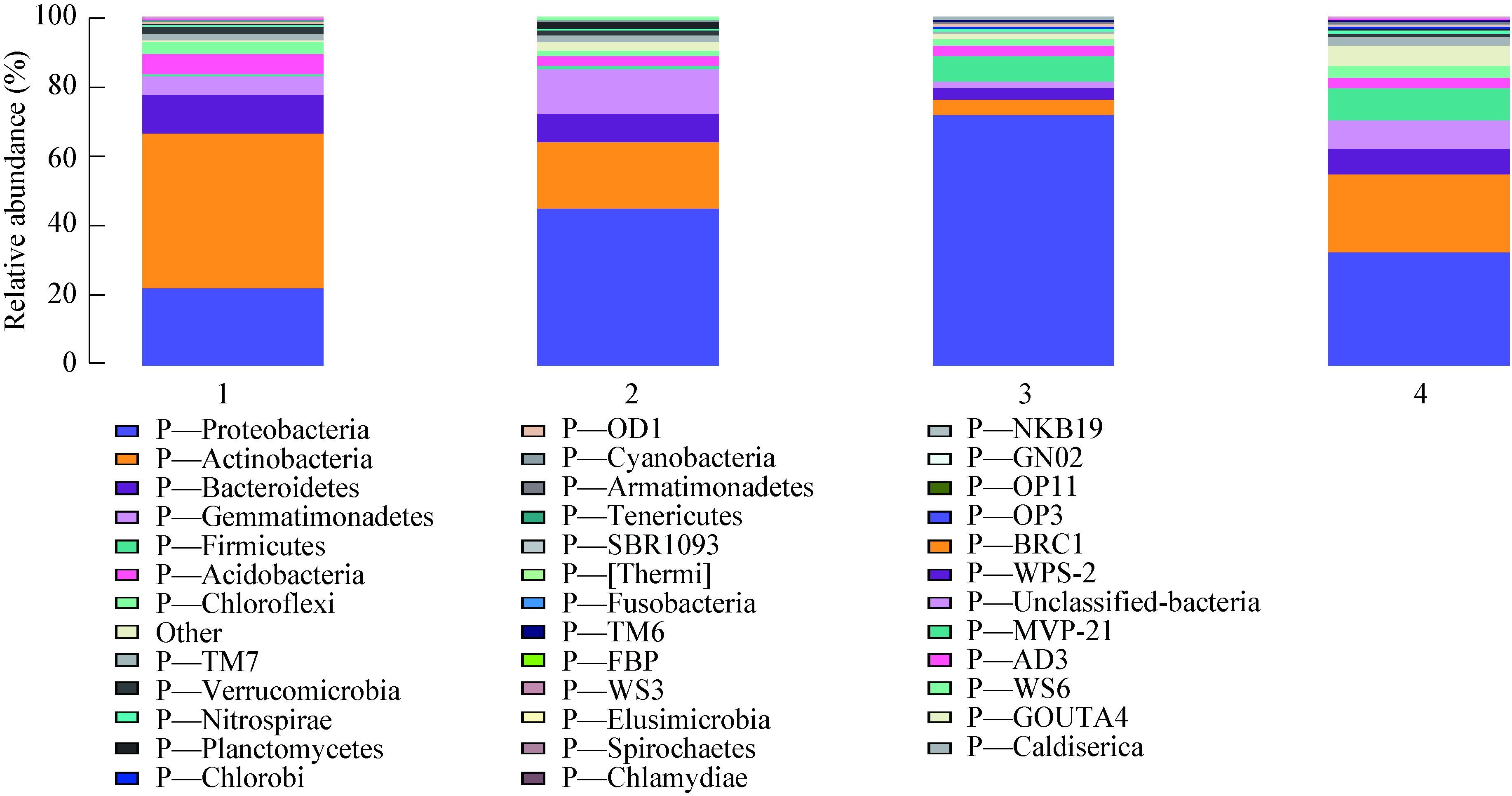
2.4.1 细菌门分类水平的比较:根据图 3,在门分类水平上共得到38种细菌类群,在这些样本中含量最高的门是变形菌门(Proteobacteria),在4类生境中相对丰度分别为22.27%、45.58%、72.44%、32.74%,其次是放线菌门(Actinobacteria)相对丰度分别为44.52%、19.16%、41.12%、22.21%,拟杆菌门(Bacteroidetes)相对丰度分别为11.54%、7.98%、3.59%、7.73%,而其他菌类比例相对较低,例如芽单胞菌门(Gemmatimonadetes)、厚壁菌门(Firmicutes)、酸杆菌门(Acidobacteria)、绿弯菌门(Chloroflexi)、疣微菌门(Verrucomicrobia)等。这表明在泥火山土壤细菌中,变形菌门、放线菌门、拟杆菌门为三大优势菌群,而其他细菌类群为非优势菌群。
|
| 图 3 细菌门分类水平的比较 Figure 3 Comparison of bacteria groups at phylum level |
|
|
2.4.2 细菌属分类水平的比较:根据图 4,在属分类水平上共得到72种细菌类群,其中含量最高的是未分类细菌,在4类生境中相对丰度分别为81.38%、74.34%、81.55%、64.12%;其次是Other、Halomonas (盐单胞菌属)、Kaistobacter、Rhodoplanes (红游动菌属)、Pseudomonas (假单胞菌属)、Streptococcus (链球菌属)、Rubrobacter (红色杆菌属)、Staphylococcus (葡萄球菌属)等,这些菌属的丰度比例相对较低。

|
| 图 4 细菌属分类水平的比较 Figure 4 Comparison of bacteria groups at genus level |
|
|
2.5.1 OTU水平的PCA图:PCA (Principal component analysis) 分析,即主成分分析,是一种分析和简化数据集的技术。通过分析不同样品OTU (97%相似性)组成可以反映样品的差异和距离,如果两个样品距离越近,则表示这两个样品的组成越相似。从图 5可以看出,第一成分的差异贡献值是23.21%,第二成分的差异贡献值是15.35%,其中生境A的3个样品距离最近,说明生境A的OTU群落结构组成在季节动态方面是相似的,这与PCoA分析的结果一致;而生境D的这一组较为分散,说明在季节变化方面OTU组成差异较大,其中生境C和生境D距离较近,说明两者在一部分OTU上有相似的组成成分。

|
| 图 5 基于OTU丰度的PCA分析 Figure 5 Principal component analysis based on OTU level |
|
|
2.5.2 丰度较高的前20个OTU heatmap图: Heatmap图通过颜色变化来反映二维矩阵或表格中的数据信息,将高丰度和低丰度的物种分块聚集,通过颜色梯度及相似程度来反映多个样品在各分类水平上群落组成的相似性和差异性。图 6是在属分类水平上12个样品的群落组成的Heatmap图,每小格代表所在样品中某种属的相对丰度。颜色越红代表相对丰度越高。由图 6可知,在属分类水平上,Shigella flexneri (X96963) (0.006%-64.470%)、OTU116 (0.005%-14.010%)、Halomonas (AY268080) (0.02%-10.90%)、OTU130 (0.022%-10.850%)、Pseudomonas (AB021404) (0.011%-5.170%)相对丰度较高。从群落组成上来看,生境A的7月份和11月份组成相似;生境C 4月份和生境D 11月份组成相似;生境B的4月和7月组成相似;生境B11月份和生境D的7月份组成相似,说明不同样本之间有交叉,且有相似之处。此外,可以明显看到heatmap图中出现大面积的白色方块,这是因为这些菌的含量非常低,导致没有数值。

|
| 图 6 基于丰度比较高的前20个OTU的Heatmap图 Figure 6 Heatmap based on the top 20 OTU abundance of bacteria genera |
|
|
本研究利用Illumina MiSeq测序技术通过对16S rRNA基因的V3-V4高变区的测序分析,成功检测了泥火山土壤细菌群落结构的多样性,获得了大量、全面且深入的菌群信息。12个样品共获得有效序列的总数为287 467,平均测序读长在420-480 bp之间。在97%的相似度水平下共产生有效OTU个数为29 005,通过与RDP数据库[18]进行比对,在门水平上得到优势菌群均为Proteobacteria、Actinobacteria、Bacteroidetes,其中Proteobacteria的丰度最高,这与Roesch等[19]对土壤微生物和Zhang等[20]对污泥微生物群落的研究相一致;在属水平上,含量最高的是未分类细菌,这说明泥火山样品中蕴含着丰富的微生物资源,很多物种是我们现在还未知的,而这些未知的物种为我们探究物种的多样性提供了很好的资源。此外,还有一些功能菌属被检测出,例如,Sphingomonas (鞘氨醇单胞菌属)是一类丰富的新型微生物资源,可用于芳香化合物的生物降解,该属菌株凭借自身的高代谢能力与多功能的生理特性,在环境保护及工业生产方面具有巨大的应用潜力,但是由于对鞘氨醇单胞菌的认识较晚,该菌的生态价值及经济价值很少被关注,对它的研究也停留在初级阶段[21]。这一类菌属的发现可以为我们提供一个新思路,在后续研究中可以通过筛选新的培养基及分离程序,进一步研究可培养微生物及其功能,从而进一步发现有利用价值的资源。
MiSeq测序结果通过丰度指数(Chao1) 和多样性指数(Shannon、Simpson)反映了细菌的群落结构多样性,其中生境D的Shannon指数和Chao1指数最高,生境B和生境C相对较低;在土壤理化因子测定结果中,生境C的含水量、有机质、全磷、全氮和全钾含量最高,生境D的土壤养分(全氮、全磷、全钾)含量相对较低,并且通过细菌群落多样性与理化因子的相关性分析,结果表明泥火山细菌群落多样性随着土壤养分的增加而基本呈下降趋势,说明了物种多样性指数与理化因子之间呈负相关关系,这与陈芳[22]的研究结果一致;群落结构多样性与含水量、全磷的相关性较高,说明泥火山微生物活动对磷循环有着很大的影响,并且磷在生物遗传中也扮演着重要的角色,是影响微生物多样性的一个重要因素,这也曾在高慧琴等[23]的研究中提及过。
OTU水平的PCA分析表明,除了生境A的群落组成在时空演替方面没有差异,生境B和生境C的群落结构均受到了季节变化的影响,尤其是生境D,其3个样品较为分散,说明微生物群落结构的差异较大,群落组成明显不同,而生境C和生境D距离较近,表明两者在一部分OTU上有相似的组成成分,说明这两个生境之间群落组成有相似之处。而且,在Heatmap图中,也更进一步验证了PCA的结果,并发现Shigella flexneri、OTU116、Halomonas、OTU116、Pseudomonas的相对丰度较高。
传统测序技术(Sanger测序)由于其读长长、准确率高,能根据测得的全长16S rRNA基因序列注释到种,但Sanger测序通量较低,大规模测序成本高,操作复杂。Illumina MiSeq高通量测序技术,它能够产生大量Reads,一般每个样品可以保证测定40 000-60 000个序列数,覆盖深度非常大,对物种多样性的分析十分有利,并且在一般规模实验中,它的成本比率更易掌控[24]。但是高通量测序读长短,不可能将16S rRNA基因的9个可变区全部测序,所以往往只选择1-3个可变区作为测序区域。自从Woese等将16S rRNA基因作为原核生物系统发育和分子生态学研究的经典标准以来[25-26],16S rRNA基因便成了细菌群落结构分析常用的系统进化标记分子。然而,由于发现该基因不同区域存在不同程度的异质性,不同可变区的选择对菌群具有偏向性,因而利用不同区域的序列进行高通量测序会造成不同程度的多样性高估,而相关研究[27]指出,基因异质性趋向于集中在特殊的区域,V1和V6区域的高估程度最高,V4和V5区域则显示了最低的高估程度。因此,在本实验中,结合相关方面的文献选择了泥火山细菌16S rRNA基因的V3-V4可变区作为研究对象,从而降低了泥火山细菌多样性分析的高估程度,使得测序结果更加全面和准确。
| [1] | Fan WP, Zheng LQ, Gong JH, et al. Forming of mud volcano and relation with hydrocarbon[J]. Tuha Oil & Gas 2007, 12(1) : 43–47. (in Chinese) 范卫平, 郑雷清, 龚建华, 等. 泥火山的形成及其与油气的关系[J]. 吐哈油气 2007, 12(1) : 43–47. |
| [2] | Ma XL, Wang Y, Yang HM, et al. The bacterial diversity from mud volcano in Xinjiang by culture-independent approach[J]. Acta Ecologica Sinica 2009, 29(7) : 3722–3728. (in Chinese) 马小龙, 王芸, 杨红梅, 等. 新疆泥火山细菌遗传多样性[J]. 生态学报 2009, 29(7) : 3722–3728. |
| [3] | Lebrun JD, Trinsoutrot-Gattin I, Vinceslas-Akpa M, et al. Assessing impacts of copper on soil enzyme activities in regard to their natural spatiotemporal variation under long-term different land uses[J]. Soil Biology and Biochemistry 2012(49) : 150–156. |
| [4] | Chen FL, Zhang K, Zheng H, et al. Analyzing the effect of mixed decomposition of conifer and broadleaf litters on soil microbial communities by using RCR-DGGE[J]. Chinese Journal of Applied & Environmental Biology 2011, 17(2) : 145–150. (in Chinese) 陈法霖, 张凯, 郑华, 等. PCR-DGGE技术解析针叶和阔叶凋落物混合分解对土壤微生物群落结构的影响[J]. 应用与环境生物学报 2011, 17(2) : 145–150. |
| [5] | Tang J, Xu QR, Wang LM, et al. Soil bacterial community diversity under different stages of degradation in Zoige wetland[J]. Microbiology China 2011, 38(5) : 677–686. (in Chinese) 唐杰, 徐青锐, 王立明, 等. 若尔盖高原湿地不同退化阶段的土壤细菌群落多样性[J]. 微生物学通报 2011, 38(5) : 677–686. |
| [6] | Chen GH, Mi BB, Li Y, et al. The effect of transgenic cucumber with double strands RNA of mapk on diversity of rhizosphere bacteria[J]. Acta Ecologica Sinica 2013, 33(4) : 1091–1097. (in Chinese) 陈国华, 弭宝彬, 李莹, 等. 转mapk双链 RNA干扰表达载体黄瓜对根际土壤细菌多样性的影响[J]. 生态学报 2013, 33(4) : 1091–1097. DOI:10.5846/stxb |
| [7] | Loman NJ, Misra RV, Dallman TJ, et al. Performance comparison of bench top high -throughput sequencing platforms[J]. Nature Biotechnology 2012, 30(5) : 434–439. DOI:10.1038/nbt.2198 |
| [8] | Yang SZ, Wen X, Jin HJ, et al. Pyrosequencing investigation into the bacterial community in permafrost soils along the China-Russia crude oil pipeline (CRCOP)[J]. PLoS One 2012, 7(12) : e52730. DOI:10.1371/journal.pone.0052730 |
| [9] | Liu B, Faller LL, Klitgord N, et al. Deep sequencing of the oral microbiome reveals signatures of periodontal disease[J]. PLoS One 2012, 7(6) : e37919. DOI:10.1371/journal.pone.0037919 |
| [10] | Duan Z, Xiao W, Wang YX, et al. Application of 454 sequencing technique in microbial ecology[J]. Journal of Microbiology 2011, 31(5) : 76–81. (in Chinese) 段曌, 肖炜, 王永霞, 等. 454测序技术在微生物生态学研究中的应用[J]. 微生物学杂志 2011, 31(5) : 76–81. |
| [11] | Po ka J, Rebecchi A, Pisacane V, et al. Bacterial diversity in typical Italian salami at different ripening stages as revealed by high-throughput sequencing of 16S rRNA amplicons[J]. Food Microbiology 2015(46) : 342–356. |
| [12] | Nelson MC, Morrison HG, Benjamino J, et al. Analysis, optimization and verification of Illumina-generated 16S rRNA gene amplicon surveys[J]. PLoS One 2014, 9(4) : e94249. DOI:10.1371/journal.pone.0094249 |
| [13] | Bao SD. Soil Agricultural Chemistry Analysis[M].3rd Edition. Beijing: China Agriculture Press, 1981: 30 -102. (in Chinese) 鲍士旦. 土壤农化分析[M].第3版. 北京: 中国农业出版社, 1981,: 30 -102. |
| [14] | Chao AN. Nonparametric estimation of the number of classes in a population[J]. Scandinavian Journal of Statistics 1984, 11(4) : 265–270. |
| [15] | Hill MO. Diversity and evenness: a unifying notation and its consequences[J]. Ecology 1973, 54(2) : 427–432. DOI:10.2307/1934352 |
| [16] | Esty WW. The efficiency of Good's nonparametric coverage estimator[J]. The Annals of Statistics 1986, 14(3) : 1257–1260. DOI:10.1214/aos/1176350066 |
| [17] | Landau S, Everitt BS. A Handbook of Statistical Analyses Using SPSS[M]. Florida: Chapman & Hall, 2004 . |
| [18] | Wang Q, Garrity GM, Tiedje JM, et al. Naïve Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J]. Applied and Environmental Microbiology 2007, 73(16) : 5261–5267. DOI:10.1128/AEM.00062-07 |
| [19] | Roesch LFW, Fulthorpe RR, Riva A, et al. Pyrosequencing enumerates and contrasts soil microbial diversity[J]. The ISME Journal 2007, 1(4) : 283–290. |
| [20] | Zhang T, Shao MF, Ye L. 454 Pyrosequencing reveals bacterial diversity of activated sludge from 14 sewage treatment plants[J]. The ISME Journal 2012, 6(6) : 1137–1147. DOI:10.1038/ismej.2011.188 |
| [21] | Hu J, He XH, Li DP, et al. Progress in research of sphingomonas[J]. Chinese Journal of Applied & Environmental Biology 2007, 13(3) : 431–437. (in Chinese) 胡杰, 何晓红, 李大平, 等. 鞘氨醇单胞菌研究进展[J]. 应用与环境生物学报 2007, 13(3) : 431–437. |
| [22] | Chen F. Study on the relationship between Plant diversity and soil Properties in Mangshan Nature Reserve[D]. Changsha: Master's Thesis of Hunan Agricultural University, 2011 (in Chinese) 陈芳. 莽山植物物种多样性及其土壤理化性质关系的研究[D]. 长沙: 湖南农业大学硕士学位论文, 2011 |
| [23] | Gao HQ, Liu L, Fang ZJ. Analysis of microbial diversity in Superficial deposit of lakes in Lixia River Area in Summer[EB/OL]. Beijing: China Science And Technology Papers Online, [2010-12]. http://www.paper.edu.cn/releasepaper/content/201012-264 (in Chinese) 高慧琴, 刘凌, 方泽建. 里下河地区湖群夏季表层沉积物微生物多样性分析[EB/OL]. 北京: 中国科技论文在线, [2010-12]. http://www.paper.edu.cn/releasepaper/content/201012-264 |
| [24] | Bokulich NA, Subramanian S, Faith JJ, et al. Quality-filtering vastly improves diversity estimates from Illumina amplicon sequencing[J]. Nature Methods 2013, 10(1) : 57–59. |
| [25] | Woese CR. Bacterial evolution[J]. Microbiological Reviews 1987, 51(2) : 221–271. |
| [26] | Woese CR, Kandler O, Wheelis ML. Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya[J]. Proceedings of the National Academy of Sciences United States of America 1990, 87(12) : 4576–4579. DOI:10.1073/pnas.87.12.4576 |
| [27] | Sun DL, Jiang X, Wu QL, et al. Intragenomic heterogeneity of 16S rRNA genes causes overestimation of prokaryotic diversity[J]. Applied and Environmental Microbiology 2013, 79(19) : 5962–5969. DOI:10.1128/AEM.01282-13 |
 2016, Vol. 43
2016, Vol. 43