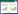扩展功能
文章信息
- 马箭, 陆震鸣, 张晓娟, 王松涛, 沈才洪, 史劲松, 许正宏
- MA Jian, LU Zhen-Ming, ZHANG Xiao-Juan, WANG Song-Tao, SHEN Cai-Hong, SHI Jing-Song, XU Zheng-Hong
- 泸型酒酒醅中梭菌群落的发酵演替规律和功能预测
- Role of Clostridia community in fermented grains during Luzhou-flavor liquor production
- 微生物学通报, 2016, 43(10): 2322-2329
- Microbiology China, 2016, 43(10): 2322-2329
- DOI: 10.13344/j.microbiol.china.160110
-
文章历史
- 收稿日期: 2016-01-28
- 接受日期: 2016-04-12
- 优先数字出版日期(www.cnki.net): 2016-05-04
2. 国家固态酿造工程技术研究中心 四川 泸州 646000 ;
3. 中国科学院天津工业生物技术研究所 天津市工业生物系统与过程工程重点实验室 天津 300308
2. National Engineering Research Center of Solid-State Brewing, Luzhou, Sichuan 646000, China ;
3. Tianjin Key Laboratory for Industrial Biological Systems and Bioprocessing Engineering, Tianjin Institute of Industrial Biotechnology, Chinese Academy of Sciences, Tianjin 300308, China
泸型酒(又称浓香型白酒)产量占中国白酒70%以上,且因其窖香浓郁、绵软甘冽等风味特征而被消费者广泛接受[1]。泸型酒的生产以高粱为主要原料,以大曲为糖化发酵剂,采用泥窖进行多菌种混合循环固态发酵。通过分离培养和分子生物学手段,发现窖池中存在的微生物主要包括酵母、霉菌、细菌、古菌,并呈现出极高的多样性[2-3]。在发酵阶段,长期驯化形成的独特而复杂的窖内微生物群落是泸型酒独特风味形成的关键[4]。
梭菌(Clostridia)是一系列厌氧的厚壁菌门(Firmicutes)微生物的统称,其典型为梭状芽孢杆菌属(Clostridium)微生物。在浓香型白酒酿造环境,尤其是窖泥中,梭菌被认为与己酸、丁酸等酒体重要风味物质的合成密切相关[5]。在前期研究中,已有部分梭状芽孢杆菌属微生物,如克鲁氏梭菌(Clostridium kluyveri)、泸型梭菌(Clostridium lushun)等从窖泥中被分离并鉴定[6, 7]。有趣的是,虽然已有关于窖泥中的梭菌群落的研究[8],但是就作为发酵主体的酒醅来说,其中的梭菌群落演替目前尚未有针对性的文献报道。
本研究主要利用分子生物学手段,针对酒醅中梭菌群落的丰度变化、结构演替以及功能组成变化进行分析,以期总结酒醅发酵过程中梭菌的演变规律,完善对梭菌在酿造过程中所起作用的认识,为完整理解浓香型白酒酿造机制提供支持。
1 材料与方法 1.1 样品采集本研究使用的酒醅样品采自四川泸州老窖集团酿酒基地某连续生产的窖池(长×宽× 高=2.5 m×2.0 m×3.0 m)。采样时间点选择入窖后0、7、14、20、30和40 d。采样方式为在窖池近中心部位进行三点采样,每个点采样约1.0 g。所有平行样当场充分混匀后迅速放入无菌保鲜袋中,-20 ℃保存。
1.2 主要试剂和仪器引物购自上海生工生物工程有限公司;PCR试剂、琼脂糖凝胶DNA回收试剂盒、pMD-19T载体、2×SYBR Premix ExTaq预混液购自大连宝生物公司;PowerSoil®DNA提取试剂盒购自美国MOBIO公司。
NanoDrop 3300分光光度计,美国Thermo Fisher Scientific公司;荧光定量PCR仪Bio-Rad CFX Real-Time System,美国Bio-Rad公司;454平台测序仪Genome Sequencer FLX Titanium,美国Roche公司;Illumina平台测序仪Illumina MiSeq Benchtop Sequencer,美国Illumin a公司。
1.3 宏基因组提取每份样品准确称取0.2 g进行宏基因组提取:采用PowerSoil®DNA提取试剂盒,操作步骤参照说明书。每一个发酵时间点设两个平行,提取后等体积混合。对DNA浓度和质量进行测定,分装并冻存于-80 ℃。
1.4 梭菌实时荧光定量PCR条件及标准曲线 制作每个DNA样品设3个复孔,采用20 μL体系:预混液10 μL,10 μmol/L上下游引物(SJ-F/SJ-R)[8]各1 μL,模板(统一稀释至10 mg/L) 2 μL,ddH2O 7 μL。反应条件:94 ℃ 5min; 94 ℃ 15 s,58 ℃ 25 s,共50个循环。熔解曲线绘制:55 ℃梯度升温至95 ℃,每0.5 min读板一次。
以本实验室分离的Clostridium kluyveri菌株CKL01的16S rRNA基因扩增产物(27F/1492R)连接载体,转化大肠杆菌JM109。提取质粒,测定吸光值并换算为质粒拷贝数,10倍梯度稀释为模板制作标准曲线[9]。
1.5 16S rRNA基因序列扩增及高通量测序采用细菌通用引物P1/P2扩增V1/V3高变区(大肠杆菌16S rRNA基因位点5-534)[10],退火温度为55 ℃;针对梭菌的引物SJ-F/SJ-R扩增V4/V5高变区(大肠杆菌16S rRNA基因位点679-952),退火温度为56.5 ℃。在每条引物5′端加上18 bp条码序列作为样本标记。文库构建时每份样品设置2个扩增平行,测定浓度后混合,上机测序。V1/V3区扩增产物采用454平台测序仪进行测序;V4/V5区扩增产物采用Illumina平台测序仪进行测序(上海美吉)。
1.6 高通量测序数据统计分析测序下机文件使用Mothur进行融合,并依据条码序列区分样品。质检并滤除符合以下其中任意一项的序列:短于150 bp、平均质量得分小于20、包含模糊碱基及包含8个以上连续相同碱基。使用UCLUST算法(http://drive5.com/usearch/manual/ uclust_algo.html)依照97%相似度进行OTU划分,并依照80%置信度采用Greengenes和RDP数据库对各OTU进行分类学注释。
对SJ-F/SJ-R引物测序结果,根据各OTU相对丰度变化采用Person距离进行相似度计算,并依据average-linkage对样品进行聚类。不同聚类的标志性OTU通过LEfSe (LDA Effect Size)分析找出[11]。各样品序列的注释结果运用PICRUSt (Phylogenetic investigation of communities by reconstruction of unobserved states)进行功能预测[12]。
2 结果与分析 2.1 不同发酵时间酒醅中梭菌丰度变化以2.52×107 copies/μL为起始浓度,设6组连续10倍梯度稀释的质粒模板,制作标准曲线。扩增曲线显示Cq值均落在10-30之间,数值可信;熔解曲线于约84 ℃显示单一峰,证明扩增特异性良好。以Cq值为横坐标,模板浓度对数为纵坐标,线性拟合得到标准曲线方程,R2为0.997 65,证明标准曲线线性良好。见图 1。

|
| 图 1 SJ-F/SJ-R 引物对Clostridium kluyveri 进行实时荧光定量PCR Figure 1 Real-time quantitative PCR of Clostridium kluyveri using primer pair SJ-F/SJ-R Note: A–C: amplification curve, melting curve and standard curve |
|
|
依据标准曲线计算样品DNA中梭菌16S rRNA基因拷贝数,并换算为不同发酵时间每克酒醅样品中梭菌16S rRNA基因拷贝数。结果显示酒醅中梭菌16S rRNA基因拷贝数随发酵时间呈先上升后下降趋势;在发酵14 d和20 d达到最大值3.46×107 copies/g (图 2)。
|
| 图 2 不同发酵时间酒醅梭菌16S rRNA 基因拷贝数(A)及占总细菌相对丰度(B) Figure 2 16S rRNA gene copy number (A) and relative abundance (B) of Clostridia community in different samples |
|
|
各样品V3/V4区测序质检合格的Reads数统一至4 454,并对其中梭菌的相对丰度进行统计。结果显示酒醅中梭菌属于低丰度微生物,占总细菌的相对丰度在0.04%-6.67%间浮动,呈现先上升后下降趋势(图 2)。在0 d样品中相对丰度最小,20 d样品中相对丰度最大;入窖7 d内梭菌相对丰度上升最为显著。
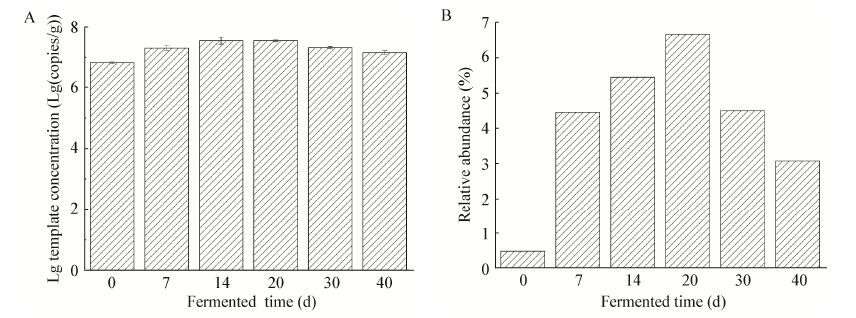
2.2 不同发酵时间酒醅梭菌群落结构变化由于酒醅中梭菌相对丰度较低,因此采用SJ-F/SJ-R引物选择性扩增梭菌V4/V5高变区。各样品质检合格的Reads数统一至20 000,依97%相似度划分共得到152个相对丰度大于千分之一的OTU。依照其中前100个OTU在各样品中的相对丰度变化对样品聚类(图 3),发现在发酵7 d样品中梭菌群落结构与其他样品有较大差异;此外,30 d、40 d样品形成一个聚类,而0、14、20 d样品形成另一聚类。其中14 d样品又与0 d、20 d样品具有一定差异。聚类结果表明:酒醅梭菌群落结构在入窖一周内发生了较为剧烈的变化,随后逐渐回复,并在发酵第三周左右恢复至与入窖时接近。而在发酵后期,伴随着生物量的衰减,梭菌群落结构又发生了一定程度的变化。
|
| 图 3 SJ-F/SJ-R 测序结果前100 个OTU 在不同样品中的分布 Figure 3 Distribution of the most abundant 100 OTUs of SJ-F/SJ-R sequences in different samples |
|
|
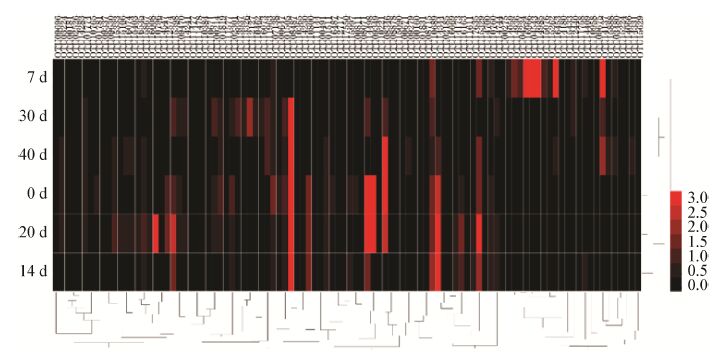
由于发现发酵7 d样品梭菌群落结构较其他样品有较大差异,因此以发酵7 d样品为一组,其他样品为另一组,运用LEfSe分析差异OTU (图 4)。结果显示有17个OTU在发酵7 d样品与其他样品中相对丰度有显著差异;其中OTU 7298、OTU 9349、OTU 14137、OTU 6251在发酵7 d样品中相对丰度显著高于其他样品,而其余13个OTU则相反。BLAST结果显示各OTU代表序列大部分与GenBank现有收录序列相似度较低(表 1),可能为一些分类地位尚不明确的微生物。
|
| 图 4 发酵7 d 样品与其他样品差异OTU Figure 4 Biomarkers of sample 7 d and other samples on OTU-level |
|
|
| OTU编号 OTU No. | 参考菌株 Reference strain | E值 E-value | 相似度 Identity (%) | 登录号 Accession No. |
| 7298 | Hydrogenisporaethanolica LX-B | 2.00E-122 | 95 | NR_125455.1 |
| 9349 | Clostridium butyricum JCM 1391 | 1.00E-138 | 99 | NR_113244.1 |
| 14137 | Clostridium tagluense A121 | 3.00E-115 | 94 | NR_043698.1 |
| 6251 | Sporomusaaerivorans TMAO3 | 4.00E-119 | 95 | NR_028991.1 |
| 214 | Desulfosporosinusacidiphilus SJ4 | 9.00E-121 | 95 | NR_074132.1 |
| 1505 | Clostridium tyrobutyricum ATCC 25755 | 9.00E-131 | 97 | NR_044718.2 |
| 4543 | Clostridium tyrobutyricum ATCC 25755 | 2.00E-137 | 99 | NR_044718.2 |
| 10796 | Clostridium butyricum JCM 1391 | 3.00E-130 | 97 | NR_113244.1 |
| 10800 | Eubacteriumpyruvativorans I-6 | 5.00E-128 | 97 | NR_042074.1 |
| 16163 | Sedimentibacterhydroxybenzoicus JW/Z-1 | 1.00E-133 | 98 | NR_029146.1 |
| 15867 | Sedimentibacterhydroxybenzoicus JW/Z-1 | 1.00E-118 | 95 | NR_029146.1 |
| 1511 | [Clostridium] populeti 743A | 3.00E-130 | 97 | NR_026103.1 |
| 14985 | Catabacterhongkongensis HKU16 | 4.00E-119 | 95 | NR_115269.1 |
| 16966 | AlkaliphilusoremlandiiOhILAs | 3.00E-120 | 95 | NR_074435.1 |
| 1819 | Oxobacterpfennigii DSM 3222 | 2.00E-112 | 93 | NR_117690.2 |
| 11477 | Clostridium sporogenes JCM 1416 | 7.00E-122 | 95 | NR_113245.1 |
| 1726 | Hydrogenisporaethanolica LX-B | 9.00E-126 | 96 | NR_125455.1 |
运用PICRUSt对不同发酵时间点样品梭菌功能组成进行预测(图 5),发现在主要代谢分类上碳水化合物代谢、氨基酸代谢、辅因子及维生素代谢以及核酸代谢有较多基因被预测。与发酵0 d相比,发酵7 d酒醅中预测到基因数大幅度减少,而后逐渐回复,在发酵20 d时基本与入窖时持平;在发酵后期预测到基因数再次减少。这与梭菌群落结构的变化趋势相符合。
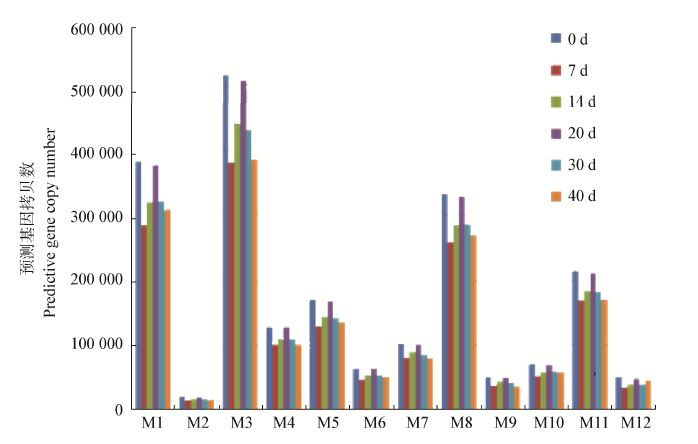
|
| 图 5 梭菌群落各代谢分类功能预测 Figure 5 Functional prediction of Clostridia community in catalog ‘metabolism’ 注:M1:氨基酸代谢;M2:其他次级代谢物生物合成;M3:碳水化合物代谢;M4:能量代谢;M5:酶家族;M6:多糖生物合成及代谢;M7:脂质代谢;M8:辅因子及维生素代谢;M9:其他氨基酸代谢;M10:萜类及聚酮代谢;M11:核酸代谢;M12:外源性化合物生物降解及代谢. Note:M1: Amino acid metabolism; M2: Biosynthesis of other secondary metabolites; M3: Carbohydrate metabolism; M4: Energy metabolism; M5: Enzyme families; M6: Glycan biosynthesis and metabolism; M7: Lipid metabolism; M8: Metabolism of Co-factors and vitamins; M9: Metabolism of other amino acids; M10: Metabolism of terpenoids and polyketides; M11: Nucleotide metabolism; M12: Xenobiotics biodegradation and metabolism. |
|
|
单独对碳水化合物功能预测结果进行分析,发现各代谢途径上的预测基因数量变化趋势也基本与梭菌群落结构变化趋势相符合(图 6);其中氨糖与核糖代谢、磷酸戊糖途径显著高于其他代谢途径,其次是三羧酸循环、糖酵解、丙酸及丁酸代谢途径上的一些基因。
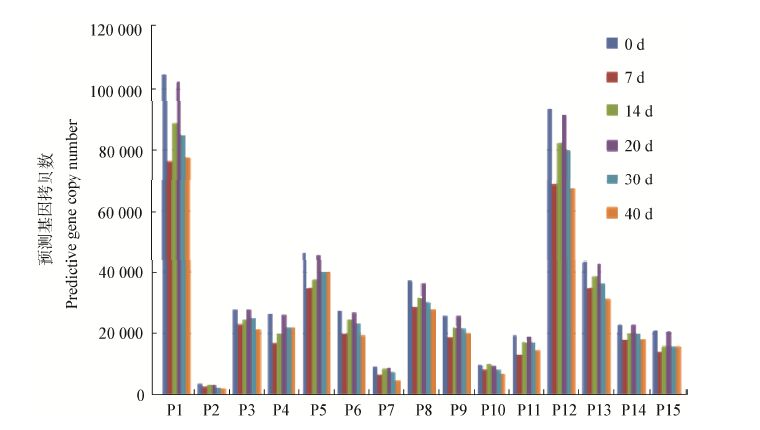
|
| 图 6 梭菌群落碳水化合物代谢个途径功能预测 Figure 6 Functional prediction of Clostridia community in catalog ‘carbohydrate metabolism’ 注: P1:氨糖与核糖代谢;P2:抗坏血酸代谢;P3:丁酸代谢;P4:五碳支链酸代谢;P5:三羧酸循环;P6:果糖和甘露糖代谢;P7:半乳糖代谢;P8:糖酵解;P9:乙醛酸盐代谢;P10:磷酸肌醇代谢;P11:戊糖与葡萄糖醛酸转换;P12:磷酸戊糖途径;P13:丙酸代谢;P14:丙酮酸代谢;P15:淀粉和蔗糖代谢. Note: P1: Amino sugar and nucleotide sugar metabolism; P2: Ascorbate and aldarate metabolism; P3: Butanoate metabolism; P4: C5-Branched dibasic acid metabolism; P5: Citrate cycle (TCA cycle); P6: Fructose and mannose metabolism; P7: Galactose metabolism; P8: Glycolysis/Gluconeogenesis; P9: Glyoxylate and dicarboxylate metabolism; P10: Inositol phosphate metabolism; P11: Pentose and glucuronateinterconversions; P12: Pentose phosphate pathway; P13: Propanoate metabolism; P14: Pyruvate metabolism; P15: Starch and sucrose metabolism. |
|
|
在浓香型白酒中,己酸和丁酸是合成主体风味化合物己酸乙酯和丁酸乙酯的重要前体[13]。前期研究发现包括梭状芽孢杆菌在内的各种梭菌与白酒中己酸和丁酸的合成具有密切关系[5]。由于梭菌在窖泥中相对丰度较高[14],而酒醅中则是乳酸菌占有绝对优势[15, 16],因此目前为止绝大多数梭菌相关研究集中于窖泥[17];考虑到酒醅是窖内发酵的主体,酒醅微生物产生的风味物质会在酒醅蒸馏阶段进入酒体[18],因此本研究就酒醅中的梭菌群落进行了考察。
通过实时荧光定量PCR和高通量测序数据分析,我们发现酒醅发酵过程中梭菌的16S rRNA基因拷贝数在106-108之间,平均相对丰度占总细菌的4.10%左右,属于低丰度微生物。同时梭菌的生物量和在细菌群落中的相对丰度均呈现先上升后下降趋势,在14-21 d内达到最高。前期的生物量上升可能因为封窖后窖池内逐渐形成的厌氧环境有利于梭菌的繁殖。另外,发酵过程中酒醅酸度的逐级增加会导致许多不耐酸的微生物消亡,而梭菌具有一定的耐受和代谢有机酸的能力[19],这可能是发酵7 d内梭菌相对丰度大幅升高的重要原因。
针对梭菌进行高通量测序,并依据优势OTU对样本进行聚类,我们发现发酵7 d样品的梭菌群落结构与其他样品显著不同。LEfSe分析发现发酵7 d样品与其他样品相比存在17个主要差异OTU,代表序列BLAST结果显示主要为一些分类地位尚不明确的微生物,有待进一步研究。
本研究采用了PICRUSt分析对酒醅中梭菌群落进行了功能预测,尤其是碳水化合物代谢相关途径的功能预测,结果显示梭菌能够利用多种底物,并对酒体风味物质的合成起到了一定作用。结合梭菌生物量的变化,我们发现在发酵14-21 d时有较多相关基因被预测到。此外,在不考虑生物量影响的前提下,梭菌的代谢功能组成变化基本与群落结构变化趋势一致,说明梭菌群落结构的变化是造成其整体代谢功能组成变化的重要原因。
综上所述,本研究综合生物量、群落结构及功能组成三个方面,对泸型酒发酵过程酒醅中梭菌群落进行了考察,为深入理解梭菌在泸型酒酿造中的作用提供了理论依据。
| [1] | Tao Y, Li JB, Rui JP, et al. Prokaryotic communities in pit mud from different-aged cellars used for the production of Chinese strong-flavored liquor[J]. Applied and Environmental Microbiology , 2014, 80 (7) : 2254–2260. DOI:10.1128/AEM.04070-13 |
| [2] | Li K, Zhang Q, Zhong XT, et al. Microbial diversity and succession in the Chinese Luzhou-flavor liquor fermenting cover lees as evaluated by SSU rRNA profiles[J]. Indian Journal of Microbiology , 2013, 53 (4) : 425–431. DOI:10.1007/s12088-013-0382-3 |
| [3] | Wang CD, Chen Q, Wang Q, et al. Long-term batch brewing accumulates adaptive microbes, which comprehensively produce more flavorful Chinese liquors[J]. Food Research International , 2014, 62 : 894–901. DOI:10.1016/j.foodres.2014.05.017 |
| [4] | Hou XG, Wang JY, Li XS, et al. The research progress on functional aroma-producing microorganisms in Zaopei and pit mud of Chinese strong-flavor liquor[J]. Microbiology China , 2013, 40 (7) : 1257–1265. (in chinese) 侯小歌, 王俊英, 李学思, 等. 浓香型白酒糟醅及窖泥产香功能菌的研究进展[J]. 微生物学通报 , 2013, 40 (7) : 1257–1265. |
| [5] | Lei GD, Yao WC, Tang YM, et al. Research on the distribution of important functional microbial communities in LuzhouLaojiao pit mud and their metabolic products[J]. Liquor-Making Science & Technology , 2012 (11) : 54–57. (in chinese) 雷光电, 姚万春, 唐玉明, 等. 泸州老窖窖泥中重要功能菌群分布及代谢产物研究[J]. 酿酒科技 , 2012 (11) : 54–57. |
| [6] | Hu XL, Du H, Xu Y. Identification and quantification of the caproic acid-producing bacterium Clostridium kluyveri in the fermentation of pit mud used for Chinese strong-aroma type liquor production[J]. International Journal of Food Microbiology , 2015, 214 : 116–122. DOI:10.1016/j.ijfoodmicro.2015.07.032 |
| [7] | Wu YY. Applied theory & practice of Lu-type Clostridium lushun fermentation[J]. Liquor-Making Science & Technology , 2007 (11) : 131–132. (in chinese) 吴衍庸. 泸型梭菌己酸发酵应用的理论与实践[J]. 酿酒科技 , 2007 (11) : 131–132. |
| [8] | Hu XL, Wang HY, Wu Q, et al. Development, validation and application of specific primers for analyzing the Clostridial diversity in dark fermentation pit mud by PCR-DGGE[J]. Bioresource Technology , 2014, 163 : 40–47. DOI:10.1016/j.biortech.2014.04.008 |
| [9] | Tao JL, Lu ZM, Wang ZM, et al. Detection of the variation of microorganisms in acetic acid fermentation of Zhenjiang aromatic vinegar through real-time quantitative PCR[J]. Food and Fermentation Industries , 2013, 39 (2) : 156–160. (in chinese) 陶京兰, 陆震鸣, 王宗敏, 等. 实时荧光定量PCR监测镇江香醋醋酸发酵过程中微生物变化[J]. 食品与发酵工业 , 2013, 39 (2) : 156–160. |
| [10] | Huang S, Yang F, Zeng XW, et al. Preliminary characterization of the oral microbiota of Chinese adults with and without gingivitis[J]. BMC Oral Health , 2011, 11 : 33. DOI:10.1186/1472-6831-11-33 |
| [11] | Segata N, Izard J, Waldron L, et al. Metagenomic biomarker discovery and explanation[J]. Genome Biology , 2011, 12 (6) : R60. DOI:10.1186/gb-2011-12-6-r60 |
| [12] | Langille MGI, Zaneveld J, Caporaso JG, et al. Predictive functional profiling of microbial communities using 16S rRNA marker gene sequences[J]. Nature Biotechnology , 2013, 31 (9) : 814–821. DOI:10.1038/nbt.2676 |
| [13] | Yan SB, Wang SC, Wei GG, et al. Investigation of the main parameters during the fermentation of Chinese Luzhou-flavour liquor[J]. Journal of the Institute of Brewing , 2015, 121 (1) : 145–154. DOI:10.1002/jib.v121.1 |
| [14] | Liu MK, Zhao K, Tang YM, et al. Analysis of Clostridium cluster I community diversity in pit mud used in manufacture of Chinese Luzhou-flavor liquor[J]. Food Science and Biotechnology , 2015, 24 (3) : 995–1000. DOI:10.1007/s10068-015-0127-7 |
| [15] | Zhang WX, Qiao ZW, Hu C, et al. Analysis of bacterial community in fermented grains during the production of Chinese strong aromatic spirits by PCR technique[J]. Journal of Sichuan University (Engineering Science Edition) , 2005, 37 (5) : 82–87. (in chinese) 张文学, 乔宗伟, 胡承, 等. PCR技术对浓香型白酒糟醅细菌菌群的解析[J]. 四川大学学报: 工程科学版 , 2005, 37 (5) : 82–87. |
| [16] | Zhang WX, Qiao ZW, Shigematsu T, et al. Analysis of the bacterial community in Zaopei during production of Chinese Luzhou-flavor liquor[J]. Journal of the Institute of Brewing , 2005, 111 (2) : 215–222. DOI:10.1002/jib.2005.111.issue-2 |
| [17] | Zheng J, Liang R, Zhang LQ, et al. Characterization of microbial communities in strong aromatic liquor fermentation pit muds of different ages assessed by combined DGGE and PLFA analyses[J]. Food Research International , 2013, 54 (1) : 660–666. DOI:10.1016/j.foodres.2013.07.058 |
| [18] | Lang ZW, Lu ZM, Gong JS, et al. Compositional discrepancy of volatile compounds in fermented grains and distilled grains of Luzhou-flavor liquor[J]. Food and Fermentation Industries , 2015, 41 (7) : 34–37. (in chinese) 郎召伟, 陆震鸣, 龚劲松, 等. 泸型酒蒸馏前后酒醅中挥发性物质的差异性分析[J]. 食品与发酵工业 , 2015, 41 (7) : 34–37. |
| [19] | Vital M, Gao JR, Rizzo M, et al. Diet is a major factor governing the fecal butyrate-producing community structure across Mammalia, Aves and Reptilia[J]. The ISME Journal , 2015, 9 (4) : 832–843. DOI:10.1038/ismej.2014.179 |
 2016, Vol. 43
2016, Vol. 43