扩展功能
文章信息
- 黄新新, 何苗, 韩伟, 沈文淑, 顾鸣, 蔡强
- HUANG Xin-Xin, HE Miao, HAN Wei, SHEN Wen-Sh, GU Ming, CAI Qiang
- 迟缓爱德华氏菌EcoRⅠ核糖体自动化分型与聚类分析
- Characterization of Edwardsiella tarda by automated EcoRⅠ ribotyping and cluster analysis
- 微生物学通报, 2016, 43(10): 2249-2253
- Microbiology China, 2016, 43(10): 2249-2253
- DOI: 10.13344/j.microbiol.china.150881
-
文章历史
- 收稿日期: 2015-11-06
- 接受日期: 2016-03-08
- 优先数字出版日期(www.cnki.net): 2016-03-09
2. 清华大学环境学院 北京 100084 ;
3. 浙江清华长三角研究院 浙江 嘉兴 314006
2. School of Environment, Tsinghua University, Beijing 100084, China ;
3. Yangtze Delta Region Institute of Tsinghua University, Jiaxing, Zhejiang 314006, China
迟缓爱德华氏菌(Edwardsiella tarda,Et)对多种动物及人致病,尤其对特种水产品有重要致病性,超过20多种海水和淡水鱼类可感染,引起脓肿、全身败血症等致死性病变,对全球水产养殖造成了巨大的经济损失[1]。此外,E. tarda也可引起人类感染,是一种重要的人畜共患病。E. tarda自1959年首次在日本由Sakazaki和Murata分离后,迅速在全世界各地蔓延开,包括亚洲、欧洲、非洲和美洲[2-3]。我国动物疫病病种目录将E. tarda列为有害生物,是我国和其他国家在水产品进出口贸易中必须检疫的微生物。E. tarda分型研究对于探讨菌株遗传多样性及流行病学分析有着重要意义。杜邦公司RiboprinterTM全自动微生物鉴定系统可实现全自动化核糖体分型,细菌裂解、DNA酶切、凝胶电泳和数据分析均做到了全部标准化。本实验采用1998年-2010年从不同区域及宿主来源分离的26株E. tarda,研究其核糖体分型特性并进行聚类分析。
1 材料与方法 1.1 菌株E.tarda标准菌株ATCC15947、ATCC23692购自Oxiod公司,其余菌株分别从斑点叉尾鮰、日本鳗、多宝鱼、比目鱼、斑醴等宿主体内分离(表 1)。所有菌株均经过革兰氏染色、氧化酶试验、VITECK 32GN1+生化鉴定和16S rRNA基因序列同源性分析。置于-80 °C甘油肉汤保存。
| 宿主 Host | 菌株 Strains | 来源 Source | 鱼类致病性 Mortality |
| 比目鱼 Flounder | BYK00679 | 安徽 | Y |
| TX1 | 山东 | Y | |
| 多宝鱼 Turbot | EIB202 | 山东 | Y |
| TJ090818 | 天津 | Y | |
| L-49231 | 辽宁 | Y | |
| WY37 | 山东 | Y | |
| ET-QD | 山东 | Y | |
| 斑点叉尾鮰 Channel catfish | GD091027 | 广东 | Y |
| 花鳗 Eel | manE29L | 广东 | Y |
| ET080729 | 广东 | Y | |
| 日本鳗 Japanese eel | 080813-2 | 福建 | Y |
| 美国鳗 American eel | ET081126R | 福建 | Y |
| 斑鳢 Channa maculata | ET-YZH | 江苏 | Y |
| 红慈鲷 Red cichlids | ET-0711059 | 上海 | Y |
| 鲷 Porgy | ET-CD | 上海 | Y |
| 未知 Unknown | BYK00685 | 江西 | Y |
| 马魮脂鲤 Serpae tetra | PPD130/91 | 新加坡 | Y |
| 抹香鲸 Sperm whale | ETB | 比利时 | Y |
| 奥斯卡鱼 Oscar fish | DT | 广东 | N |
| 人 Human | ET-1 | 美国 | N |
| ET-11 | 美国 | N | |
| ET-13 | 美国 | N | |
| ET-0605 | 江苏 | N | |
| ET-89602 | 江苏 | N | |
| ATCC15947 | 美国 | N | |
| ATCC23692 | 美国 | N |
RiboprinterTM全自动微生物鉴定系统、RiboprinterTM系统EcoRⅠ试剂盒,杜邦中国集团有限公司;BioNumerics数据库分析软件,比利时Applied Maths Inc;脑心浸液和胰蛋白胨大豆琼脂,北京陆桥技术股份有限公司。
1.3 Riboprinter操作规程1.3.1 样品准备:挑取平皿中培养24 h的单个纯菌落,放入加有缓冲液(试剂盒提供)的离心管中,搅拌数秒,制备成混浊菌悬液。取30 μL菌悬液于微量反应板内(试剂盒配置),置于细菌热处理器,加热约25 min。将经热处理的样品内分别加入A、B裂解液5 μL备用。
1.3.2 上样:按照仪器显示加样及有关试剂标识进行加样和填充有关试剂,随后仪器自动运行。整体过程如下:
(1) 制备DNA:细菌破壁,去蛋白并用限制性内切酶EcoRⅠ将DNA切成片段。
(2) 分离并转移DNA片段:依据DNA片段分子量大小进行电泳分离,并转移至硝酸纤维素膜上,与杜邦公司设计的探针杂交。
(3) 膜处理及检测:酶标抗体与探针特异性结合,探针包含编码高度保守的16S rRNA基因和23S rRNA基因的DNA片段及间隔序列。加入化学发光底物,底物与酶反应产生的光信号被CCD相机记录并储存为“条形码”图像数据。
(4) 数据处理:系统依据每个样品的图像数据产生一个Riboprinter模式,并与储存在系统中的其他Riboprinter模式进行比较来鉴定样品。
1.3.3 数据统计分析:获得的图像数据使用BioNumerics数据库软件进行处理,聚类图使用非加权配对算数平均法(UPGMA)构建,条带位置优化度设为0.5%。
1.3.4 核糖体指纹图谱数据库的建立:在BioNumerics软件中依次建立数据库、安装Riboprinter输入程序、初始化数据库后导入Riboprinter指纹图谱文本文件。录入实验菌株的背景信息,建立E.tarda核糖体指纹图谱数据库。
2 结果与分析 2.1 菌株鉴定结果每个菌株产生的Riboprinter模式以85%相似度为界与数据库中的标准菌株进行比对鉴定,只有ATCC15947及BYK00685超过0.85,分别为0.95和0.90。其余菌株的比对结果分别为:ET-1:0.74;ET-11:0.72;ET-YZH:0.73;ET-89602:0.76;manE29L:0.83;DT:0.83;ETB:0.65。剩余 17株包括ATCC23692均未能在Riboprinter系统中显示鉴定的菌株为E. tarda。
2.2 Riboprinter分型及聚类分析2株E.tarda标准株和24株分离株的核糖体分型条带模式如图 1所示,共有21种核糖体带型。其中18种带型分别对应1株Et,为PPD130/91、TX1、WY37、L-49231、TJ090818、080813-2、ET-CD、ET081126R、ET-YZH、ETB、ATCC15947、ET-0711059、ET-13、BYK00685、ET0605、DT、manE29L、ATCC23692;2种带型对应3株菌株,即BYK00679、EIB202、ET-QD和ET-1、ET-11、ET-89602;1种带型对应2株菌株:ET-080729、GD-091027。所有菌株的核糖体DNA被限制性内切酶EcoRⅠ消化后条带数为10-12条左右,酶切条带分子量范围位于1-50 kb之间。经相似度矩阵分析,不同带型之间的最低相似度为20.0% (ATCC23692与080813-2)。使用BioNumerics软件对结果进行分群和多维尺度分析,可见26株E.tarda的核糖体分型主要分为3个群。对进化树上的菌株进一步分析发现,来自相同地域或相同宿主的菌株同源性更近,更趋于分布在同一或相近基因树上。例如:来自山东、辽宁、天津等北方地区的菌株EIB202、ET-QD、TX1、WY37、L-49231、TJ090818聚集于第一群;来自广东、福建、上海的ET-090729、GD-091027、080813-2、ET081126R、ET-CD聚集于第二群;所有的人源株ET-1、ET-11、ET-13、ET-0605、ET-89602与其他的鱼源株则分布于第三群。
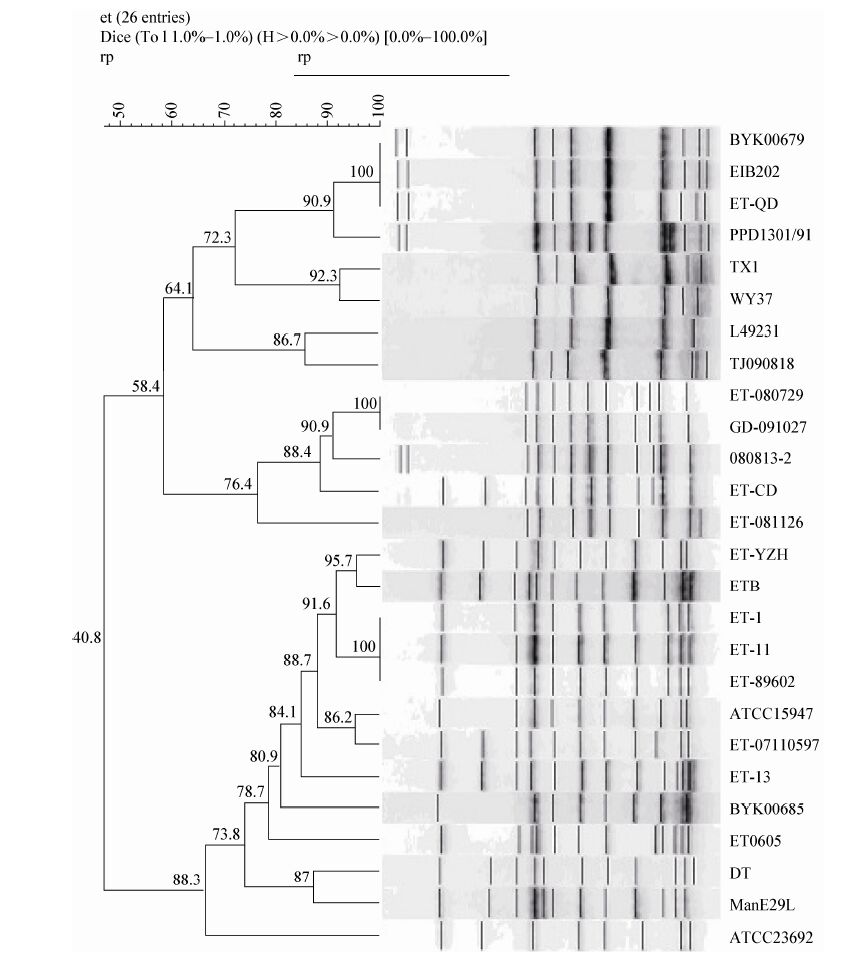
|
| 图 1 迟缓爱德华氏菌核糖体分型聚类分析图 Figure 1 Edwardsiella tarda ribotyping clustering analysis |
|
|
利用生化表型差异进行E. tarda分型的研究虽有报道[4],但在血清型分类及使用方面远不及沙门氏菌及李斯特菌等病原菌成熟[5- 6]。采用蛋白质组学分析E.tarda鞭毛蛋白、胞外产物及菌体蛋白指纹或利用免疫印迹法研究E.tarda菌株异质性近年来也有涉及[7-8]。表型分型技术的缺陷和分子生物学的迅猛发展引发了基因分型技术的产生,在此基础上建立的众多技术依据其不同的性能特点和实验室的具体情况已逐步应用于细菌检测和溯源分析。脉冲场凝胶电泳(pulsed-field gel electrophoresis,PFGE)及多位点序列分型(multi locus sequence typing,MLST)是目前国内外流行病学研究广泛接受的方法之一[9]。与其他方法相比具有分辨率高、高灵敏性的优点,能在细菌基因组很庞大的情况下,尽可能反映较多的变异信息,被誉为细菌分子分型方法的金标准。然而这些方法存在的缺点是耗时费力,也缺乏标准化的操作手法。
RiboprinterTM全自动微生物鉴定系统基于Southern杂交的自动化核糖体分型,其大容量专家数据库包含了8 528条标准菌株信息,含 1 740多个种以及290多个属的微生物DNA信息,涵盖病原菌、环境菌、益生菌、致腐菌、质控菌及其他多种微生物,全部经ATCC,JCM和DSMZ验证。已被广泛用于流行病学调研及食源性微生物溯源追踪。其分型结果基本与PFGE及MLST方法吻合,均显示菌株遗传多样性与宿主及地理来源有着密切的关联性[9]。Yang等[10]将33株E.tarda用PFGE进行了分型,表明北方系与南方系分属两个群,人源株与DT株在另一个分支上。本次研究与Yang等[10]所选用的E.tarda菌株有部分相同,结果有高度一致性:来自山东、辽宁、天津等北方的菌株汇成一群;广东、福建、上海等南方来源的菌株汇成第二个群;人源株及DT等其他鱼源菌株分布于第三个群。值得注意的是,来自安徽的BYK00679及来自新加坡的PPD130/91在两种研究方法中均归属于北方系,表明这两株菌株最初可能均由中国的北方地区传入。在本研究中,有较多的鱼源菌株与人源株归入一群;BYK00685 (江西株)未归于南方系,表明两种方法有少许的差异。但从本试验的结果看,如果以85%相似度为界与数据库中的标准菌株进行比对鉴定,只有ATCC15947及BYK00685超过0.85。因而在分型中Ribotype法将BYK00685归入人源株一群是更为合理科学的。
RiboprinterTM全自动微生物鉴定系统在提供简便、标准化、客观及省力等多功能性能的同时,也存在一些相关问题。比如仪器购买价格昂贵,运行成本高。另外,由于有效使用该系统需要良好的识别数据库,因而对于一些菌株必需由用户构建自己的数据库以增加原先制造商所提供的库容量。对于此次研究来说,26株E.tarda仅有2株与原数据库匹配相似度大于0.85,其余7株菌虽能鉴别出是E.tarda,但相似度很低,其余17株甚至ATCC23692都未能鉴别出。因而从整体来看,该系统在分子分型方便更有优势,而在鉴定菌株方面需结合其他方法。同时在分型聚类分析中,为进一步提高分辨率及可信度,同时使用PvuⅡ酶切可得到更好的验证效果。
| [1] | Mohanty BR, Sahoo PK. Edwardsiellosis in fish: a brief review[J]. Journal of Biosciences , 2007, 32 (S3) : 1331–1344. DOI:10.1007/s12038-007-0143-8 |
| [2] | White FH, Simpson CF, Williams LE Jr. Isolation of Edwardsiella tarda from aquatic animal species and surface waters in Florida[J]. Journal of Wildlife Diseases , 1973, 9 (3) : 204–208. DOI:10.7589/0090-3558-9.3.204 |
| [3] | Wang YM, Wang QY, Xiao JF, et al. Genetic relationships of Edwardsiella strains isolated in China aquaculture revealed by rep-PCR genomic fingerprinting and investigation of Edwardsiella virulence genes[J]. Journal of Applied Microbiology , 2011, 111 (6) : 1337–1348. DOI:10.1111/j.1365-2672.2011.05166.x |
| [4] | Maiti NK, Mandal A, Mohanty S, et al. Phenotypic and genetic characterization of Edwardsiella tarda isolated from pond sediments[J]. Comparative Immunology, Microbiology Infectious Diseases , 2009, 32 (1) : 1–8. DOI:10.1016/j.cimid.2007.11.001 |
| [5] | Chen Y, Ross WH, Gray MJ, et al. Attributing risk to Listeria monocytogenes subgroups: dose response in relation to genetic lineages[J]. Journal of Food Protection , 2006, 69 (2) : 335–344. |
| [6] | Corcoran D, Clancy D, Mahony MO, et al. Comparison of Listeria monocytogenes strain types in Irish smoked salmon and other foods[J]. International Journal of Hygiene and Environmental Health , 2006, 209 (6) : 527–534. DOI:10.1016/j.ijheh.2006.06.001 |
| [7] | Tan YP, Lin Q, Wang XH, et al. Comparative proteomic analysis of extracellular proteins of Edwardsiella tarda[J]. Infection and Immunity , 2002, 70 (11) : 6475–6480. DOI:10.1128/IAI.70.11.6475-6480.2002 |
| [8] | Liu XJ, Kang LQ, Liu YJ, et al. Characterization of the Edwardsiella tarda proteome in response to different environmental stresses[J]. Journal of Proteomics , 2013, 80 : 320–333. DOI:10.1016/j.jprot.2013.01.022 |
| [9] | Abayneh T, Colquhoun DJ, S?rum H. Multi-locus sequence analysis (MLSA) of Edwardsiella tarda isolates from fish[J]. Veterinary Microbiology , 2012, 158 (3/4) : 367–375. |
| [10] | Yang MJ, Shao S, Xiao JF, et al. Phylogenetic investigation of Edwardsiella tarda with multilocus sequence typing (MLST) and pulsed field gel electrophoresis (PFGE) typing methods[J]. Aquaculture , 2013, 410-411 : 79–85. DOI:10.1016/j.aquaculture.2013.05.024 |
 2016, Vol. 43
2016, Vol. 43