扩展功能
文章信息
- 肖景惠, 李美玉, 孙淼, 王晓辉
- XIAO Jing-hui, LI Mei-yu, SUN Miao, WANG Xiao-hui
- 一种几丁质酶基因的克隆表达及酶解产物分析
- Cloning and expression of a novel chitinase gene and the analysis of its hydrolysis products
- 微生物学通报, 2016, 43(10): 2179-2186
- Microbiology China, 2016, 43(10): 2179-2186
- DOI: 10.13344/j.microbiol.china.150851
-
文章历史
- 收稿日期: 2015-10-31
- 接受日期: 2016-02-23
- 优先数字出版日期(www.cnki.net): 2016-08-04
几丁质(chitin),俗称甲壳素,是一种可从虾蟹壳等废弃物中提取的天然多糖,其结构为N-乙酰-D-氨基葡萄糖(约50%-100%)和少量D-氨基葡萄糖通过β-1,4-糖苷键(约0-50%)连接而成,自然界中每年由生物体合成的甲壳素为1011 t[1],是海洋环境中含量最丰富,自然界中仅次于纤维素的第二大可再生的生物质来源[2]。几丁质酶(chitinase,chi,EC3.2.1.14)可降解几丁质制备高附加值几丁寡糖,几丁寡糖具有良好的生物相容性、生物降解性,而且还表现出调节免疫力、抗菌、抗肿瘤、诱导植物抗病性和促进植物生长等生物活性,可广泛应用于医药工业、食品工业、农业和养殖业等众多领域[3-4]。
CAZy数据库(http://www.cazy.org/Glycoside- Hydrolases.html)基于氨基酸序列保守性,将几丁质酶归为糖苷水解酶(glycosyl hydrolases,GH)第18、19、23和48家族。18家族的几丁质酶广泛存在于细菌、古生菌、病毒到高等生物如植物、昆虫及哺乳动物体内,具有相同的催化机理以及水解β-1,4-糖苷键连接的几丁质多聚物的酶学活性[5]。但不同生物中的几丁质酶发挥不同的生理功能和生态作用,导致几丁质酶的酶学性质会存在一定的差异。本研究几丁质酶来自于海洋细菌Pseudoalteromonas sp. DL-6 (CGMCC No. 8580),本实验室早期研究报道[6-7]菌株DL-6为典型的耐冷菌,至少产生并向胞外分泌3种以上不同分子质量的几丁质酶。截止目前,国内外仅有10多种海洋几丁质酶系基因被克隆、测序及初步的生化性质表征[8-12],仅解析了1种来源于海洋细菌Moritella marina的MmChi60的结晶结构[13]。本研究拟克隆Pseudoalteromonas sp. DL-6的几丁质酶基因chiA,构建其pET系列的原核表达载体,表达纯化该重组蛋白,并对其酶解产物进行解析,为今后工业化制备几丁寡糖奠定理论基础。
1 材料与方法 1.1 材料1.1.1 菌株及培养基:假交替单胞菌(Pseudoalteromonas sp. DL-6)菌株由本实验室分离并保存;大肠埃希氏菌(Escherichia coli,E. coli Top10)和E. coli BL21(DE3)为本实验室保存,分别为基因克隆和表达宿主菌。LB (Luria-Bertani)液体培养基(g/L)[14]:酵母提取物5.0,胰蛋白胨10.0,氯化钠10.0,pH 7.2。LB固体培养基:在LB液体培养基的基础上,琼脂粉含量为1.5%。培养基在用之前1×105 Pa灭菌20 min。
1.1.2 载体:质粒pMDl9-T用作克隆载体,pET28a用作表达载体;引物合成和基因测序由生工生物工程(上海)股份有限公司完成。
1.1.3 引物:如表 1所示。
1.2 主要试剂和仪器DNA限制性内切酶、T4 DNA连接酶、TaKaRa
| 引物名称 Primer name | 引物序列 Primer sequence (5′→3′) | 用途 Use |
| chiA-F | ATGAGCTCACGKAAAATAATAAMAAATGCC | 正向引物,扩增全长目的基因的保守片段 |
| chiA-R | TTACAGGCTACAACTTAARSTCCAATC | 反向引物,扩增全长目的基因的保守片段 |
| chiAF | CCCGGATCCGTCAACTGATTGCAGTACATTAACG | 去除信号肽,克隆chiA的正向引物,加BamHⅠ位点 |
| chiAR | CCGCTCGAGCAGGCTACAACTTAAAGTCCAATCTG | 去除终止密码子,克隆chiA的反向引物,加XhoⅠ位点 |
LATaqTM DNA polymerase、pMD™19-T vector cloning kit等购自宝生物工程(大连)有限公司;DNA marker购自上海玉博生物科技有限公司;Bradford蛋白浓度测定试剂盒购自碧云天生物技术有限公司;Ni-NTA His·Bind Resins购自美国Novagen公司;其它生化试剂和抗生素均为进口或国产生化或分析纯级试剂。SDS-PAGE电泳仪Power Pac 300 购自美国Bio-Rad Laboratories公司;Hybaid PCR仪购自上海普迪生物技术有限公司;荧光分光光度计购自美国Molecular Devices公司;电喷雾质谱仪(ESI-MS)购自美国Bruker Daltonics公司。
1.3 方法1.3.1 chiA基因的克隆与转化:NCBI数据库检索同属的几丁质酶基因序列,结果发现基因组测序的假交替单胞菌属Pseudoalteromonas undina NCIMB 2128、Pseudoalteromonas citrea NCIMB 1889、Pseudoalteromonas sp. SM 9913和Pseudoalteromonas rubra ATCC 29570的基因组中存在假定的几丁质酶A基因,序列比对后设计简并引物chiA-F/chiA-R (表 1)。
按照TaKaRa公司的细菌基因组DNA提取试剂盒提取Pseudoalteromonas sp. DL-6的基因组DNA,以其为模板进行PCR扩增。50 μL体系:dNTP mixture (各2.5 mmol/L) 8.0 μL,10×LATaq bufferⅡ (Mg2+ plus) 5.0 μL,LATaqTM DNA polymerase (5 U/μL) 0.5 μL,引物chiA-F/chiA-R (10 μmol/L)各2.0 μL,模板1 μL,补加超纯水至 50 μL。PCR扩增条件:98 ℃ 1 min;98 ℃ 10 s,55 ℃ 15 s,72 ℃ 3 min,30个循环;72 ℃ 5 min。PCR扩增的目的片段连接至pMD19-T载体,并将其转化至E. coli Top10,通过含有 100 mg/L氨苄青霉素(Amp)的选择平板对重组子进行筛选。通过质粒提取试剂盒提取重组质粒,由生工生物工程(上海)股份有限公司测序后,基因序列提交至Genbank数据库。
采用Vector NTI Suite软件进行开放阅读框(Open reading frames,ORF)分析。通过NCBI (National Center for Biotechnology Information,http://www.ncbi.nlm.nih.gov/)上的Basic Local Alignment Search Tool (BLAST,http://blast.ncbi. nlm.nih.gov/Blast.cgi)完成序列的同源分析。采用SignalP 4.1进行信号肽预测。采用在线的Simple Modular Architecture Research Tool (SMART,http://smart.embl-heidelberg.de/)进行序列结构域分析。采用Vector NTI Suite 软件的AlignX程序进行氨基酸序列的多重比对。
1.3.2 重组几丁质酶的表达与纯化:以重组质粒pMD19-T-chiA为模板,通过正向引物chiAF和反向引物chiAR使用TaKaRa LATaqTM DNA polymerase扩增得到不含信号肽的低温几丁质酶基因序列。将该DNA序列连接到pMDl9-T载体,构建pMD19-T-chiA质粒转化至E. coli Top10,得到阳性转化子。用限制性内切酶BamHⅠ和XhoⅠ将chiA基因片段从pMDl9-T-chiA上切下,连接到pET-28a表达载体上,转化至E. coli BL21(DE3),通过菌落PCR、酶切筛选鉴定阳性克隆子,命名为pET28a-chiA。
将重组E. coli BL21(DE3)以1%接种量接入含卡那霉素(50 mg/L)的LB液体培养基[14]中,37 ℃、220 r/min培养过夜。当OD600达到0.8左右,以 0.2 mmol/L IPTG 16 ℃、100 r/min诱导6 h。诱导表达结束后,4 ℃、5 000 r/min离心5 min收集菌体,洗涤菌体并用缓冲液重悬菌体,冰上进行超声破壁。收集上清液,即为粗蛋白。采用Ni-NTA亲和层析柱纯化几丁质酶。所有纯化步骤在4 ℃下进行。
1.3.3 SDS-PAGE电泳:SDS-PAGE电泳采用浓缩胶5%,分离胶12%,考马斯亮蓝R-250染色显示蛋白带。
1.3.4 几丁质酶的酶活测定[15]:利用4-甲基伞形酮荧光底物4MU-(GlcNAc)2进行酶活测定,酶活测定体系:175 μL 50 mmol/L Tris-HCl缓冲液(pH 8.0),20 μL 1 mmol/L4MU-(GlcNAc)2,5 μL酶液,将反应体系在20 ℃温育10 min,加入1.8 mL 0.2 mol/L Na2CO3溶液,混匀后用荧光分光光度计测定荧光强度,激发波长为370 nm,发射波长为448 nm。设定3个重复。
酶活力单位(U)定义为在上述实验条件下,每分钟释放1 μmol 4-甲基伞形酮所需的酶量。比活力用每毫克蛋白所含的酶活力单位数表示。
1.3.5 蛋白质浓度测定:蛋白浓度通过Bradford蛋白浓度测定试剂盒测定。
1.3.6 酶的降解产物分析:1 mL反应体系中加入0.5 μmol/L纯化的ChiA (168.68 U),1 g/L的胶体几丁质,于DH-II旋转混合仪20 ℃反应24 h的酶解产物经C18柱料除盐、Sevage法除蛋白,然后浓缩、干燥并复溶于甲醇中。利用基质辅助电喷雾质谱(ESI-MS)分析酶解反应的产物组成 。
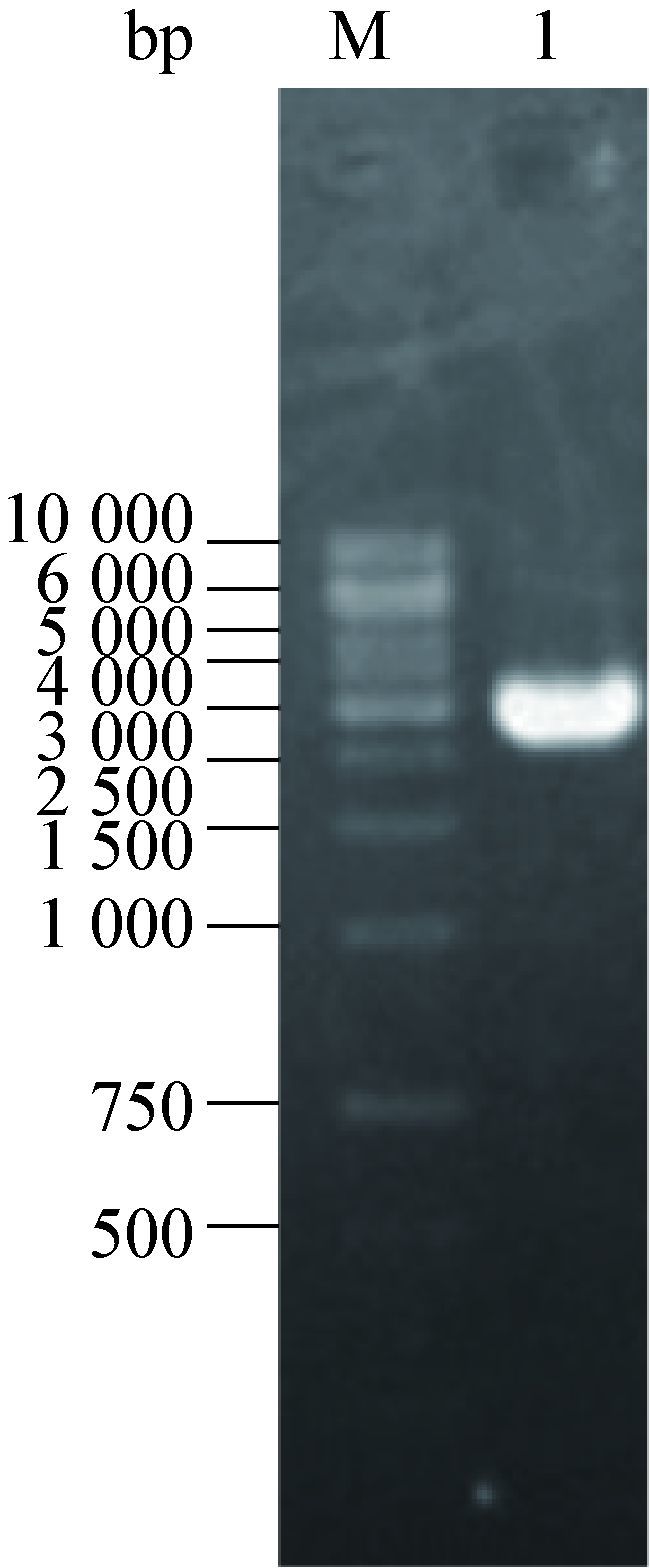
2 结果与分析 2.1 chiA基因的克隆与鉴定2.1.1 chiA基因全序列的电泳检测:以Pseudoalteromonas sp. DL-6菌株基因组DNA为模板,利用简并引物扩增出目的片段(图 1)。测序结果表明该基因全长3 160 bp,编码1 053个氨基酸,其理论分子量为113.544 85 kD,预测值为4.49,将其命名为chiA,提交NCBI数据库,登录号为KF234015。
|
| 图 1 几丁质酶chiA的琼脂糖凝胶电泳分析 Figure 1 Argarose gel electrophoresis of the chiA gene Note: M: λ DNA/Hind III marker; 1: The PCR products of the chiA gene. |
|
|
SMART数据库分析chiA基因编码氨基酸序列的保守结构域。chiA编码氨基酸的N端1-27位氨基酸构成信号肽,N端34-79位和C端1 012-1 053位氨基酸构成类型3几丁质结合域(type-3 chitin-binding domains,ChtBD3),N端102-181位和194-280位氨基酸为成人多囊肾病结构域(polycystic kidney disease,PKD),323-786位氨基酸为18家族几丁质酶催化域(GH18 catalytic domain,Glyco_18)。
2.1.2 chiA编码氨基酸序列分析:BLAST检索结果显示chiA的氨基酸序列与来源于深海菌株Pseudoalteromonas undina NCIMB 2128 (登录号WP_010389313.1)和Pseudoalteromonas sp. SM9913 (登录号YP_004065201.1) chiA序列一致性分别为95%和87%,两株深海菌株都已完成全基因组测序,其几丁质酶基因是通过序列比对预测结果,目前还未见功能研究的文献报道。因此深入研究本课题chiA对于深海环境中几丁质的降解代谢也具有一定参考价值。chiA与来源于Pseudoalteromonas sp. S9、Pseudoalteromonas piscicida、Hahella chejuensis KCTC 2396和Simiduia agarivoransSA1=DSM 21679进行功能研究的几丁质酶分别具有71%、68%、51%和56%的一致性。
为分析chiA编码序列保守氨基酸残基信息,利用Align X软件与已知晶体结构的几丁质酶的氨基酸序列比对分析(图 2)。几丁质酶比对分析所选序列包括粘质沙雷氏菌(Serratia marcescens)几丁质酶A (PDB:1EDQ)、哈氏弧菌(Vibrio harveyi)几丁质酶(PDB:3B8S)、海洋细菌(Moritella marina)低温几丁质酶MmChi60 (PDB:4HMC)、环状芽孢杆菌(Bacillus circulans Wl-12)几丁质酶A1 (PDB:1ITX)。尽管不同来源几丁质酶的功能及编码氨基酸序列差异较大,但催化域关键氨基酸高度保守。ChiA具有18家族糖苷水解酶的负责结合底物及催化降解功能的典型保守模块SxGG和DxxDxDxE[5](图 2黑框),这说明chiA具有18家族几丁质酶“底物辅助保留机理(Substrate-assisted retaining mechanism)”的催化机制。
本研究chiA为18家族几丁质酶,其催化模块“DxxDxDxE”中谷氨酸(Glu,E)活性位点为酸碱催化氨基酸(图 2

|
| 图 2 chiA编码产物的多重序列比对 Figure 2 Multiple sequence alignments of the chiA |
|
|
2.1.3 几丁质酶在大肠杆菌的表达与纯化:将测序正确的重组质粒pET28a-chiA在E. coli BL21(DE3)中异源表达。通过SDS-PAGE分析目标蛋白表达情况。如图 3A所示,泳道2约110 kD蛋白带与ChiA理论分子量(110 592.53 Da,去除信号肽)相一致,由此推测IPTG成功诱导大肠杆菌表达目的蛋白。

|
| 图 3 重组几丁质酶ChiA的诱导表达 Figure 3 The inducible expression of the recombinant ChiA Note: M: PageRuler Prestained Protein Ladder; 1: Cell extract of strain E. coli BL21(DE3)/pET28a without IPTG; 2: Cell extract of strain E. coli BL21(DE3)/pET28a-chiA with IPTG; 3: Cell extract of strain E. coli BL21(DE3)/pET28a-chiA; 4: Soluble fractions of cell extract of strain E. coli BL21(DE3)/pET28a-chiA; 5: Insoluble fractions of cell extract of strain E. coli BL21(DE3)/pET28a-chiA; 6: Cell extract of strain E. coli BL21(DE3)/pET28a; 7: Soluble fractions of cell extract of strain E. coli BL21(DE3)/pET28a; 8: Insoluble fractions of cell extract of strain E. coli BL21(DE3)/pET28a. |
|
|
以阴性对照菌株E. coliBL21(DE3)/pET28a相比,基因工程菌株E. coli BL21(DE3)/pET28a-chiA在约110 kD位置出现特异蛋白谱带(图 3B),与预测的重组蛋白质条带大小(110 592.53 Da)基本一致,说明ChiA在E. coli BL21(DE3)中可溶性表 达。
通过Ni+-NTA亲和层析法纯化重组蛋白(图 4)。纯化ChiA总蛋白含量为33.74 mg,比活力为42.17 U/mg,总活力为168.68 U,总回收率为63.25%,纯化倍数为7.32 (表 2)。

|
| 图 4 重组几丁质酶ChiA的分离纯化 Figure 4 Purification of the recomninant ChiA Note: M: PageRuler Prestained Protein Ladder; 1: Soluble fractions of cell extract of strain E. coli BL21(DE3)/ pET28a-chiA; 2: Fractions that flowed through Ni-NTA resin; 3: Wash with bind buffer; 4: Wash with 20 mmol/L imidazole; 5: Wash with 40 mmol/L; 6: Elution with 60 mmol/L imidazole imidazole; 7: Elution with 120 mmol/L imidazole; 8: Elution with 150 mmol/L imidazole; 9: Elution with 300 mmol/L imidazole. |
|
|
| 纯化步骤 Purification procedure | 总蛋白量 Total protein quantity (mg) | 总活力 Total activity (U) | 比活力 Specific activity (U/mg) | 纯化倍数 Purification fold | 得率 Yield (%) |
| 破菌后上清液 Cell extract | 46.31 | 266.70 | 5.76 | 1.00 | 100 |
| Ni-NTA柱 Column of Ni-NTA | 33.74 | 168.68 | 42.17 | 7.32 | 63.25 |
利用ESI-MS分析ChiA酶解胶体几丁质的产物,推测ChiA的降解模式。如图 5所示,在正离子模式下,m/z为244、447/463、650、852/871的离子峰代表聚合度为2-5寡糖的钠或钾或氢加合物[(GlcNAc)2-9-H2O+Na/K/H]。ChiA降解胶体几丁质的酶解产物中几丁二糖离子峰(m/z 447)的相对丰度达到最大值100%。三糖(m/z 650)的相对丰度超过30%,四糖(m/z 852)的相对丰度超过50%。从结果
可见ChiA为一种18家族的内切几丁质酶,随机水解底物生成几丁寡糖。
3 讨论结构域分析表明ChiA为多结构的几丁质酶,包含催化域、ChtBD3、PKD等结构域。Watanabe等[17]报道ChtBD3结构域在酶降解几丁质中发挥关键作用,ChtBD3更利于几丁质酶催化域与底物结合,可持续作用几丁质底物并具有脱乙酰功能,但具体机理并未完全阐述清楚。Orikoshi等[18]定点突变菌株Alteromonas sp. strain O-7几丁质酶A的 N端PKD结构域关键芳香族氨基酸,结果表明其中W30和W67与几丁质底物紧密结合而实现高效降解,此外,PKD结构域可以与不溶底物粉状几丁质、纤维素及壳聚糖特异结合。然而,目前已研究的大多数几丁质酶只有一个ChtBD3或PKD结构域,但本研究ChiA包含两个ChtBD3和两个PKD,这种特殊的结构可能更利于酶在海洋等低温环境下结合不溶晶体底物、促进酶的水解功能。

|
| 图 5 ESI-MS分析ChiA降解胶体几丁质的酶解产物 Figure 5 ESI-MS spectra of the ChiA hydrolysis products of colloidal chitin Note: DP indicates the degree of polymerisation of chitin oligosaccharides from the enzymatic reaction. |
|
|
E. coli表达系统优点在于遗传背景研究深入清楚、繁殖快、成本低、表达量高以及有大量可利用的表达载体、宿主和纯化系统等特点,成为目前应用最广的表达体系[19]。但海洋环境来源的微生物大多具有冷适应性,分泌的酶因最适作用温度较低,异源表达较困难[20],且本研究重组蛋白ChiA分子量约110 kD,也为体外正确折叠造成一定困难,极大地限制了其在工业上的应用。因此,通过在大肠杆菌中采用16 ℃低温诱导、低浓度IPTG (0.2 mmol/L)、低摇床转数(100 r/min)并更换表达载体等方法成功获得满足研究需求的重组蛋白,并分离纯化获得纯酶。
目前已报道[11, 21-22]的细菌内切几丁质酶降解胶体几丁质可生成聚合度为2-4或4-9的寡糖,而ChiA可降解胶体几丁质产生聚合度2-5的几丁寡糖。通过酶解产物分析表明ChiA为一种来源于海洋环境的18家族内切几丁质酶。目前本实验室深入研究其生化性质,为酶的晶体解析,探讨其降解机制、规模化制备几丁寡糖应用于食品、医药和农业等领域奠定基础。
| [1] | Vaaje-Kolstad G, Horn SJ, Sørlie M, et al. The chitinolytic machinery of Serratia marcescens- a model system for enzymatic degradation of recalcitrant polysaccharides[J]. FEBS Journal , 2013, 280 (13) : 3028–3049. DOI:10.1111/febs.12181 |
| [2] | Souza CP, Almeida BC, Colwell RR, et al. The importance of chitin in the marine environment[J]. Marine Biotechnology , 2011, 13 (5) : 823–830. DOI:10.1007/s10126-011-9388-1 |
| [3] | Chavan SB, Deshpande MV. Chitinolytic enzymes: an appraisal as a product of commercial potential[J]. Biotechnology Progress , 2013, 29 (4) : 833–846. DOI:10.1002/btpr.1732 |
| [4] | Zhang C, Kim SK. Chapter 28-application of marine microbial enzymes in the food and pharmaceutical industries[J]. Advances in Food and Nutrition Research , 2012, 65 : 423–435. DOI:10.1016/B978-0-12-416003-3.00028-7 |
| [5] | Adrangi S, Faramarzi MA. From bacteria to human: a journey into the world of chitinases[J]. Biotechnology Advances , 2013, 31 (8) : 1786–1795. DOI:10.1016/j.biotechadv.2013.09.012 |
| [6] | Wang XH, Zhao Y, Zhao XM, et al. Screening, Identification and zymogram analysis of a marine cold-adapted Chitinase-producing strain[J]. Acta Agriculturae Boreali-occidentalis Sinica , 2014, 23 (9) : 92–97. (in chinese) 王晓辉, 赵勇, 赵小明, 等. 海洋低温几丁质酶菌株筛选、鉴定及酶谱分析[J]. 西北农业学报 , 2014, 23 (9) : 92–97. |
| [7] | Wang XH, Yue M. Optimization of fermentation conditions for chitinase production by the Pseudoalteromonas sp. DL-6 using the response surface methodology[J]. Science and Technology of Food Industry , 2014, 35 (12) : 312–315. (in chinese) 王晓辉, 岳敏. 交替假单胞菌产几丁质酶的响应面优化[J]. 食品工业科技 , 2014, 35 (12) : 312–315. |
| [8] | Ramli ANM, Mahadi NM, Rabu A, et al. Molecular cloning, expression and biochemical characterisation of a cold-adapted novel recombinant chitinase from Glaciozyma antarctica PI12[J]. Microbial Cell Factories , 2011, 10 : 94. DOI:10.1186/1475-2859-10-94 |
| [9] | Park HJ, Kim D, Kim IH, et al. Characteristics of cold-adaptive endochitinase from Antarctic bacterium Sanguibacter antarcticus KOPRI 21702[J]. Enzyme and Microbial Technology , 2009, 45 (5) : 391–396. DOI:10.1016/j.enzmictec.2009.07.002 |
| [10] | Han Y, Yang BJ, Zhang FL, et al. Characterization of antifungal chitinase from marine Streptomyces sp. DA11 associated with South China Sea sponge Craniella australiensis[J]. Marine Biotechnology , 2009, 11 (1) : 132–140. DOI:10.1007/s10126-008-9126-5 |
| [11] | Jang MS, Lee YM, Cho YS, et al. Overexpression and characterization of a novel chitinase gene from a marine bacterium Pseudomonas sp. BK1[J]. Indian Journal of Biochemistry & Biophysics , 20054, 2 (6) : 339–344. |
| [12] | Orikoshi H, Nakayama S, Miyamoto K, et al. Roles of four chitinases (ChiA, ChiB, ChiC, and ChiD) in the chitin degradation system of marine bacterium Alteromonas sp. strain O-7[J]. Applied and Environmental Microbiology , 2005, 71 (4) : 1811–1815. DOI:10.1128/AEM.71.4.1811-1815.2005 |
| [13] | Malecki PH, Raczynska JE, Vorgias CE, et al. Structure of a complete four-domain chitinase from Moritella marina, a marine psychrophilic bacterium[J]. Acta Crystallographica Section D , 2013, 69 (5) : 821–829. DOI:10.1107/S0907444913002011 |
| [14] | Sambrook J, Russell DW. Molecular Cloning: a Laboratory Manual[M].3rd Edition. New York: Cold Spring Harbor Laboratory Press, 2001: 2344 . |
| [15] | Hoell IA, Klemsdal SS, Vaaje-Kolstad G, et al. Overexpression and characterization of a novel chitinase from Trichoderma atroviride strain P1[J]. Biochim Biophys Acta , 2005, 1748 (2) : 180–190. DOI:10.1016/j.bbapap.2005.01.002 |
| [16] | Uchiyama T, Katouno F, Nikaidou N, et al. Roles of the exposed aromatic residues in crystalline chitin hydrolysis by chitinase a from Serratia marcescens 2170[J]. Journal of Biological Chemistry , 2001, 276 (44) : 41343–41349. DOI:10.1074/jbc.M103610200 |
| [17] | Watanabe T, Ariga Y, Sato U, et al. Aromatic residues within the substrate-binding cleft of Bacillus circulans chitinase A1 are essential for hydrolysis of crystalline chitin[J]. Biochemical Journal , 2003, 376 (Pt 1) : 237–244. |
| [18] | Orikoshi H, Nakayama S, Hanato C, et al. Role of the N- terminal polycystic kidney disease domain in chitin degradation by chitinase A from a marine bacterium, Alteromonas sp. strain O-7[J]. Journal of Applied Microbiology , 2005, 99 (3) : 551–557. DOI:10.1111/jam.2005.99.issue-3 |
| [19] | Terpe K. Overview of bacterial expression systems for heterologous protein production: from molecular and biochemical fundamentals to commercial systems[J]. Applied Microbiology and Biotechnology , 2006, 72 (2) : 211–222. DOI:10.1007/s00253-006-0465-8 |
| [20] | Baker KN, Rendall MH, Patel A, et al. Rapid monitoring of recombinant protein products: a comparison of current technologies[J]. Trends Biotechnology , 2002, 20 (4) : 149–156. DOI:10.1016/S0167-7799(01)01914-X |
| [21] | Bhattacharya D, Nagpure A, Gupta RK. Bacterial chitinases: properties and potential[J]. Critical Reviews in Biotechnology , 2007, 27 (1) : 21–28. DOI:10.1080/07388550601168223 |
| [22] | Wang SL, Liu CP, Liang TW. Fermented and enzymatic production of chitin/chitosan oligosaccharides by extracellular chitinases from Bacillus cereus TKU027[J]. Carbohydrate Polymers , 2012, 90 (3) : 1305–1313. DOI:10.1016/j.carbpol.2012.06.077 |
 2016, Vol. 43
2016, Vol. 43