扩展功能
文章信息
- 秦余, 李茜茜, 郑雨焕, 丁光玲, 郭霞
- QIN Yu, LI Qian-Qian, ZHENG Yu-Huan, DING Guang-Ling, GUO Xia
- 3种野生牛肝菌的菌塘土壤细菌多样性
- Bacteria diversity of shiroes soil growing three species of wild Boletus
- 微生物学通报, 2016, 43(10): 2148-2153
- Microbiology China, 2016, 43(10): 2148-2153
- DOI: 10.13344/j.microbiol.china.150823
-
文章历史
- 收稿日期: 2015-10-26
- 接受日期: 2016-03-01
- 优先数字出版日期(www.cnki.net): 2016-03-02
牛肝菌是一类外生菌根菌,营养价值丰富,味道鲜美,具有非常高的经济以及食药用价值。李泰辉和宋斌对我国牛肝菌资源进行了系统的调查,发现我国已知牛肝菌199种,牛肝菌资源非常丰富[1]。目前仅有极少数牛肝菌种类,例如远东疣柄牛肝菌可以实现仿生栽培[2],暗褐网柄牛肝菌与小粒咖啡苗形成的人工菌塘中获得了发育成熟的子实体[3],但是,绝大数牛肝菌种类尚不能人工栽培或人工栽培十分困难。多数外生菌根菌很难用人工栽培法和宿主建立共生关系,其中关键的因素主要集中在菌根菌、宿主、环境三者的联系。菌塘是由菌丝与植物根系及土壤颗粒相互交错扭结而成的团状结构,是菌丝体与宿主植物共生并进行营养交换的场所,也是其进行核配与分化并形成子实体原基的地方,是菌根菌完成其整个生活不可缺少的场所。牛肝菌菌丝生长缓慢,容易退化,人工培养非常困难,与宿主植物形成菌根效率低。于富强等将牛肝菌采集地土壤浸出液作为分离培养基的成分,牛肝菌分离成功率从41.7%-58.3%,提高了70%[4],并指出人工培养基质接近牛肝菌的野外生存营养环境,是成功分离的重要因素之一。为什么添加牛肝菌菌塘土壤浸出液能促进牛肝菌菌丝生长呢?可能是菌塘中分离的微生物对菌根菌的菌丝生长和菌根的合成有一定的积极作用,土壤中的微生物分泌了某些代谢产物促进了菌丝生长,而人工培养菌丝因为缺乏这些特异代谢物导致生长缓慢。近年来,研究发现植物根圈促生细菌不仅能提高菌根真菌对植物根部的侵染效率[5],还能通过与外生菌根真菌的互作改良土壤结构和促进植物营养吸收[6]。但是,牛肝菌菌塘土壤中到底都有哪些细菌类群,以及优势菌群是哪些类群都鲜有报道。
本文以重庆石柱县当地盛产并且十分受当地人喜欢的3种牛肝菌[远东疣柄牛肝菌(Le)、美味牛肝菌(Be)和小孢粉孢牛肝菌(Tm,煮熟过水后食用)菌塘土壤]为研究对象,利用高通量测序技术研究菌塘细菌的多样性,以期为研究牛肝菌的菌塘生态功能、人工菌塘的建立、菌根菌人工培植和人工林地驯化提供理论基础。
1 材料与方法 1.1 材料1.1.1 样品采集:2014年8月于平均海拔1 550 m的重庆石柱黄水森林公园采集用于研究的3种牛肝菌菌塘土样。采样点位于针阔叶混交林,乔木主要为云南松(Pinus yunnanensis Franch.)、云南油杉(Keteleeria evelyniana Mast.)、青冈(Cyclobalanopsis glauca),菌塘枯枝落叶层厚度为1.5 cm-2.2 cm。采其菌塘正下方5 cm-10 cm深处的菌塘土壤。每 一种牛肝菌选择5个其生长旺盛的菌塘,首先用灭菌且锋利的小刀轻轻拨开牛肝菌菌塘表面的土壤,用灭菌的镊子将菌塘的土壤夹起放入灭菌的布袋中混匀后,迅速放入冰盒内,尽快带回实验室。将每种牛肝菌5个菌塘的土壤分别放入搅拌机中混匀后,分别取混合土壤用于DNA提取。
1.1.2 主要试剂和仪器:DNA提取试剂盒,广州美基生物科技有限公司;PCR扩增仪、凝胶成像仪,美国Bio-Rad公司。
1.2 土壤总DNA的提取用HiPure Soil DNA Kits (Magen,广州)专用土壤DNA提取试剂盒提取土壤总DNA。
1.3 高通量测序分析提取各样本细菌总DNA,扩增16S rRNA基因序列的v3-v4可变区,引物F:5′-GTACTCCTAC GGGAGGCAGCA-3′,引物R:5′-GTGGACTACHV GGGTWTCTAAT-3′。PCR反应体系:含15 mmol/L MgCl2的10×Buffer 10.0 μL,2.5 mmol/L dNTPs 2.0 μL,10 μmol/L引物各5.0 μL,5 U/μL Taq酶1.0 μL,DNA模板4.0 μL,灭菌去离子水73.0 μL,总体积100 μL。PCR反应条件:94 ℃ 5 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 30 s,30个循环;72 ℃ 10 min。扩增好的DNA序列,采用Illumina MiSeq第二代高通量测序平台测定16S高变区域的序列,测序长度2×250 bp (委托北京华诺时代科技有限公司完成)。
1.4 数据分析对获得的测序数据,采用COPE软件及其内部程序进行数据拼接和过滤,0.97相似度下利用Qiime (v1.8.0)软件提交到RDP数据库搜索比对。数据的统计分析以每个样品中可操作的分类单元(Operational taxonomic unit,OTU)作为分类和计算的依据[7-8]。在本文中,定义一个OTU为不少于120个有效碱基,且碱基差异小于3%,即序列中碱基差异少于3个的序列群,一个OTU在结果分析中被视为代表 1种细菌。
| 样品 Sample | 样品中有效序列总数 Total numbers of valid sequences in each sample | 样品中有效OTU总数 Total OTUs in each sample | OTU总数/序列 The ratio of total OTUs/total sequences |
| Le | 17 852 | 2 770 | 0.155 |
| Be | 20 399 | 2 677 | 0.131 |
| Tm | 7 681 | 1 441 | 0.188 |
| 注:Le、Be和Tm分别代表远东疣柄牛肝菌、美味牛肝菌和小孢粉孢牛肝菌. | |||
| Note: Le,Be and Tm represent Leccinum extremiorientale,Boletus edulis and Tylopilus microsporus. | |||
对各土壤样品中高通量测序的数据进行统计(表 1),OTU数值越大,说明物种多样性越丰富。从表中可见,从Le、Be和Tm样品中分别读取了20 399、17 852、7 681个序列,其中优质序列OTU分别为2 677、2 770、1 441个。3种牛肝菌的菌塘OTU数量存在一定差异,Le菌塘的OTU数值最大,其次是Be的菌塘,最低是Tm的菌塘。从各个样品的序列总数和OTU总数的比值来看,3种牛肝菌的测序序列数可基本覆盖OTU的多样性,其中,Le和Be样品的测序深度要好于Tm样品。
从3种样品中随机抽取的测序条数如图 1横轴所示,纵轴则表示基于该测序条数能构建的OTU数量,绘制了各样品高通量测序结果的稀释性曲线。当曲线趋于平坦时,说明测序数据量合理,更多的数据量对发现新OTU的边际贡献很小;反之则表明继续测序还可能产生较多新的OTU。该分析是基于OTU序列差异水平在0.03即相似度97%的水平上进行运算的。从图上分析,除了Tm样品以外,Le和Be样品曲线逐渐趋于平缓,说明有效序列可以基本覆盖OTU的多样性。其中,Le和Be样品的测序质量优于Tm样品。
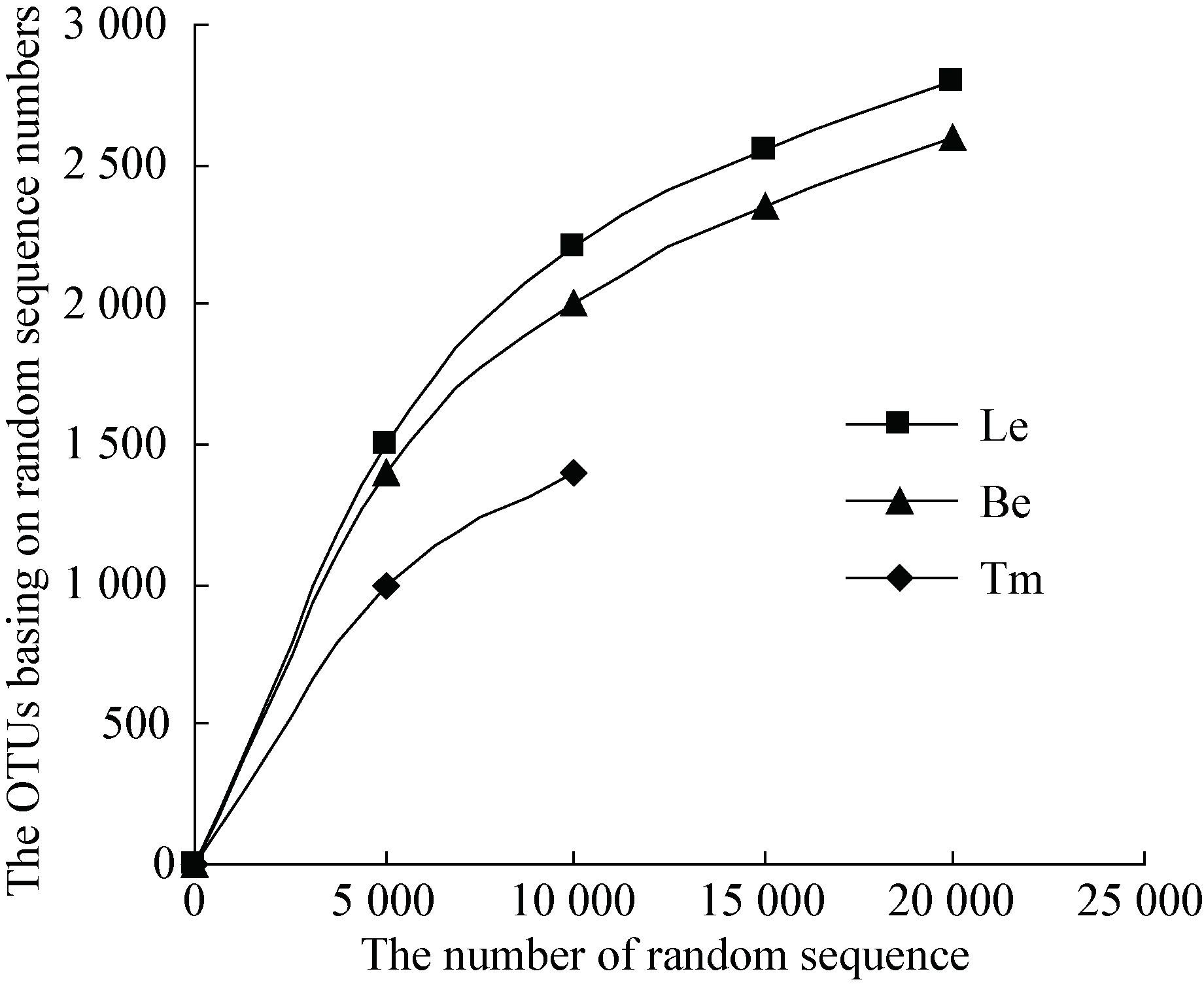
|
| 图 1 3种样品高通量测序结果的稀释曲线 Figure 1 The dilution curves of high throughput sequencing results of three samples |
|
|
在0.97的相似度下,得到了每个样品的OTU个数,利用R (v3.1.1)画图软件绘出Venn图,结果可以展示多样品相同和各自特有OTU数目,直观展示样品间OTU的重叠情况。结合OTU所代表的物种,可以找出不同环境中的核心微生物。如图 2所示,Le样品中含有细菌种类2 770个,Be样品中2 677个,Tm样品中含有1 441个。其中,Le和Be有相同种类1 708个,即31.3%的种类是相同。Be与Tm共有1 011个,共有种类约21.5%;Le和Tm共有916个,共有种类约24.6%。三者共有OTU 654个,即9.50%的种类同时出现在3种牛肝菌的菌塘中。

|
| 图 2 3种样品中细菌多样性的相关性分析 Figure 2 The similarity analysis of bacteria diversity among three samples 注:图中数字为各样品中的OTU数量. Note:The number in each figure is the OTU amount in three samples. |
|
|
将OTU的序列提交16S ribosomal RNA sequences (Bacteria and Archaea) RDP数据库中进行同源性比对,结果发现3种牛肝菌菌塘土壤细菌菌群丰富,Le菌塘土样中共检测到细菌门26个,属72个;Be菌塘土样则为门26个,属53个;Tm菌塘土样则为门22个,属44个。Le菌塘土样中的主要门分别为变形杆菌门(53.6%)、酸杆菌门(33.8%)、拟杆菌门(4.7%)、放线杆菌门(1.7%);Be中主要类群为酸杆菌门(40.7%)、变形杆菌门(38.1%)、拟杆菌门(10.3%)、放线杆菌门(3.1%);Tm中酸杆菌门(65.3%)、变形杆菌门(23.8%)、拟杆菌门(4.9%)为主要类群。
在属的层次下对各样品物种丰度进行统计,3个样品所含物种基本相似,但物种分布差异较大,图 3对所含主要种属进行了分析和归类。3种牛肝菌菌塘土壤细菌中优势物种在数据库中并没有相关的已知信息可供参考。Le主要属中,69.8%为未知属,即与未培养细菌的序列相似性较高,此外,还有伯克氏菌属Burkholderia (16.0%)、酸杆菌门中的CandidatusSolibacter (5.9%)、酸孢菌属Acidocella (3.3%)。Be中主要的属分布在未知类群(83.0%),CandidatusSolibacter (8.0%),伯克氏菌属Burkholderia (2.1%)。Tm中主要属分别为未知类群(80.9%)、CandidatusSolibacter (12.6%)。
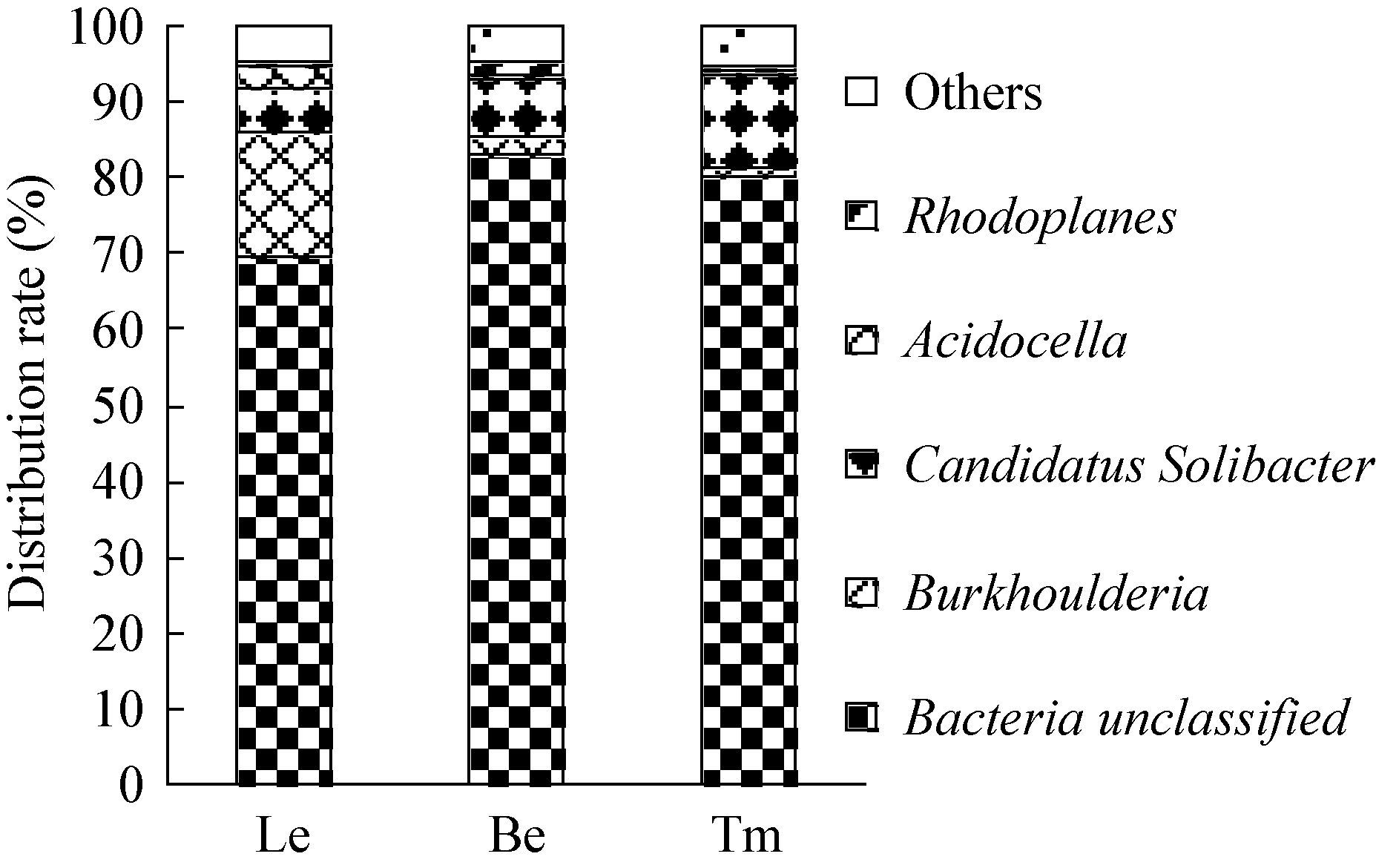
|
| 图 3 3种样品中的主要属的分布图 Figure 3 Major genus distribution of three samples |
|
|
对各样品中OTU优势度最高的前10位进行丰度分析,并将每个样品中优势度最高的前10种OTU的序列提交16S ribosomal RNA sequences (Bacteria and Archaea) RDP数据库中进行同源性比对,选取比对结果中相似度最高的已知菌株序列作为参考。结果发现,在菌塘中的优势细菌在数据库中几乎没有已知序列可供比对。
3 讨论3种牛肝菌菌塘中变形杆菌门、酸杆菌门、拟杆菌门为优势类群。牛肝菌菌塘土壤中含有较多的变形菌门和拟杆菌门,这与从云南松口蘑菌塘中分离到可培养细菌类群变形菌门(占58.43%)、厚壁菌门(占26.40%)、拟杆菌门(占10.67%),其次还有放线菌门(占4.49%)比较一致[9]。Candidatus Solibacter和Burkholderia是3种牛肝菌的优势属,与松口蘑菌塘可分离微生物中也含有大量伯克氏菌属Burkholderia细菌相似。从这些优势属或者优势门中分离菌根促生细菌应该是一种可行并且快捷的途径。
菌根菌和宿主共生的微环境菌塘,具有特定的菌落结构和丰富的细菌多样性。菌塘土壤细菌种类与普通林区土壤细菌菌群存在较大的差异,例如宝天曼森林土壤以变形菌门(29%)、酸杆菌门(18.5%)、疣微菌门(10%)等为主[10]。与菜地土壤微生物类群相比,其主要是α-和γ-变形杆菌亚门微生物[11],与牛肝菌菌塘细菌类群也存在较大差异。这种差异不仅与土壤所处环境有关,还与具体的检测方法有关。文献关于菌塘促生细菌的分离报道中,芽孢杆菌属报道较多。例如,芽孢杆菌属细菌能提高粘盖牛肝菌[12]和彩色豆马勃的菌丝生长和菌根生成[13]。蜡状芽孢杆菌对美味牛肝菌菌丝的促生作用均得到证实[5]。但是,实验中牛肝菌菌塘中的厚壁菌门微生物确有检测到,但并不是优势菌群,3种样品中都有少量OTU属于厚壁菌门的链球菌属和芽孢杆菌属。可能是因为由于土壤生态系统的复杂性,某些细菌与土壤颗粒结合紧密,在离心去除土壤颗粒的同时,丢失了一部分土壤微生物类群。放线菌类群可以通过在菌塘中释放抗生素,抑制其它致病微生物生长,从而促进菌根菌与宿主共生。例如,链霉菌属细菌能促进毒蝇鹅膏菌丝生长,提高共生植物云杉二级根菌根形成率1.2-1.7倍[14]。3种牛肝菌中也均检测到少量放线菌属细菌,但是数量较少均不是优势种群。菌塘中发现大量优势菌群,与未培养微生物同缘关系。可能是因为,虽然人们已分离培养了大量细菌种类,但是已发现的细菌物种数约占自然界存在种类的10%。这是一类目前认识不足的微生物类群,也可能与特殊的微环境有关[15]。菌塘是一个复杂且特殊的生态系统,菌根菌子实体的形成,与菌塘微生物密切相关。此外,还有温度、降雨、植被、光照等环境因素的影响,以及采集方式和管理模式等因素[16]。本研究仅检测了菌塘细菌多样性,要获得牛肝菌菌塘微生物多样性的完整信息,还需要对菌塘真菌以及原生动物的多样性,环境因子对菌塘微生物多样性的影响进行研究。
致谢: 感谢重庆师范大学生命科学学院王汉臣副教授和长江师范学院陈今朝教授在牛肝菌鉴定中的帮助。| [1] | Li TH, Song B. Species and distributions of Chinese edible Boletes[J]. Acta Edulis Fungi , 2002, 9 (2) : 22–30. (in chinese) 李泰辉, 宋斌. 中国食用牛肝菌的种类及其分布[J]. 食用菌学报 , 2002, 9 (2) : 22–30. |
| [2] | Chen YH, Chen ZM, Lin GH. Cultivation of Leccinum extremioriorientale[J]. Journal of Fujian Agriculture and Forestry University (Natural Science Edition) , 2012, 31 (4) : 532–535. (in chinese) 陈宇航, 陈政明, 林国华. 远东疣柄牛肝菌仿生栽培[J]. 福建农林大学学报: 自然科学版 , 2012, 31 (4) : 532–535. |
| [3] | Ji KP, Zhang CX, Zeng Y, et al. Artificial fungal colony and its fruiting of Phlebopus portentosus (Boletaceae) in Pot[J]. Acta Botanica Yunnanica , 2007, 29 (5) : 554–558. (in chinese) 纪开萍, 张春霞, 曾雁, 等. 盆栽条件下暗褐网柄牛肝菌人工菌塘及其子实体的培养[J]. 云南植物研究 , 2007, 29 (5) : 554–558. |
| [4] | Yu FQ, Ji DG, Liu PG. Studies on pure culture of the ectomycorrhizal fungi associated with Pinus yunnanensis[J]. Bulletin of Botanical Research , 2003, 23 (1) : 66–71. (in chinese) 于富强, 纪大干, 刘培贵. 云南松外生菌根真菌分离培养研究[J]. 植物研究 , 2003, 23 (1) : 66–71. |
| [5] | Sheng JM, Wu XQ, Hou LL, et al. Isolation and identification of a MHB strain from the rhizosphere soil of Pinus thunbergi inoculated with Boletus edulis[J]. Chinese Journal of Applied and Environmental Biology , 2010, 16 (5) : 701–704. (in chinese) 盛江梅, 吴小芹, 侯亮亮, 等. 一株黑松-美味牛肝菌菌根辅助细菌的筛选及鉴定[J]. 应用与环境生物学报 , 2010, 16 (5) : 701–704. |
| [6] | Kataoka R, Taniguchi T, Futai K. Fungal selectivity of two mycorrhiza helper bacteria on five mycorrhizal fungi associated with Pinus thunbergii[J]. World Journal of Microbiology and Biotechnology , 2009, 25 (10) : 1815–1819. DOI:10.1007/s11274-009-0082-7 |
| [7] | Jiao JK, Mo BH. Bacteria diversity of cheeses with high coverage by Illumina MiSeq Platform[J]. China Brewing , 2014, 33 (5) : 34–38. (in chinese) 焦晶凯, 莫蓓红. Illumina MiSeq平台高覆盖率测定干酪中的细菌微生物多样性[J]. 中国酿造 , 2014, 33 (5) : 34–38. |
| [8] | Jervis-Bardy J, Leong LEX, Marri S, et al. Deriving accurate microbiota profiles from human samples with low bacterial content through post-sequencing processing of Illumina MiSeq data[J]. Microbiome , 2015, 3 (1) : 1–11. DOI:10.1186/s40168-014-0066-1 |
| [9] | Jiang H, He CG, Yu FQ, et al. Bacterial diversity cultured from shiros of Tricholoma matsutake[J]. Chinese Journal of Ecology , 2015, 34 (1) : 150–156. (in chinese) 姜华, 何承刚, 于富强, 等. 松口蘑(Tricholoma matsutake)菌塘土壤可培养细菌多样性[J]. 生态学杂志 , 2015, 34 (1) : 150–156. |
| [10] | Bai XX, Shi RJ, You YM, et al. Bacterial community structure and diversity in soils of different forest ages and types in Baotianman forest, Henan Province, China[J]. Chinese Journal of Applied Ecology , 2015, 26 (8) : 2273–2281. (in chinese) 白晓旭, 史荣久, 尤业明, 等. 河南宝天曼不同林龄与林型森林土壤的细菌群落结构与多样性[J]. 应用生态学报 , 2015, 26 (8) : 2273–2281. |
| [11] | Teng QH, Cao H, Cui ZL, et al. PCR-RFLP analysis of bacterial 16S rDNA from a typical garden soil in Taihu region[J]. Biodiversity Science , 2006, 14 (4) : 345–351. (in chinese) 滕齐辉, 曹慧, 崔中利, 等. 太湖地区典型菜地土壤微生物16S rDNA的PCR-RFLP分析[J]. 生物多样性 , 2006, 14 (4) : 345–351. DOI:10.1360/biodiv.060003 |
| [12] | Frey-Klett P, Garbaye J, Tarkka M. The mycorrhiza helper bacteria revisited[J]. New Phytologist , 2007, 176 (1) : 22–36. DOI:10.1111/nph.2007.176.issue-1 |
| [13] | Zhao L, Wu XQ, Ye JR, et al. Isolation and characterization of a mycorrhiza helper bacterium from rhizosphere soils of poplar stands[J]. Biology and Fertility of Soils , 2014, 50 (4) : 593–601. DOI:10.1007/s00374-013-0880-9 |
| [14] | Schrey SD, Schellhammer M, Ecke M, et al. Mycorrhiza helper bacterium Streptomyces AcH 505 induces differential gene expression in the ectomycorrhizal fungus Amanita muscaria[J]. New Phytologist , 2005, 168 (1) : 205–216. DOI:10.1111/j.1469-8137.2005.01518.x |
| [15] | Guo LD. Progress of microbial species diversity research in China[J]. Biodiversity Science , 2012, 20 (5) : 572–580. (in chinese) 郭良栋. 中国微生物物种多样性研究进展[J]. 生物多样性 , 2012, 20 (5) : 572–580. |
| [16] | Li XL, Jin X, Li Q, et al. Effects of ecological environment factors on soil microorganisms of Tricholoma matsutake shiro in Sichuan Province[J]. Chinese Journal of Applied and Environmental Biology , 2015, 21 (1) : 164–169. (in chinese) 李小林, 金鑫, 李强, 等. 生态环境因子对四川松茸菌塘土壤微生物的影响[J]. 应用与环境生物学报 , 2015, 21 (1) : 164–169. |
 2016, Vol. 43
2016, Vol. 43