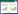扩展功能
文章信息
- 张晓欢, 崔文璟, 周哲敏
- ZHANG Xiao-huan, CUI Wen-jing, ZHOU Zhe-Min
- 高分子量腈水合酶在大肠杆菌中的表达策略及重组菌的细胞催化
- Strategy of high molecular mass nitrile hydratase expression in Escherichia coli and the whole-cell catalysis by the recombinant strains
- 微生物学通报, 2016, 43(10): 2121-2128
- Microbiology China, 2016, 43(10): 2121-2128
- DOI: 10.13344/j.microbiol.china.150912
-
文章历史
- 收稿日期: 2015-11-13
- 接受日期: 2016-03-01
- 优先数字出版日期(www.cnki.net): 2016-03-10
腈水合酶(nitrile hydratase,简称NHase,EC 4.2.1.84)是一种将腈类物质水合转化为更有利用价值的酰胺类化合物的金属酶[1],在工业上主要用于丙烯酰胺和烟酰胺的生产[2]。大部分NHase由α和β两个亚基构成,一般以α2β2四聚体形式存在,亚基分子量均为23 kD左右[3]。常见NHase大多数来源于红球菌属(Rhodococcus)、假诺卡氏菌属(Pseudonocardia)、诺卡氏菌属(Nocardia)[3]。另外,在一些真核生物中也存在[4-5]。NHase活性中心含有不同的金属离子,一般可分为铁型腈水合酶(Fe-NHase)和钴型腈水合酶(Co-NHase)。Fe-NHase活性中心含有一个非血红素铁离子[6],Co-NHase含有非咕啉态钴离子[1],并且金属离子都位于α亚基保守序列-Cys-X-Leu-Cys(SO2H)-Ser-Cys(SOH)上,其中包含两个被氧化的半胱氨酸残基:被深度氧化的硫酰基(-SO2-)和被浅度氧化的亚硫酰基(-SO-)[6-8]。另外,近几年的研究还发现了一些比较特殊的NHase,其中含有Cu2+或者Zn2+[9]。生物体内,NHase通常与腈水解酶[10]、酰胺酶[6]这两种关键酶一起发挥作用进行腈类物质代谢。
在化学工业中,NHase被应用于酰胺类物质,如丙烯酰胺、尼克酰胺以及5-氰基戊酰胺的生产已有数十年[2]。其中,将表达NHase的细胞通过固定化来进行生物催化生产丙烯酰胺的实例尤为典型。工业用丙烯酰胺的生产菌株经历了三代,第一代为红球菌Rhodococcus sp. N774[11-12],第二代为假单胞菌Pseudomonas chlororaphis B23[13-14],玫瑰色红球菌Rhodococcus rhodochrous J1是第三代[15-16]。在R. rhodochrous J1中同时存在两种不同类型的腈水合酶,按其分子量分为高分子量腈水合酶(H-NHase)和低分子量腈水合酶(L-NHase)。基因nhhBAG编码的H-NHase以多聚体结构形式存在[16];基因nhlBAE编码的L-NHase以α2β2四聚体结构形式存在[17]。
先前的研究表明,虽然R. rhodochrous J1来源的H-NHase和L-NHase已分别实现在红球菌属Rhodococcus rhodochrous ATCC12674[18]和Rhodococcus facians DSM43985[19]的异源表达,但是,因为它们同属红球菌属表达系统,因此,有与R. rhodochrous J1相同的缺陷:培养时间长,表达量低[20-21]。而相对红球菌属表达系统,大肠杆菌表达系统具有明显的优势:菌体生长速度快,蛋白表达量高。目前,L-NHase已在大肠杆菌表达系统中实现高效表达[22],但H-NHase在大肠杆菌中的表达量极低,且粗酶液酶活仅为3.47×10-3 U/mg[23],目前尚无其在大肠杆菌中成功大量表达的报道。
本研究对编码R. rhodochrous J1来源的H-NHase基因nhhBAG非编码区中SD序列和间隔区的长度进行改造后,并进一步根据大肠杆菌偏好性将密码子进行优化,最终实现其在大肠杆菌表达系统中的高效表达。另外,重组菌全细胞催化性质也体现了重组大肠杆菌巨大的生产潜力。
1 材料与方法 1.1 材料1.1.1 菌株与质粒:目的基因nhhBrbsArbsG由生工生物工程(上海)股份有限公司化学合成,表达载体pET24a(+)及宿主菌E. coliJM109、E. coli BL21(DE3)均为本实验室保存。
1.1.2 培养基:大肠杆菌E. coli BL21(DE3)表达培养基2YT (g/L):胰蛋白胨16.0,酵母提取物10.0、NaCl 5.0。菌体浓度OD600达到1.0左右时,加入0.1 g/L CoCl3·6H2O和0.6 mmol/L IPTG。
1.1.3 主要试剂和仪器:质粒小量提取试剂盒购自天根生化科技(北京)有限公司;Protein marker (Low/Broad)、T4 DNA连接酶、限制性内切酶等购自TaKaRa宝生物工程(大连)有限公司;琼脂糖凝胶回收试剂盒购自东洋纺(上海)生物科技有限公司;测序由上海生工生物工程公司进行;3-cyanopyridine (3-氰基吡啶,烟腈)购自梯希爱(上海)化成工业发展有限公司;尼克酰胺标准样品、标准蛋白质marker (MW-MARKER PROTEINS)购自日本和光纯药工业株式会社。
蛋白质电泳仪、核酸电泳仪及凝胶成像仪购自Bio-Rad公司;AKTA快速纯化液相色谱系统、HiTrap Q HP 1 mL蛋白纯化预装柱购自美国通用电气医疗集团;高效液相色谱仪HPLC、色谱柱HITACHI LaChrom C18 (250ACHI LaChrom,5 μm)购自日立(HITACHI)公司;UV-1800PC型紫外可见分光光度计购自上海美谱达有限公司。
1.2 方法1.2.1 表达载体pET24a(+)-nhhBrbsArbsG的构建:以R. rhodochrous J1来源的H-NHase基因nhhBAG序列(Genbank登录号为D67027.1)为模板,如图 1所示,编码H-NHase亚基NhhA的基因nhhA及调控基因nhhG上游原SD序列均被强的SD序列AAGGA代替,并将基因nhhB和nhhA之间以及nhhA和nhhG之间的间隔序列调整为GATATAGAT,完成表达元件的优化,构建目的基因nhhBrbsArbsG。随后,根据大肠杆菌密码子偏好性对基因nhhBrbsArbsG序列中含有的稀有密码子进行优化,并在目的基因的5′-和-3′分别设计Nde Ⅰ和EcoR Ⅰ限制性内切酶位点。设计得到的目的基因由上海生工生物工程有限公司合成。将合成的目的序列经 Nde Ⅰ/EcoR Ⅰ酶切后插入pET24a(+)质粒,构建重组表达载体pET24a(+)-nhhBrbsArbsG。表达载体pET24a(+)-nhhBrbsArbsG转化至E. coliBL21(DE3),从含有卡那霉素(Kan,50 μg/mL)的2YT抗性平板上挑取阳性重组子。培养后提取重组质粒经Nde Ⅰ/ EcoR Ⅰ双酶切验证并进行测序鉴定后保存备用。

|
| 图 1 nhhBrbsArbsG基因结构示意图 Figure 1 Genetic organization for the construction of nhhBrbsArbsG 注:较细下划线处为终止密码子或起始密码子,较粗下划线处为SD序列. Note:Fine underlined for stop codon or initiation codon,coarse underlined for the SD sequence. |
|
|
1.2.2 重组质粒在大肠杆菌表达系统中的表达:将重组菌E. coli BL21(DE3)/pET24a(+)-nhhBrbsArbsG接种到2YT液体培养基,37 ℃、200 r/min培养过夜,以1%接菌量转接至含有30 mL 2YT培养液的250 mL三角瓶中,培养至OD600约为1.0,加入终浓度为0.6 mmol/L的IPTG和0.1 g/L的CoCl2·6H2O,24 ℃诱导表达18 h,SDS-PAGE检测H-NHase的表达量,HPLC检测酶活。
1.2.3 H-NHase的纯化:用10 mmol/L磷酸钾缓冲液(potassium phosphate buffer,KPB:1.83% K2HPO4,0.27% KH2PO4,pH 7.5)悬浮诱导表达后的菌体,冰浴超声破碎菌体细胞(功率300 W,破碎2 s,间歇7 s,破碎时间30 min),12 000 r/min高速离心10 min,取上清,即粗酶液。粗酶液经硫酸铵盐析沉淀进行初步分离,收集40%-65%之间的沉淀蛋白10 mmol/L KPB复溶于透析袋中,在缓冲液 10 mmol/L KPB [含0.5 mmol/L二流苏糖醇(Dighiothreitol,DTT)]中4 ℃过夜透析。透析后的粗酶液经0.22 μm微孔滤膜过滤,取4 mL注入强阴离子交换层析柱Hitrap Q HP进行分离。目的蛋白通过高盐缓冲液(10 mmol/L KPB+1 mol/L KCl,pH 7.5)线性洗脱(洗脱范围0.2-0.6 mol/L KCl)得到。收集到的目的蛋白经超滤离心脱盐浓缩后得到一定浓度的纯酶。此部分实验通过AKTA pure蛋白质快速纯化系统在4 ℃进行[24]。
1.2.4 重组蛋白H-NHase的分子量测定:利用凝胶过滤分子排阻层析(Size-exclusion chromatography,SEC)测定H-NHase的相对分子量。10 mmol/L KPB (含0.15 mmol/L KCl,pH 7.5)为缓冲液,流速 0.5 mL/min。100 μL标准蛋白质marker [MW-MARKER PROTEINS:(1) 谷氨酸脱氢酶(酵母),290 kD;(2) 乳酸脱氢酶(猪心脏),142 kD;(3) 烯醇化酶(酵母),67 kD;(4) 肌激酶(酵母),32 kD;(5) 细胞色素C (马心脏),12.4 kD]在缓冲液的驱动下进入凝胶柱Superdex 200 10/300,根据蛋白分子量大小不同而依次流出。同样地,100 μL目的蛋白以相同的缓冲液及流速经过凝胶过滤层析柱,通过对比待测样品和标准蛋白质marker的出峰体积,计算出目的蛋白分子量。
1.2.5 重组H-NHase的酶活测定:H-NHase活性测定依据之前建立的方法[21]进行。单位酶活力定义为:25 ℃条件下单位毫克纯酶每分钟催化产生烟酰胺的量(U/mg)。蛋白浓度由Bradford法测定。
将10 μL酶液或菌体细胞加入90 μL 10 mmol/L的磷酸缓冲液中,400 μL 500 mmol/L烟腈为底物,25 ℃反应10 min,500 μL乙腈终止反应。反应液经0.22 μm微孔滤膜过滤,载入C18色谱柱进行HPLC分析,流动相为乙腈水(乙腈׃水=1׃2)的混合溶液。
1.2.6 重组型基因工程菌全细胞催化条件的确定:为确定全细胞催化最适pH,用0.5 mol/L的Na2HPO4/NaH2PO4配制成pH分别为5.5、6.0、6.5、7.0、7.5、8.0、8.5、9.0的磷酸缓冲液重悬细胞,测定不同pH条件下重组菌细胞酶活,分析不同pH条件下细胞酶活变化。为确定细胞催化最适反应温度,分别将反应体系于15、20、25、30、35、40 ℃金属浴测定酶活,测定不同温度条件下细胞催化的酶活;为确定重组菌细胞催化的最适底物浓度,以50、100、200、400、800 mmol/L的烟腈为底物,在25 ℃、pH 7.5条件下分别测定细胞酶活,测定不同底物浓度条件下细胞催化的酶活。
1.2.7 底物流加细胞催化工艺的建立:模拟烟酰胺的生产工艺,设计底物烟腈流加工艺试验,测定烟酰胺的积累量[25],考察在此工艺下的烟酰胺生物转化过程:1 mol/L的烟腈经蠕动泵以1.0 mL/min的流速加入20 mL OD600为5.0的菌液中并不断搅拌,每10 min取样并测定产物的积累量,1 h后停止流加。流加过程中,体系温度维持在20-25 ℃范围。
1.2.8 数据分析方法:使用SPSS 19.0软件对数据进行差异显著性分析,并使用Regression-Lineaner对各因子进行线性回归分析。试验均做3个平行。结果以“平均数±标准差”形式表示。文中涉及数据分析的图表中,均已进行差异显著性分析。
2 结果与分析 2.1 重组载体pET24a(+)-nhhBrbsArbsG在大肠杆菌中的表达将重组大肠杆菌E. coli BL21(DE3)/ pET24a(+)-nhhBrbsArbsG活化后,接种到2YT液体培养基培养至OD600约为1.0,加入终浓度为 0.6 mmol/L的IPTG和0.1 g/L的CoCl3·6H2O于24 ℃诱导表达18 h,收集细胞冰浴破碎。如图 2所示,α亚基和β亚基均有与理论相对分子量(α:22.7 kD;β:26.3 kD)相对应的条带[16]。破碎上清粗酶液的活性为85.5±4.3 U/mg[23]。表明H-NHase通过改造已实现在大肠杆菌中的过量表达。

|
| 图 2 H-NHase在E. coli BL21(DE3)中的表达 Figure 2 Expressions of H-NHase in E. coli BL21(DE3) 注:M:Marker;1:BL21(DE3)/pET24a(+)破碎上清;2:BL21(DE3)/pET24a(+) -nhhBrbsArbsG破碎上清. Note:M: Marker; 1: supernatants of BL21(DE3)/pET24a(+) expression after crushing as a comparison; 2: supernatants of BL21(DE3)/pET24a(+)-nhhBrbsArbsG expression after crushing. |
|
|
重组菌E. coli BL21(DE3)/pET24a(+)- nhhBrbsArbsG发酵培养后,收集菌体细胞后超声破碎。经硫酸铵盐析和强阴离子交换层析柱Hitrap Q HP纯化,超滤脱盐浓缩得到一定浓度H-NHase纯酶,如图 3所示,重组H-NHase达到电泳纯。以烟腈为底物测得重组酶的比酶活为234.0±11.7 U/mg,与野生菌R. rhodochrous J1中H-NHase和在R. rhodochrous ATCC12674中重组表达的H-NHase的比酶活处同一水平[16, 26]。
|
| 图 3 H-NHase分离纯化电泳图 Figure 3 SDS-PAGE of the purified H-NHase 注:1:Marker;2:高分子量型腈水合酶纯酶. Note:1: Marker; 2: purified H-NHase. |
|
|
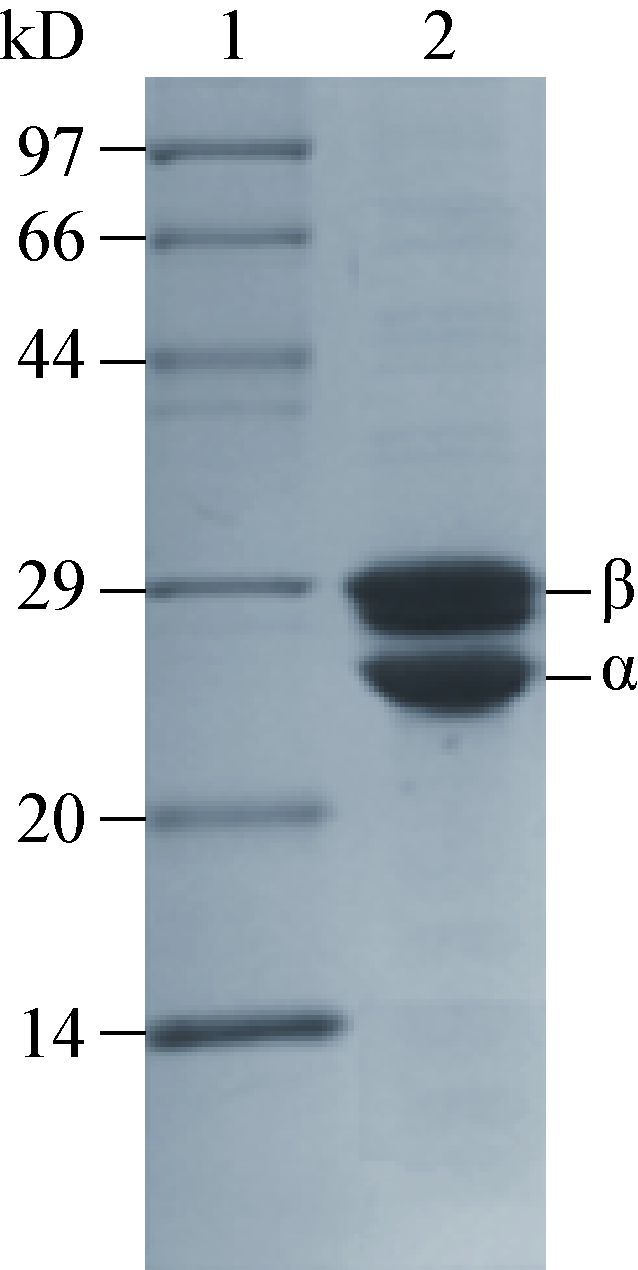
通过SEC对重组H-NHase的分子量进行鉴定。标准蛋白marker的分子量和洗脱体积分别为:(1) 谷氨酸脱氢酶(酵母),290 kD,11.17 mL;(2) 乳酸脱氢酶(猪心脏),142 kD,12.85 mL;(3) 烯醇化酶(酵母),67 kD,13.99 mL;(4) 肌激酶(酵母),32 kD,15.57 mL;(5) 细胞色素C (马心脏),12.4 kD,17.17 mL。如图 4所示,以洗脱体积与蛋白分子量的对数绘制标准曲线。随后,H-NHase纯酶以相同缓冲液经凝胶过滤层析柱,洗脱体积为10.55 mL,
|
| 图 4 H-NHase分子量测定 Figure 4 Molecular mass determination of H-NHase |
|
|
对照标准曲线,计算得出H-NHase的分子量为504.5±9.8 kD。进而,依据α,β亚基理论分子量(α:22.7 kD;β:26.3 kD)[16]推断出重组型H-NHase与野生型H-NHase聚体形式一致[16]。
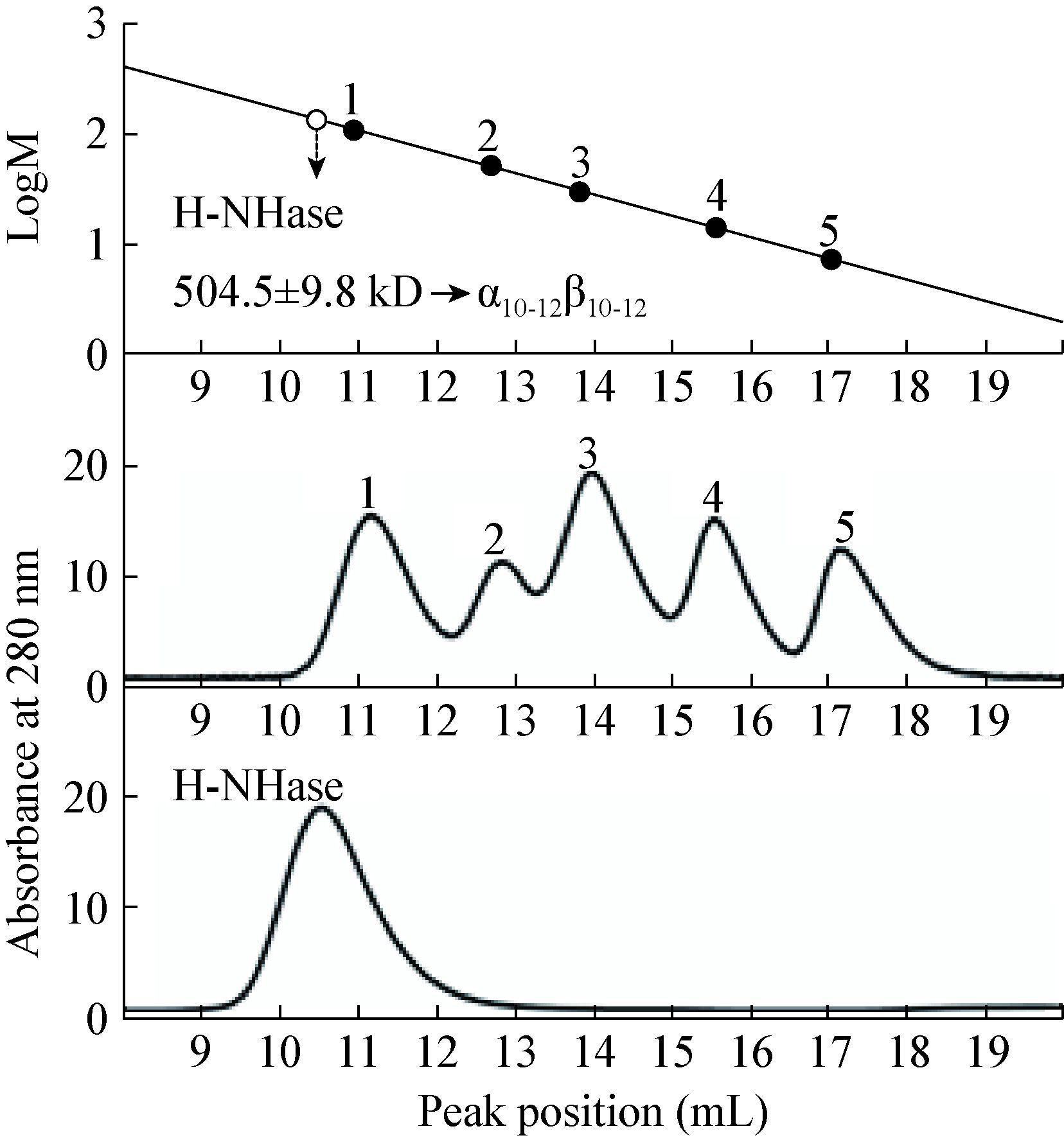
2.3 重组基因工程菌全细胞催化最适条件的确定2.3.1 pH对全细胞NHase的影响:反应体系的pH对酶的活性通常有较大的影响,不同来源的NHase有各自最适的pH[3]。为确定重组基因工程菌 E. coli-pET24a(+)-nhhBrbsArbsG全细胞催化烟腈的最适反应pH,选定8个不同pH条件的磷酸缓冲液(pH 5.5、6.0、6.5、7.0、7.5、8.0、8.5、9.0)进行实验。检测不同pH条件下重组菌E. coli-pET24a(+)- nhhBrbsArbsG细胞酶活的变化,结果表明,在pH 7.5条件下细胞催化烟腈的酶活最高,如图 5A所示,表明此pH值为最适pH。
2.3.2 温度对全细胞NHase的影响:酶在不同温度下进行酶促反应时,其活性有显著差别。同样,全细胞在不同反应温度下其活性也是不同的。为确定重组基因工程菌E. coli-pET24a(+)-nhhBrbsArbsG细胞的最适反应温度,分别测得不同温度下(15、20、25、30、35、40 ℃)重组菌细胞酶活。如图 5B所示,随着反应温度的升高,重组菌细胞酶活逐渐升高,在25 ℃时达到最大,随后降低,表明25 ℃为细胞催化的最适温度。
|
| 图 5 重组菌全细胞催化条件优化 Figure 5 Optimal condition for the whole-cell catalysis of recombinant strains E. coliBL21(DE3)/pET24a(+)- nhhBrbsArbsG 注:A:pH对重组菌细胞酶活的影响;B:温度对重组菌细胞酶活的影响;C:底物浓度对重组菌细胞酶活的影响. Note:A: Effect of pH on the activity of the recombinant strains; B: effect of temperature on the activity of the recombinant strains; C: effect of substrate concentrationon the activity of the recombinant strains. |
|
|
2.3.3 底物浓度对全细胞NHase的影响:在反应体系中,当酶没有达到饱和时,反应速率随底物浓度增大而增大,饱和时则不变。相似地,需要确定全细胞催化过程中酶活与底物浓度之间的关系。由 图 5C可知,底物浓度小于400 mmol/L时,细胞酶活随底物浓度的增加而升高,在400 mmol/L时达到最大,之后保持相对稳定。
2.4 生物法合成烟酰胺的流加工艺重组基因工程菌E. coli BL21(DE3)/pET24a(+)- nhhBrbsArbsG在最适反应条件(pH 7.5,25 ℃,400 mmol/L烟腈为底物)下酶活达256.0±10.4 U/mL (图 5)。基于此最适条件,通过使用底物恒速流加的方法模拟了烟酰胺的生产工艺。如图 6所示,1 mol/L的烟腈作为底物,以1 mL/min的流速向重组菌液中流加,随着底物的消耗,产物也不断积累,在1 h时停止流加,90%以上底物被重组菌细胞水合生成相应的烟酰胺,只有少量剩余。停止流加后10 min,剩余底物完全转化为烟酰胺。烟酰胺的终浓度达到748.8±2.4mmol/L,底物转化率为99.9%。结果表明,采用底物恒速流加策略可以提高烟酰胺的最终产量,有利于后期产物的提取。

|
| 图 6 细胞催化烟腈水合生成烟酰胺 Figure 6 Hydration reaction of 3-Cyanopyridine to nicotinamide catalyzed by the whole-cell 注:随着烟腈的流加,产物的量不断积累. -■-:重组大肠杆菌流加反应体系中产物烟酰胺的生成量(mmol/L);-□-:对应的底物烟腈的剩余量(mmol/L);- - -:1 h时停止流加. 重组大肠杆菌E. coli BL21(DE3)/pET24a(+)-nhhBrbsArbsG体系中产物烟酰胺的浓度在1 h内从0增加到650 mmol/L左右,70 min时剩余底物完全转化. 实验进行3次重复. Note: The product nicotinamide accumulated gradually,when immersed in dynamically increased concentrations of 3-Cyanopyridine. -■- for the concentration of nicotinamide in the feeding system including the recombinant strains E. coli BL21(DE3)/pET24a(+)-nhhBrbsArbsG,-□- for the concentration of the corresponding residual 3-Cyanopyridine,- - - for the stop of the feeding at 1 h. The concentration of nicotinamide in the system including the recombinant strains E. coli BL21(DE3)/pET24a(+)-nhhBrbsArbsG increased from 0 to about 650 mmol/L,and the residual substrate was all biocatalyzed. The experiments were performed in triplicate. |
|
|
本研究从重组蛋白表达量、重组酶活性及其蛋白质分子量这三个方面,探讨了重组大肠杆菌E. coli BL21(DE3)异源表达高分子量型腈水合酶H-NHase的酶学性质和催化能力,基于此建立了底物恒速流加的全细胞生物催化方法,高效地转化底物烟腈生产烟酰胺。之前Kobayashi等[23]虽尝试将R. rhodochrous J1来源的两种腈水合酶H-NHase和L-NHase在Escherichia coli中表达,但表达量均不高,酶活极低[23]。本研究中,我们对野生型nhhBAG基因的表达元件进行了三方面的改造。(1) 我们对各亚基上游的SD序列进行优化。将原始序列替换成大肠杆菌最偏好的SD序列AAGGA,与原核生物中16S rRNA基因的-3′端的嘌呤互补,进而增强蛋白质的翻译起始效率[27]。(2) 对各亚基之间间隔序列的长度进行优化。起始密码子AUG到SD序列的距离强烈影响翻译起始的效率[28-29]。其距离不同,所产生的调控翻译起始的水平也各不相同,我们在本实验中将原始序列替换成了常见的9 bp间隔序列GATATAGAT。(3) 将表达元件优化后的基因nhhBrbsArbsG进行大肠杆菌密码子偏好性优化。大肠杆菌的密码子使用具有简并性,即一个氨基酸可由两个以上的密码子编码。但是,大肠杆菌并不是同等地使用每个密码子来编码氨基酸。对某一个氨基酸而言,大肠杆菌通常倾向于使用它所对应的同义密码子中的一种或数种,而不是均衡地或随机地使用每一种同义密码子来编码它,即大肠杆菌的密码子使用具有偏好性[30-32]。通过这三方面的改造,我们实现了红球菌R. rhodochrous J1来源的H-NHase在大肠杆菌中的过量表达。另外,相对红球菌中H-NHase繁琐的纯化步骤[18],重组大肠杆菌中H-NHase的纯化方法实现了很大程度上的简化。H-NHase重组酶的活性、分子量大小以及蛋白质四级结构缔合方式等酶学性质与野生酶一致,表明H-NHase在大肠杆菌实现了高水平、高质量表达。本研究为腈水合酶基因工程改造及进一步提高腈水合酶在大肠杆菌中的表达水平奠定了基础,同时也为一些异源重组表达效果不好的多亚基蛋白质的高效表达奠定了一定的实践基础。
红球菌重组基因表达系统完成一个发酵周期至少需要90 h[20-21]。相比之下,利用重组表达系统完成发酵培养只需20 h,大大缩短了发酵周期。这一研究解决了野生型产腈水合酶的红球菌R. rhodochrous J1培养时间长、表达量低等问题。在大规模发酵生产腈水合酶中可以显著降低发酵成本,同时重组菌高效的底物生物转化效率也体现了重组大肠杆菌巨大的生产潜力和更高的工业价值。
4 结论本研究实现了红球菌R. rhodochrous J1来源的高分子量型腈水合酶H-NHase在重组型基因工程菌E. coli BL21(DE3)的过量表达。通过离子交换层析完成H-NHase的纯化,比酶活为234.0±11.7 U/mg,相对分子质量为504.5±9.8 kD (α10-12β10-12)。另外,重组菌E. coliBL21(DE3)/pET24a(+)-nhhBrbsArbsG全细胞催化条件pH 7.5,25 ℃,400 mmol/L烟腈为底物,重组菌细胞酶活256.0±10.4 U/mL。重组菌进行全细胞催化,在模拟底物流加工艺中,底物的转化率达99.9%。
| [1] | Kobayashi M, Shimizu S. Cobalt proteins[J]. European Journal of Biochemistry , 1999, 261 (1) : 1–9. DOI:10.1046/j.1432-1327.1999.00186.x |
| [2] | Yamada H, Kobayashi M. Nitrile hydratase and its application to industrial production of acrylamide[J]. Bioscience, Biotechnology, and Biochemistry , 1996, 60 (9) : 1391–1400. DOI:10.1271/bbb.60.1391 |
| [3] | Prasad S, Bhalla TC. Nitrile hydratases (NHases): at the interface of academia and industry[J]. Biotechnology Advances , 2010, 28 (6) : 725–741. DOI:10.1016/j.biotechadv.2010.05.020 |
| [4] | Foerstner KU, Doerks T, Muller J, et al. A nitrile hydratase in the eukaryote Monosiga Brevicollis[J]. PLoS One , 2008, 3 (12) : e3976. DOI:10.1371/journal.pone.0003976 |
| [5] | Marron AO, Akam M, Walker G. Nitrile hydratase genes are present in multiple eukaryotic supergroups[J]. PLoS One , 2012, 7 (4) : e32867. DOI:10.1371/journal.pone.0032867 |
| [6] | Noguchi T, Nojiri M, Takei KI, et al. Protonation structures of Cys-Sulfinic and Cys-Sulfenic acids in the photosensitive nitrile hydratase revealed by fourier transform infrared spectroscopy[J]. Biochemistry , 2003, 42 (40) : 11642–11650. DOI:10.1021/bi035260i |
| [7] | Nagashima S, Nakasako M, Dohmae N, et al. Novel non-heme iron center of nitrile hydratase with a claw setting of oxygen atoms[J]. Nature Structural & Molecular Biology , 1998, 5 (5) : 347–351. |
| [8] | Murakami T, Nojiri M, Nakayama H, et al. Post-translational modification is essential for catalytic activity of nitrile hydratase[J]. Protein Science , 2000, 9 (5) : 1024–1030. DOI:10.1110/(ISSN)1469-896X |
| [9] | Okamoto S, Eltis LD. Purification and characterization of a novel nitrile hydratase from Rhodococcus sp. RHA1[J]. Molecular Microbiology , 2007, 65 (3) : 828–838. DOI:10.1111/mmi.2007.65.issue-3 |
| [10] | Kobayashi M, Fujiwara Y, Goda M, et al. Identification of active sites in amidase evolutionary relationship between amide bond-and peptide bond-cleaving enzymes[J]. Proceedings of the National Academy of Sciences of the United States of America , 1997, 94 (22) : 11986–11991. DOI:10.1073/pnas.94.22.11986 |
| [11] | Watanabe I, Satoh Y, Enomoto K. Screening, isolation and taxonomical properties of microorganisms having acrylonitrile-hydrating activity[J]. Agricultural and Biological Chemistry , 1987, 51 (12) : 3193–3199. |
| [12] | Watanabe I, Satoh Y, Enomoto K, et al. Optimal conditions for cultivation of Rhodococcus sp. N-774 and for conversion of acrylonitrile to acrylamide by resting cells[J]. Agricultural and Biological Chemistry , 1987, 51 (12) : 3201–3206. |
| [13] | Nagasawa T, Yamada H. Microbial transformations of nitriles[J]. Trends in Biotechnology , 1989, 7 (6) : 153–158. DOI:10.1016/0167-7799(89)90026-7 |
| [14] | Nagasawa T, Ryuno K, Yamada H. Superiority of Pseudomonas chlororaphis B23 nitrile hydratase as a catalyst for the enzymatic production of acrylamide[J]. Experientia , 1989, 45 (11/12) : 1066–1070. |
| [15] | Nagasawa T, Takeuchi K, Yamada H. Occurrence of a cobalt-induced and cobalt-containing nitrile hydratase in Rhodococcus rhodochrous J1[J]. Biochemical and Biophysical Research Communications , 1988, 155 (2) : 1008–1016. DOI:10.1016/S0006-291X(88)80597-7 |
| [16] | Nagasawa T, Takeuchi K, Yamada H. Characterization of a new cobalt-containing nitrile hydratase purified from urea-induced cells of Rhodococcus rhodochrous J1[J]. European Journal of Biochemistry , 1991, 196 (3) : 581–589. DOI:10.1111/ejb.1991.196.issue-3 |
| [17] | Wieser M, Takeuchi K, Wada Y, et al. Low-molecular-mass nitrile hydratase from Rhodococcus rhodochrous J1: purification, substrate specificity and comparison with the analogous high-molecular-mass enzyme[J]. FEMS Microbiology Letters , 1998, 169 (1) : 17–22. DOI:10.1111/fml.1998.169.issue-1 |
| [18] | Komeda H, Kobayashi M, Shimizu S. Characterization of the gene cluster of high-molecular-mass nitrile hydratase (H-NHase) induced by its reaction product in Rhodococcus rhodochrous J1[J]. Proceedings of the National Academy of Sciences of the United States of America , 1996, 93 (9) : 4267–4272. DOI:10.1073/pnas.93.9.4267 |
| [19] | Zhou ZM, Hashimoto Y, Kobayashi M. Self-subunit swapping chaperone needed for the maturation of multimeric metalloenzyme nitrile hydratase by a subunit exchange mechanism also carries out the oxidation of the metal ligand cysteine residues and insertion of cobalt[J]. The Journal of Biological Chemistry , 2009, 284 (22) : 14930–14938. DOI:10.1074/jbc.M808464200 |
| [20] | Nagasawa T, Takeuchi K, Nardi-Dei V, et al. Optimum culture conditions for the production of cobalt-containing nitrile hydratase by Rhodococcus rhodochrous J1[J]. Applied Microbiology and Biotechnology , 1991, 34 (6) : 783–788. |
| [21] | Zhou ZM, Hashimoto Y, Shiraki K, et al. Discovery of posttranslational maturation by self-subunit swapping[J]. Proceedings of the National Academy of Sciences of the United States of America , 2008, 105 (39) : 14849–14854. DOI:10.1073/pnas.0803428105 |
| [22] | Yu YC, Cui WJ, Liu Y, et al. Strategy for over-expression of nitrile hydratase from Rhodococcus rhodochrous J1 in Escherichia coli[J]. Industrial Microbiology , 2014, 44 (2) : 14–19. (in chinese) 余越春, 崔文璟, 刘义, 等. Rhodococcus rhodochrous J1腈水合酶在大肠杆菌中的表达策略[J]. 工业微生物 , 2014, 44 (2) : 14–19. |
| [23] | Kobayashi M, Nishiyama M, Nagasawa T, et al. Cloning, nucleotide sequence and expression in Escherichia coli of two cobalt-containing nitrile hydratase genes from Rhodococcus rhodochrous J1[J]. Biochimica et Biophysica Acta (BBA) - Gene Structure and Expression , 1991, 1129 (1) : 23–33. DOI:10.1016/0167-4781(91)90208-4 |
| [24] | Cui YT, Cui WJ, Liu ZM, et al. Improvement of stability of nitrile hydratase via protein fragment swapping[J]. Biochemical and Biophysical Research Communications , 2014, 450 (1) : 401–408. DOI:10.1016/j.bbrc.2014.05.127 |
| [25] | Tian Y, Chen J, Yu H, et al. Overproduction of the Escherichia coli chaperones GroEL-GroES in Rhodococcus ruber improves the activity and stability of cell catalysts harboring a nitrile hydratase[J]. Journal of Mcrobiology & Biotechnology , 2016, 26 (2) : 337–346. |
| [26] | Zhou ZM, Hashimoto Y, Cui TW, et al. Unique biogenesis of high-molecular mass multimeric metalloenzyme nitrile hydratase: intermediates and a proposed mechanism for self-subunit swapping maturation[J]. Biochemistry , 2010, 49 (44) : 9638–9648. DOI:10.1021/bi100651v |
| [27] | Mironova RS, Xu J, AbouHaidar MG, et al. Efficiency of a novel non-shine-dalgarno and a shine-dalgarno consensus sequence to initiate translation in Escherichia coli of genes with different downstream box composition[J]. Microbiological Research , 1999, 154 (1) : 35–41. DOI:10.1016/S0944-5013(99)80032-9 |
| [28] | Steitz JA, Jakes K. How ribosomes select initiator regions in mRNA: base pair formation between the 3' terminus of 16S rRNA and the mRNA during initiation of protein synthesis in Escherichia coli[J]. Proceedings of the National Academy of Sciences of the United States of America , 1975, 72 (12) : 4734–4738. DOI:10.1073/pnas.72.12.4734 |
| [29] | Chen HY, Bjerknes M, Kumar R, et al. Determination of the optimal aligned spacing between the Shine-Dalgarno sequence and the translation initiation codon of Escherichia coli mRNAs[J]. Nucleic Acids Research , 1994, 22 (23) : 4953–4957. DOI:10.1093/nar/22.23.4953 |
| [30] | Bulmer M. The selection-mutation-drift theory of synonymous codon usage[J]. Genetics , 1991, 129 (3) : 897–907. |
| [31] | Sakai H, Imamura C, Osada Y, et al. Correlation between Shine-Dalgarno sequence conservation and codon usage of bacterial genes[J]. Journal of Molecular Evolution , 2001, 52 (2) : 164–170. |
| [32] | Gustafsson C, Govindarajan S, Minshull J. Codon bias and heterologous protein expression[J]. Trends in Biotechnology , 2004, 22 (7) : 346–353. DOI:10.1016/j.tibtech.2004.04.006 |
 2016, Vol. 43
2016, Vol. 43