扩展功能
文章信息
- 余君涵, 马雯雯, 王智文, 陈涛, 赵学明
- YU Jun-Han, MA Wen-Wen, WANG Zhi-Wen, CHEN Tao, ZHAO Xue-Ming
- 人工合成启动子文库研究进展
- Progress in synthetic promoter library
- 微生物学通报, 2016, 43(1): 198-204
- Microbiology China, 2016, 43(1): 198-204
- 10.13344/j.microbiol.china.150267
-
文章历史
- 收稿日期: 2015-03-31
- 接受日期: 2015-05-11
- 优先数字出版日期(www.cnki.net): 2015-06-05
2. 教育部系统生物工程重点实验室 天津 300072;
3. 天津化学化工协同创新中心 天津 300072
2.Key Laboratory of Systems Bioengineering,Ministry of Education,Tianjin 300072,China;
3.Collaborative Innovation Center of Chemical Science and Engineering,Tianjin 300072,China
近年来,随着合成生物学的发展,人工合成功能元件在代谢工程领域显示出巨大应用潜力[1]。细胞的代谢通量主要受到转录水平的调控,而启动子是转录水平的重要调控元件[2],因此,启动子被列为合成生物学的重要功能元件之一[3, 4]。
随着代谢工程研究的深入,人们发现对代谢途径的过度扰动,不仅不会提高目的产物的产量,反而会增加细胞的代谢负担[5]。为了调节代谢网络的通量平衡,提高目标产物产量、降低副产物生成,需要对代谢途径进行更为精细和定量的调控,使代谢途径中各个基因的表达强度实现最佳组合,这往往需要一系列表达强度差异可达几个数量级的启动子。然而,内源性启动子很难实现这种较为连续、完整的调控。对于诱导型启动子,添加不同浓度的诱导物在一定范围内可以实现不同强度的表达,但是超过或低于诱导范围则只能实现单一的“是或非”的表达模式[5]。对于组成型启动子,当序列确定时,基因表达的效率一定,无法进行人为调控。因此,研究者试图合成及表征更多的不同强度、不同功能的启动子,对细胞的代谢进行更为精细的调控,人工合成启动子文库也就应运而生。本文综述了人工合成启动子文库(Synthetic promoter library,SPL)的研究进展,评述了不同启动子文库构建策略;讨论了人工合成启动子文库在代谢工程领域的应用,并展望了人工合成启动子文库的发展方向。
目前,文库构建策略主要包括非保守序列随机化、易错PCR、杂合启动子工程、理性设计转录因子结合位点(Transcription factor-binding sites,TFBSs)、启动子与RBS组合。表 1对人工合成启动子文库的构建方法及效果进行了简单的概括。
| 构建策略 Construction strategy | 选用元件 Elements utilized | 文库大小 Library range (fold) | 参考文献 Reference |
| 非保守序列随机化 Randomization of non-conserved sequences | 31 conserved, 2 semi-conserved (W or R) and 22 random nucleotides | 400 | [6] |
| 24 conserved, 13 semi-conserved (W, R or D), 20 random nucleotides | 349 | [8] | |
| −10 consensus TATAAT, −35 consensus TTGACA, 27 random nucleotides | 23 | [9] | |
| 16 conserved, 3 semi-conserved | 160 | [10] | |
| 70 random nucleotides, 5′UTR, ribosome binding site (AGGA) | 12 | [11] | |
| −10 consensus gngnTA(c/t)aaTgg, E. coli −35 consensus | 6 | [12] | |
| ermEp1 UP element, −10 and −35 consensus sequences | 159 | [7] | |
| PFY1p core promoter, Reb1p TFBS, poly-dT element, 26 conserved and 48 random nucleotides | 9 | [13] | |
| Two CT boxes, twp RPG boxes, TATA box, 83 random nucleotides | 5 286 | [14] | |
| Jet promoter, 61 conserved and 69 random nucleotides | 10 | [15] | |
| 易错PCR Ep-PCR | Phage PL-λ | 196 | [16] |
| TEF1 promoter | 15 | [17] | |
| 杂合启动子工程 Hybrid promoter engineering | Core-tac-promoter | 4.4 | [18] |
| UAS1B enhancer from PXPR2 and LEU2 minimal core promoter | 32 | [19] | |
| UAS1B enhancer from PXPR2, LEU2 minimal core promoter, TEF core promoter series | 400 | [20] | |
| Gal4p TFBSs and PLEUM core promoter | 50 | [21] | |
| apoE and ABP enhancers, ADH6, hAAT, CYP, SV40, and FIX core promoters | 975 | [22] | |
| p47phox elements, SP46 | 94.6 | [23] | |
| NFκB, E-box, C/EBPα, GC-box, E4F1, CRE, CMV core promoter element | 100 | [24] | |
| 理性设计TFBSs Rational design of TFBSs | AOX1 promoter | 28 | [25] |
| 启动子与RBS组合 Combination of promoter and RBS | 114 promoters and 111 RBSs | 1 000 | [26] |
原核生物的启动子结构一般包括−35区“TTGACA”,−10区“TATAAT”,−35区和−10区之间的间隔区域及−35区上游区域(UP element)。真核生物的启动子结构较原核生物复杂,主要包括核心启动子元件和上游启动子元件两部分,其中核心启动子元件由转录起始点和TATA盒两部分组成,上游启动子元件通常包括位于−70 bp附近的CAAT盒和GC盒以及距转录起始点更远的上游增强子。
保守序列是RNA聚合酶直接结合的区域,其序列的改变对启动子的活性影响显著。非保守序列虽然不直接结合转录蛋白,但其序列或长度的改变会对启动子的三维结构产生影响,改变关键基团的相对方位,影响相关蛋白的识别与特异性结合,从而改变基因的表达水平。因此,非保守序列的改变可获得不同强度的启动子。
非保守序列随机化一般有3种不同的设计方案:(1) 保留菌种的保守或半保守序列,其余序列随机化;(2) 保留启动子的保守序列或半保守序列,其余序列随机化;(3) 完全随机化,即整个启动子结构全部采用简并序列。
虽然多数原核生物的启动子都包含−10区和−35区2个保守序列,但对于某一菌种而言,仍存在其特有的保守序列。因此,利用非保守序列随机化的方法构建启动子文库时,首先统计出目标菌种的保守序列,根据统计结果进行随机序列的设计。Jensen等[6]根据乳酸乳球菌中特有的启动子保守序列,保留了−10区、−35区、−10区上游1 bp的TG结构、−35区邻近的ATTC结构、−44位点的AGTTT和+1至+8之间的GTACTGTT,其余序列随机化,最终筛选出38个不同活性的启动子,表达活性差异达400倍。基于某一特定的启动子序列,进行随机化设计,也是常用的构建方法。Siegl等[7]保留了ErmEp1启动子的−10区和−35区序列,
其余序列随机化,所得文库的活性在ErmEp1原始活性的2%-319%之间。进而利用文库中筛选出的强启动子Promoter 21表达RppA基因,使淡黄霉素产量提高了3.3倍。此外,启动子序列完全随机化的构建方法已在谷氨酸棒杆菌中得以实现。2013年,Yim等[11]利用完全随机化在谷氨酸棒杆菌中构建了启动子文库,并从中筛选出高活性的启动子PH36,将其用于木聚糖内切酶基因XynA的表达,最终在5 L发酵罐中实现了746 mg/L的产量。除此之外,研究人员也将序列随机化的方法引入酿酒酵母[14]及哺乳动物细胞[15]中进行了启动子文库的构建。
序列随机化的半理性设计在原核生物中的应用较为广泛。随机化区域的可选择性强,增加了启动子序列的多样性。该方法设计简便,但也存在一定的缺陷,如引物序列较长,成本较高,且这种半理性随机化的方法具有一定的盲目性,不同的宿主细胞对于不同启动子序列的识别有一定的差异,所构建的文库强度较难预测,需要高通量技术进行筛选。
2 易错 PCR易错PCR通过调整PCR反应体系中镁离子浓度、加入锰离子、改变4种dNTPs浓度、加入保真性差的DNA聚合酶等,来增加扩增的突变率,从而引入突变位点。易错PCR是简单、有效的体外随机诱变技术,是获得序列多样性的有效方法。
Alper等[16]通过添加核酸类似物8-oxo-dGTP和dPTP对噬菌体PL-λ启动子进行易错PCR扩增,最终得到了22个不同强度的启动子,启动子的强度分布范围可达196倍。Kagiya等[27]向PCR体系中添加0.50 mmol/L的MnCl2,将一段来源于Hela细胞的无活性DNA序列进行4轮易错PCR,在212 bp的碱基序列中引入了7个碱基转换和7个碱基颠换突变,最终产生了一个在大肠杆菌中的高活性启动子,其活性甚至超过了Tac启动子。Nevoigt等[17]通过添加核酸类似物8-oxo-dGTP和dPTP将随机突变引入DAN1启动子,获得了在微氧条件下也能够诱导表达的启动子突变体,其活性是野生型启动子的1.8−2.9倍。此外,他们在酿酒酵母TEF1启动子的基础上,构建了表达强度差异为15倍的启动子文库,并从该文库中挑选出5个不同强度的启动子与GPD1的原始启动子进行替换,探究了3-磷酸甘油醛脱氢酶的活性对甘油产量的影响[28]。
易错PCR引入的突变是随机突变,可以发生在启动子区域的任何位置,为构建较大的文库提供了保证。但仍存在一些问题:(1) 碱基突变具有倾向性,影响启动子序列的多样性;(2) 突变效果依赖于目的片段的碱基种类及长度,需进行条件优化;(3) 突变结果多是无活性突变,筛选较为复杂。
3 杂合启动子工程杂合启动子工程是指将不同来源的上游原件或核心启动子区域进行重构。杂合启动子在原核生物中早有体现,如常用的大肠杆菌Tac启动子的就是通过融合Trp和Lac启动子的核心启动子区域获得[29];RhaPBAD是阿拉伯糖启动子与鼠李糖启动子融合的产物[30]。杂合启动子往往综合了亲本启动子的优势,具有高于2个亲本的强度。
在真核生物的启动子结构中,TATA框与RNA聚合酶结合并决定转录起始,但单一的RNA聚合酶与TATA框相结合只能引起较低水平的转录。上游启动子元件(Upstream promoter element,UPE),即酵母中的上游激活序列(Upstream activate sequence,UAS)对转录起始有较大的影响[31]。UPE与对应的转录因子结合(例如,GC区与转录因子Sp1结合,CAAT区与转录因子CTF结合),直接作用于靶位点,有利于稳定起始前复合物(Pre-initiation complex,PIC)的结构,进而加速转录起始过程。由于这些转录因子彼此独立行使功能,可作用于任何含有特异性结合位点的启动子,因此,通过在核心启动子上游引入不同来源及不同数量的UPE,即可获得不同表达活性的启动子,如图 1所示。

|
| 图 1 杂合启动子工程示意图 Figure 1 Schematic plot of hybrid promoter project |
目前,研究人员已通过串联不同数量UPE的方法,在多种真核生物细胞中构建了杂合启动子文库。Mutalik等[32]首次在解脂耶罗维亚酵母(Yarrowia lipolytica)中将XPR2启动子上游序列元件UAS1B串联至LEU2核心启动子上游,分别串联1−4个UAS1B构建了杂合启动子hp1d、hp2d、hp3d、hp4d。研究发现,随着UAS1B数量的增加,启动子强度逐级递增。在此基础上,Blazeck等[20]进一步将1−32个UAS1B元件分别串联至LEU2启动子中,构建了一个活性变化范围达400倍的启动子文库。此外,串联不同来源的UAS元件也是研究人员积极寻求的方向。Blazeck等[21]将分别来自CLB2、CIT1、GAL1、TEF基因的UAS元件按照不同的组合分别与不同的核心启动子元件进行融合,成功构建了杂合启动子文库,使酵母中最强启动子PGPD的转录活性提高了2.5倍。Brown等[24]利用启动子杂合的方法实现了CHO细胞中启动子文库的构建。
杂合启动子工程可以在强启动子基础上进一步提高转录活性,解决了某些菌株中高活性启动子缺乏的难题。但该方法也存在局限性:(1) 人工串联的UPE数量有限,文库的活性变化范围受到影响;(2) 特定UPE的添加使启动子活性变化呈阶梯状,文库的精细度受到影响;(3) 无法“量体裁衣”,不能针对具体的核心启动子筛选出适当来源及数量的UPE。表征不同来源的UPE并探究其相应转录因子与PIC的作用机理将成为杂合启动子工程未来发展的重点。
4 理性设计TFBSs无论是真核生物,还是原核生物的启动子结构中均含有不同的短核酸序列,这些结构所介导的胞内转录因子的结合实现了对启动子活性的调控。探究启动子元件的作用机理是理性设计TFBSs的一个重要方面。Murphy等[33]通过改变TetO2抑制因子结合位点在PGAL1中的位置及数量,确定了其对基因表达水平的影响机制。作者发现TetO2位点与TATA框的距离越近、叠加数量越多,转录抑制效果越强。Hartner等[25]报道了利用改变TFBSs的方法在巴斯德毕赤酵母(Pichia pastoris)中构建启动子文库。通过将AOX1启动子的TFBSs进行删除或倍增的突变操作后,所得文库中启动子的活性在原启动子活性的6%-160%之间变化。
相较于前3种启动子文库构建方法,理性改变TFBSs更具有针对性,是文库构建发展的方向。但目前该方法的应用较少,这归因于启动子与转录因子的作用机制还不够清晰。随着转录调控机制研究的进一步深入,以及高通量表征技术的发展,转录因子结合位点的表征将会越来越清晰,靶向突变也将会越来越合理,最终将获得所需表达强度的启动子。
5 启动子与RBS组合启动子和RBS分别是转录水平和翻译水平的重要调控元件。Salis等开发的RBS计算器(RBS calculator),可以对RBS序列进行强度预测。本实验室结合RBS计算器设计出RBS文库,对核黄素代谢通路的RibF的表达量进行了不同程度的弱化,获得了核黄素产量提高了77%的克隆[34]。此外,针对另一个出发菌株,同样采用RBS文库对核黄素代谢通路的5个基因的表达强度进行了组合优化,短期内就获得了核黄素产量提高了2.67倍的克隆[35]。因为RBS序列很短,人工设计的RBS文库可以通过引物设计和PCR扩增与启动子文库进行连接,从而构建新型启动子文库。Kosuri等[26]将114个启动子和111个RBS序列进行排列组合,以绿色荧光蛋白作为报告基因,最终得到了4个数量级的启动子强度变化范围。相较于前4种文库构建方法,启动子与RBS组合是双文库的集成,所构建的启动子文库范围更为广阔,可实现转录和翻译水平的双层次的调控。
6 结论与展望随着合成生物学的发展,从头设计并构建新的生物元件、模块和系统,实现生物系统的标准化是合成生物学家追求的目标。人工合成启动子文库作为合成生物学的重要元件,在代谢途径精细调控方面已表现出巨大的应用潜力。越来越多的研究表明,代谢途径的完全理性设计通常无法实现最优化,因此,一种“半理性”的工程思想“组合优化法”应运而生[36, 37](图 2)。Liu等[38]利用双启动子系统对n-乙酰葡糖胺合成路径进行改造。通过不同强度的启动子对该路径中2个关键酶基因进行组合优化,将n-乙酰葡糖胺的产量提高了32.4%。
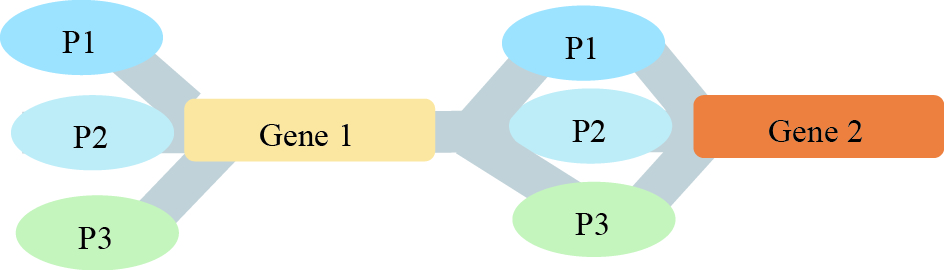
|
| 图 2 组合优化示意图 Figure 2 Schematic plot of combinatorial optimization |
构建启动子文库是“组合优化”策略的典型代表,是一种对系统功能元件进行集中突变或修饰来获得最优表型的方法。总体上讲,人工合成启动子文库将会向着以下2个方向发展:(1) 启动子序列或元件的设计更加理性化;(2) 结合工程化思想,实现启动子强度的模型预测。
随着系统生物学各项技术的发展,核酸序列的功能性被进一步揭示,DNA-蛋白质相互作用机理进一步明确,更加有助于阐明启动子序列中核心元件的结构、功能及其对启动子强度影响的大小,从而可以更加理性地设计启动子序列及各个作用元件,减小启动子构建的盲目性,降低对高通量筛选技术的要求。
在合成生物学研究中,“抽提”是重要的工程化研究方法,用计算机等其他工程领域的复杂层次思想处理生物学系统,使生物系统的模拟成为现实。例如,Park等[39]利用计算子模型确定了造血干细胞中转录调控模式。随着计算机模型预测的发展,最终将会实现代谢网络中启动子表达强度的可预测性,从而在代谢途径优化的过程中,对启动子的选择更加理性。
总之,随着合成生物学和生物信息学技术的发展,各种不同来源数据的累积都将为理性设计启动子文库提供依据,加之多种组合优化技术和工程模拟思想的日臻成熟,也将为人工合成启动子文库与其他调控元件在代谢工程中共同发挥作用提供条件。人工合成启动子文库将为最终合成新的生物系统和构建新的生物功能提供有力支持。
| [1] | Khalil AS, Collins JJ. Synthetic biology: applications come of age[J]. Nature Reviews Genetics, 2010, 11(5): 367-379 |
| [2] | Keasling JD. Synthetic biology for synthetic chemistry[J]. ACS Chemical Biology, 2008, 3(1): 64-76 |
| [3] | Keasling JD. Synthetic biology and the development of tools for metabolic engineering[J]. Metabolic Engineering, 2012, 14(3): 189-195 |
| [4] | Young E, Alper H. Synthetic biology: tools to design, build, and optimize cellular processes[J]. Journal of Biomedicine and Biotechnology, 2010, 2010: 130781 |
| [5] | Mnaimneh S, Davierwala AP, Haynes J, et al. Exploration of essential gene functions via titratable promoter alleles[J]. Cell, 2004, 118(1): 31-44 |
| [6] | Jensen PR, Hammer K. The sequence of spacers between the consensus sequences modulates the strength of prokaryotic promoters[J]. Applied and Environmental Microbiology, 1998, 64(1): 82-87 |
| [7] | Siegl T, Tokovenko B, Myronovskyi M, et al. Design, construction and characterisation of a synthetic promoter library for fine-tuned gene expression in actinomycetes[J]. Metabolic Engineering, 2013, 19: 98-106 |
| [8] | de Mey M, Maertens J, Lequeux GJ, et al. Construction and model-based analysis of a promoter library for E. coli: an indispensable tool for metabolic engineering[J]. BMC Biotechnology, 2007, 7: 34 |
| [9] | Braatsch S, Helmark S, Kranz H, et al. Escherichia coli strains with promoter libraries constructed by Red/ET recombination pave the way for transcriptional fine-tuning[J]. Biotechniques, 2008, 45(3): 335-337 |
| [10] | Rud I, Jensen PR, Naterstad K, et al. A synthetic promoter library for constitutive gene expression in Lactobacillus plantarum[J]. Microbiology, 2006, 152(Pt 4): 1011-1019 |
| [11] | Yim SS, An SJ, Kang M, et al. Isolation of fully synthetic promoters for high-level gene expression in Corynebacterium glutamicum[J]. Biotechnology and Bioengineering, 2013, 110(11): 2959-2969 |
| [12] | Rytter JV, Helmark S, Chen J, et al. Synthetic promoter libraries for Corynebacterium glutamicum[J]. Applied Microbiology and Biotechnology, 2014, 98(6): 2617-2623 |
| [13] | Blount BA, Weenink T, Vasylechko S, et al. Rational diversification of a promoter providing fine-tuned expression and orthogonal regulation for synthetic biology[J]. PLoS One, 2012, 7(3): e33279 |
| [14] | Jeppsson M, Johansson B, Jensen PR, et al. The level of glucose-6-phosphate dehydrogenase activity strongly influences xylose fermentation and inhibitor sensitivity in recombinant Saccharomyces cerevisiae strains[J]. Yeast, 2003, 20(15): 1263-1272 |
| [15] | Tornøe J, Kusk P, Johansen TE, et al. Generation of a synthetic mammalian promoter library by modification of sequences spacing transcription factor binding sites[J]. Gene, 2002, 297(1/2): 21-32 |
| [16] | Alper H, Fischer C, Nevoigt E, et al. Tuning genetic control through promoter engineering[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(36): 12678-12683 |
| [17] | Nevoigt E, Kohnke J, Fischer CR, et al. Engineering of promoter replacement cassettes for fine-tuning of gene expression in Saccharomyces cerevisiae[J]. Applied and Environmental Microbiology, 2006, 72(8): 5266-5273 |
| [18] | Li MJ, Wang JS, Geng YP, et al. A strategy of gene overexpression based on tandem repetitive promoters in Escherichia coli[J]. Microbial Cell Factories, 2012, 11: 19 |
| [19] | Madzak C, Treton B, Blanchin-Roland S. Strong hybrid promoters and integrative expression/secretion vectors for quasi-constitutive expression of heterologous proteins in the yeast Yarrowia lipolytica[J]. Journal of Molecular Microbiology and Biotechnology, 2000, 2(2): 207-216 |
| [20] | Blazeck J, Liu LQ, Redden H, et al. Tuning gene expression in Yarrowia lipolytica by a hybrid promoter approach[J]. Applied and Environmental Microbiology, 2011, 77(22): 7905-7914 |
| [21] | Blazeck J, Garg R, Reed B, et al. Controlling promoter strength and regulation in Saccharomyces cerevisiae using synthetic hybrid promoters[J]. Biotechnology and Bioengineering, 2012, 109(11): 2884-2895 |
| [22] | Gehrke S, Jérôme V, Müller R. Chimeric transcriptional control units for improved liver-specific transgene expression[J]. Gene, 2003, 322: 137-143 |
| [23] | Kang WS, Kwon JS, Kim HB, et al. A macrophage-specific synthetic promoter for therapeutic application of adiponectin[J]. Gene Therapy, 2014, 21(4): 353-362 |
| [24] | Brown AJ, Sweeney B, Mainwaring DO, et al. Synthetic promoters for CHO cell engineering[J]. Biotechnology and Bioengineering, 2014, 111(8): 1638-1647 |
| [25] | Hartner FS, Ruth C, Langenegger D, et al. Promoter library designed for fine-tuned gene expression in Pichia pastoris[J]. Nucleic Acids Research, 2008, 36(12): e76 |
| [26] | Kosuri S, Goodman DB, Cambray G, et al. Composability of regulatory sequences controlling transcription and translation in Escherichia coli[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(34): 14024-14029 |
| [27] | Kagiya G, Ogawa R, Hatashita M, et al. Generation of a strong promoter for Escherichia coli from eukaryotic genome DNA[J]. Journal of Biotechnology, 2005, 115(3): 239-248 |
| [28] | Nevoigt E, Fischer C, Mucha O, et al. Engineering promoter regulation[J]. Biotechnology and Bioengineering, 2007, 96(3): 550-558 |
| [29] | de Boer HA, Comstock LJ, Vasser M. The tac promoter: a functional hybrid derived from the trp and lac promoters[J]. Proceedings of the National Academy of Sciences of the United States of America, 1983, 80(1): 21-25 |
| [30] | Haldimann A, Daniels LL, Wanner BL. Use of new methods for construction of tightly regulated arabinose and rhamnose promoter fusions in studies of the Escherichia coli phosphate regulon[J]. Journal of Bacteriology, 1998, 180(5): 1277-1286 |
| [31] | Hahn S, Young ET. Transcriptional regulation in Saccharomyces cerevisiae: transcription factor regulation and function, mechanisms of initiation, and roles of activators and coactivators[J]. Genetics, 2011, 189(3): 705-736 |
| [32] | Mutalik VK, Guimaraes JC, Cambray G, et al. Precise and reliable gene expression via standard transcription and translation initiation elements[J]. Nature Methods, 2013, 10(4): 354-360 |
| [33] | Murphy KF, Balázsi G, Collins JJ. Combinatorial promoter design for engineering noisy gene expression[J]. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(31): 12726-12731 |
| [34] | Lin Z, Xu Z, Li Y, et al. Metabolic engineering of Escherichia coli for the production of riboflavin[J]. Microbial Cell Factories, 2014, 13: 104 |
| [35] | Li YF, Gu Q, Lin ZQ, et al. Multiplex iterative plasmid engineering for combinatorial optimization of metabolic pathways and diversification of protein coding sequences[J]. ACS Synthetic Biology, 2013, 2(11): 651-661 |
| [36] | Gu Q, Li YF, Chen T. Combinatorial optimization of synthetic biological systems[J]. Chinese Journal of Biotechnology, 2013, 29(8): 1064-1074 (in Chinese) |
| [37] | Yadav VG, de Mey M, Lim CG, et al. The future of metabolic engineering and synthetic biology: towards a systematic practice[J]. Metabolic Engineering, 2012, 14(3): 233-241 |
| [38] | Liu YF, Zhu YQ, Li JH, et al. Modular pathway engineering of Bacillus subtilis for improved N-acetylglucosamine production[J]. Metabolic Engineering, 2014, 23: 42-52 |
| [39] | Park SJ, Umemoto T, Saito-Adachi M, et al. Computational promoter modeling identifies the modes of transcriptional regulation in hematopoietic stem cells[J]. PLoS One, 2014, 9(4): e93853 |
| [36] | 顾群, 李一凡, 陈涛. 合成生物系统的组合优化[J]. 生物工程学报, 2013, 29(8): 1064-1074 |
 2016, Vol. 43
2016, Vol. 43




