扩展功能
文章信息
- 刘朝, 乔建军, 朱宏吉
- LIU Zhao, QIAO Jian-Jun, ZHU Hong-Ji
- 乳酸菌肽聚糖的研究进展
- Research progress in peptidoglycan of lactic acid bacteria
- 微生物学通报, 2016, 43(1): 188-197
- Microbiology China, 2016, 43(1): 188-197
- 10.13344/j.microbiol.china.150222
-
文章历史
- 收稿日期: 2015-03-18
- 接受日期: 2015-05-11
- 优先数字出版日期(www.cnki.net): 2015-05-13
乳酸菌(Lactic acid bacteria,LAB)是一类能够利用可发酵碳水化合物产生大量乳酸的无芽孢、革兰氏阳性细菌。在工业上应用较多的主要有乳球菌属(Lactococcus)、乳杆菌属(Lactobacillus)、链球菌属(Streptococcus)、肠球菌属(Enterococcus)、酒球菌属(Oenococcus)、小球菌属(Pediococcus)和明串珠菌属(Leuconostoc)等[1, 2]。乳酸菌能提高食物的营养价值和附加值、改善食物风味,因此常用于制造酸奶、乳酪等发酵产品[3]。乳酸菌还能调节宿主胃肠道正常菌落比例、改善胃肠道功能,并能增强宿主免疫力,是广泛存在于人体胃肠道内的益生菌[4]。另外,有研究证明乳酸菌可在人体肠道定殖,因此可用作疗效蛋白或核酸疫苗的肠道黏膜靶向运送载体[5]。乳酸菌的上述功能应用都与其细胞壁组分和结构有关。噬菌体感染是工业发酵牛奶面临的重大威胁之一,而乳酸菌细胞壁正是由于含有噬菌体受体成为了近些年研究的热点[6]。同时风味物质的形成是奶酪发酵后期成熟过程的关键因素,近年研究还聚焦在构建细胞壁可控水解的乳酸菌发酵剂,以实时控制乳酸菌的自溶释放出脂肪酶、蛋白酶等胞内物质,这些有利于风味物质的形成和奶酪的成熟[7, 8]。
乳酸菌细胞壁是以多层肽聚糖为骨架的较厚封闭网状结构,磷壁酸、多糖及相关蛋白等特殊组分穿插其中,此外某些乳酸菌外壁还有次晶态的S层蛋白[9]。细胞壁在细菌生长过程中起着多重作用。一方面具有一定的机械强度以保护细菌细胞免受内部渗透压和外部环境的影响,从而使细菌保持特征性形态;另一方面具有一定的柔韧性以适应细菌细胞在生长和分裂过程中的形态变化。细胞壁作为细菌细胞接触外界环境的第一道保护层,它不仅在抵御环境胁迫中发挥着重要作用,还能够影响细菌与噬菌体或真核宿主细胞之间的联系[6]。研究表明在产乳酸菌素(Nisin)的乳酸乳球菌中,野生型低Nisin耐受菌株与突变型高Nisin耐受菌株相比,二者细胞壁有明显差异[10, 11]。
肽聚糖(Peptidoglycan,PG)是乳酸菌细胞壁的主要组成成分,除了大家所熟知的上述功能外,它作为乳酸菌中含量最丰富的微生物相关保守分子(Microbe-associated molecular patterns,MAMPs)之一,在益生菌与宿主的联系中起重要作用[12, 13],并对宿主免疫反应具有一定的调节作用和抗炎作用[14]。因为MAMPs可被宿主天然免疫系统中存在的相关模式识别受体识别,通过激活不同的信号传导途径释放各种免疫调控物质,从而发挥免疫功能[15]。此外,乳酸菌肽聚糖可作为功能蛋白的结合组件使乳酸菌成为理想的靶向载体。比如肽聚糖水解酶有特异性结合位点,将它与功能蛋白进行融合可构建表面功能展示系统[16]。本文就目前研究较多的乳酸菌肽聚糖的结构、代谢调控及其生物学活性加以综述,将为深入了解乳酸菌各功能应用的作用机制提供基础依据,并为挖掘新的应用拓宽研究思路。
1 乳酸菌肽聚糖的组成和结构特点乳酸菌肽聚糖是由糖链、四肽尾及肽桥(相邻2条四肽尾间的交联方式)聚合而成的多层网状大分子结构。乳酸菌的肽聚糖糖链都是由N-乙酰葡萄糖胺(GlcNAc)和N-乙酰胞壁酸(MurNAc)通过β-1,4糖苷键交替连接而成[17],但是四肽尾氨基酸组成和肽桥构造因菌种而异[18]。四肽尾共价连结在MurNAc的乳酰基上,它在乳酸菌新生肽聚糖的氨基酸组成是1-L-丙氨酸-2-γ-D-谷氨酸-3-X-4-D-丙氨酸-5-Y(1-L-Ala-2-γ-D-Glu-3-X-4-D-Ala-5-Y),最后一个氨基酸Y在成熟大分子中会被水解掉,其中X是一个二氨基氨基酸,通常是L-赖氨酸(L-Lys)、内消旋二氨基庚二酸(mDAP)或L-鸟氨酸(L-Orn),而Y是D-丙氨酸(D-Ala)或D-乳酸(D- Lac)[19]。例如在植物乳杆菌(Lactobacillus plantarum)中X是mDAP,Y是D-Lac[20],在发酵乳杆菌(Lactobacilus fermentum)中X是L-Orn[21],在以乳酸乳球菌(Lactococcus lactis)为代表的其他大多数乳酸菌中X是L-Lys[2]。相邻四肽尾间通过一条四肽尾的第4位氨基酸与另一条四肽尾的第3位氨基酸进行4-3交联形成肽桥,肽桥可分为直接交联和间接交联2种方式,直接交联仅存在于植物乳杆菌等少数乳酸菌中[20]。乳酸菌中常见的间接肽桥是由一个D-氨基酸或多个L-氨基酸构成,如乳酸乳球菌和干酪乳杆菌(Lactobacillus casei)等大部分乳酸菌的肽桥是D-天冬氨酸(D-Asp)[22, 23, 24],而粪肠球菌(Enterococcus faecalis)的是L-二丙氨酸(L-Ala-L-Ala)[25]。如图 1所示,GlcNAc和MurNAc组成的双糖、连在MurNAc上的肽尾以及D-Asp间接肽桥形成了乳酸菌肽聚糖的典型基本结构单位。
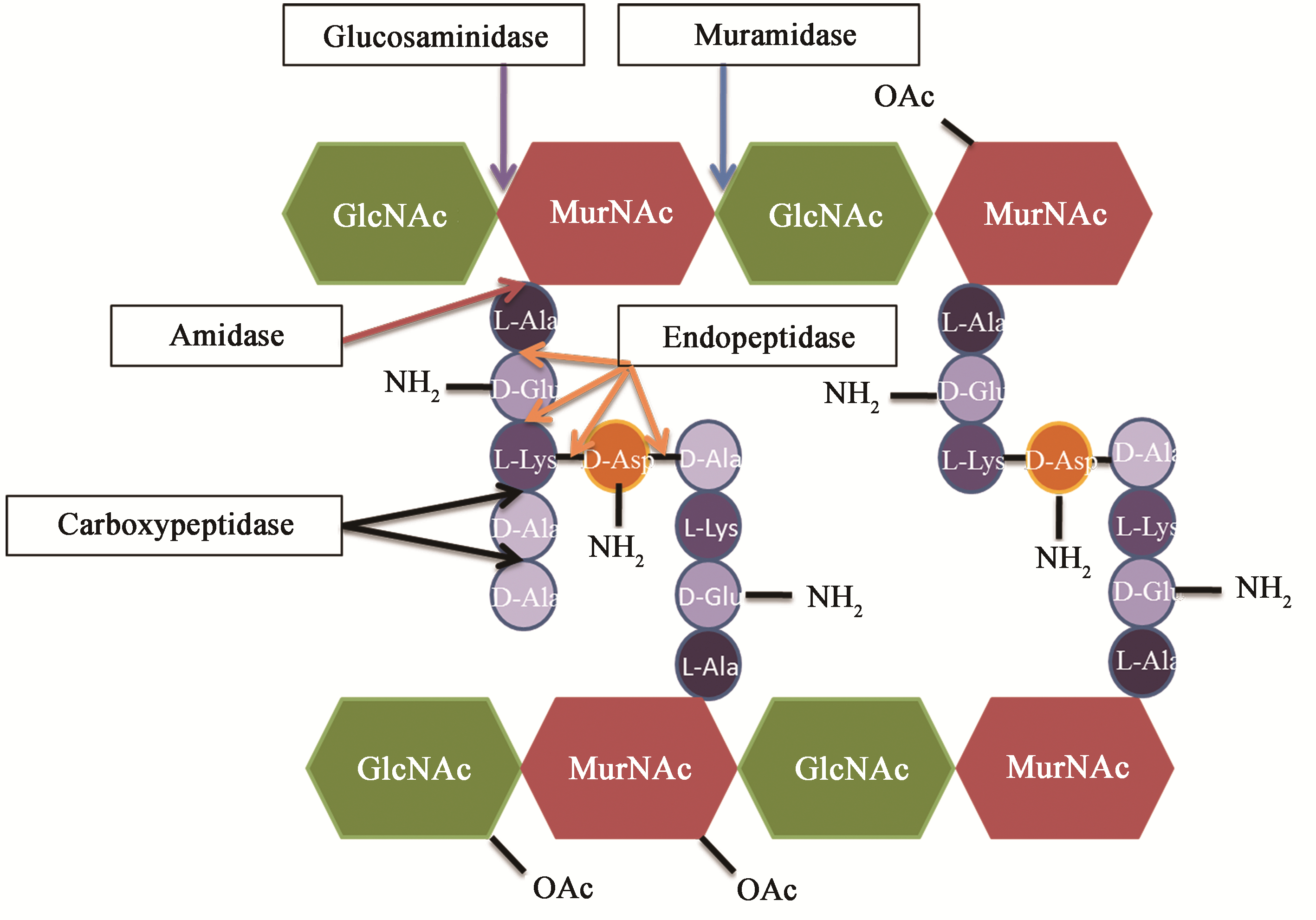
|
| 图 1 肽聚糖的典型化学结构 Figure 1 The representativechemical structure of peptidoglycan |
交联度和糖链长度是肽聚糖结构的2个重要特征,它们不仅具有菌株特异性,还与生长条件紧密相关。立体的交联使肽聚糖形成多层网状结构,交联度的大小表征网状结构的紧密程度。有研究表明乳酸乳球菌MG1363的交联度是35.8%[22],干酪乳杆菌BL23的是34%[23],鼠李糖乳杆菌GG (Lactobacillusrhamnosus GG)的是36.5%[24],植物乳杆菌NZ7100的是37.5%[26]。另一方面,Wheeler等利用分子排阻高效液相色谱(Size-exclusion HPLC)对乳酸乳球菌MG1363和粪肠球菌JH2-2的肽聚糖糖链长度进行检测,发现大于25双糖单位的长糖链占主导,超过100双糖单位的长糖链约为8%−14%[27]。Andre等采用能与肽聚糖结合的LysM域探针对乳酸乳球菌突变株(WPS-)纳米级肽聚糖结构进行相对作用力测定,根据原子力显微镜得到的力曲线谱图分析结果得出肽聚糖是以平行于细胞短轴类螺旋线方式进行组装的[28]。
2 乳酸菌肽聚糖的代谢及其调控 2.1 肽聚糖的生物合成肽聚糖的生物合成是一个涉及多步酶促反应的复杂过程,其合成可根据反应发生部位的不同而分成在细胞质中、细胞膜上和细胞膜外3个阶段(图 2)。
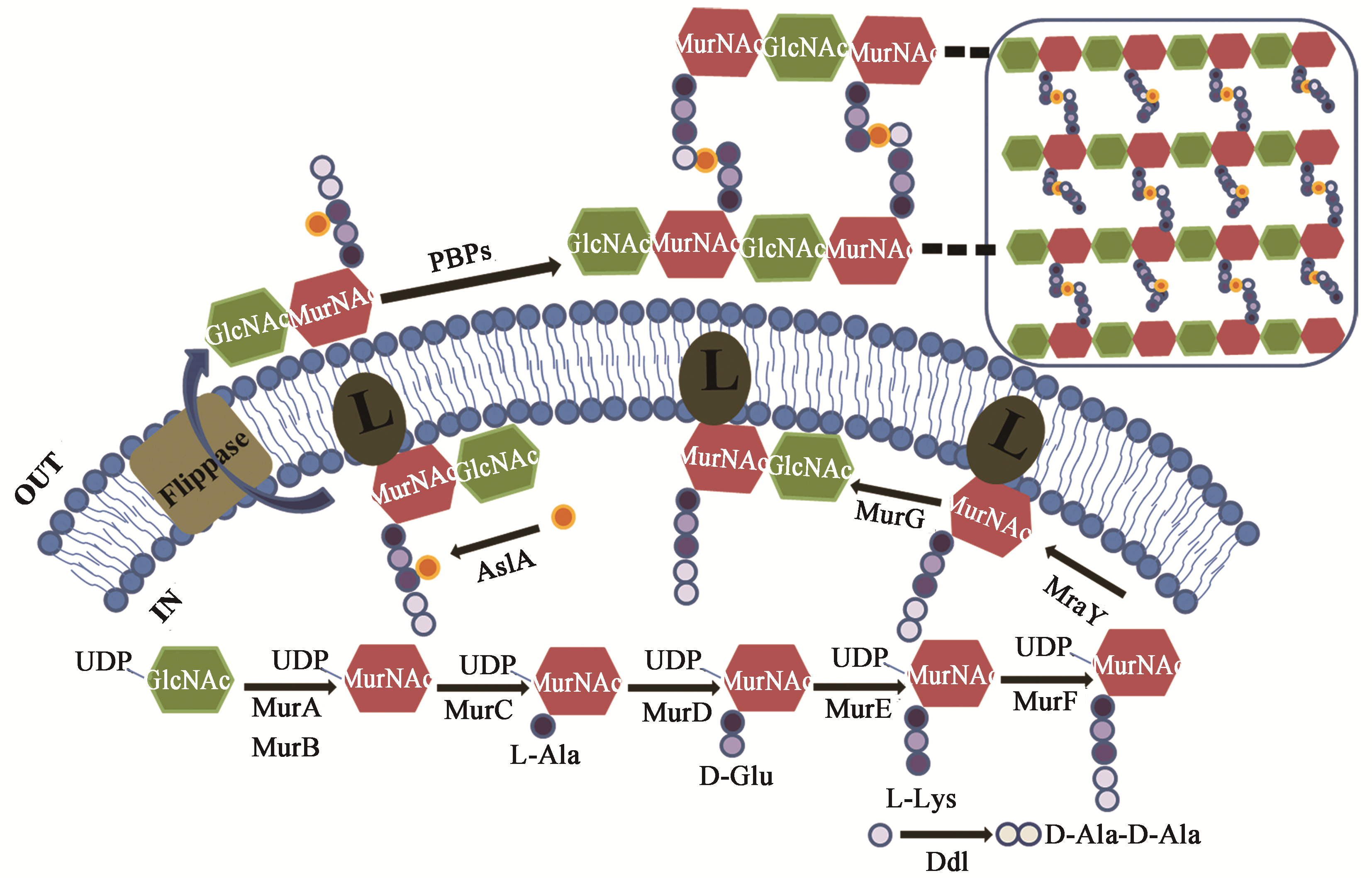
|
| 图 2 肽聚糖的生物合成过程 Figure 2 The biosynthesisprocess of peptidoglycan |
(1) 第一阶段:在细胞质中合成UDP-MurNAc五肽。GlcNAc和MurNAc的尿苷二酸衍生物(UDP-GlcNAc,UDP-MurNAc)是合成肽聚糖的活性前体,UDP-GlcNAc经过2步连续的酶促反应生成UDP-MurNAc:MurA催化UDP-GlcNAc和磷酸烯醇式丙酮酸反应生成无机磷和尿苷二磷酸-N-乙酰葡萄糖胺烯醇式丙酮酸(UDP-GlcNAcEP),后者随后被其还原酶MurB还原为UDP-MurNAc。随后4个Mur连接酶逐步催化合成UDP-MurNAc五肽[29]。首先在MurC催化下UDP-MurNAc的乳酰基连接上L-Ala,其次在MurD催化下上一步产物连接上D-Glu,再次在MurE催化下连接上L-Lys或者mDAP,最后在MurF催化下连接上二肽形式(D-Ala-D-Ala)或二缩肽形式(D-Ala-D-Lac)的2个残基,而二肽或二缩肽的合成是在D-D-连接酶(Ddl)作用下完成的。其中自然界中存在的L-Ala和L-Glu分别需要经过特异消旋酶Alr和MurI的作用转换成
肽聚糖中存在的D型[29]。此外,植物乳杆菌中存在一种Aad酶,能水解Ddl连接酶合成的D-Ala-D-Ala二肽,这个过程保证新合成UDP-MurNAc五肽肽尾的最后一个氨基酸都是D-Lac[30]。Deghorain等将植物乳杆菌中的Ddl连接酶基因在乳酸乳球菌中进行异源表达后,理论存在的D-Ala-D-Ala二肽形式肽尾被实际检测的D-Ala-D-Lac二缩肽形式肽尾部分或全部取代,并进一步证明了UDP-MurNAc五肽肽尾最后一个残基的改变会引起整个肽聚糖结构和细胞形态发生明显变化[31]。
(2) 第二阶段:在细胞膜上进行移位。UDP-MurNAc五肽经质膜移位酶MraY的催化作用连接到一种称为细菌萜醇(即十一碳二烯磷酸酯)的类脂载体上,生成十一碳二烯-焦磷酰-胞壁酰五肽或称Lipid I。随后,糖基转移酶MurG将UDP-GlcNAc连在Lipid I的MurNAc部分,同时释放出UDP,形成十一碳二烯-焦磷酰-双糖五肽或称Lipid II[32]。另一个发生在质膜上的重要酶促反应是Lipid II上肽侧链的合成,该肽侧链在细胞膜外合成阶段形成间接肽桥,因此其合成存在菌种特异性[33]。D-Asp是乳酸菌中最常见的间接肽桥,由racD编码的天冬氨酸消旋酶作用于L-Asp而生成。D-Asp经ATP-Grasp家族中的天冬氨酸连接酶(AslA)催化连接到Lipid II五肽尾的第3个氨基酸(L-Lys)。Veiga等通过基因组分析鉴定出yxbA在乳酸乳球菌中同源编码AslA,于是将该基因更名为aslA,同时研究者们将该基因敲除又用重组质粒作回补,将L-Ser-L-Ala和L-Ala-L-Ala代替D-Asp作为AslA的催化底物,证明aslA是菌株生长所必需的基因,但它所编码酶的底物选择特异性低[34]。L-Ala-L-Ala是粪肠球菌中的间接肽桥,Bouhss等研究表明粪肠球菌JH2-2中LipidII上的肽侧链L-Ala-L-Ala是BppA1和BppA2分别将丙酰基-tRNA的L-Ala逐步连接到LipidII上合成得到的[25]。有或无侧链的Lipid II再经过一个翻转酶的作用转运至细胞膜外侧。Mohammadi等首次提出内膜蛋白FtsW或其同源蛋白RodA是负责Lipid II跨膜转运的翻转酶[35],但是最近Sham等证实在大肠杆菌(Escherichia coli)肽聚糖合成过程中Lipid II的翻转酶是内膜转运蛋白MurJ而不是FtsW或RodA[36],乳酸菌中相关翻转酶的鉴定有待进一步的研究发现。
(3) 第三阶段:在细胞膜外进行交联。肽聚糖单体通过转糖基作用和转肽作用发生聚合形成肽聚糖网状结构。在转糖基过程中,Lipid II上的双糖单位连接到已存在的肽聚糖糖链中,使糖链在横向上延伸一个双糖单位,类脂载体失去一个无机磷而转移至细胞膜内侧开启新一轮的循环。再经过转肽作用,新加入肽聚糖单体肽尾与其相邻肽尾间一条肽尾(供体链)第4位的D-Ala羧基和另一条肽尾(受体链)第3位的二氨基氨基酸或其附加侧链氨基酸自由氨基之间形成肽键,并释放出供体链的D-Ala或D-Lac和受体链的D-Ala-D-Ala二肽或D-Ala-D-Lac二缩肽,使这个新加入肽聚糖单体的肽尾与其相邻肽尾发生纵向交联。青霉素结合蛋白(PBPs)是参与该阶段肽聚糖组装的主要蛋白,它们在各乳酸菌中的数量存在差异。根据分子量大小的不同,PBPs可主要分为2类:高分子量(HMW) PBPs和低分子量(LMW) PBPs[37]。David的研究表明乳酸乳球菌中存在6种青霉素结合蛋白,其中PBP1a、PBP1b、PBP2a、PBP2b和PBPx这5种属于HMW,而D-丙氨酸-D-丙氨酸-羧肽酶DacA这一种属于LMW[38]。Courtin等的研究证明乳酸乳球菌中还存在一种L,D-羧肽酶(DacB),它负责切割肽尾中3-L-Lys-4-D-Ala间肽键[22]。D,D-羧肽酶DacA和L,D-羧肽酶DacB参与肽聚糖交联过程中四肽尾和三肽尾的产生。另外,PBPs还可根据其所起作用不同分为2类:A类既负责转糖基作用又参与转肽作用,而B类只参与转肽作用[37]。
2.2 肽聚糖的结构修饰在大部分乳酸菌中,成熟肽聚糖的部分基本结构会经过修饰,如糖链的O-乙酰化或N-去乙酰化,或者肽尾氨基酸的酰胺化(图 1),这些结构修饰在细菌生长过程中会影响肽聚糖水解酶(PGHs)对肽聚糖的水解[39],以下将具体介绍肽聚糖的各种化学基团修饰。
在大多数乳酸菌中肽聚糖糖链的O-乙酰化特指MurNAc残基的C6位羟基发生乙酰化而形成2,6-N,O-二乙酰衍生物,催化该步反应的O-乙酰转移酶OtaA最先在金黄色葡萄球菌(Staphylococcusaureus)中被发现[40]。进一步研究证明,OtaA在乳酸菌等革兰氏阳性菌中高度保守[20, 41, 42],该酶有N端跨膜螺旋域和C端转乙酰基功能域,在细胞膜外侧利用乙酰-CoA提供的乙酰基进行新合成肽聚糖MurNAc残基的O-乙酰化[42]。研究人员在对常见乳酸菌的肽聚糖结构进行鉴定分析时发现,乳酸乳球菌MG1363中MurNAc的O-乙酰化程度仅为3.2%[23],干酪乳杆菌BL23中为30%[23],鼠李糖乳杆菌GG中为37%[24],植物乳杆菌NZ7100中为39%[20]。另外,GlcNAc的O-乙酰化仅在植物乳杆菌、清酒乳杆菌(Lactobacillus sake)和类肠膜魏斯氏菌(Weissellaparamesenteroides)等少数细菌中被发现[19],负责GlcNAc的O-乙酰化的酶是另一个特异性O-乙酰转移酶OtaB,这个酶和植物乳杆菌的OtaA有相似的N端和C端结构域,但二者氨基酸序列相似度仅为21%[20]。Bernard等在植物乳杆菌中的研究表明肽聚糖的O-乙酰化会影响其自溶,GlcNAc的O-乙酰化会抑制N-乙酰葡萄糖胺糖苷酶Acm2的活性,MurNAc的O-乙酰化反而会激活N-乙酰胞壁酸-L-丙氨酸酰胺酶LytH的作用[20]。Veiga等发现乳酸乳球菌在应对溶菌酶或抗生素胁迫时,首先激活二组分系统(TCS) CesSR,CesSR再激活一个全局转录因子SpxB,SpxB再激活oatA的表达,最后oatA的表达会增强肽聚糖MurNAc的O-乙酰化从而增强菌株对溶菌酶或抗生素的耐受性[43]。
GlcNAc的N-去乙酰化是肽聚糖骨架中存在葡萄糖胺(图 1中GlcNH2)的原因,催化该过程的酶PgdA最先在肺炎链球菌(Streptococcuspneumoniae)中被鉴定[44]。Meyrand等在乳酸乳球菌IL1403中鉴定出编码GlcNAc的N-去乙酰酶PgdA的基因xynD,重命名为pgdA,同时还发现增加GlcNAc的N-去乙酰化程度可减少主要自溶素AcmA对肽聚糖的水解作用,从而降低自溶率[45]。干酪乳杆菌BL23基因组中存在pgdA的同源序列,却在实验条件下未检测到N-去乙酰化的GlcNAc[23]。Kobayashi等在枯草芽孢杆菌(Bacillus subtilis)中发现的MurNAc的N-去乙酰酶[46]及其同源蛋白目前在乳酸菌中未见报道。
肽聚糖肽尾的第2位D-Glu和第3位mDAP以及肽桥的D-Asp常发生酰胺化(图 1),这些反应在细胞内由特定的酶催化进行[33]。在乳酸乳球菌中,肽桥侧链D-Asp是在被天冬氨酸连接酶(AsnA)添加至肽聚糖前体之后才被酰胺化,该步骤所涉及的酶是天冬氨酸合成酶(AsnH)[47](图 2)。同时该研究还证明乳酸乳球菌肽桥D-Asp的酰胺化不仅会影响PGHs活性,还能增强对Nisin等阳离子抗菌剂的耐受性,这可能与细胞壁上负电荷的减少有关[47]。有研究发现乳酸乳球菌中约75%的D-Asp肽桥发生酰胺化[34],而干酪乳杆菌中接近100%发生酰胺化[23]。Bernard等证明植物乳杆菌中约94%的mDAP都被酰胺化,并首次鉴定出与该步酰胺化相关的酶AsnB1,还提出mDAP酰胺化可能在控制分离过程中起重要作用[20]。肽尾D-Glu的酰胺化存在于乳酸菌等多种细菌中,金黄色葡萄球菌中参与该步反应的酶是谷氨酸酰胺转移酶GatD和MurT,而乳酸菌中相关酶还未被发现[48, 49]。同时Figueiredo等的研究还证明肽尾D-Glu的酰胺化会影响菌株对β-内酰胺抗生素和溶菌酶的敏感性,从而影响菌株生长[48]。D-Glu的酰胺化在常见的4种乳酸菌中几乎都接近100%[20, 22, 23, 24],而且通过基因组分析都鉴定出了gatD和murT的同源基因。
2.3 肽聚糖的水解代谢肽聚糖水解酶(PGHs)是一类能特异性水解细胞壁肽聚糖肽键的酶。根据作用位点的不同,PGHs主要分为以下几类:(1) N-乙酰胞壁酸酶(Muramidases),它可以切断MurNAc和GlcNAc之间的β1-4糖苷键,使MurNAc的还原基团解离出来;(2) N-乙酰葡萄糖胺酶(Glucosaminidases),它作用于GlcNAc和MurNAc之间β1-4糖苷键解离出GlcNAc的还原基团;(3) N-乙酰胞壁酰-L-丙氨酸酰胺酶(Amidases),它主要水解MurNAc和肽尾第一位L-Ala之间的酰胺键;(4) 肽酶(Peptidase),它主要作用于肽尾或肽桥氨基酸之间的肽键,包括D型氨基酸和D型氨基酸间的内肽酶(Endopeptidase)、D型氨基酸和D型氨基酸间的羧肽酶(Carboxypeptidase)以及L型氨基酸和D型氨基酸间的羧肽酶[50](图 1)。
目前多种乳酸菌中部分PGHs已经被鉴定。在对乳酸乳球菌研究中依次发现了4个同源的葡萄糖胺酶AcmA、AcmB、AcmC、AcmD和内肽酶YjgB,它们在菌体不同生长阶段的表达水平、酶活性以及在细胞分裂过程中所起的作用都各不相同[51]。AcmA有3个氨基酸重复序列组成的C端LysM域和N端催化域,前者是与细胞壁结合的识别位点,后者是N-乙酰葡萄糖胺酶活性位点[52]。AcmB有3个结构域,分别为中心催化活性域、C端重复序列结构域和N端富含丝氨酸/苏氨酸/脯氨酸/天冬氨酸(Ser/Thr/Pro/Asp)结构域[53]。AcmC只有一个N端催化活性结构域,AcmD与AcmA相似有C端识别结构域和N端催化活性域,内肽酶YjgB有信号肽结构域和催化活性结构域[52]。Redko等通过将乳酸乳球菌中唯一的内肽酶YjgB失活后发现细菌的表型并没有发生明显变化,由此推断乳酸乳球菌可能含有多于上述5种PGHs[54]。干酪乳杆菌BL23中基因组分析得到编码PGHs的基因13个,再利用菌体在不同生长阶段的蛋白质组学分析结合SDS-PAGE和LC-MS/MS发现该菌株在生长过程中合成7种PGHs,分别为胞壁质酶和羧肽酶各1个,内肽酶和酰胺酶各2个,以及内肽酶或酰胺酶(未确定) 1个,其中γ-D-谷氨酰-L-赖氨酰基-内肽酶是主要的PGHs[23]。有研究提出PGHs在细胞分裂中起重要作用,而且不同菌种利用各不相同的PGHs作用于细胞分裂[55]。乳酸乳球菌MG1363中AcmA和AcmB均参与菌体的细胞分裂和自溶过程,AcmD单独作用时对菌体自溶不产生影响,但与AcmA联合作用时会加速菌体自溶,同时AcmD在细胞分裂中也起着重要作用[56]。植物乳杆菌WCFS1中Acm2是主要的自溶素,它不仅是菌体分裂过程中的必需酶,而且在自溶中也发挥着重要作用,另外D,L-内肽酶LytA决定着菌体形态,它是维持菌体形态和保证细胞壁完整性所不可或缺的[57]。
2.4 肽聚糖的代谢调控肽聚糖的调控是指对肽聚糖的合成和水解过程进行协调平衡,以满足菌体进行正常的生理活动,乳酸菌中的这种调控主要就是调控肽聚糖水解酶(PGHs)的活性。研究表明某些乳酸菌的PGHs活性会受到蛋白酶的调控。乳酸乳球菌MG1363产生的一种与细胞壁结合的蛋白酶PrtP会水解其主要的自溶素AcmA[58],另外乳酸乳球菌IL1403的胞外蛋白酶HtrA也可以降解AcmA[59]。Buist等在乳酸乳球菌MG1363中敲除编码蛋白酶PrtP基因后发现菌株的自溶率明显提高,随后表达PrtPⅠ型蛋白酶(分泌型)、PrtPⅢ型蛋白酶(细胞壁锚定型)或共表达PrtPⅠ-PrtPⅢ型蛋白酶作为回补,酶谱分析结果显示AcmA的降解由低至高分别为表达PrtPⅠ型蛋白酶、共表达PⅠ-PⅢ型蛋白酶、表达PrtPⅢ型蛋白酶[58]。乳酸菌中某些PGHs的活性还受它在细胞壁的锚定位点影响。Steen等将乳酸乳球菌中自溶素AcmA的C端LysM域去掉而保留N端催化域,发现该AcmA衍生物既不能与乳酸菌细胞壁结合也不能裂解乳酸菌,然而将AcmA的C端LysM域与恶性疟原虫裂殖子表面蛋白MSA2的C端进行融合,免疫荧光标记显示该融合蛋白能特异性结合到包括乳酸乳球菌在内的多种革兰氏阳性菌的细胞壁上,再对乳酸乳球菌细胞壁进行纯化及进一步处理,发现肽聚糖是融合蛋白结合的组件并且有特异的结合位点[60]。细胞壁的磷壁酸或多糖等组分不仅通过对肽聚糖的屏蔽作用影响PGHs在细胞壁的定位从而调节菌体自溶特性[61],而且乳酸菌的自溶还会受到磷壁酸D-丙氨酰酯化修饰的影响[62]。Palumbo等在植物乳杆菌NCIMB8826中将主要负责磷壁酸D-丙氨酰酯化的dlt操纵子插入失活后发现,尽管磷壁酸组分的D-丙氨酰酯化修饰程度明显降低,但是磷壁酸的糖基化以及它的长度明显提高,而且突变菌株的生长速率下降而菌体自溶程度提高。该研究提出dlt突变菌株的高自溶是由于磷壁酸D-丙氨酰酯化修饰程度的降低激活了植物乳杆菌主要自溶素Acm2的活性[62]。此外,Rolain等首次提出植物乳杆菌中Acm2的O-糖基化会控制该酶的活性[63]。该研究发现Acm2的糖基化是发生在富含丙氨酸/丝氨酸/组氨酸(Ala/Ser/His,AST)的N端结构域,即在Ser或His残基上连接有21个GlcNAc单体。该研究结果表明Acm2酶N端AST域不发生糖基化反而会增强它的酶活力,这可能是因为O-糖基化改变Acm2结构域的构象或者影响域间的作用,从而使Acm2的N端AST域阻碍它的催化活性域与底物的结合[63]。
目前,针对编码PGHs的基因在基因水平、转录水平以及翻译水平等调控机制方面的研究大都集中在研究较多的模式菌株中,乳酸菌中相关研究还未见报道。Dubrac等首次提出金黄色葡萄球菌中二组分系统WalK/WalR可正向调控9个PGHs基因的表达从而影响自溶素的活性,另外WalK/WalR缺陷细胞不仅对溶菌酶的耐受性提高,而且表现为肽聚糖的交联度和糖链长度会增加[64]。Smith等的研究综述了枯草芽孢杆菌中自溶素在转录水平的调控,处于稳定期菌株自溶素基因lytC、lytD和lytF的表达主要受到σD的调控,约20%的lytC表达又会直接被σA所调控,而lytE的表达同时受到σA和σH的调控,这些调控机制与枯草芽孢杆菌的生理活动密切相关[65]。
3 乳酸菌肽聚糖的生物学活性肽聚糖是乳酸菌细胞壁中的重要组分,可能参与乳酸菌多种生物学功能,如免疫增强功能、抗感染、抗肿瘤及抗过敏等[66, 67]。乳酸菌肽聚糖是人类免疫系统识别的激活剂,它是通过诱导非特异性免疫因子或特异性免疫因子的释放或表达来刺激机体免疫系统发挥功能。现有研究表明某些乳酸菌肽聚糖可以诱导促炎性和抗炎性细胞因子的产生,如促炎性白细胞介素(IL-1、IL-6、IL-12)、肿瘤坏死因子-α (TNF-α)、γ干扰素(IFN-γ)、抗炎性白细胞介素(IL-10)和转化生长因子β[68]等,其中IL-12和IFN-γ对巨噬细胞和自然杀伤细胞的功能具有增强作用,这可能是乳酸菌抗感染和抗癌的作用机制。在天然免疫中,微生物相关保守分子是乳酸菌的主要免疫调节成分,宿主细胞利用相关受体可以识别入侵细菌的肽聚糖等微生物相关分子模式,通过不同的信号传导途径引发免疫反应,目前已发现的信号通路有信号传导途径(Toll途径)和免疫缺陷途径(Imd途径)。乳杆菌肽聚糖通过与Toll样受体(Toll-like receptors, TLRs)结合激活机体抗原递呈细胞并通过表达表面受体调节它们的功能,同时促使机体分泌细胞因子和趋化因子,从而引发机体的免疫反应,肽聚糖可能是乳杆菌免疫赋活的重要组分[69]。姚光国等给小鼠腹腔注射乳酸菌肽聚糖提取物后,参照对照组发现实验组巨噬细胞吞噬率和吞噬指数均明显提高、血清溶菌酶活性显著增强,由此表明乳酸菌肽聚糖不仅能够激活巨噬细胞,还对血清溶菌酶有明显的刺激作用,从而增强机体非特异性免疫功能[70]。还有研究发现,乳酸菌肽聚糖也能通过诱导产生相应的细胞因子影响Th1/Th2失衡,从而对过敏产生一定的抑制作用。马冬雪等首先用牛乳β-乳球蛋白腹腔注射BALB/c小鼠建立过敏症模型,然后分离致敏小鼠的脾淋巴细胞与活/死乳杆菌体外共同孵育,结果发现乳杆菌可提高致敏小鼠脾淋巴细胞的IFN-γ/IL-4比值,进而纠正Th2占优势的Th1/Th2失衡,下调抗体分泌量[71]。李丹等同样建立上述过敏症模型成功后,分离致敏小鼠的脾淋巴细胞与不同剂量的嗜酸乳杆菌肽聚糖提取物共同孵育,结果显示嗜酸乳杆菌肽聚糖提取物体外刺激致敏脾细胞后可显著抑制IgE的产生,上调CD3+、CD4+细胞数及CD4+/CD8+比值,上调Th1型因子(IFN-γ,T-bet mRNA)及Treg型因子(TGF-β,Foxp3 mRNA)的表达,下调Th2型因子(IL-4,GATA-3 mRNA)和Th17型因子(IL-17,RORγt mRNA)的表达,由此进一步表明乳杆菌肽聚糖体外刺激可有效纠正致敏脾淋巴细胞的Th1/Th2及Treg/Th17失衡[72]。因此,与肽聚糖相关的免疫识别研究是一项非常复杂的工作,其准确的免疫调节作用机制有待进一步深入研究。
4 展望随着现代技术手段的进步,乳酸菌肽聚糖的结构及其合成研究在过去二十年中取得显著进步,不仅分离纯化得到了多种常见乳酸菌肽聚糖的组分,而且阐明了乳酸菌肽聚糖精准结构甚至种间和种内的差异。随后,得力于基因工程手段的发展,鉴定得到了多种乳酸菌中肽聚糖合成、修饰以及水解途径所涉及的基因信息。这些成果为乳酸菌在食品和健康方面的应用奠定了基础。然而,乳酸菌如何多层次调控肽聚糖水解酶活性和菌体自溶,噬菌体如何专一吸附到靶向细菌表面以及益生菌乳酸菌和宿主之间如何联系,这些分子机制都是基于乳酸菌肽聚糖研究的前沿方向。目前乳酸菌肽聚糖与抗生素和溶菌酶耐受性关系的研究较多[44, 47, 48, 49],而乳酸菌肽聚糖对乳酸菌其他方面耐受能力(比如酸、温度、重金属等)的影响尚需进一步研究。此外,乳酸菌肽聚糖生物学活性方面的研究也取得了不错的进展,例如乳酸菌肽聚糖抗感染、抗肿瘤以及抗过敏等方面的活性相继被发现,并由此被人们用于畜禽饲料或药物添加剂,与此相关的作用机制还有待被挖掘。肽聚糖要作为合格的人类食物添加剂和医药产品还需要进一步的研究。随着人们对乳酸菌肽聚糖的结构、代谢及其生物学活性的进一步探索发现,其应用前景将更为广阔,其多种有益的生物学功能将会得到更加广泛的应用。
| [1] | Salminen S, von Wright A, Ouwehand AC, et al. Lactic Acid Bacteria: Microbiological and Functional Aspects[M]. Florida: The Chemical Rubber Company Press, 2004: 1-2 |
| [2] | Makarova K, Slesarev A, Wolf Y, et al. Comparative genomics of the lactic acid bacteria[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(42): 15611-15616 |
| [3] | Sgarbi E, Lazzi C, Tabanelli G, et al. Nonstarter lactic acid bacteria volatilomes produced using cheese components[J]. Journal of Dairy Science, 2013, 96(7): 4223-4234 |
| [4] | da Silva ST, dos Santos CA, Bressan J. Intestinal microbiota; relevance to obesity and modulation by prebiotics and probiotics[J]. Nutrición Hospitalaria, 2013, 28(4): 1039-1048 |
| [5] | Bermúdez-Humarán LG, Kharrat P, Chatel JM, et al. Lactococci and lactobacilli as mucosal delivery vectors for therapeutic proteins and DNA vaccines[J]. Microbial Cell Factories, 2011, 10(Suppl 1): S4 |
| [6] | Chapot-Chartier MP. Interactions of the cell-wall glycopolymers of lactic acid bacteria with their bacteriophages[J]. Frontiers in Microbiology, 2014, 5: 236 |
| [7] | Xu Y, Kong J. Construction and potential application of controlled autolytic systems for Lactobacillus casei in cheese manufacture[J]. Journal of Food Protection, 2013, 76(7): 1187-1193 |
| [8] | Xu Y. A high-yield membrane protein expression system and autolysis in lactic acid bacteria[D]. Jinan: Doctoral Dissertation of Shandong University, 2012 (in Chinese) 徐毅. 乳酸菌高效膜蛋白表达系统的构建及其自溶性质的研究[D]. 济南: 山东大学博士学位论文, 2012 |
| [9] | Schär-Zammaretti P, Ubbink J. The cell wall of lactic acid bacteria: surface constituents and macromolecular conformations[J]. Biophysical Journal, 2003, 85(6): 4076-4092 |
| [10] | Zhang YF, Liu SY, Du YH, et al. Genome shuffling of Lactococcus lactis subspecies lactis YF11 for improving nisin Z production and comparative analysis[J]. Journal of Dairy Science, 2014, 97(5): 2528-2541 |
| [11] | Kramer NE, Hasper HE, van den Bogaard PTC, et al. Increased D-alanylation of lipoteichoic acid and a thickened septum are main determinants in the nisin resistance mechanism of Lactococcus lactis[J]. Microbiology, 2008, 154(6): 1755-1762 |
| [12] | Lebeer S, Vanderleyden J, De Keersmaecker SCJ. Host interactions of probiotic bacterial surface molecules: comparison with commensals and pathogens[J]. Nature Reviews Microbiology, 2010, 8(3): 171-184 |
| [13] | Tripathi P, Beaussart A, Andre G, et al. Towards a nanoscale view of lactic acid bacteria[J]. Micron, 2012, 43(12): 1323-1330 |
| [14] | Huang SL, Xing HX, Gong H, et al. Immune stimulation and influencing factors of prebiotic peptidoglycan[J]. Chinese Journal of Microecology, 2014, 26(5): 604-605, 608 (in Chinese) 黄少磊, 邢会霞, 龚虹, 等. 益生菌肽聚糖对机体的免疫刺激作用及其影响因素[J]. 中国微生态学杂志, 2014, 26(5): 604-605, 608 |
| [15] | Cario E, Gerken G, Podolsky DK. Toll-like receptor 2 controls mucosal inflammation by regulating epithelial barrier function[J]. Gastroenterology, 2007, 132(4): 1359-1374 |
| [16] | Kobierecka P, Wyszyńska A, Maruszewska M, et al. Lactic acid bacteria as a surface display platform for Campylobacter jejuni antigens[J]. Journal of Molecular Microbiology and Biotechnology, 2015, 25(1): 1-10 |
| [17] | Vollmer W, Blanot D, de Pedro MA. Peptidoglycan structure and architecture[J]. FEMS Microbiology Reviews, 2008, 32(2): 149-167 |
| [18] | Inamura S, Fukase K, Kusumoto S. Synthetic study of peptidoglycan partial structures. Synthesis of tetrasaccharide and octasaccharide fragments[J]. Tetrahedron Letters, 2001, 42(43): 7613-7616 |
| [19] | Chapot-Chartier MP, Kulakauskas S. Cell wall structure and function in lactic acid bacteria[J]. Microbial Cell Factories, 2014, 13(Suppl 1): S9 |
| [20] | Bernard E, Rolain T, Courtin P, et al. Characterization of O-Acetylation of N-Acetylglucosamine: a novel structural variation of bacterial peptidoglycan[J]. Journal of Biological Chemistry, 2011, 286(27): 23950-23958 |
| [21] | Šimelyte E, Rimpiläinen M, Lehtonen L, et al. Bacterial cell wall-induced arthritis: chemical composition and tissue distribution of four Lactobacillus strains[J]. Infection and Immunity, 2000, 68(6): 3535-3540 |
| [22] | Courtin P, Miranda G, Guillot A, et al. Peptidoglycan structure analysis of Lactococcus lactis reveals the presence of an L, D-carboxypeptidase involved in peptidoglycan maturation[J]. Journal of Bacteriology, 2006, 188(14): 5293-5298 |
| [23] | Regulski K, Courtin P, Meyrand M, et al. Analysis of the peptidoglycan hydrolase complement of Lactobacillus casei and characterization of the major γ-D-Glutamyl-L-Lysyl-Endopeptidase[J]. PLoS One, 2012, 7(2): e32301 |
| [24] | Claes IJJ, Schoofs G, Regulski K, et al. Genetic and biochemical characterization of the cell wall hydrolase activity of the major secreted protein of Lactobacillus rhamnosus GG[J]. PLoS One, 2012, 7(2): e31588 |
| [25] | Bouhss A, Josseaume N, Severin A, et al. Synthesis of the L-alanyl-L-alanine cross-bridge of Enterococcus faecalis peptidoglycan[J]. Journal of Biological Chemistry, 2002, 277(48): 45935-45941 |
| [26] | Typas A, Banzhaf M, Gross CA, et al. From the regulation of peptidoglycan synthesis to bacterial growth and morphology[J]. Nature Reviews Microbiology, 2011, 10(2): 123-136 |
| [27] | Wheeler R, Mesnage S, Boneca IG, et al. Super-resolution microscopy reveals cell wall dynamics and peptidoglycan architecture in ovococcal bacteria[J]. Molecular Microbiology, 2011, 82(5): 1096-1109 |
| [28] | Andre G, Kulakauskas S, Chapot-Chartier MP, et al. Imaging the nanoscale organization of peptidoglycan in living Lactococcus lactis cells[J]. Nature Communications, 2010, 1: 27 |
| [29] | Barreteau H, Kovač A, Boniface A, et al. Cytoplasmic steps of peptidoglycan biosynthesis[J]. FEMS Microbiology Reviews, 2008, 32(2): 168-207 |
| [30] | Deghorain M, Goffin P, Fontaine L, et al. Selectivity for D-lactate incorporation into the peptidoglycan precursors of Lactobacillus plantarum: role of Aad, a VanX-like D-alanyl-D-alanine dipeptidase[J]. Journal of Bacteriology, 2007, 189(11): 4332-4337 |
| [31] | Deghorain M, Fontaine L, David B, et al. Functional and morphological adaptation to peptidoglycan precursor alteration in Lactococcus lactis[J]. Journal of Biological Chemistry, 2010, 285(31): 24003-24013 |
| [32] | Bouhss A, Trunkfield AE, Bugg TDH, et al. The biosynthesis of peptidoglycan lipid-linked intermediates[J]. FEMS Microbiology Reviews, 2008, 32(2): 208-233 |
| [33] | van Heijenoort J. Lipid intermediates in the biosynthesis of bacterial peptidoglycan[J]. Microbiology and Molecular Biology Reviews, 2007, 71(4): 620-635 |
| [34] | Veiga P, Piquet S, Maisons A, et al. Identification of an essential gene responsible for D-Asp incorporation in the Lactococcus lactis peptidoglycan crossbridge[J]. Molecular Microbiology, 2006, 62(6): 1713-1724 |
| [35] | Mohammadi T, van Dam V, Sijbrandi R, et al. Identification of FtsW as a transporter of lipid-linked cell wall precursors across the membrane[J]. The EMBO Journal, 2011, 30(8): 1425-1432 |
| [36] | Sham LT, Butler EK, Lebar MD, et al. MurJ is the flippase of lipid-linked precursors for peptidoglycan biogenesis[J]. Science, 2014, 345(6193): 220-222 |
| [37] | Sauvage E, Kerff F, Terrak M, et al. The penicillin-binding proteins: structure and role in peptidoglycan biosynthesis[J]. FEMS Microbiology Reviews, 2008, 32(2): 234-258 |
| [38] | David B. Role of PBPs in the ovoid Lactococcus lactis cell cycle[D]. London: UCL, 2013 |
| [39] | Vollmer W. Structural variation in the glycan strands of bacterial peptidoglycan[J]. FEMS Microbiology Reviews, 2008, 32(2): 287-306 |
| [40] | Bera A, Herbert S, Jakob A, et al. Why are pathogenic staphylococci so lysozyme resistant? The peptidoglycan O-acetyltransferase OatA is the major determinant for lysozyme resistance of Staphylococcus aureus[J]. Molecular Microbiology, 2005, 55(3): 778-787 |
| [41] | Bernard E, Rolain T, David B, et al. Dual role for the O-acetyltransferase OatA in peptidoglycan modification and control of cell septation in Lactobacillus plantarum[J]. PLoS One, 2012, 7(10): e47893 |
| [42] | Moynihan PJ, Clarke AJ. Assay for peptidoglycan O-acetyltransferase: A potential new antibacterial target[J]. Analytical Biochemistry, 2013, 439(2): 73-79 |
| [43] | Veiga P, Bulbarela-Sampieri C, Furlan S, et al. SpxB regulates O-acetylation-dependent resistance of Lactococcus lactis peptidoglycan to hydrolysis[J]. Journal of Biological Chemistry, 2007, 282(27): 19342-19354 |
| [44] | Vollmer W, Tomasz A. The pgdA gene encodes for a peptidoglycan N-acetylglucosamine deacetylase in Streptococcus pneumoniae[J]. Journal of Biological Chemistry, 2000, 275(27): 20496-20501 |
| [45] | Meyrand M, Boughammoura A, Courtin P, et al. Peptidoglycan N-acetylglucosamine deacetylation decreases autolysis in Lactococcus lactis[J]. Microbiology, 2007, 153(10): 3275-3285 |
| [46] | Kobayashi K, Sudiarta IP, Kodama T, et al. Identification and characterization of a novel polysaccharide deacetylase C (PdaC) from Bacillus subtilis[J]. Journal of Biological Chemistry, 2012, 287(13): 9765-9776 |
| [47] | Veiga P, Erkelenz M, Bernard E, et al. Identification of the asparagine synthase responsible for D-Asp amidation in the Lactococcus lactis peptidoglycan interpeptide crossbridge[J]. Journal of Bacteriology, 2009, 191(11): 3752-3757 |
| [48] | Figueiredo TA, Sobral RG, Ludovice AM, et al. Identification of genetic determinants and enzymes involved with the amidation of glutamic acid residues in the peptidoglycan of Staphylococcus aureus[J]. PLoS Pathogens, 2012, 8(1): e1002508 |
| [49] | Münch D, Roemer T, Lee SH, et al. Identification and in vitro analysis of the GatD/MurT enzyme-complex catalyzing lipid Ⅱ amidation in Staphylococcus aureus[J]. PLoS Pathogens, 2012, 8(1): e1002509 |
| [50] | Vollmer W, Joris B, Charlier P, et al. Bacterial peptidoglycan (murein) hydrolases[J]. FEMS Microbiology Reviews, 2008, 32(2): 259-286 |
| [51] | Huard C, Miranda G, Redko Y, et al. Analysis of the peptidoglycan hydrolase complement of Lactococcus lactis: identification of a third N-acetylglucosaminidase, AcmC[J]. Applied and Environmental Microbiology, 2004, 70(6): 3493-3499 |
| [52] | Steen A, Buist G, Horsburgh GJ, et al. AcmA of Lactococcus lactis is an N-acetylglucosaminidase with an optimal number of LysM domains for proper functioning[J]. FEBS Journal, 2005, 272(11): 2854-2868 |
| [53] | Huard C, Miranda G, Wessner F, et al. Characterization of AcmB, an N-acetylglucosaminidase autolysin from Lactococcus lactis[J]. Microbiology, 2003, 149(3): 695-705 |
| [54] | Redko Y, Courtin P, Mézange C, et al. Lactococcus lactis gene yjgB encodes a γ-D-glutaminyl-L-lysyl-endopeptidase which hydrolyzes peptidoglycan[J]. Applied and Environmental Microbiology, 2007, 73(18): 5825-5831 |
| [55] | Layec S, Decaris B, Leblond-Bourget N. Diversity of Firmicutes peptidoglycan hydrolases and specificities of those involved in daughter cell separation[J]. Research in Microbiology, 2008, 159(7/8): 507-515 |
| [56] | Visweswaran GRR, Steen A, Leenhouts K, et al. AcmD, a homolog of the major autolysin AcmA of Lactococcus lactis, binds to the cell wall and contributes to cell separation and autolysis[J]. PLoS One, 2013, 8(8): e72167 |
| [57] | Rolain T, Bernard E, Courtin P, et al. Identification of key peptidoglycan hydrolases for morphogenesis, autolysis, and peptidoglycan composition of Lactobacillus plantarum WCFS1[J]. Microbial Cell Factories, 2012, 11(1): 137 |
| [58] | Buist G, Venema G, Kok J. Autolysis of Lactococcus lactis is influenced by proteolysis[J]. Journal of Bacteriology, 1998, 180(22): 5947-5953 |
| [59] | Poquet I, Saint V, Seznec E, et al. HtrA is the unique surface housekeeping protease in Lactococcus lactis and is required for natural protein processing[J]. Molecular Microbiology, 2000, 35(5): 1042-1051 |
| [60] | Steen A, Buist G, Leenhouts KJ, et al. Cell wall attachment of a widely distributed peptidoglycan binding domain is hindered by cell wall constituents[J]. Journal of Biological Chemistry, 2003, 278(26): 23874-23881 |
| [61] | Hanson BR, Neely MN. Coordinate regulation of Gram-positive cell surface components[J]. Current Opinion in Microbiology, 2012, 15(2): 204-210 |
| [62] | Palumbo E, Deghorain M, Cocconcelli PS, et al. D-Alanyl ester depletion of teichoic acids in Lactobacillus plantarum results in a major modification of lipoteichoic acid composition and cell wall perforations at the septum mediated by the Acm2 autolysin[J]. Journal of Bacteriology, 2006, 188(10): 3709-3715 |
| [63] | Rolain T, Bernard E, Beaussart A, et al. O-glycosylation as a novel control mechanism of peptidoglycan hydrolase activity[J]. Journal of Biological Chemistry, 2013, 288(31): 22233-22247 |
| [64] | Dubrac S, Boneca IG, Poupel O, et al. New insights into the WalK/WalR (YycG/YycF) essential signal transduction pathway reveal a major role in controlling cell wall metabolism and biofilm formation in Staphylococcus aureus[J]. Journal of Bacteriology, 2007, 189(22): 8257-8269 |
| [65] | Smith TJ, Blackman SA, Foster SJ. Autolysins of Bacillus subtilis: multiple enzymes with multiple functions[J]. Microbiology, 2000, 146(Pt 2): 249-262 |
| [66] | Frkanec R, Tomašić J. High performance liquid chromatography in analysis of compounds comprising the elements of bacterial peptidoglycan structure with immunological activity[J]. Journal of Liquid Chromatography & Related Technologies, 2007, 31(1): 107-133 |
| [67] | Jin PP, Deng YJ. Research progress on Lactobacillus peptidoglycan immune function[J]. Chinese Journal of Microecology, 2013, 25(4): 485-487 (in Chinese) 金盼盼, 邓燕杰. 乳酸杆菌肽聚糖免疫作用的研究进展[J]. 中国微生态学杂志, 2013, 25(4): 485-487 |
| [68] | Christensen HR, Frøkiær H, Pestka JJ. Lactobacilli differentially modulate expression of cytokines and maturation surface markers in murine dendritic cells[J]. The Journal of Immunology, 2002, 168(1): 171-178 |
| [69] | Wells JM. Immunomodulatory mechanisms of lactobacilli[J]. Microbial Cell Factories, 2011, 10(Suppl 1): S17 |
| [70] | Yao GG, Yao W, Lu Y, et al. Partial immunoactivies of peptidoglycan from lactic acid bacteria[J]. Microbiology China, 2007, 34(1): 105-107 (in Chinese) 姚光国, 姚文, 陆扬, 等. 乳酸菌肽聚糖部分免疫增强作用的研究[J]. 微生物学通报, 2007, 34(1): 105-107 |
| [71] | Ma DX, Meng XC, Gao XJ, et al. Effect of lactobacilli on production of Th1/Th2 cytokine and antibody in lymphocyte of bovine β-lactoglobulin-sensitized mice in vitro[J]. Microbiology China, 2011, 38(7): 1063-1069 (in Chinese) 马冬雪, 孟祥晨, 高学军, 等. 乳杆菌对牛乳β-乳球蛋白致敏小鼠淋巴细胞分泌Th1/Th2型细胞因子和抗体的体外影响 [J]. 微生物学通报, 2011, 38(7): 1063-1069 |
| [72] | Li D, Li AL, Ji XM, et al. Effect of whole peptidoglycan from lactobacilli on the imbalance of Th1/Th2 and Treg/Th17 in lymphocyte of bovine β-Lactoglobulin-sensitized mice in vitro[J]. Microbiology China, 2014, 41(7): 1334-1341 (in Chinese) 李丹, 李艾黎, 季晓梅, 等. 乳杆菌肽聚糖对β-乳球蛋白致敏小鼠脾细胞Th1/Th2及Treg/Th17失衡的体外影响[J]. 微生物学通报, 2014, 41(7): 1334-1341 |
 2016, Vol. 43
2016, Vol. 43




