扩展功能
文章信息
- 周毅, 文斌, 孙豪, 段玲, 傅祥超, 曾东, 倪学勤, 汪平
- ZHOU Yi, WEN Bi, SUN Hao, 段 玲, 傅 祥超, ZENG Dong, NI Xue-Qin, WANG Ping
- 应用PCR-DGGE 和Real-Time PCR 分析健康与腹泻獭兔盲肠菌群
- Application of PCR-DGGE and Real-Time PCR to analyze the cecum microflora of health and diarrhea rex rabbit
- 微生物学通报, 2016, 43(1): 131-139
- Microbiology China, 2016, 43(1): 131-139
- 10.13344/j.microbiol.china.150276
-
文章历史
- 收稿日期: 2015-04-02
- 接受日期: 2015-06-25
- 优先数字出版日期(www.cnki.net): 2015-07-03
2. 四川省草原科学研究院 四川 成都 611731
3. 西南民族大学生命科学与技术学院 四川 成都 610041
2. Sichuan Academy of Grassland Science, Chengdu, Sichuan 611731, China
3. College of Life Science &Technology, Southwest University for Nationalities, Chengdu, Sichuan 610041,China
盲肠在兔消化吸收中具有十分重要的作用,主要原因是有数量巨大、结构复杂的微生物在其中定殖。成年兔每克盲肠内容物中含有109−1010个拟杆菌属细菌[1],而双歧杆菌属、梭菌属、链球菌属、肠杆菌属等的总量为1010−1012 CFU/g[2]。这些微生物通过分泌消化酶或者微生物自身代谢活动代谢尿素、氨、肽类及分解纤维素,并能够产生各种挥发性脂肪酸、氨基酸、维生素等[3];在提高免疫[4]、调整肠道微生态[5]等方面具有十分重要的作用。由于兔特殊的消化道生理结构[6],在断奶后极易发生腹泻,而且一旦腹泻往往会导致死亡,给兔养殖业带来极大损失。腹泻与兔肠道菌群关系密切[7],但是,已有的研究主要是采用活菌计数的方法分析部分细菌数量的差异,具有一定的局限,不能较全面地反映整个菌群结构的变化,而且活菌计数对严格 厌氧的细菌不能培养,检测结果有一定的误差。本研究拟采集腹泻兔和健康兔盲肠内容物,采用现代分子技术,在DNA水平上比较和分析盲肠细菌数量和结构的差异,全面反映腹泻发生后肠道细菌群落的变化情况,为微生态防治断奶兔腹泻提供理论依据。
1 材料与方法 1.1 材料 1.1.1 样品采集和处理:从四川省草原科学研究院獭兔原种场,采集同一批次1.5月龄的健康獭兔5只,临床表现基本一致的腹泻獭兔5只,处死后连同肠内容物结扎盲肠肠段2 cm,迅速置于液氮中保存,健康、腹泻獭兔分别对应编号为HR (Health rabbit,HR) 1−5、DR (Diarrhea rabbit,DR) 1−5。 1.1.2 主要试剂:DGGE成套试剂,Bio-Rad公司;银染药品等相关试剂均为分析纯;2X Taq MasterMix (含染料),康为世纪生物科技有限公司;SYBR Premix ExTaqⅡ,宝生物工程(大连)有限公司;OMEGA粪样提取试剂盒[OMEGA® StoolDNA Kit (50)],成都昊优生物科技有限责任公司;PCR引物由英潍捷基(上海)贸易有限公司合成。 1.2 盲肠总DNA提取使用OMEGA粪样提取试剂盒[OMEGA® Stool DNA Kit (50)],按照操作手册分别提取各肠道内容物总DNA,用核酸浓度测定仪(NanoDrop®ND-1000 UV-Vis Spectrophotometer)测定浓度(OD260/OD280=1.8−2.2),置−30 °C备用。
1.3 16SrRNA基因V3区PCR扩增使用提取的DNA扩增细菌16S rRNA基因的V3区(339−539 bp),引物序列:上游引物带GC发夹:5′-CGCCCGGGGCGCGCCCCGGGCGGGGCG GGGGCACGGGGGGACTCCTACGGGAGGCAGCAGT-3′;下游引物为:5′-GTATTACCGCGGCTGGC AC-3′[8]。反应体系(25 μL):2×TaqMasterMix (CWBIO) 12.5 μL,上下游引物(10 μmol/L)各1 μL,模板DNA 1 μL,最后用ddH2O补足25 μL,设一个阴性对照组。反应条件:94 °C 4 min;94 °C 30 s,58 °C 30 s,72 °C 2 min,30个循环;72 °C 10 min。用1.0%琼脂糖凝胶电泳检测PCR产物片段大小和浓度。
1.4 PCR-DGGE凝胶电泳PCR-DGGE采用Bio-Rad® DcodeUniversal Detection System进行,凝胶浓度梯度为35%−65%,变性方向与电泳方向一致。电泳缓冲液为1×TAE,100 V、60 °C条件下电泳16 h。采用硝酸银染色后用Bio-Rad®GS800 Calibrated Densitometer对凝胶进行成像处理。
1.5 荧光定量Real-Time PCRReal-TimePCR (RT-PCR)采用Bio-Rad® CFX 96荧光定量PCR仪进行,各菌引物及标准曲线见表 1。反应体系(25 μL):SYBR Premix ExTaq Ⅱ 12.5 μL,上下游引物(10 μmol/L)各1 μL,模板1 μL,ddH2O 9.5 μL。反应条件:94 °C 4 min;94 °C 30 s,引物最佳退火温度(表 1) 30 s,72 °C 30 s,40个循环;72 °C 1 min;95 °C 1 min,50 °C 1 min,60 °C 5 s,95 °C。在退火温度结束后照相采集荧光值,溶解曲线每隔0.5 °C照相采集一次荧光值。结束后数据导出到Excel表格中整理分析,参照标准曲线计算出各菌绝对含量。
| 细菌种属 Bacterial species | 引物序列 Primer sequence (5′→3′) | 回归方程/退火温度 Regression curve/Tm | 参考文献 reference |
| 总细菌(130 bp) Total bacteria | F-CGGYCCAGACTCCTACGGG R-TTACCGCGGCTGCTGGCAC | Y=14.354−0.260 7X R2=0.997 Tm=60 °C | [9] |
| 普雷沃氏菌(151 bp) Prevotella | F-GGTGTCGGCTTAAGTGCCAT R-CGGAYGTAAGGGCCGTGC | Y=14.39−0.280 7X R2=0.998 Tm=55 °C | [9] |
| 溶纤维丁酸弧菌(158 bp) Butyrivibrio fibrisolvens | F-GTTCGGCTGGGCACTCT R-CACCGCGACATTCTGATTC | Y=−0.290 7X+14.290 R2=0.997 Tm=60 °C | [10] |
| 肠杆菌科(230 bp) Enterobacteriaceae family | F-CATTGACGTTACCCGCAGAAGAAGC R-CTCTACGAGACTCAAGCTTGC | Y=13.87−0.192 7X R2=0.995 Tm=52 °C | [10] |
| 肠球菌属(144 bp) Enterococcus spp. | F-CCCTTATTGTTAGTTGCCATCATT R-ACTCGTTGTACTTCCCATTGT | Y=−0.254 2X+14.356 R2=0.999 Tm=52 °C | [7] |
| 双歧杆菌属(243 bp) Bifidobacterium spp. | F-TCGCGTCYGGTGTGAAAG R-CCACATCCAGCRTCCAC | Y=13.837−0.204 5X R2=0.998 Tm=62 °C | [9] |
| 梭菌类群I (120 bp) Clostridium cluster I | F-ATGCAAGTCGAGCGAKG R-TATGCGGTATTAATCTYCCTTT | Y=13.376−0.226 6X R2=0.998 Tm=60 °C | [9] |
| 梭菌类群IV (230 bp) Clostridium leptum group | F-GCACAAGCAGTGGAGT R-CTTCCTCCGTTTTGTCAA | Y=14.288−0.247 2X R2=0.992 Tm=60 °C | [11] |
| 梭菌类群XIVa (440 bp) Clostridium cluster XIVa | F-AAATGACGGTACCTGACTAA R-CTTTGAGTTTCATTCTTGCGAA | Y=14.748−0.261 5X R2=0.997 Tm=60°C | [11] |
| 黄色瘤胃球菌(295 bp) Ruminococcus lavefaciens | F-GGACGATAATGACGGTACTT R-GCAATCYGAACTGGGACAAT | Y=14.211−0.273 9X R2=0.998 Tm=56 °C | [12] |
| 白色瘤胃球菌(175 bp) Ruminococcus albus | F-CCCTAAAAGCAGTCTTAGTTCG R-CCTCCTTGCGGTTAGAACA | Y=14.164−0.269 1X R2=0.998 Tm=52 °C | [13] |
| 普拉氏梭杆菌(158 bp) Fusobacterium rausnitzii | F-CCCTTCAGTGCCGCAGT R-GTCGCAGGATGTCAAGAC | Y=−0.300 4X+14.394 R2=0.995 Tm=60 °C | [9] |
| 沙门氏菌(160 bp) Salmonella species | F-TCGTCATTCCATTACCTACC R-AAACGTTGAAAAACTGAGGA | Tm=59 °C | [14] |
| 埃希氏大肠杆菌(113 bp) Escherichia coli | F-CATGCCGCGTGTATGAAGAA R-CGGGTAACGTCAATGAGCAAA | Tm=59 °C | [15] |
采用Quantity one对PCR-DGGE图谱进行处理后导出数据,使用Excel表格进行整理,NTSYS 2.1软件采用UPGMA法进行聚类分析;采用SPSS 19.0进行独立样本t检验进行显著性分析;应用GraphPadPrism 6作图。
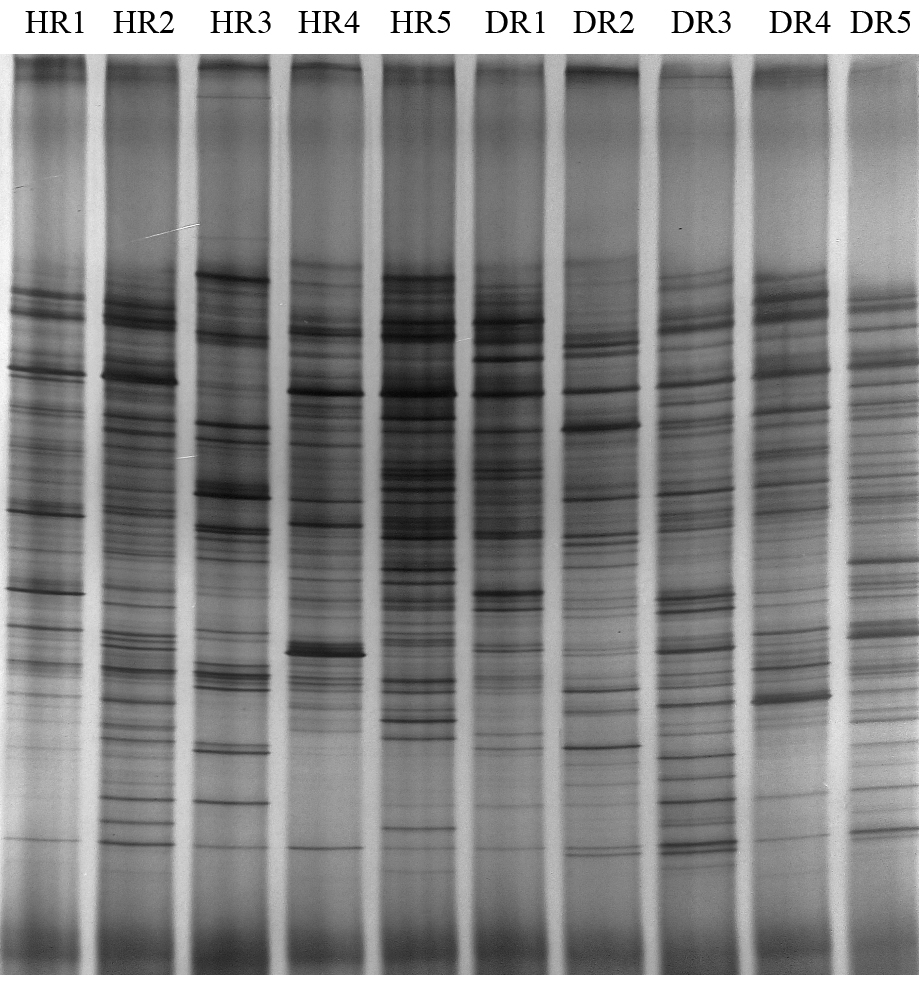
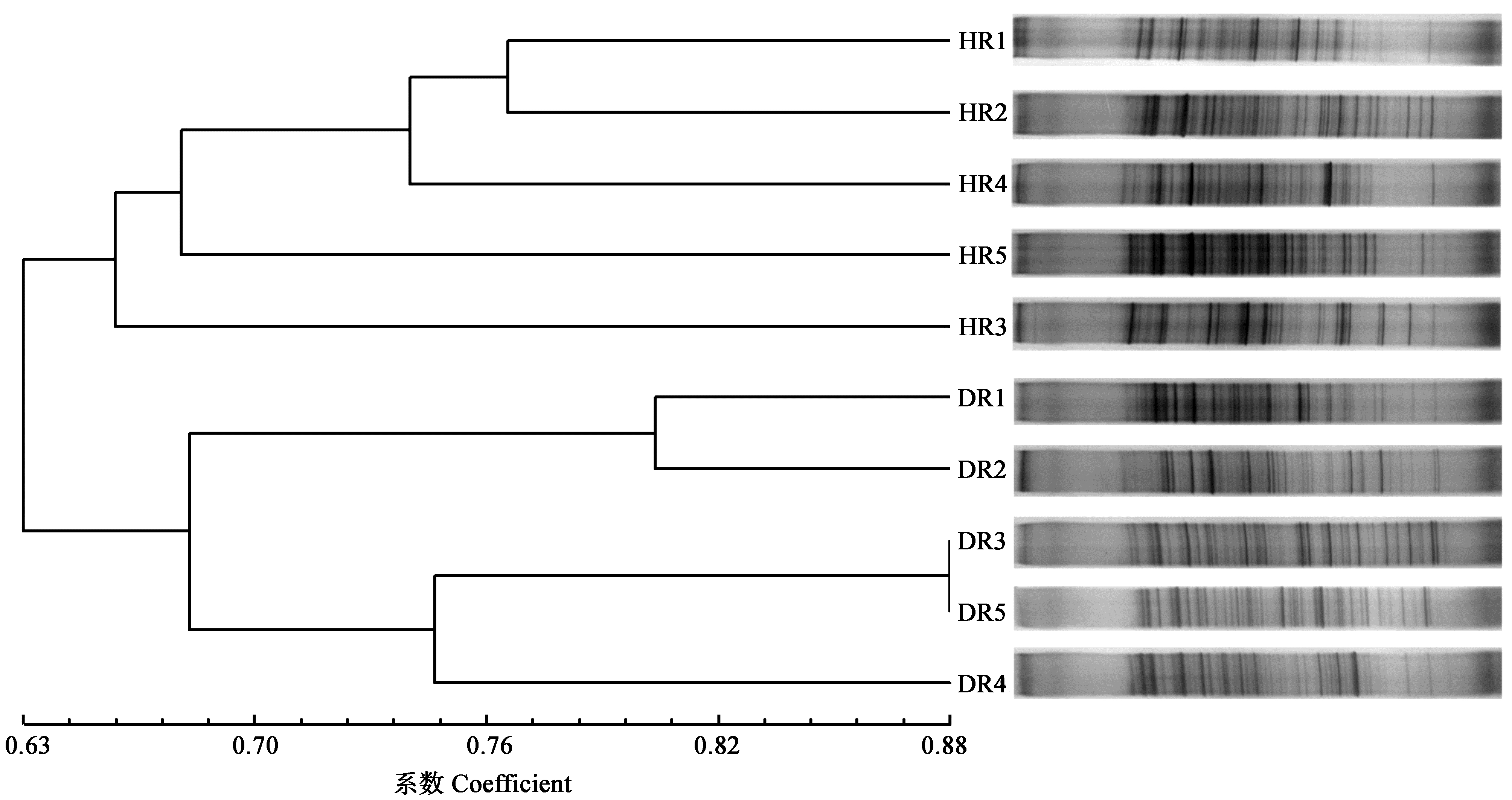
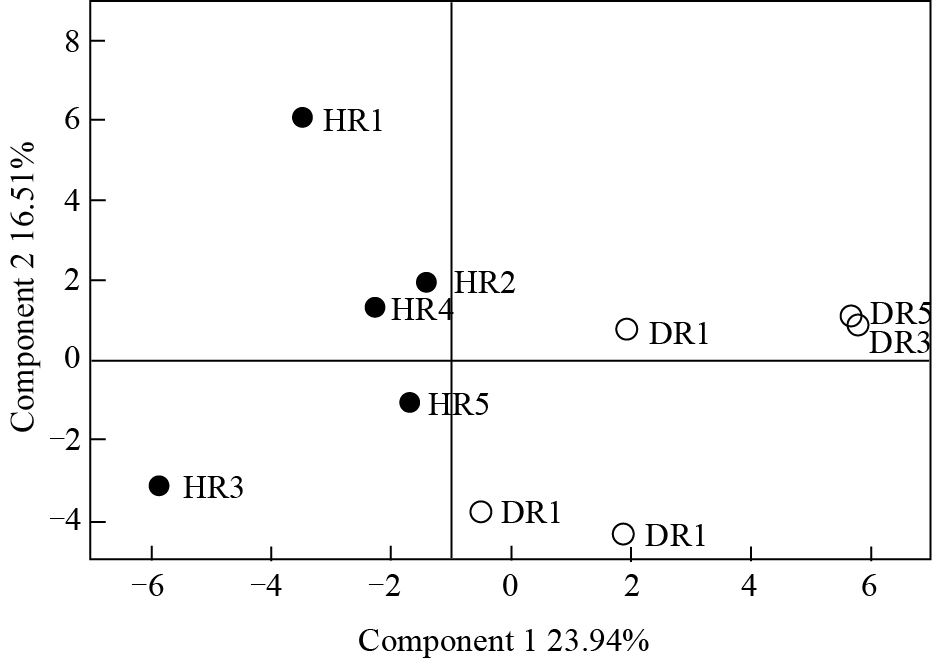
2 结果与分析 2.1 PCR-DGGE结果在PCR-DGGE图谱(图 1)中,条带颜色的深浅与该菌在样本中含量成正比。从图 1中可以看出,无论是腹泻獭兔还是健康獭兔都产生了丰富的条带,健康和腹泻獭兔肠道细菌丰富度分别达到47.40和49.60,深色条带为该样本中的优势菌群。PCR-DGGE条带UPGMA法聚类分析(图 2)显示,健康和腹泻獭兔组盲肠样品被明显区分为两组,两组样品之间的相似性为0.63,说明獭兔发生腹泻后微生物菌群结构部分发生变化。但分析各健康和腹泻獭兔肠道细菌DGGE电泳图谱多样性指数、均匀度、丰富度(表 2),两组之间差异均不显著(P>0.05)。说明腹泻时獭兔盲肠微生物结构发生变化,但是变化不显著。DGGE条带主成分分析(图 3)与聚类分析的结果相同,PC1 (23.94%)将健康和腹泻獭兔区分为左右两部分。因此,腹泻发生后肠道微生物结构部分发生改变,但PCR-DGGE图谱的条带差异不显著。
|
|
图 1
健康和腹泻獭兔PCR-DGGE图谱
Figure 1
PCR-DGGE profile of health & diarrhea rex rabbit
注:HR 1−5、DR 1−5分别表示健康獭兔、腹泻獭兔. Note: HR 1−5 (Health rabbit 1−5) & DR 1−5 (Diarrhea rabbit 1−5)represented health rabbit & diarrhea rabbit respectively. |
|
|
图 2
健康与腹泻獭兔肠道菌群PCR-DGGE条带聚类分析图
Figure 2
Cluster analysis figurefor PCR-DGGE bands of cecum floraof health & diarrhea rex rabbite
注:聚类分析图谱由NTSYS2.1生成,数据分析采用DICE系数,聚类分析使用UPGMA法;HR 1−5和DR 1−5分别表示健康獭兔1−5、腹泻獭兔1−5. Note:Clustering analysis graph generated by NTSYS2.1, DICE coefficient was used toanalysis the data, the clustering analysis by UPGMA; HR 1−5 & DR 1−5 represented health rabbit 1−5 & diarrhea rabbit 1−5 respectively. |
|
| 图 3 健康与腹泻獭兔肠道菌群PCR-DGGE条带主成分分析结果 Figure 3 The PCA result ofPCR-DGGE analysis in health & diarrhea rex rabbit cecum microbial flora |
| 分组 Group | 多样性指数 Diversity Index | 均匀度 Evenness | 丰富度 Richness |
| 腹泻獭兔 Diarrhea Rabbit | 3.90±0.04 | 0.90±0.01 | 49.60±1.81 |
| 健康獭兔 Health Rabbit | 3.85±0.12 | 0.89±0.03 | 47.40±5.60 |
Note: Independent sample t test wasused for analysis, P=0.05, n=5; Data using mean ± standard deviation, there is nosignificant difference diarrhea compared with the healthy group(P>0.05).
独立样本t检验(表 3)结果可以看出,与健康獭兔比较,腹泻獭兔盲肠微生物总量明显减少(P<0.01),肠杆菌科(P<0.01)、肠球菌属(P<0.01)、白色瘤胃球菌(P<0.01)、梭菌类群ⅩⅣa (P<0.01)、梭菌类群Ⅳ (P<0.01)、双歧杆菌属(P<0.01)、溶纤维丁酸弧菌(P<0.01)、普拉梭杆菌(P<0.01)均显著降低,普雷沃氏菌(P>0.05)、梭菌类群Ⅰ(P>0.05)这2种菌数量变化不明显。黄色瘤胃球菌和沙门氏菌在健康和腹泻獭兔中均未检出。在分析各菌群在总菌中的比例时,腹泻组肠杆菌科比例显著升高(P<0.05),进一步检测埃希氏大肠杆菌数量,腹泻獭兔Cq值显著小于健康獭兔(腹泻组VS健康组= 22.05±1.21 VS26.71±3.16,P<0.05),其他细菌变化不明显。主成分分析(图 4)显示,PC1能够解释RT-PCR中所有数据的74.76%,并且将腹泻和健康组分为左右部分,说明两者之间差异明显。可见,腹泻造成獭兔盲肠细菌数量急剧降低,但主要菌群比例变化不显著,与DGGE条带多样性分析结果一致。
| 细菌种属 Bacterial species | 健康獭兔 Health rabbit (logcopy/g) | 腹泻獭兔 Diarrhea rabbit (logcopy/g) | 细菌种属 Bacterial species | 健康獭兔 Health rabbit (logcopy/g) | 腹泻獭兔 Diarrhea rabbit (logcopy/g) |
| 总菌 Total bacteria | 11.73±0.32 | 10.95±0.13** | 白色瘤胃球菌 Ruminococcus albus | 8.78±0.21 | 7.74±0.54** |
| 普雷沃氏菌 Prevotella | 10.04±0.67 | 9.42±0.24 | 溶纤维丁酸弧菌 Butyrivibrio fibrisolvens | 8.33±0.27 | 7.30±0.33** |
| 肠杆菌科 Enterobacteriacae family | 10.26±0.15 | 9.79±0.06** | 普拉氏梭杆菌 Fusobacterium rausnitzii | 11.15±0.29 | 10.48±0.07** |
| 肠球菌属 Enterococcus spp. | 7.29±0.41 | 5.66±0.58** | 梭菌类群Ⅰ Clostridium cluster Ⅰ | 6.22±0.21 | 5.60±1.00 |
| 双歧杆菌属 Bifidobacterium spp. | 7.80±0.06 | 6.98±0.20** | 梭菌ⅩⅣa Clostridium cluster ⅩⅣa | 10.67±0.28 | 10.02±0.03** |
| 梭菌类群Ⅳ Clostridium leptum group | 10.97±0.38 | 10.30±0.23** |
Note:Independent sample t test was used for analysis, P=0.05, n=5.Significant difference for the diarrhea compared with health group, *:significant differences (P<0.05),**: significantdifferences (P<0.01).

|
| 图 4 健康与腹泻肠道菌群Real-Time PCR主成分分析结果 Figure 4 The PCA result of Real-Time PCR on health & diarrhea rex rabbit cecum microbialflor |
肠道微生物在提供营养物质、维生素,抵抗病原菌入侵,与肠道上皮细胞交互增强宿主免疫力等方面起着十分重要的作用[10],研究发现采食无菌饲料的兔子其肠粘膜淋巴细胞密度降低,派氏结变小,免疫球蛋白数量降低[11]。然而在动物饲料中添加适量的益生菌则可以提高动物生长性能[12]、增强动物免疫力[4]、调整宿主肠道微生态平衡[5],因此肠道微生物在维持宿主健康状态方面发挥着十分重要的作用。
通过分析PCR-DGGE图谱发现,腹泻獭兔和健康獭兔肠道菌群相似性为0.63,腹泻獭兔盲肠中菌群结构发生一定改变,但是这种结构的改变并没有在多样性指数等方面表现出显著的差异(P>0.05),这与苏勇等[13]报道的健康与腹泻仔猪肠菌群多样性差异不显著一致。而腹泻獭兔盲肠菌群PCR-DGGE条带丰富度在数量上略高于健康獭兔,这和通常认为的腹泻发生后肠道微生物结构趋于简单的结论不符[7, 14]。从PCR-DGGE条带聚类分析和主成分分析结果来看,均可以清晰的将健康和腹泻獭兔区分开,说明腹泻和健康獭兔盲肠微生物的差异是存在的,可能是由于腹泻发生时部分原来的优势菌群数量下降至PCR-DGGE不能检测水平,而原来的PCR-DGGE未能检测的细菌数量则上升,这种此消彼长最终导致PCR-DGGE图谱的多样性检测结果并没有显著差异。Takahashi等[15]在给仔猪饲喂乳酸杆菌后,采用RAPD-PCR及16S rRNA基因测序技术也发现在对照组中未检测到的其他乳酸杆菌在试验组中则被检测到。
RT-PCR分析显示,腹泻獭兔盲肠中普雷沃氏菌和梭菌类群Ⅰ等[16]主要微生物数量并没有发生明显变化(P>0.05),这也说明肠道微生态系统在一定程度上具有良好的稳定性,这种稳定性是肠道微生态发挥其生理功能的重要前提[17]。肠球菌属作为肠道内正常菌群的重要组成部分,其大多为需氧菌[18],在肠道中占据独特的生态位点,通过消耗氧气控制肠道中需氧菌的数量,与其他菌群协同作用保持肠道健康[19],它们数量的减少(P<0.01)会引起肠道菌群结构紊乱。梭菌类群Ⅳ和ⅩⅣa、白色瘤胃球菌、溶纤维丁酸弧菌、普拉梭杆菌等这些肠道主要微生物的数量均明显减少(P<0.01),这些微生物在盲肠中参与纤维素代谢为宿主提供能量,产生的挥发性脂肪酸不仅能够抑制有害细菌的繁殖,还能够预防和治疗肠道炎症。在人肠道中产丁酸的微生物多为梭菌类群Ⅳ和ⅩⅣa,丁酸在促进肠道黏膜修复和防治腹泻方面有着很好的效果[20],而据报道,梭菌属的变化与肠道黏膜的完整性也密切相关,拟杆菌中的一些细菌则与肠道上皮细胞作用,通过NF-κB途径促进促炎症因子的表达[21]。可见在獭兔腹泻发生时,这些与獭兔盲肠消化代谢能力、肠黏膜修复能力、肠道免疫力密切相关的微生物数量降低(P<0.01),而这些有益微生物数量的降低进一步加重獭兔腹泻发生,最终引起獭兔机体损伤。双歧杆菌作为肠道中重要的益生菌,甚至可以作为肠道健康的指示菌[22]。不仅可以代谢产生无机酸、参与维生素代谢过程,还能够通过与肠道相关淋巴组织(GALT)作用,激活细胞增殖分化、激活巨噬细胞、分泌IgA,在发挥肠道菌群的营养和免疫方面起着十分重要的作用[23]。在腹泻发生后,獭兔盲肠内双歧杆菌数量出现明显降低(P<0.01),这对于獭兔缓解腹泻症状、恢复健康状态极为不利。由此可见,腹泻发生时肠道内细菌数量发生巨大变化,从较高分类水平分析腹泻獭兔盲肠细菌数量均为降低,但分析肠杆菌科在总菌中的比例却发现腹泻组肠杆菌科占比显著增加(P<0.05),进一步降低分类水平检测埃希氏大肠杆菌,却发现腹泻组Cq值小于健康组(P<0.05),即腹泻组中大肠杆菌数量上升,这与采用常规活菌技术检测腹泻动物大肠杆菌数量上升相吻合[24]。说明腹泻发生后肠道菌群结构发生变化,主要体现在某些肠道细菌数量上的变化即有益微生物数量下降、致病性微生物数量上升,具体发生变化的微生物种类还需要进一步分析。
本试验综合运用PCR-DGGE和Real-Time PCR方法,对健康和腹泻獭兔盲肠微生物进行分析,发现獭兔发生腹泻后盲肠菌群有益菌数量减少,有害细菌数量增加;肠道菌群结构在一定程度上有复杂化的趋势。
| [1] | Gouet P, Fonty G. Evolution de la microflore digestive du lapin holoxénique de la naissance au sevrage[J]. Annales de Biologie Animale, Biochimie, Biophysique, 1973, 13(4): 733-735 |
| [2] | de Blas C, Wiseman J. Nutrition of the Rabbit[M]. 2nd Edition. Oxon, UK: CABI, 2010: 4-6 |
| [3] | Emaldi O, Crociani F, Matteuzzi D, et al. A note on the total viable counts and selective enumeration of anaerobic bacteria in the caecal content, soft and hard faeces of rabbit[J]. Journal of Applied Bacteriology, 1979, 46(1): 169-172 |
| [4] | Macfarlane S, Hopkins MJ, Macfarlane GT. Bacterial growth and metabolism on surfaces in the large intestine[J]. Microbial Ecology in Health and Disease, 2000, 12(2): 64-72 |
| [5] | Ji DD. Adhesion evaluation of Lactobacillus pentosus strains isolated from Chinese foods which are able to modulate intestinal flora[D]. Beijing: Master’s Thesis of Beijing Forestry University, 2009 (in Chinese) 冀朵朵. 戊糖乳杆菌的黏附能力及对肠道菌群的调节[D]. 北京: 北京林业大学硕士学位论文, 2009 |
| [6] | Zomborsky-Kovács M, Gyarmati T, Párizs T, et al. Some physiological properties of the digestive tract in traditionally reared and exclusively milk-fed young rabbits[J]. World Rabbit Science, 2000, 8: 499-506 |
| [7] | Niu ZX, Guo YK. Observation on the normal microflora in the stomach and intestinal mucosa of healthy and diarrhoeal rabbits with electron-microscop[J]. Acta Veterinaria et Zootechnica Sinica, 1995, 26(6): 565-568 (in Chinese) 牛钟相, 郭延奎. 健康兔与腹泻兔胃肠道正常微生物群超微观察[J]. 畜牧兽医学报, 1995, 26(6): 565-568 |
| [8] | Walter J, Hertel C, Tannock GW, et al. Detection of Lactobacillus, Pediococcus, Leuconostoc, and Weissella species in human feces by using group-specific PCR primers and denaturing gradient gel electrophoresis[J]. Applied and Environmental Microbiology, 2001, 67(6): 2578-2585 |
| [9] | Lee DH, Zo YG, Kim SJ. Nonradioactive method to study genetic profiles of natural bacterial communities by PCR-single-strand-conformation polymorphism[J]. Applied and Environmental Microbiology, 1996, 62(9): 3112-3120 |
| [10] | Hooper LV, Wong MH, Thelin A, et al. Molecular analysis of commensal host-microbial relationships in the intestine[J]. Science, 2001, 291(5505): 881-884 |
| [11] | Fortun-Lamothe L, Boullier S. A review on the interactions between gut microflora and digestive mucosal immunity. possible ways to improve the health of rabbits[J]. Livestock Science, 2007, 107(1): 1-18 |
| [12] | Cao WN, Ren ZJ, Li YL, et al. Effects of lactic acid bacteria on growth performance, meat quality and mRNA relative expression of insulin-like growth factor-1, insulin-like growth factor-1 receptor and growth hormone receptor in muscle of Rex Rabbits[J]. Chinese Journal of Animal Nutrition, 2014, 26(9): 2902-2910 (in Chinese) 曹玮娜, 任战军, 李亚力, 等. 乳酸菌对獭兔生长性能、肉品质及肌肉胰岛素样生长因子-1、胰岛素样生长因子1受体、生长激素受体mRNA 相对表达量的影响[J]. 动物营养学报, 2014, 26(9): 2902-2910 |
| [13] | Su Y, Xie F, Zhu WY. Comparison of concentration of volatile fatty acid and microbiota in faeces between healthy and diarrhoeal piglets[J]. Acta Microbiologica Sinica, 2011, 51(12): 1632-1638 (in Chinese) 苏勇, 谢飞, 朱伟云. 健康与腹泻仔猪粪样中挥发性脂肪酸和菌群区系的比较[J]. 微生物学报, 2011, 51(12): 1632-1638 |
| [14] | Yao W, Zhu WY, Mao SY. Tracking of the development of fecal bacterial community of diarrhea piglets by 16S rDNA techniques[J]. Acta Microbiologica Sinica, 2006, 46(1): 150-153 (in Chinese) 姚文, 朱伟云, 毛胜勇. 16S rDNA 技术研究新生腹泻仔猪粪样细菌区系的多样性变化[J]. 微生物学报, 2006, 46(1): 150-153 |
| [15] | Takahashi S, Efawa Y, Simojo N, et al. Oral administration of Lactobacillus plantarum strain Lq80 to weaning piglets stimulates the growth of indigenous lactobacilli to modify the lactobacillal population[J]. The Journal of General and Applied Microbiology, 2007, 53(6): 325-332 |
| [16] | RinttiläT, Kassinen A, Malinen E, et al. Development of an extensive set of 16S rDNA-targeted primers for quantification of real-time PCR[J]. Journal of Applied Microbiology, 2004, 97(6): 1166-1177 |
| [17] | Larsson E, Tremaroli V, Lee YS, et al. Analysis of gut microbial regulation of host gene expression along the length of the gut and regulation of gut microbial ecology through MyD88[J]. Gut, 2012, 61(8): 1124-1131 |
| [18] | Mohamed JA, Huang DB. Biofilm formation by enterococci[J]. Journal of Medical Microbiology, 2007, 56(12): 1581-1588 |
| [19] | He MQ. Animal Microecology[M]. Beijing: China Agriculture Press, 1994: 45-46 (in Chinese) 何明清. 动物微生态学[M]. 北京: 中国农业出版社, 1994: 45-46 |
| [20] | van Immerseel F, Ducatelle R, de Vos M, et al. Butyric acid-producing anaerobic bacteria as a novel probiotic treatment approach for inflammatory bowel disease[J]. Journal of Medical Microbiology, 2010, 59(2): 141-143 |
| [21] | Kelly D, Campbell JI, King TP, et al. Commensal anaerobic gut bacteria attenuate inflammation by regulating nuclear-cytoplasmic shuttling of PPAR-γ and RelA[J]. Nature Immunology, 2003, 5(1): 104-112 |
| [22] | Li PL. Screening and physiological function of Bifidobacteria from macrobian[D]. Beijing: Doctoral Dissertation of China Agricultural University, 2004 (in Chinese) 李平兰. 长寿老人源双歧杆菌优良菌株的筛选及生理功能研究[D]. 北京: 中国农业大学博士学位论文, 2004 |
| [23] | Li JJ, Chen QS. Recent advances in research on the role of Bifidobacteria in regulating and improving gut-associated diseases[J]. Food Science, 2012, 32(23): 326-332 (in Chinese) 李俊洁, 陈庆森. 双歧杆菌调理和改善肠道相关疾病作用的研究进展[J]. 食品科学, 2012, 32(23): 326-332 |
| [24] | He MQ. The origin of animal microecology preparation in our country, the development strategy and application prospects[J]. Chinese Journal of Microecology, 2001, 13(3): 166-167, 176 (in Chinese) 何明清. 我国动物微生态制剂的起源、发展战略及应用前景 [J]. 中国微生态学杂志, 2001, 13(3): 166-167, 176 |
 2016, Vol. 43
2016, Vol. 43




