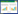扩展功能
文章信息
- 张星星, 刘亚鹏, 袁博, 赵吉睿, 王瑞刚, 冯福应
- ZHANG Xing-Xing, LIU Ya-Peng, YUAN Bo, ZHAO Ji-Rui, WANG Rui-Gang, FENG Fu-Ying
- 含好氧不产氧光合基因簇和Xanthorhodopsin-like 基因的Sphingomonas sp. MIM37:基因组及光促生长分析
- Sphingomonas sp.MIM37 possessing aerobic anoxygenic photosynthetic gene cluster and xanthorhodopsin-like gene:its genome draft and growth stimulation by illumination
- 微生物学通报, 2015, 42(8): 1520-1528
- Microbiology China, 2015, 42(8): 1520-1528
- 10.13344/j.microbiol.china.140863
-
文章历史
- 收稿日期: 2014-11-02
- 接受日期: 2015-02-11
- 优先数字出版日期(www.cnki.net): 2015-03-04
2.内蒙古师范大学 内蒙古 呼和浩特 010018
3.天津市环境监测中心 天津 300191
2. Inner Mongolia Normal University,Huhhot,Inner Mongolia 010018,China
3. Tianjin Environmental Monitoring Center,Tianjin 300191,China
不产氧光合作用是所有光合作用的祖先[1]。自发现以来,不产氧光合作用一直是微生物学、生态与环境学以及其他一些交叉学术领域关注的热 点[2]。传统上根据生长对氧的需求,不产氧光合细菌又可分为厌氧不产氧光合细菌(anaerobic anoxygenic phototrophic bacteria,AnAnPB)和好氧不产氧光合细菌(aerobic anoxygenic phototrophic bacteria,AAnPB)[3]。但是,自2000年发现变形菌视紫质(proteorhodopsin,PR)以来[4],越来越多的学者视“由视紫质介导的捕获和利用光能合成ATP”为另外一种广泛存在的光能利用途径[5, 6]。而Kang等首次报道了分离自海洋的细菌Fulvimarina pelagi同时具有好氧不产氧光合基因簇(photosynthetic gene cluster,PGC)和xanthorhodopsin (XR)光合基因[7]。但在此之后,未见有关PGC和视紫质同时存在的研究报道。我们不禁要问:PGC和视紫质是否同时存在于其他类群微生物,而作用又如何?
本文从内蒙古荒漠淡水湖泊分离得到一株兼具PGC和视紫质的细菌Sphingomonas sp. MIM37,对其进行了基因组测序和光照对生长影响的分析,以期为更充分认识和理解光能利用途径与光合生物的多样性以及光合基因进化和功能等提供线索。
1 材料与方法 1.1 菌株分离及培养2013年5月采集内蒙古腾格里沙漠天鹅湖(40°00′N,101°35′E)湖区表层(0-20 cm)水样。分离采用固体培养基稀释涂布法,置于光照培养箱培养,经反复划线分离纯化得到纯培养物。分离培养用1/2 R2A固体培养基,光促生长分析用1/4 R2A液体培养基。1/n R2A培养基是指将R2A培养基营养成分稀释n倍(固体培养基中的琼脂用量相同)。R2A培养基按Reasoner和Geldreich[8]的方法配置。
1.2 形态观察挑取分离纯化后的单菌落,按四区划线法在1/2 R2A固体培养基上划线,25 °C培养48 h,刮取第三区菌体,进行革兰氏染色并观察细菌形态。
1.3 DNA的提取和基因扩增采用CTAB法进行菌株DNA提取[9]。16S rRNA基因扩增引物为27F (5′-AGAGTTTGATCMTGG CTCAG-3′)和1492R (5′-GGTTACCTTGTTACGAC TT-3′)[9],光合基因pufM扩增引物为pufM_uniFfresh (5′-GGNAAYYTGTTYTAYAACC-3′)和pufM_uni Rfresh (5′-CCCATSGTCCANCKCCARAA-3′)[rid="b6">6],视紫质基因扩增引物为PRpf1 (5′-TAYCGYTAYGTN GAYTGG-3′)和PRpr1 (5′-ATYGGRTANACRCCCC A-3′)[6];PCR体系和条件均按照相应引物文献所述进行。PCR产物回收纯化后直接送上海生工生物工程有限公司测序。
1.4 基因序列及系统发育分析对测序所得序列进行同源性比较:16S rRNA和pufM基因用Blastn;因核酸序列变异大,视紫质基因用Blastx;以来源于纯培养物的、一致性高或模式菌株的序列为参比进行系统发育分析。16S rRNA和pufM基因核酸序列基于Kimura-2-Parameter模型,而视紫质蛋白氨基酸序列则基于JTT Matrix模型,利用MEGA 6.06软件[10]、采用邻接法(N-J)构建系统发育树,重复验证数为1 0 00。
1.5 光促生长分析为确定相关光合基因是否表达、并用于将光能转化为细胞可利用的化学能(ATP),进行了光促生长分析:以液体1/2 R2A培养基、经暗培养(诱导光合色素合成)至对数生长期,取100 μl的培养物接种于含100 ml寡营养液体培养基(1/4 R2A)的500 ml三角瓶中,分别在光照(光暗周期为12 h:12 h)与黑暗条件下振荡培养,每组设置3个重复。培养温度为25 °C,光照强度为30 mmol/(m2·s)。每隔6 h测定波长600 nm处的光吸收值来衡量细胞浓度变化。取对数期末、刚进入平台期的菌体,进行DAPI染色,利用荧光显微镜观察细胞,选择大小占多数的细胞测量其长(l)和宽(w),每个处理测量30个细胞、计算相应平均值,细胞体积V以圆柱体体积衡量(即πw2/4xl),最后比较光暗培养条件下细胞体积大小。
1.6 基因组测序及注释分析采用Illumina Hiseq2000测序技术完成菌株的基因组扫描测序,构建Illumina PE文库(片段长度为300-500 bp的文库),测序得到的原始图像数据经过Base Calling转化为序列数据,利用SOAPdenovo拼接软件对优化序列进行多个K-mer参数的拼接,得到最优的组装结果。以GapCloser软件对组装结果进行局部内洞填充和碱基校正。依据拼接序列的总长、Scaffold的数量以及Scaffold N50等技术指标,对多个K-mer的组装结果进行综合评定,从而选择最优K-mer作为最终的组装结果。应用RAST在线软件完成基因组注释。基因组序列已提交至GenBank,登录号为JROH00000000。
利用GCDC2.0 (http://ggdc.dsmz.de/)估算基因组相似性(模拟DDH;DDH,DNA-DNA Hybridization)[11]。
2 结果与分析 2.1 菌株基本生物学特性分离得到一株编号为MIM37的细菌,革兰氏染色为阴性,短杆状,运动。最适生长pH为7.5,最适盐度为0,最适温度为25-30 °C。培养于温度28 °C和光暗交替(12 h:12 h)条件下,在1/2 R2A平板上,菌落呈橘红色,菌落圆形隆起,边缘整齐,湿润,易挑起,菌落较大。
2.2 同源性和系统发育分析基因同源性(Blastn)分析表明,MIM37的16S rRNA基因与Sphingomonas sp. LH-CAB13的相似性最高(99%),而与模式菌株的相似性最高为97%、其序列来自Sphingomonas japonica KC7T;pufM与Methylobacterium extorquens AM1的基因相似性最高(79%)。在与Sphingomonas及其近缘属的模式种构建的系统发育树(图1A)中,MIM37与Sphingomonas属的种很好地聚在一起(自展值达85%)。在与近缘的AAnPB一起构建的pufM基因系统发育树中,MIM37也与来自Sphingomonas属的细菌处于同一进化支(图1B)。视紫质基因的预测蛋白序列与Sphingomonas elodea的XR相似性最高(75%),系统发育分析表明其与来源于Sphingomonas分类相近菌属的细菌亲缘关系最近、而与其他类群微生物的相距较远(图1C)。
|
|
图 1
16S rRNA 基因(A)、pufM 基因(B)及视紫质蛋白(C)序列系统发育树
Figure 1 Phylogenetic tree based on 16S rRNA gene (A),pufM (B) and rhodopsin protein sequences (C)
注:Outgroup,A中,Haloarcula vallismortis JCM 8877(AB355982);B中,Chloroflexus aurantiacus (X07847);C中,Rhodopsin[Halobacterium salinarum](AF306937).图中粗体表示相应基因组同时含好氧不产氧光合基因簇和视紫质基因. Note:Haloarcula vallismortis JCM 8877(AB355982),Chloroflexus aurantiacus (X07847),and rhodopsin[Halobacterium salinarum](AF306937) was used as outgroup for phylogenetic tree A,B and C,respectively.Letters in bold denote the corresponding genome contains anoxygenic photosynthetic gene cluster and rhodopsin gene simultaneously. |
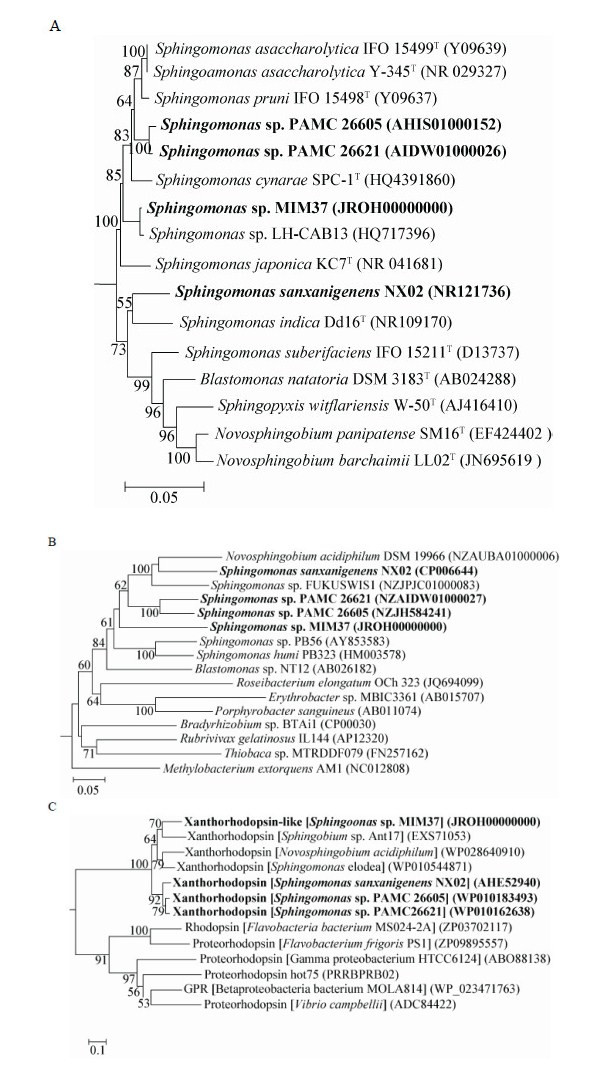
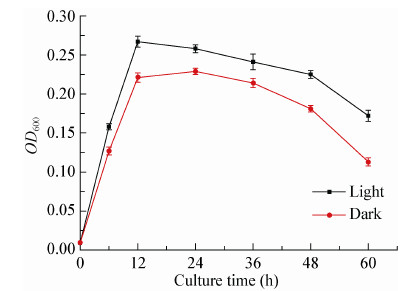
在寡营养(1/4 R2A培养基)条件下,分别在光照与黑暗下对菌株进行培养,它们的生长曲线(图2)表明:两种培养条件下的生长曲线具有相似的走势;生长初期(6 h前),两种条件下的生长没有明显差异;而之后,光照培养下的细胞浓度明显高于黑暗培养下的;约在12 h时,即对数期末期,两种培养条件下的细胞浓度均达到最大,此时光照培养下的细胞浓度为0.267 (OD600)、为同时刻黑暗培养下细胞浓度的1.2倍;在细胞浓度下降过程中同一培养时间,光照培养下细胞浓度是黑暗条件下的1.1-1.5倍。而在对数期末期,光照培养下的细胞体积也明显大于黑暗培养下的,细胞平均长、宽和体积分别相差约2.5、1.5和5.6倍(图3)。
|
| 图 2 光、暗条件下细菌生长曲线 Figure 2 Growth curve of bacteria in light and dark conditions |
|
| 图 3 光(A)、暗(B)条件下细胞大小比较 Figure 3 Cell size comparison in light (A) and dark (B) conditions |
测序所得菌株MIM37的基因组长度为 3 695 881 bp,包含154个Contigs,129个Scaffolds,G+C含量64.47%,基因组覆盖度318.5x,Contig N50值为71 636 bp,编码序列3 377条,有49种tRNA。编码两类光合系统的光合基因同时存在,具有完整的AAnPB光合基因簇(PGC)及视紫质蛋白(XR-like),未发现固碳的卡尔文循环中关键酶类如Rubisco酶相关基因。MIM37的PGC基因排列顺序cycA-ORF-tspO-bchPG-ppsR-ppaA-bchFNBHLM-lha A-puhAB-acsF-ORF-pufBALMC-bchCXYZ-crtCDF- ORF-bchID,其保守区域基因排列方式为正向bchFNBHLM-lhaA-puh,反向crtF-bchCXYZ-puf (图4A)。基因组中也存在编码视紫质色素——视黄醛合成关键基因blh,以及与之相关的还有β-胡萝卜素合成基因crtY等;未发现与固氮作用相关的基因,但含有完整好氧呼吸电子传递链组分、一些硫代谢基因(如硫氧还蛋白)和孢子形成与萌发的基因。核心的分解代谢途径主要有糖酵解途径、Entner-Doudoroff途径、磷酸戊糖途径、三羧酸循环等。MIM37还具有合成铁载体酶基因,含有温和噬菌体侵染、复制和组装等相关组分以及一些基因转移有关因子。此外,MIM37还具有重金属还原、微囊藻毒素和多环芳烃类等多种致癌物降解功能,还具有同时合成聚羟基脂肪酸酯(polyhydroxyalkanoate,PHA)和多聚磷(polyphosphate,polyP)的潜力等。
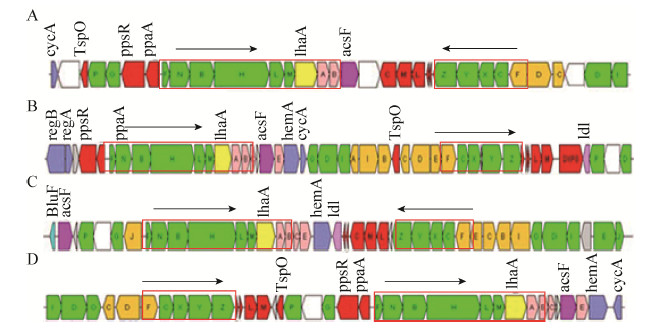
|
|
图 4 光合基因簇组成和排列结构
Figure 4 Photosynthetic gene cluster structure and arrangement
注:A:MIM37;B:Loktanella vestfoldensis SKA53;C:Congregibacter litoralis KT71;D:Citromicrobium sp.JL354.绿色:bch (细菌叶绿素合成相关)基因;红色:puf (光反应中心和捕光复合物蛋白相关)及调节基因;粉色:puh (光反应中心和捕光复合物蛋白相关)基因;橙色:crt (类胡萝卜素合成相关);蓝色:hem (血红素合成相关)和cyc (细胞色素合成相关)基因;黄色:lhaA (捕光蛋白复合体组装蛋白相关)基因;灰色:假设基因;白色:不确定或不相关基因;箭头代表假设的转录方向.B,C和D所示参考自Zheng等[12]. Note:A:MIM37;B:Loktanella vestfoldensis SKA53;C:Congregibacter litoralis KT71;D:Citromicrobium sp.JL354.Green:bch genes (related to bacteriochlorophyll biosynthesis);Red:puf (related to photosynthetic reaction unit fixed) and regulators genes;Pink:puh genes (related to photosynthetic reaction unit and light harvesting complexes);Orange:crt genes (related to carotinoid biosynthesis);Blue:hem (related to heme biosynthesis) and cyc gene (related to cytochrome biosynthesis);Yellow:LhaA gene (related to light harvesting complex assembling protein);Blank:uncertain or unrelated genes;Grey:hypothetical protein.The horizontal arrows represent putative transcripts.B,C and D refered from Zheng et al[12]. |
基因组相似性分析表明,MIM37与Sphingomonas wittichii RW1 (GenBank登录号为NC_009511)同源性最高,但是它们的16S rRNA基因相似性和基因组模拟DDH却分别只有93%和32%,而与同时含有PGC和视紫质的Sphingomonas sanxanigenens NX02、Sphingomonas sp. PAMC 26621和Sphingomonas sp. PAMC 26605基因组的模拟DDH最高只有20%。
3 讨论藻类和蓝细菌进行的光合作用是水生生态系统中能量和有机碳主要的来源。而最近的研究表明一些异养微生物可捕获光能、产生ATP来补充能量需求,但并不固定碳。这样的光合生物包括好氧不产氧光合细菌(AAnPB)和含视紫质的细菌,它们广泛地分布于各种环境中、且丰度很高[4, 5, 6, 7, 8, 9, 10, 11, 12, 13],在全球能量循环和碳循环中发挥重要作用,在营养贫瘠的环境中光合作用赋予其更强的竞争力[13, 14]。
本研究从内蒙古高原荒漠淡水湖泊天鹅湖分离得到一株细菌MIM37,经形态和系统发育鉴定为鞘脂单胞菌属(Sphingomonas)菌种。Sphingomonas最早由Yabuuchi等提出;而现在普遍使用的是Takeuchi等建议的将Sphingomonas属进一步分成4个属,即Sphingomonas、Sphingobium、Novosphingobium和Sphingopyxis (分类学命名),合称sphingomonads (非分类学命名)[15]。Sphingomonads类细菌在水环境中无处不在,也因具有非凡的降解多种致癌物的能力而备受关注[16]。同时,很多Sphingomonads类细菌或含有AAnPB的光合基因簇(photosynthetic gene cluster,PGC),或含有视紫质基因而成为光合细菌中重要的一类。基因组DNA的相似性(DNA-DNA hybridization,DDH)大于70%属于同种、而小于70%可能是新种[17, 18]。而基于基因组序列的模拟DDH与传统基于杂交技术的DDH有很强的相关性,其阈值70%可用于种的界定[10]。MIM37与现有的Sphingomonas spp. (含最近缘的模式种)基因组模拟DDH均低于70%,说明MIM37可能为Sphingomonas属的新种。
虽然在GenBank数据库中检索到3株Sphingomonas sp. (图1)中同时存在PGC和视紫质基因,但在其中相关功能基因注释并不明确(有许多hypothetic protein),或许因为相关作者并未注意到而未报道PGC和视紫质的同时存在[19]。而光合基因PCR扩增和测序以及基因组测序均表明MIM37同时含有PGC和具有编码视紫质蛋白xanthorodopsin-like (XR-like)的基因。这些光合功能基因与看家基因16S rRNA得出的系统发育分析结果较相似。功能基因与保守基因相似的系统发育分析意味着功能基因可能并非基因水平转移的结 果[20]。所以,我们推测同时含PGC和XR-like的MIM37中的光合相关基因可能并非通过基因转移、从Sphingomonas之外的其他类群微生物获得的。按排列顺序,PGC可分为三类:正向bchFNBHLM- LhaA-puh,正向crtF-bchCXYZ-puf;正向bchFNBHLM-LhaA-puh,反向crtF-bchCXYZ-puf;正向crtF-bchCXYZ-puf,正向bchFNBHLM- LhaA-puh[11](图4B、C、D)。MIM37具有完整的PGC,为第二类排列。而其他同时含PGC和视紫质的基因组中PGC的排列也属于第二类。视紫红质(rhodopsin)是一类吸光色素膜蛋白,与色素视黄醛(retinal)通过共价键结合而形成质子泵型或感光型的视紫红质。质子泵型可在光的驱动下合成ATP,为细胞提供能量[21]。质子泵型视紫质105位氨基酸不同导致其吸收峰明显不同而分为两类,即蓝光吸收型(Gln 105)和绿光吸收型(Leu 105)[22, 23]。MIM37中的视紫质基因长度为627 bp,其编码的105位氨基酸为Leu,蛋白序列与质子泵型xanthanorhodopsin (XR)最相似。因此,MIM37的XR-like应该为绿色吸收型。在微生物中,15,15-单氧酶催化β-胡萝卜素裂解而成视黄醛[24]。β-胡萝卜素合成基因为crt家族,如crtY等[25],这些相关基因也存在于MIM37中。McCarren等指出视紫质与视黄醛合成关键基因bhl紧邻、并倾向于同步转 移[26]。在MIM37中也发现紧邻视紫质蛋白基因上游存在一个可能为bhl的基因,由其预测的蛋白与Thioalkalimicrobiumcyclicum ALM1已明确注释的15,15-单氧酶相似性最高,但相似性较低(仅30%),其功能还需要体外表达等实验证实。在系统发育树中,MIM37的视紫质与来自于Sphingomonas属的很鲁棒地(自展值100%)形成一支、明显与其他的视紫质分布位置不同,并且视紫质同源性与其它来源的最高也只有70%左右。因此,我们建议将此类视紫质命名为sphingorhodopsin。
光不能作为AAnPB的唯一能量来源,需要消耗有机碳来维持其代谢和生长。光能仅可为AAnPB的异养代谢方式补充所需要总能量的20%,但是光能却能使其吸收有机物质的效率提高一倍[13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27];光照也能显著促进含质子泵型视紫质细菌在寡营养等逆境下的生长[28]。但光对细菌叶绿素的合成有强烈抑制作用[29],光也对一些含视紫质基因微生物生长的影响有限[30, 31, 32]。在活体细胞中,细菌叶绿素和视黄醛的吸收峰明显不同:细菌叶绿素在 800-870 nm处有最大吸收[17],而视黄醛在490 nm (蓝光型视紫质)或525 nm (绿光型视紫质)附近吸收最明显[23]。可见,它们的同时存在可使细胞吸收不同波段的光,使光的吸收最大化。虽然,MIM37活细胞和色素浸提液的吸收光谱并没有检测到明显的细菌叶绿素和视黄醛的吸收峰(数据未列),这可能与光合色素合成量很低有关[13]。同时,在本研究中,也不能判定是PGC、还是XR-like单独作用,或是二者同时作用,但光对寡营养条件下MIM37的生长显然产生了明显的促进作用。这一特性或许可用于促进污染物降解研究和应用,但相关光促进机制还需进一步深入 研究。
| [1] | Xiong J,Fischer WM,Inoue K,et al.Molecular evidence for the early evolution of photosynthesis[J].Science,2000,289(5485): 1724-1730 |
| [2] | Yang SP,Lin ZH,Cui XH,et al.Current taxonomy ofanoxygenic phototrophic bacteria-a review[J].Acta Microbiologica Sinica,2008,48(11):1562-1566 (in Chinese) 杨素萍, 林志华, 崔小华, 等.不产氧光合细菌的分类学进展 [J].微生物学报,2008,48(11):1562-1566 |
| [3] | Yurkov VV,Beatty JT.Aerobic anoxygenic phototrophic bacteria[J].Microbiology and Molecular Biology Reviews,1998, 62(3):695-724 |
| [4] | Beja O,Aravind L,Koonin EV,et al.Bacterial rhodopsin: evidence for a new type of phototrophy in the sea[J].Science, 2000,289(5486):1902-1906 |
| [5] | Hohmann-Marriott MF,Blankenship RE.Evolution of photosynthesis[J].Annual Review of Plant Biology,2011,62: 515-548 |
| [6] | Martinez-Garcia M,Swan BK,Poulton NJ,et al.High-throughput single-cell sequencing identifies photoheterotrophs and chemoautotrophs in freshwater bacterioplankton[J].The ISME Journal,2012,6(1):113-123 |
| [7] | Kang I,Oh HM,Lim SI,et al.Genome sequence of Fulvimarina pelagi HTCC2506T,a Mn(Ⅱ)-oxidizing alphaproteobacterium possessing an aerobic anoxygenic photosynthetic gene cluster and Xanthorhodopsin[J].Journal of Bacteriology,2010,192(18): 4798-4799 |
| [8] | Reasoner DJ,Geldreich EE.A new medium for the enumeration and subculture of bacteria from potable water[J].Applied Environmental Microbiology,1985,49(1):1-7 |
| [9] | Wisotzkey JD,Jurtshuk P Jr,Fox GE.PCR amplification of 16S rDNA from lyophilized cell cultures facilitates studies in molecular systematics[J].Current Microbiology,1990,21:325-327 |
| [10] | Tamura K,Stecher G,Peterson D,et al.MEGA6:molecular evolutionary genetics analysis version 6.0[J].Molecular Biology and Evolution,2013,30(12):2725-2729 |
| [11] | Meier-Kolthoff JP,Auch AF,Klenk HP,et al.Genome sequence-based species delimitation with confidence intervals and improved distance functions[J].BMC Bioinformatics,2013, 14:60 |
| [12] | Zheng Q,Zhang R,Koblizek M,et al.Diverse arrangement of photosynthetic gene clusters in aerobic anoxygenic phototrophic bacteria[J].PLoS One,2011,6(9):e25050 |
| [13] | Hunter N,Daldal F,Thurnauer M,et al.The Purple Phototrophic Bacteria[M].Dordrecht:Springer,2009,28:31-55 |
| [14] | Jiao NZ,Luo TW,Zhang Y,et al.Microbial carbon pump in the ocean-from microbial ecological process to carbon cycle mechanism[J].Journal of Xiamen University (Natural Science Edition),2011,50(2):387-401 (in Chinese) 焦念志, 骆庭伟, 张瑶, 等.海洋微型生物碳泵-从微型生物 生态过程到碳循环机制效应[J].厦门大学学报:自然科学版, 2011,50(2):387-401 |
| [15] | Yabuuchi E,Yano I,Oyaizu H,et al.Proposals of Sphingomonas paucimobilis gen.nov.and comb.nov.,Sphingomonas parapaucimobilis sp.nov.,Sphingomonas yanoikuyae sp.nov., Sphingomonas adhaesiva sp.nov.,Sphingomonas capsulata comb.nov.,and two genospecies of the genus Sphingomonas[J].Microbiology and Immunology,1990,34(2):99-119 |
| [16] | Vaz-Moreira I,Nunes OC,Manaia CM.Diversity and antibiotic resistance patterns of Sphingomonadaceae isolates from drinking water[J].Applied Environmental Microbiology,2011,77(16):5697-5706 |
| [17] | Stackebrandt E,Goebel BM.Taxonomic note:a place for DNA-DNA reassociation and 16S rRNA sequence analysis in the present species definition in bacteriology[J].International Journal of Systematic Bacteriology,1994,44(4):846-849 |
| [18] | Wayne LG,Brenner DJ,Colwell RR,et al.International committee on systematic bacteriology announcement of the report of the ad hoc Committee on Reconciliation of Approaches to Bacterial Systematics[J].Journal of Applied Bacteriology, 1988,64(4):283-284 |
| [19] | Lee H,Shin SC,Lee J,et al.Genome sequence of Sphingomonas sp.strain PAMC 26621,an Arctic-lichen-associated bacterium isolated from a Cetraria sp.[J].Journal of Bacteriology,2012, 194(11):3030 |
| [20] | Wang Q,Yue JP,Zhang TC,et al.Horizontal gene trensfer provides new insights into biological evolution[J].Chinese Science Bulletin,2014(21):2055-2064 (in Chinese) 王洽, 乐霁培, 张体操, 等.水平基因转移在生物进化中的作 用[J].科学通报,2014(21):2055-2064 |
| [21] | Wang N,Lan YL,Wang WW.The review and prospect of proteorhodopsin in ten years[J].Acta Biophysica Sinica,2010, 26(11):1036-1046 (in Chinese) 王宁, 兰艳黎, 王伟武.变形菌视紫红质十年的回顾与展望 [J].生物物理学报,2010,26(11):1036-1046 |
| [22] | Man D,Wang W,Sabehi G,et al.Diversification and spectral tuning in marine proteorhodopsins[J].European Molecular Biology Organization,2003,22(8):1725-1731 |
| [23] | Wang WW,Sineshchekov OA,Spudich EN,et al.Spectroscopic and photochemical characterization of a deep ocean proteorhodopsin[J].Journal of Biological Chemistry,2003, 278(36):33985-33991 |
| [24] | Giuliano G,Al-Babili S,Lintig J.Carotenoid oxygenases:cleave it or leave it[J].Trends in Plant Science,2003,8(4):145-149 |
| [25] | Armstrong GA.Genetics of eubacterial carotenoid biosynthesis: a colorful tale[J].Annual Review of Plant Biology,1997,51: 629-659 |
| [26] | McCarren J,DeLong EF.Proteorhodopsin photosystem gene clusters exhibit co-evolutionary trends and shared ancestry among diverse marine microbial phyla[J].Environmental Microbiology,2007,9(4):846-858 |
| [27] | Soora M,Cypionka H.Light enhances survival of Dinoroseobacter shibae during long-term starvation[J].PLoS One,2013,8(12):e83960 |
| [28] | Palovaara J,Akram N,Baltar F,et al.Stimulation of growth by proteorhodopsin phototrophy involves regulation of central metabolic pathways in marine planktonic bacteria[J].Proceedings of the National Academy of Aciences of the United States of America,2014,111(35):E3650- E3658 |
| [29] | Jiao NZ,Sieracki ME,Zhang Y,et al.Aerobic anoxygenic phototrophic bacteria and their roles in marine ecosystems[J].Chinese Science Bulletin,2003,48(6):530-534 (in Chinese) 焦念志,Sieracki ME, 张瑶, 等.好氧不产氧光合异养细菌 及其在海洋生态系统中的作用[J].科学通报,2003,48(6): 530-534 |
| [30] | Giovannoni SJ,Bibbs L,Cho JC,et al.Proteorhodopsin in the ubiquitous marine bacterium SAR11[J].Nature,2005,438(7064): 82-85 |
| [31] | Gonzalez JM,Fernandez-Gomez B,Fernandez-Guerra A,et al.Genome analysis of the proteorhodopsin-containing marine bacterium Polaribacter sp.MED152 (Flavobacteria)[J].Proceedings of the National Academy of Sciences of the United States of America,2008,105(25):8724-8729 |
| [32] | Stingl U,Desiderio RA,Cho JC,et al.The SAR92 clade:an abundant coastal clade of culturable marine bacteria possessing proteorhodopsin[J].Applied and Environment Microbiology, 2007,73(7):2290-2296 |
 2015, Vol. 42
2015, Vol. 42