扩展功能
文章信息
- 赵东磊, 李金玉, 方淑梅, 韦江司, 王伟, 梁喜龙
- ZHAO Dong-Lei, LI Jin-Yu, FANG Shu-Mei, WEI Jiang-Si, WANG Wei, LIANG Xi-Long
- 部分模式真菌中Prp5 蛋白生物信息学分析
- The bioinformatics analysis of Prp5 proteins in some model fungi
- 微生物学通报, 2015, 42(8): 1458-1465
- Microbiology China, 2015, 42(8): 1458-1465
- 10.13344/j.microbiol.china.140588
-
文章历史
- 收稿日期: 2014-08-02
- 接受日期: 2015-03-25
- 优先数字出版日期(www.cnki.net): 2015-05-26
在真核生物基因表达的过程中,基因首先转录形成含有外显子与内含子的前体mRNA (Pre-mRNA),然后Pre-mRNA再通过剪接复合体(spliceosome)催化的两步转酯反应切除内含子并将相邻的两个外显子连接,此过程即为Pre-mRNA剪接(Pre-mRNA splicing)[1]。Pre-mRNA剪接包括组成性剪接和选择性剪接两种方式,是所有真核生物基因表达通路中的重要一步,更是真核生物生长发育与进化的调控点[2,3,4]。特别是在选择性剪接过程中,依靠剪接复合体和剪接因子,非编码的内含子可根据需要被准确高效地去除而形成多种成熟的mRNA,进而翻译出多种具有正常功能的蛋白 质[5,6]。这样同一个基因就可以通过选择性剪接产生多种功能有差异的蛋白质。所以,Pre-mRNA的选择性剪接增加了高等生物基因表达的复杂性,其对于高等生物的细胞分化与器官发育等复杂的生物学过程具有重要作用。
为此,前人已对Pre-mRNA的剪接过程进行了深入研究[7]。通常情况下,内含子中保守的5′剪接位点,富含嘧啶区的分支点和3′剪接位点对于高效准确的剪接是非常重要的[8]。mRNA前体的5′剪接位点由U1 snRNA以碱基互补的方式进行识别,3′剪接位点由结合在该位点上游富含嘧啶区的U2AF识别,而且U2AF剪接因子还会引导U2 snRNP与分支点相结合形成剪接前体,并进一步与U4、U5、U6 snRNP三聚体相结合,形成60S的剪接体,此时内含子弯曲成套索状,上下游的外显子相互靠近,结构调整,并发生转酯反应,进行Pre-mRNA分子的剪接[9,10]。
Prp5蛋白属于DEAD/H蛋白超家族中的一员,是一类RNA依赖的ATP水解酶,同时又是RNA解螺旋酶,其在Pre-mRNA分子剪接过程中对5′和3′剪接位点的选择发挥重要作用[11]。前人通过对酿酒酵母(Saccharomyces cerevisiae)与裂殖酵母(Schizosaccharomyces pombe)等研究表明,Prp5蛋白具有与U1和U2 snRNPs相互作用的结构域,能参与前剪接体(pre-spliceosome)的组装,并对RNA剪接的保守性和灵活性产生重要的调控作用,若体系中缺少Prp5蛋白,剪接体则不能形成[10,11,12,13,14]。目前,虽然对Prp5蛋白的功能研究相对较多,但对于其在不同生物中结构及理化性质的差异性和保守性研究鲜见报道。例如,Dalbadie-Mcfarland等[15]仅通过生物信息学手段预测出了酿酒酵母Prp5蛋白的分子量为96 kD。为此,本文采用生物信息学方法对部分模式真菌中的Prp5蛋白进行了研究,旨在深入分析模式真菌中Prp5蛋白的基本特征及其差异性,从而为进一步试验研究非模式真菌中的Prp5蛋白在RNA剪接中的作用及其他独特的生物学功能提供基础。
1 材料与方法 1.1 模式真菌基本信息的获取与理化特性分析研究对象为7种模式真菌,具体包括稻瘟菌(Magnaporthe oryzae)、裂殖酵母(Schizosaccharomyces pombe)、酿酒酵母(Saccharomyces cerevisiae)、粗糙脉胞菌(Neurospora crassa)、构巢曲霉(Aspergillus nidulans)、新生隐球菌(Cryptococcus neoformans)、白色念珠菌(Candida albicans)。在稻瘟菌数据库(http://www.broadinstitute. org/annotation/genome/magnaporthe_grisea)中先搜索到Prp5蛋白质序列,然后通过NCBI数据库(http://www.ncbi.nlm.nih.gov)与白色念珠菌基因组数据库(http://www.candidagenome.org/)利用同源比对确定酿酒酵母、粗糙脉胞菌、构巢曲霉、新生隐球菌和白色念珠菌的Prp5蛋白质序列,同源比对参数为默认。由于稻瘟菌中的Prp5与裂殖酵母中的Prp5 (NP_595604)一致性非常低,仅为4.91%,但与Prp11一致性较高,达到63%,因此本文选用裂殖酵母的Prp11蛋白质序列代替其他生物的Prp5蛋白进行相关分析。各种模式真菌中Prp5/Prp11蛋白的同源比对情况如表 1所示。蛋白质的分子量、等电点、亲水性及稳定性分析均采用ExPASy (http://web.expasy.org/protparam/)在线软件进行。
| 生物名称 Species | 蛋白名称 Protein | 登录号 GenBank accession number | 一致性 Homology (%) | 覆盖度 Query coverage (%) | E值 E value |
| 稻瘟菌 Magnaporthe oryzae | MOPrp5 | ELQ33225 | 100 | 100 | 0 |
| 裂殖酵母 Schizosaccharomyces pombe | SPPrp5 | NP_595604 | 4.91 | 100 | 10 |
| 裂殖酵母 Schizosaccharomyces pombe | SPPrp11 | NP_587856 | 63 | 67 | 0 |
| 酿酒酵母 Saccharomyces cerevisiae | SCPrp5 | YBR237W | 63 | 67 | 0 |
| 粗糙脉胞菌 Neurospora crassa | NCPrp5 | XP_965469 | 70 | 83 | 0 |
| 构巢曲霉 Aspergillus nidulans | ANPrp5 | XP_658870 | 57 | 83 | 0 |
| 白色念珠菌 Candida albicans | CAPrp5 | ORF19.6831 | 48 | 78 | 4e-165 |
| 新生隐球菌 Cryptococcus neoformans | CNPrp5 | AFR93204 | 54 | 64 | 0 |
Note: Proteins are named by initials of genus name and specific name plus Prp protein. The same below.
下载模式真菌Prp5 (裂殖酵母为Prp11)蛋白质序列,利用在线网站NPS@SOMPA (https://npsa- prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)预测各种蛋白质二级结构。利用NCBI网站和Sanger (http://pfam.sanger.ac.uk/)在线软件分析蛋白质序列中可能形成结构域的区域,然后利用DomainDraw (http://domaindraw.imb.uq.edu.au/)在线软件按比例绘制结构域模式图。
1.3 进化树的构建利用ClustalX 2.0软件进行多序列比对,通过MEGA 5.22软件中的Neighbor-Joining法绘制系统进化树,其中参数设置为Test of Phylogeny:Bootstrap;Replications:1 000;Model/Method:pPoisson model。
1.4 蛋白质三级结构分析蛋白质三级结构预测分析主要采用SWISS MODEL (http://swissmodel.expasy.org/)在线软件进行,参数为默认,建模所用同源性模板为4ljy.1.A。预测的蛋白质三级结构获取后下载其PDB文件,然后利用蛋白质三级结构显示与加工软件PyMOL 1.7进行处理与显示[16]。
2 结果与分析 2.1 Prp5蛋白和基因基本信息的获取及理化特性分析通过同源比对(一致性均高于45%),利用相关数据库或网站获取稻瘟菌、酿酒酵母、粗糙脉胞菌、构巢曲霉、新生隐球菌及白色念珠菌的Prp5蛋白序列,裂殖酵母的Prp11蛋白序列,然后利用ExPASy在线软件分析它们的理化特性,具体结果如表 2所示。由表 2可知,稻瘟菌Prp5的分子量最大,酿酒酵母Prp5的分子量最小。稻瘟菌、粗糙脉胞菌、构巢曲霉的Prp5蛋白及裂殖酵母Prp11蛋白均呈碱性,其中粗糙脉胞菌的等电点最高(pI=8.69);而人类致病菌,新生隐球菌与白色念球菌的Prp5蛋白显酸性,其中新生隐球菌的等电点最低(pI=6.00);所有蛋白均为不稳定的亲水性蛋白。这些研究结果初步表明真菌中的Prp5为不稳定的亲水性蛋白,其理化特性存在一定的差异。
| 生物名称 Species | 蛋白名称 Protein | 登录号 Accession number | 蛋白长度 Length of proteins (aa) | 分子量 Molecular (kD) | 等电点 Isoelectric point | 一致性 Homology (%) | 覆盖度 Query coverage (%) | E值 E value |
| 稻瘟菌 Magnaporthe oryzae | MOPrp5 | ELQ33225 | 1 200 | 131.69 | 8.52 | 100 | 100 | 0 |
| 裂殖酵母 Schizosaccharomyces pombe | SPPrp11 | NP_587856 | 1 014 | 114.21 | 7.89 | 63 | 67 | 0 |
| 酿酒酵母 Saccharomyces cerevisiae | SCPrp5 | YBR237W | 849 | 96.359 | 8.22 | 63 | 67 | 0 |
| 粗糙脉胞菌 Neurospora crassa | NCPrp5 | XP_965469 | 1 194 | 131.30 | 8.69 | 70 | 83 | 0 |
| 构巢曲霉 Aspergillus nidulans | ANPrp5 | XP_658870 | 1 173 | 129.99 | 8.43 | 57 | 83 | 0 |
| 白色念珠菌 Candida albicans | CAPrp5 | ORF19.6831 | 884 | 100.40 | 6.09 | 48 | 78 | 4e-165 |
| 新生隐球菌 Cryptococcus neoformans | CNPrp5 | AFR93204 | 1 071 | 119.77 | 6.00 | 54 | 64 | 0 |
为明确7种模式真菌中Prp5蛋白的进化关系与保守性,本文利用MEGA 5.22软件进行了系统进化树的构建,结果如图 1所示。从图 1中可以看出,稻瘟菌和粗糙脉胞菌位于相同结点,遗传距离相同,表明二者的同源性最高,亲缘关系也最近。而二者与构巢曲霉、裂殖酵母、新生隐球菌、白色念珠菌和酿酒酵母的亲缘关系依次渐远。同时,以HSPrp5 (Homo sapiens-人)和RNPrp5 (Rattus norvegicus)为外群,根据Prp5蛋白的进化关系与保守性,7种模式真菌被分为2个类群。

|
| 图 1 7种模式真菌Prp5/Prp11蛋白的进化分析 Figure 1 Phylogenetic analysis of Prp5/Prp11 protein from seven model fungi |
蛋白质二级结构是进一步形成高级构象的基础,其中α螺旋和β折叠是蛋白质最常见的二级结构形式。本研究中各模式真菌Prp5蛋白的二级结构信息如表 3所示,β转角在各模式真菌中所占比例相似,而α螺旋、β折叠和无规卷曲所占比例差异较大,其中α螺旋结构在白色念珠菌中所占比例最高,为44.23%,β折叠所占比例以酿酒酵母最高,比含量最低的稻瘟菌高7.35%。
| 蛋白 Protein | α螺旋 α-helix (%) | β折叠 β-strand (%) | β转角 β-turn (%) | 无规卷曲 Coil (%) |
| MOPrp5 | 36.33 | 12.08 | 8.33 | 43.25 |
| SPPrp11 | 39.45 | 15.29 | 8.38 | 36.88 |
| SCPrp5 | 40.05 | 19.43 | 8.24 | 32.27 |
| NCPrp5 | 36.26 | 12.90 | 8.71 | 42.13 |
| ANPrp5 | 38.87 | 13.04 | 8.78 | 39.30 |
| CAPrp5 | 44.23 | 14.48 | 7.58 | 33.71 |
| CNPrp5 | 38.19 | 14.57 | 7.75 | 39.50 |
为了进一步确定Prp5蛋白的结构及可能的功能,利用Sanger及DomainDraw在线软件对7种模式真菌中的Prp5蛋白结构域进行了预测,其结果见图 2。7种模式真菌的Prp5蛋白均具有DEAD和Helicase_C结构域。对这两个功能结构域的氨基酸进行分析发现,DEAD结构域中氨基酸残基数量在175−177之间,其中碱性氨基酸所占比例在12.6%−14.6%之间,酸性氨基酸所占比例在8.6%−10.3%之间,Helicase_C结构域中氨基酸残基数量在78−79之间,碱性氨基酸所占比例在16.5%−20.5%之间,酸性氨基酸所占比例除酿酒酵母(6.4%)外其他都在10.1%−14.1%之间,表明DEAD和Helicase_C结构域在除酿酒酵母外的6种生物中基本相同,因此这6种模式真菌中的Prp5蛋白可能会形成相同的三级结构,并具有相似的生物学功能。而酿酒酵母Prp5蛋白在高级结构形成及功能上可能会与其他蛋白有差异,这一分析结果在后面三级结构的预测中获得支持。另外,发现MOPrp5、NCPrp5和ANPrp5的序列长度及结构域位置基本一致,表明Prp5在这3种生物中是高度保守的。而且,从图 1中也可以看出,它们的亲缘关系也是最 近的。

|
| 图 2 部分模式真菌中Prp5/Prp11蛋白结构域模式图 Figure 2 The model graph for domains of Prp5/Prp11 proteins from some model fungi Note: MOPrp5: Prp5 in Magnaporthe oryzae; SPPrp11: Prp11 in Schizosaccharomyces pombe; SCPrp5: Prp5 in Saccharomyces cerevisiae; NCPrp5: Prp5 in Neurospora crassa; ANPrp5: Prp5 in Aspergillus nidulans; CAPrp5: Prp5 in Candida albicans; CNPrp5: Prp5 in Cryptococcus neoformans. |
为了进一步明确7种模式真菌中Prp5蛋白的三级结构,利用SWISS MODEL在线软件,以4ljy.1.A为模板对7种模式真菌中的Prp5蛋白的三级结构进行了预测。结果表明,Prp5蛋白的三级结构主要由2个结构域组成,其中DEAD结构域较稳定,而Helicase_C结构域在空间结构上存在一定的变化。从各模式真菌来看,稻瘟菌、粗糙脉胞菌和构巢曲霉菌的Prp5蛋白三级结构同源程度高,裂殖酵母的Prp11、白色念珠菌和新生隐球菌的Prp5蛋白三级结构同源程度高,这些研究结果显示出其在生物体内功能的相似性,具体结果如图 3和表 4所示。对酿酒酵母而言,其在三级结构上多出一个α螺旋,因此其Prp5蛋白在结构与功能上可能具有一定的独特性。
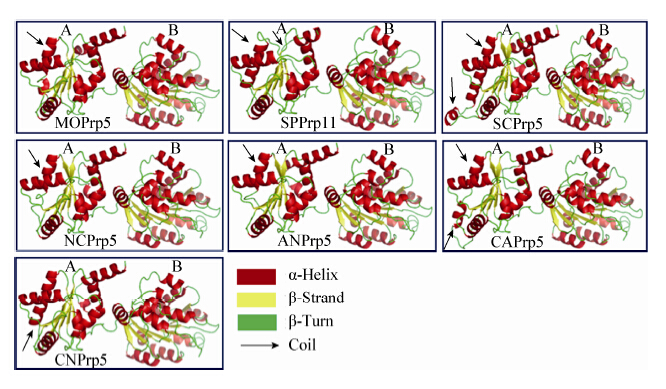
|
| 图 3 部分模式真菌中Prp5/Prp11蛋白三级结构预测模式图 Figure 3 The model graph on tertiary structure of Prp5/Prp11 from some model fungi Note: A: Helicase_C domain; B: DEAD domain. MOPrp5: Prp5 in Magnaporthe oryzae; SPPrp11: Prp11 in Schizosaccharomyces pombe; SCPrp5: Prp5 in Saccharomyces cerevisiae; NCPrp5: Prp5 in Neurospora crassa; ANPrp5: Prp5 in Aspergillus nidulans; CAPrp5: Prp5 in Candida albicans; CNPrp5: Prp5 in Cryptococcus neoformans. |
| 蛋白 Protein | 模板蛋白 Template protein | 一致性 Identity (%) | 建模质量评价 GMQE |
| MOPrp5 | 4ljy.1.A | 44.35 | 0.24 |
| SPPrp11 | 4ljy.1.A | 44.39 | 0.29 |
| SCPrp5 | 4ljy.1.A | 100.00 | 0.55 |
| NCPrp5 | 4ljy.1.A | 44.02 | 0.24 |
| ANPrp5 | 4ljy.1.A | 44.02 | 0.25 |
| CAPrp5 | 4ljy.1.A | 44.12 | 0.37 |
| CNPrp5 | c2lehB | 44.10 | 0.28 |
对几种蛋白质结合位点的氨基酸分析(表 5)显示,结合底物的氨基酸主要以Gly、Thr、Gln、Ser、Trp和Lys为主,其中Gly存在于所有蛋白质结合位点,表明其对底物结合具有重要作用。
| 蛋白 Protein | 结合位点氨基酸残基 Amino acid residues of binding sites |
| MOPrp5 | 3Gly,2Thr,Gln,2Arg,Asp,Phe,Pro,Val |
| SPPrp11 | 2Gly,3Thr,Gln,Ser,Trp,2Lys,Tyr |
| SCPrp5 | Gly,3Thr,2Gln,Ser,Lys |
| NCPrp5 | 2Gly,3Thr,Gln,Ser,Trp,2Lys,Phe |
| ANPrp5 | Gly,Ser,Leu,Ile |
| CAPrp5 | 2Gly,2Thr,Gln,2Ser,Trp,Lys, |
| CNPrp5 | 3Gly,4Thr,Gln,Ser,2Trp,Lys,Leu,Glu, 2Ala,2Arg,2Asp,Met |
前人研究表明Prp5蛋白在前剪接体形成及剪接的精确控制方面起重要作用,缺少Prp5因子,前剪接体不能形成,剪接无法继续[17]。为此本文对7种模式真菌中Prp5蛋白的基本信息、理化性质、分子进化关系、二级结构、结构域及蛋白质三级结构进行了分析,通过分析了解到这7种模式真菌中的Prp5蛋白在等电点和分子量上存在一定的差异,而在其他理化性质方面相似,这可能与不同生物在进化过程中蛋白质分子的功能进化有关。如图 1所示,等电点较小的新生隐球菌(pI=6.00)、白色念珠菌(pI=6.09)和裂殖酵母(pI=7.89)及分子量较小的酿酒酵母的Prp5蛋白与其他真菌的亲缘关系均较远。本研究结果对指导Prp5蛋白的分离提纯具有重要意义,同时对研究其他非模式真菌的Prp5蛋白也有一定的参考价值。
虽然这7种模式真菌中的Prp5蛋白在等电点和分子量上存在一定的差异,但这些差异并未构成影响蛋白质功能的主要因素,因为蛋白质的功能主要取决于其所具有的功能结构域和三级结构。前人对酿酒酵母Prp5蛋白研究表明其具有保守的DEAD (Asp-Glu-Ala-Asp)结构域[18]。拥有DEAD结构域的蛋白可参与细胞内多种生物学事件,如核糖体形成、Pre-mRNA剪接、mRNA转运、翻译起始、细胞器基因表达、RNA降解等[19]。本研究中发现7种模式真菌中的Prp5蛋白均具有DEAD和Helicase_C结构域,表明其在模式真菌中具有一定的保守性,因此该蛋白在各生物体中主要发挥相似的生物学功能。进一步对蛋白质的三级结构进行分析显示,各模式真菌中的Prp5蛋白具有相似但并不完全相同的三级结构,且这种不同主要位于Prp5蛋白的Helicase_C结构域中,结合二级结构和结构域中氨基酸组成情况的分析结果,笔者认为Prp5蛋白三级结构的这种差异源于其二级结构各类型所占百分比的差异及Helicase_C结构域中酸性氨基酸的不同。同时三级结构的差异性也表明Prp5蛋白在各模式真菌中可能还具有其独特的功能,尤其是SCPrp5蛋白,值得今后进行进一步深入的研究。
| [1] | House AE, Kristen WL. Regulation of alternative splicing: more than just the ABCs[J]. Journal of Biological Chemistry, 2008, 283(3): 1217-1221 |
| [2] | Black DL. Mechanisms of alternative pre-messenger RNA splicing[J]. Annual Review of Biochemistry, 2003, 72(1): 291-336 |
| [3] | Wang ET, Sandberg R, Luo S, et al. Alternative isoform regulation in human tissue transcriptomes[J]. Nature, 2008, 456(7221): 470-476 |
| [4] | Pan Q, Shai O, Lee LJ, et al. Deep surveying of alternative splicing complexity in the human transcriptome by highthroughput sequencing[J]. Nature Genetics, 2008, 40(12): 1413-1415 |
| [5] | Schmucker D, Clemens JC, Shu H, et al. Drosophila dscam is an axon guidance receptor exhibiting extraordinary molecular diversity[J]. Cell, 2000, 101(6): 671-684 |
| [6] | Wojtowicz WM, Flanagan JJ, Millard SS, et al. Alternative splicing of Drosophila dscam generates axon guidance receptors that exhibit isoform-specific homophilic binding[J]. Cell, 2004, 118(5): 619-633 |
| [7] | Moore MJ, Proudfoot NJ. Pre-mRNA processing reaches back to transcription and ahead to translation[J]. Cell, 2009, 136(4): 688-700 |
| [8] | Valadkhan S. SnRNAs as the catalysts of pre-mRNA splicing[J]. Current Opinion in Chemical Biology, 2005, 9(6): 603-608 |
| [9] | Hastings ML, Krainer AR. Pre-mRNA splicing in the new millennium[J]. Current Opinion in Cell Biology, 2001, 13(3): 302-309 |
| [10] | Wahl MC, Will CL, Luhrmann R. The spliceosome: design principles of a dynamic RNP machine[J]. Cell, 2009, 136(4): 701-718 |
| [11] | Xu YZ, Newnham CM, Kameoka S, et al. Prp5 bridges U1 and U2 snRNPs and enables stable U2 snRNP association with intron RNA[J]. The EMBO Journal, 2004, 23(2): 376-385 |
| [12] | Xu YZ, Query CC. Competition between the ATPase Prp5 and branch region-U2 snRNA pairing modulates the fidelity of spliceosome assembly[J]. Molecular Cell, 2007, 28(5): 838-849 |
| [13] | Chathoth KT, Barrass JD, Webb S, et al. A splicing-dependent transcriptional checkpoint associated with prespliceosome formation[J]. Molecular Cell, 2014, 53(5): 779-790 |
| [14] | Shao W, Kim HS, Cao Y, et al. A U1-U2 snRNP interaction network during intron definition[J]. Molecular and Cellular Biology, 2012, 32(2): 470-478 |
| [15] | Dalbadie-Mcfarland G, Abelson J. PRP5: a helicase-like protein required for mRNA splicing in yeast[J]. Proceedings of the National Academy of Sciences of the United States of America, 1990, 87(11): 4236-4240 |
| [16] | Biasini M, Bienert S, Waterhouse A, et al. SWISS-MODEL: modelling protein tertiary and quaternary structure using evolutionary information[J]. Nucleic Acids Research, 2014(42): 252-258 |
| [17] | Liang WW, Cheng SC. A novel mechanism for Prp5 function in prespliceosome formation and proofreading the branch site sequence[J]. Genes & Development, 2015, 29(1): 81-93 |
| [18] | Barham K, Abu AD, Tiffani KQ, et al. Probing interactions between the U2 small nuclear ribonucleoprotein and the DEAD-box protein, Prp5[J]. The Journal of Biological Chemistry, 2002, 277(23): 20221-20233 |
| [19] | Rocak S, Linder P. DEAD-box proteins: the driving forces behind RNA metabolism[J]. Nature Reviews Molecular Cell Biology, 2004, 5(3): 232-241 |
 2015, Vol. 42
2015, Vol. 42