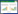扩展功能
文章信息
- 谢长校, 孙建中, 李成林, 朱道辰
- XIE Chang-Xiao, SUN Jian-Zhong, LI Cheng-Lin, ZHU Dao-Chen
- 细菌降解木质素的研究进展
- Exploring the lignin degradation by bacteria
- 微生物学通报, 2015, 42(6): 1122-1132
- Microbiology China, 2015, 42(6): 1122-1132
- 10.13344/j.microbiol.china.140686
-
文章历史
- 收稿日期: 2014-09-12
- 接受日期: 2014-11-04
- 优先数字出版日期(www.cnki.net): 2014-11-05
木质素是自然界中含量丰富的三大生物质之一,占陆生植物干重的15%−40%,其结构为由苯丙烷单元结构通过醚键和碳碳键组成的具有三维空间结构无定型芳香类化合物,与纤维素和半纤维素构成植物骨架的主要成分[1, 2]。木质素与纤维素、半纤维素结合十分稳定,不易降解,是生物质能源利用的关键瓶颈之一,同时木质素也是木材水解工业和造纸工业的副产物以及城市生活垃圾中一种较难降解的物质,采用化学试剂处理这些废弃物会造成严重的环境污染。尽管针对木质素的降解方法已经有了几十年的研究,但是仍然存在许多的问题,例如化学法不能够解决污染问题,而物理法又有高能耗的挑战,生物法对环境友好但是效率较低,因此仍然是生物能源领域研究的热点和难点。木质素在自然界中的完全降解是真菌、细菌及相应微生物群落共同作用的结果,其中真菌降解木质素的研究最为广泛和深入,但是到目前为止仍未开发出具有商业价值的生物利用木质素工业途径,以真菌为主要模式菌株开展的木质素生物降解研究长期未能突破。由于广泛的生长条件和良好的环境适应能力,细菌在木质素降解方面深受研究人员的关注,成为国际上的研究热点之一。在降解机制、新种属筛选、调控机理以及工业化的可能性等方面进行了大量深入的研究,也取得了显著的成就。在此背景下,本文对近年来木质素降解细菌的筛选、细菌降解木质素的相关机制、代谢途径及其相关酶的最新研究进展,以及木质素转化为工业产品的相关途径和机制进行综述,并探讨细菌降解木质素的工业化应用前景和未来研究发展方向。
1 木质素模型化合物降解的基本机制由于木质素属于结构非常复杂的天然聚合物,因此结构表征特别困难,是木质素研究的瓶颈之一,目前主要采用的手段包括:光谱法、光化学降解、热重分析和计算模拟等。上述方法学上的进步以及先进核磁共振方法的应用大大提高了对木质素结构表征的认识,但是天然木质素(protolignin)的结构仍未清楚。研究表明木质素是由苯丙烷单元通过醚键和碳碳键连接而成的聚酚类三维网状高分子芳香族化合物,同时含有多种活性官能团,如羟基、羰基、羧基、甲基及侧链等结构。因单体不同将木质素分为3种类型:由紫丁香基丙烷结构单体聚合而成的紫丁香基木质素(Syringyl lignin,S-木质素),由愈创木基丙烷结构单体聚合而成的愈创木基木质素(Guajacyl lignin,G-木质素)和由对羟基苯基丙烷结构单体聚合而成的对羟基苯基木质素(Hydroxy-phenyl lignin,H-木质素)[3]。
由于木质素的结构(图1)十分复杂,因此通常主要通过研究微生物对木质素模型化合物(例如藜芦醇、藜芦醛、香草醛、丁香酸等)的降解,以此来推测木质素降解的可能途径和机制[4, 5, 6, 7]。
|
| 图1 木质素结构 Figure 1 Structure of lignin |
现已基本确定,通过单电子转移机制,木质素过氧化物酶(LiP)催化氧化非酚型木质素模型化合物为其苯自由基正离子,苯自由基正离子与水或其他亲和试剂作用,随底物的不同而发生不同类型的反应,例如,当苯环上的取代基是甲氧基时,就发生脱甲氧基反应氧化形成相应的醌。β-o-4型连接方式占了所有木质素连接类型的50%,是木质素中最重要的连接方式,LiP催化β-o-4木质素模型化合 物经Cα−Cβ断裂形成藜芦醛和2-甲氧基苯酚,后者在反应条件下易于聚合,同时还有相当一部分形成Cα氧化产物。Cα−Cβ断裂是木质素降解过程中重要的反应过程,中间伴随着自由基正离子的产生。漆酶(Laccase)在氧气存在下能催化苯酚氧化生成苯氧自由基,与木质素过氧化物酶不同,漆酶不需要过氧化氢的参与,对于木质素而言,其中发生的反应包括脱甲氧基、脱羟基、C−C键断裂等。
研究细菌降解木质素的机制对木质素的降解及木质纤维素的工业化应用具有非常重要的意义。由于目前的研究主要局限于木质素模型物的研究,并不能完全反映天然木质素的代谢机制,因此深入研究细菌对天然木质素降解机理是目前需要解决的关键问题之一,也是目前国际研究的热点。
2 木质素降解相关细菌种类由于厌氧处理过程不能裂解芳香环,并且好氧过程对芳香环的裂解也比较缓慢,通常需要数天的时间,因此自然界中绝大多数细菌难以降解木质素,已经筛选到的降解木质素的细菌大多是好氧细菌,只有少数在厌氧情况下能生长,另外也有极少数细菌能在极端环境中(高温或强碱)降解木质素。
2.1 好氧细菌能够降解木质素的好氧细菌种类较多,目前主要集中在放线菌门(Actinobacteria)、变形菌门(Proteobacteria)和厚壁菌门(Firmicutes)等[8](表1)。放线菌门包括链霉菌(Streptomyces),微杆菌(Microbacterium)和红球菌(Rhodococcus)等[10, 11]。变形菌门包括假单胞菌(Pseudomonas),伯克霍尔德氏(Burkholderia)以及肠杆菌(Enterobacter)等。木质素降解菌筛选来源包括:昆虫肠道、牛胃、土壤、堆肥和海底沉积物等广泛的环境中。我们课题组近年来在南海海底沉积物和堆肥等环境中筛选到芽孢杆菌(Bacillus)、鞘氨醇单胞菌(Sphingomonas)、丛毛单胞菌(Comamonas)、微杆菌、假单胞菌、红球菌等多种能够降解木质素的细菌,为进一步研究细菌对木质素的降解提供了重要的细菌来源,通过比较不同种属细菌对木质素的降解可以总结出较完善的细菌降解木质素的机制。
| 分类 Classification | 菌名 Name |
| 放线菌 Actinobacteria | Microbacterium sp.[9], Streptomyces viridosporus T7A[10], Streptomyces coelicolor[11] |
| Rhodococcus jostii RHA1[11], Arthrobacter globiformis[11] Nocardia autotrophica[12] | |
| 变形菌 Proteobacteria | Sphingobium sp. SYK-6[7], Pseudomonas putida mt-2[11], Acinetobacter sp.[11] Brucella melitensis[9], Ochrobactrum sp.[9], Sphingomonas sp.[9], Burkholderia cepacia[13] |
| 厚壁菌 Firmicutes | Bacillus ligniniphilus L1[14], Bacillus sp. BP-7[15] |
少数厌氧细菌也能够降解木质素,例如脱硫微菌(Desulfomicrobium)是一种严格的厌氧菌,其能够利用硫酸盐、亚硫酸盐和硫代硫酸盐作为电子受体产生硫化氢。Takahiro等[16]研究发现脱硫微菌也能降解木质素。生长在白蚁肠道共生细菌螺旋原虫(Spirochaetes)在厌氧条件下能降解木质纤维素[17, 18]。厌氧微生物降解木质素的意义之一在于能够在污泥厌氧处理废水工艺中起到协同作用参与对含有木质素芳香环类物质的降解。
2.3 极端细菌极端细菌是指能够生长在高温、高压、高盐、低温、高酸碱度以及高辐射等极端条件下的细菌,往往具有一些特殊的酶和具有特殊的功能,其中有些细菌也能够降解木质素。Sung等[19]研究发现最适生长温度为75 °C的嗜热厌氧菌(Anaerocellum thermophilum DSM6725)能够直接高效利用多种未经处理的植物成分。此外,我们从南海海泥中筛选到一株能降解木质素的耐盐嗜碱芽孢杆菌L1,其生长环境非常广泛,能在温度为10−45 °C、pH为6.0−11.0条件下生长,耐盐度达到10% NaCl浓度,不仅能降解木质素还能降解许多其他芳香化合 物[14, 20]。由于实际木质素处理可能会存在高碱和高盐度等极端环境,例如造纸厂排放的废水成分复杂,碱度极高且含有大量木质素,通过筛选能够降解木质素的极端细菌,利用其独特的生理特性和降解木质素的能力处理含木质素的工业废水具有重要的意义。
3 细菌中木质素降解相关酶 3.1 漆酶漆酶是在1883年由日本学者吉田在漆树的分泌物中发现的,是一种含铜的多酚氧化酶,它能利用分子氧作为电子受体氧化多种类型的酚类化合物及非酚类化合物,将分子氧还原为水[21]。漆酶广泛存在于昆虫、植物、真菌和细菌中,具有广泛的底物作用范围,对各种酚类、芳胺及其衍生物等都具有催化氧化作用。每个漆酶分子结构中一般含有4个铜离子,根据光谱学特性可以分成3类:I型Cu2+和II型Cu2+各一个,Ⅲ型Cu2+两个。其中I型Cu2+形成单核中心,在610 nm有强烈光吸收,呈蓝色;II型Cu2+和Ⅲ型Cu2+形成一个三核中心[21, 22]。漆酶具有广泛的应用价值,可应用于制浆漂白、生物燃料、污染物降解和食品行业等。在多种细菌中已发现漆酶的存在,其中芽孢杆菌中发现的碱性漆酶克服了真菌漆酶热稳定性差,不能耐受碱性条件等缺点,并且与真菌漆酶相比,结构更简单,更适于工业化生产[23],我们近期也在耐盐嗜碱芽孢杆菌L1中发现碱性漆酶基因[24]。细菌漆酶目前所存在的问题为活性还不够高、酶产量低、工业应用中容易失活等。此外,对细菌漆酶的研究还不够深入,不同细菌来源的漆酶的蛋白结构以及分子机制不清楚。因此,深入了解漆酶的分子结构以及催化机制,通过合成生物学等手段提高漆酶的产量以及通过蛋白定向进化提高酶活是未来研究的重点方向。
3.2 过氧化物酶过氧化物酶是一类广泛存在于动物、植物、微生物中的酶,分子量为35−100 kD。目前已知基因序列的过氧化物酶约有6 000多种,其功能主要是催化过氧化氢和其他过氧化物对各种有机物和无机物的氧化作用,基于序列相似性可将真菌、细菌和植物来源的过氧化物酶归为过氧化物酶超家族。依来源不同将过氧化物酶超家族细分为3个亚家族:其中与木质素降解相关的是Class I和Class II,Class I是存在于植物、真菌、古生菌以及细菌中的胞内型过氧化物酶,包括细胞色素C过氧化物酶(CcP)、抗坏血酸过氧化物酶(APx)和过 氧化物酶(CP);Class Ⅱ是来源于担子菌纲(Homobasidiomycetes)的胞外型过氧化物酶,包括各种木质素过氧化物酶(LiP)和锰过氧化物酶(MnP)。
细菌通过分泌多种过氧化物酶降解木质素,经证实大量降解木质素的放线菌都具有胞外过氧化物酶活性,但这些酶的活性并不稳定[25]。绿孢链霉菌T7A可以分泌多种胞外过氧化物酶催化氧化β-芳基醚木质素模型物,嗜热放线菌(Thermobifida fusca)通过分泌染料脱色过氧化物酶降解木质 素[10, 26]。Mark等[27]研究发现红球菌RHA1通过分泌一种染料脱色过氧化物酶DypB来降解木质素。DypB是一种过氧化物酶,在过氧化氢的存在下具有降解木质素的活性,而且在Mn2+的作用下活性增强。因反应过程中Mn2+被氧化为Mn3+,研究人员推测其可能为MnP。MnP是代表一系列带有糖基的胞外血红素蛋白,分子量约46 kD,由一个血红素基和一个Mn2+构成活性中心,另外还有两个起稳定结构作用的Ca2+[28]。MnP只能氧化酚型木质素,在H2O2作用下,Mn2+被氧化成Mn3+,然后Mn3+氧化苯酚生成苯氧残基[29]。目前,关于细菌降解木质素产MnP 的报道非常少。Lip主要来源于真菌,Tien等[30]于1983年首次从白腐真菌属的黄孢原毛平革菌(Phanerochaete chrysosporium)的限制性培养基中发现了Lip,但最近发现细菌也能分泌Lip。链霉菌(Streptomyces cinnamomensis)能够分泌Lip降解木质素但活性较低,Jing等[31]通过优化培养基组成使Lip活性得到大幅度提升。
另外,研究表明芳醇氧化酶、乙二醛氧化酶、葡萄糖氧化酶、酚氧化酶、过氧化氢酶、新阿魏酰酯酶和对香豆酰酯酶与木质素的降解密切相关[32]。我们课题组通过研究耐盐嗜碱芽孢杆菌L1的全基因组,发现其中含有与降解木质素有关的芳基醇脱氢酶、NADH脱氢酶、乙醇酸氧化酶和超氧化物歧化酶等参与木质素降解的酶基因[15]。
4 细菌降解木质素的代谢途径由于木质素结构的复杂性,其完整代谢途径和与其有关的酶和基因尚未完全阐明,目前研究较为清楚的细菌降解木质素代谢途径主要是以鞘氨醇单胞菌SYK-6为研究对象构建的代谢途径[33]。研究木质素的代谢网络,构建木质素降解菌的酶系统非常重要,不仅有助于了解碳素循环,还有助于实现将木质素转化为有工业价值的中间代谢产物。
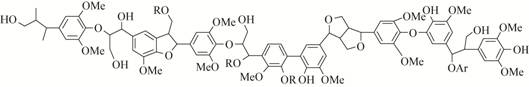
4.1 芳基醚的裂解芳基醚是木质素中最丰富的结构,其代谢途径如图2所示[34]。在脱氢酶LigD的催化作用下,α-OH被氧化成相应的酮,随后醚键在谷胱甘肽和β-eatherase酶的作用下发生裂解。代谢中产生的酮是细菌降解木质素生成的副产物,Ahmad等[11]用含木质素培养基培养恶臭假单胞菌和红球菌RHA1时,检测到培养基中有酮生成。由于γ-OH容易被氧化,酮将进一步被氧化成羧酸。
|
| 图2 芳基醚的裂解 Figure 2 β-Aryl ether cleavage |
多氯联苯是典型的联苯结构,也是一种常见的环境污染物,目前已经对其展开了深入的研究,发现有很多细菌能够降解联苯模型物,其代谢途径如图3所示[34]。LigX为脱甲基酶,作用于联苯结构上脱去甲基,生成的产物在加双氧酶LigZ的催化作用下,苯环发生裂解。随后在LigY的作用下C−C键断裂,生成5-羧基香草酸。LigW为脱羧酶,氨基酸序列比对发现其与水解酶LigY具有20%相似性,但二者催化的反应不同。在鞘氨醇单胞菌SYK-6中,LigW催化5-羧基香草酸转化成香草酸[23]。
4.3 阿魏酸代谢途径阿魏酸是木质纤维素中重要的组成成分,通过酯键与半纤维素连接在一起。在酯酶的作用下,阿魏酸从半纤维素中释放出来,从而被细菌通过两种代谢途径降解。在鞘氨醇单胞菌 SYK-6中,在ATP和辅酶A存在下,利用阿魏酰辅酶A合成酶FerA催化阿魏酸生成中间产物阿魏酰辅酶A,然后在阿魏酰辅酶A裂解酶FerB和FerB2的作用下转化成香草醛[33]。在另一些细菌中,由于含有阿魏酸脱羧酶而可以直接脱去阿魏酸中的羧基生成相应的芳香化合物。脱羧生成的4-乙烯基芳香化合物在乙烯基苯酚还原酶和NADPH的共同作用下被还原成4-乙基酚(图4)。
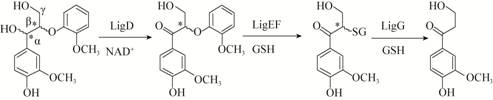
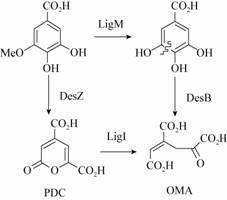
4.4 没食子酸甲酯代谢途径在木质素类芳香族化合物的生物降解中,香草酸和丁香酸是重要的中间代谢产物(图5)。丁香酸经依赖四氢叶酸的脱甲基酶催化生成没食子酸甲酯。没食子酸甲酯可以经香草酸脱甲基酶催化转化成PDC,也可以在脱甲基酶催化下形成没食子酸,没食子酸在没食子酸加双氧酶DesB的作用下经一系列反应生成OMA[33]。
|
| 图5 没食子酸甲酯代谢途径 Figure 5 Metabolic pathways of 3-methyl-gallic acid |
原儿茶酸是细菌降解芳香化合物的重要中间产物,目前知道有3种酶(原儿茶酸-3,4-加双氧酶,原儿茶酸-4,5-加双氧酶,原儿茶酸-2,3-加双氧酶)可以催化原儿茶酸中苯环的裂解。在SYK-6中细菌代谢原儿茶酸途径是原儿茶酸-4,5-加双氧酶催化使原儿茶酸苯环中第4个和第5个碳原子之间的键断裂的反应(图6)。原儿茶酸首先在原儿茶酸-4,5-加双氧酶LigAB的催化下形成4-羧基-2-羟基-粘糠酸半醛(CHMS),CHMS分子内发生缩合反应形成半缩醛形式,然后经CHMS脱氢酶 LigC氧化生成PDC,PDC在水解酶LigI的作用下水解成OMA,OMA被OMA水合酶催化生成4-羧基-4-羟基-2-氧己二酸(CHA),CHA被醛缩酶LigK催化生成丙酮酸和草酰乙酸[33]。在其他细菌中,原儿茶酸被原儿茶 酸-3,4-加双氧酶氧化生成顺己二烯二酸,然后在通过β-keto-adipate代谢途径或者ortho-cleavage代谢途径降解顺己二烯二酸。这种代谢途径已经在恶臭假单胞菌和醋酸钙不动杆菌(Acinetobacter calcoaceticus)中阐述清楚[34]。

|
| 图6 原儿茶酸代谢途径 Figure 6 Metabolic pathways of protocatechuic acid |
真菌降解二芳基丙烷木质素模型物已经被研究得非常清楚,即利用木质素过氧化物酶氧化生成香草醛,但在细菌中却存在另一种情况。研究发现,假单胞菌TMY1009在以二芳基丙烷木质素模型物为碳源时,能分泌某种酶催化该底物生成甲醛和lignostilbene (图7)。到目前为止,仍未鉴定编码该酶的基因,但是关于lignostilbene的研究已经取得一定成果。在lignostilbene加双氧酶的作用下,lignostilbene被氧化成香草醛,并且lignostilbene加双氧酶的同工酶和相关基因都已被鉴定[35, 36]。
4.7 香草酸代谢途径Paul等[37]研究发现,红球菌RHA1能够降解β-芳基醚木质素模型物,主要产物是3-(3′-甲氧基-4′-羟苯基)-3-酮基-丙醇和草酸,同时还有副产物香草酸。红球菌RHA1还能降解联苯木质素模型物生成中间代谢产物5-羧基香草酸,经脱羧形成香草酸(图8)。红球菌RHA1中vdh和vanA基因分别编码香草醛脱氢酶和香草酸去甲基化酶。研究发现,敲除vdh基因后,红球菌RHA1不能在以香草醛为单一碳源培养基上生长但能在以香草酸为单一碳源培养基上生长,表明红球菌RHA1能分泌香草醛脱氢酶使香草醛脱氢生成香草酸。而敲除了vanA基因后,红球菌RHA1既不能在以香草醛为单一碳源培养基上生长也不能在以香草酸为单一碳源培养基上生长,表明红球菌RHA1能分泌香草酸去甲基化酶使香草酸脱甲基生成3,4-二羟苯基羧酸。这说明香草酸代谢途径是红球菌RHA1降解木质素的重要 途径。
总之,对于细菌降解木质素已经有了不少的推测性研究,但是由于细菌的种类不同,其代谢途径也不完全相同,并且这类研究还不够系统,尚未完整系统地描述在细菌中木质素的代谢机制和途径。因此,建立模式细菌,在目前组学技术和先进分析手段的基础上,构建细菌降解木质素的完整代谢途径是目前急需解决的问题。
5 新技术在细菌降解木质素上的应用宏基因组测序技术有望对细菌降解木质素的研究提供有力的技术支持。通过宏基因组测序技术可以获得样品中所有微生物的DNA序列,通过对DNA序列的分析能够发现更多可以降解木质素的微生物(尤其是不可培养微生物)。Zhu等[38]通过对来自35−4 000 m不等深度的16个南海海底沉积物样品进行高通量测序,获得了超过26.5万条细菌序列,揭示了中国南海沉积物的细菌多样性生态分布,并发现一些具有对木质素和纤维素降解能力的细菌新种群,例如能够降解木质素的耐盐嗜碱芽孢杆菌L1。Shi等[39]通过对贪铜菌(Cupriavidus basilensis B-8 )进行高通量基因组测序,推测出3条可能的木质素代谢途径:β-ketoadipate代谢途径、苯酚降解途径和龙胆酸代谢途径。
转录组测序技术,利用RNA-Seq高通量测序技术测定细菌RNA序列,在整体水平上对微生物的基因表达水平和调控规律进行研究,通过绘制转录图谱,推断相应未知基因的功能,并揭示特定调节基因的作用机制。通过比较微生物在不同环境条件下的高通量转录组数据,结合宏基因组及定量PCR等其他研究手段,有助于发现环境条件对微生物代谢活性的影响以及微生物应对环境变化而进行的转录调控[40]。通过分析海量的微生物转录组数据,可以推测微生物对某类营养物或污染物代谢的可能调控路径。目前利用转录组测序技术研究细菌降解木质素的相关报道较少,但是在真菌降解木质素方面已经有了相应的成果。Shin等[41]以红橡木为唯一碳源培养黄孢原毛平革菌并经大规模平行测序技术对其转录组进行测序,发现了参与降解木质素的基因以及一些编码未知功能蛋白的基因。我们课题组目前正在以耐盐嗜碱芽孢杆菌L1为研究对象分析其以木质素为唯一碳源,在不同培养时间下的转录组数据,以期能够发现耐盐嗜碱芽孢杆菌L1降解木质素的关键基因。
蛋白质组学的研究试图比较细胞在不同生理条件下蛋白质表达的异同,对相关蛋白质进行分类和鉴定,更重要的是分析蛋白质间相互作用和蛋白质的功能[42]。但是目前,在细菌降解木质素方面的蛋白质组学研究比较少。Kristen等[43]通过2-D LC联合MS技术检测不同培养条件下肠杆菌SCF1分泌的蛋白质种类和数量,发现在含有木质素的培养基中生长的肠杆菌分泌871种独特蛋白质,并且有127种蛋白质至少上调2倍,为进一步研究细菌降解木质素的蛋白质调控机制打下基础。Chen等[44]结合基因组、转录组和蛋白组技术研究红球菌时,发现了8 947个与油脂运输、合成和代谢有关的编码基因,并且发现了177种可能参与油脂代谢的蛋白,这给研究红球菌内的脂滴提供了新的方法和技术。因此,借鉴研究红球菌脂滴时采用的组学技术的思想及之前的研究成果,利用组学技术研究细菌降解木质素的机理可能会取得某些重大突破,给细菌降解木质素的研究带来帮助。McLeod等[45]通过对红球菌RHAl的全基因组进行生物信息学分析发现了能够编码木质素降解酶的基因,Kellner等[46]从森林土壤中提取的DNA中鉴定出了超过100条不同的细菌漆酶序列,因此,结合基因组学、转录组学以及蛋白质组学等组学技术研究细菌降解木质素能发现参与木质素降解时的关键酶及基因表达情况,从而有助于阐明细菌降解木质素关键酶的调控机制,这将为细菌降解木质素的研究开创一种新的研究方法。
生物型传感器是利用某些生物活性物质具有选择性识别待测物质的能力制成的一类传感器,能够在分子水平上广泛用于检测活性物质等。Carmeron等[47]构建了荧光蛋白生物传感器,在煤层细菌群落的宏基因中发现了6个与木质素降解相关的酶基因。这种方法在环境中挖掘不可培养微生物的木质素降解相关酶基因方面非常有效。
借助上述新技术,将对细菌降解木质素的机制研究带来极大的帮助,克服以前传统手段的不足。可以预见,新的组学、生物信息学以及生物传感器等技术的应用,将为细菌降解木质素的研究迅速推向一个新的高度,并且将发现一大批木质素降解相关新酶基因。
6 细菌降解木质素的工业化应用前景木质素的预处理是木质纤维素利用的关键瓶颈之一,筛选能降解木质素的细菌可以为生物质的预处理提供有效手段。利用基因工程手段构造能够高效降解木质素的重组菌,再结合高密度培养技术有望将细菌降解木质素实现产业化。此外,造纸厂废水中含有大量的木质素及其他芳香族化合物,筛选能够高效降解污水中木质素的细菌并且利用这些细菌处理污水,将大大减小环境污染,同时使造纸厂废水得到较大程度净化。Ram等[48]从造纸厂污水中筛选出2株柠檬酸杆菌(Citrobacter IITRL1和Citrobacter IITRSU7)都能降解木质素,研究发现它们分别能使合成木质素脱色61%和64%,使硫酸盐木质素脱色49%和54%,而且两株混合菌的脱色效率更高,分别能使合成木质素和硫酸盐木质脱色85%和62%。对这两株菌加以驯化,使其对木质素的降解能力更强,有可能将其应 用到污水处理方面。真菌对硫酸盐木质素的脱色效果虽然比细菌好,但其需要消耗大量的葡萄糖而且时间较长。Zheng等[49]以6 000 mg/L硫酸盐木质素为单一碳源筛选得到一株木质素降解菌Pandoraea sp. B-6,该菌能在短时间内在高浓度木质素(2 820 mg/L)培养液中降解木质素且使COD降低46.2%。因此,可以通过合理的方法提高Pandoraea sp. B-6降解木质素的能力,使其具有应用到造纸工业及印染工业废水处理的潜力。细菌分泌的木质素降解酶能使染料脱色,如漆酶等。Niladevi等[50]研究发现沙链霉菌(Streptomyces psammoticus)分泌的漆酶在10 d内使雷玛唑亮蓝脱色80%,表明漆酶具有非常好的染料脱色能力,可以应用于印染工业的污水处理。细菌降解木质素的代谢产物含有很多有工业价值的芳香化合物(图9)[8]。Eiji等[33]研究发现,鞘氨醇单胞菌SYK-6能利用木质素模型化合物为碳源生成香草醛。此外,通过敲除红球菌RHA1中的相关基因,可以使其大量蓄积香草酸,天然香草酸是一种高值的香料,广泛用于食品和医药工业[37]。这些研究都证明细菌具有将木质素转化成有工业价值化学品的能力,但是仍存在一个难点即产物得率太低。因此若能通过控制细菌降解木质素的代谢途径,使目的产物大量产生,减少或抑制其他代谢产物的产生,就能够获得大量有用代谢产物,从而有望使其具有商业价值。
7 结论与展望与真菌相比,细菌具有良好的环境适应性和丰富的生物多样性,能适应强酸、强碱、高温等极端环境,因此,细菌降解木质素在未来工业应用上具有较大的潜力。由于目前还存在很多未解决的问题,例如细菌降解木质素的效率较低,代谢网络途径以及关键酶的调控机制等仍未完全阐明,缺乏一种较为系统的研究细菌降解木质素的方法等,所以细菌降解木质素还没有发展到实用阶段。但是目前国际上已经认识到木质素研究在生物能源领域的重要性,美国能源部已经开始大量资助木质素降解相关项目,在近几年内对木质素降解的研究可能会取得一些进展,其中食木质素细菌的研究将会成为重要的一环。
| [1] | Zakzeski J, Bruijnincx PCA, Jongerius AL, et al. The catalytic valorization of lignin for the production of renewable chemicals[J]. Chemical Reviews, 2010, 110: 3552-3559 |
| [2] | Arthur JR, Gregg TB, Mary JB, et al. Lignin valorization: Improving lignin processing in the biorefinery[J]. Science, 2014, 344: 709-719 |
| [3] | Faix O. Classification of lignins from different botanical origins by FT-IR Spectroscopy[J]. Holzforschung, 1991, 45: 21-27 |
| [4] | Xue P, Takashi E, Kaoru H, et al. Cloning of a Sphingomonas paucimobilis SYK-6 gene encoding a novel oxygenase that cleaves lignin-related biphenyl and characterization of the enzyme[J]. Applied and Environmental Microbiology, 1998, 64: 2520-2527 |
| [5] | Jörg O, Horst P, Alexander S, et al. Biochemical and genetic analyses of ferulic acid catabolism in Pseudomonas sp. strain HR199[J]. Applied and Environmental Microbiology, 1999, 65: 4837-4847 |
| [6] | Devendra N, Stephen WR. Characterization of a three-component vanillate O-demethylase from Moorella thermoacetica[J]. Journal of Bacteriology, 2001, 183: 3276-3281 |
| [7] | Hirofumi H, Eiji M, Keisuke M, et al. Characterization of the 4-carboxy-4-hydroxy-2-oxoadipate aldolase gene and operon structure of the protocatechuate 4,5-cleavage pathway genes in Sphingomonas paucimobilis SYK-6[J]. Journal of Bacteriology, 2003, 185: 41-50 |
| [8] | Timothy DHB, Mark A, Elizabeth MH, et al. The emerging role for bacteria in lignin degradation and bio-product formation[J]. Current Opinion in Biotechnology, 2011, 22: 394-400 |
| [9] | Wenzel M, Schönig I, Berchtold M, et al. Aerobic and facultatively anaerobic cellulolytic bacteria from the gut of the termite Zootermopsis angusticollis[J]. Journal of Applied Microbiology, 2002, 92(1): 32-40 |
| [10] | Rama CM, Crawford DL, Hertel G. Characterization of an extracellular lignin peroxidase of the lignocellulolytic actinomycete Streptomyces viridosporus[J]. Applied and Environmental Microbiology, 1988, 54: 3057-3063 |
| [11] | Ahmad M, Taylor CR, Pink D, et al. Development of novel assays for lignin degradation: comparative analysis of bacterial and fungal lignin degraders[J]. Molecular BioSystems, 2010, 6: 815-821 |
| [12] | Zimmermann W. Degradation of lignin by bacteria[J]. Journal of Bacteriology, 1990, 13: 119-130 |
| [13] | Kato K, Kozaki S, Sakuranaga M. Degradation of lignin compounds by bacteria from termite guts[J]. Biotechnology Letters, 1998, 20(5): 459-462 |
| [14] | Zhu DC, Tanabe SH, Xie CX, et al. Bacillus ligniniphilus sp. nov., an alkaliphilic and halotolerant bacterium isolated from sediments of South China Sea[J]. International Journal of Systematic and Evolutionary Microbiology, 2014, 64: 1712-1717 |
| [15] | Prim N, Pastor FIJ, Diaz P. Biochemical studies on cloned Bacillus sp. BP-7 phenolic acid decarboxylase PadA[J]. Applied Microbiology and Biotechnology, 2003, 63(1): 51-56 |
| [16] | Takahiro Y, Ryoko YI, Zhu JQ. Sulfate-reducing bacteria in a denitrification reactor packed with wood as a carbon source[J]. Bioresource Technology, 2011, 102(3): 2235-2241 |
| [17] | Yanxia W, Quan L, Lei Y, et al. A novel lignin degradation bacterial consortium for efficient pulping[J]. Bioresource Technology, 2013, 139: 113-119 |
| [18] | Geib SM, Filley TR, Hatcher PG, et al. Lignin degradation in wood-feeding insects[J]. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105: 12932-12937 |
| [19] | Sung JY, Irina K, Scott DHB, et al. Efficient degradation of lignocellulosic plant biomass, without pretreatment, by the thermophilic anaerobe "Anaerocellum thermophilum" DSM 6725[J]. Applied and Environmental Microbiology, 2009, 75: 4762-4769 |
| [20] | Zhu DC, Li PP, Tanabe SH, et al. Genome sequence of the alkaliphilic bacterial strain Bacillus ligninesis L1, a novel degrader of lignin[J]. Genome Announcements, 2013, 1: e00042-13 |
| [21] | Ge HH, Wu Y, Xiao YZ. Structure, catalytic mechanism and applications of laccases: a review[J]. Chinese Journal of Biotechnology, 2011, 27: 156-163 (in Chinese) 葛宏华, 武赟, 肖亚中. 漆酶空间结构、反应机理及应用[J]. 生物工程学报, 2011, 27: 156-163 |
| [22] | Garavaglia S, Cambria MT, Miglio M, et al. The structure of rigidoporus lignosus laccase containing a full complement of copper ions reveals an asymmetrical arrangement for the T3 copper pair[J]. Journal of Molecular Biology, 2004, 342(5): 1519-1531 |
| [23] | Ruijssenaars HJ, Hartmans S. A cloned Bacillus halodurans multicopper oxidase exhibiting alkaline laccase activity[J]. Applied Microbiology and Biotechnology, 2004, 65(2): 177-182 |
| [24] | Zhu DC, Sun JZ, Yang B. Lignin degradation in Bacillus ligninphilus L1[C]. Society for Industrial Microbiology and Biotechnology (SIMB) Annual Meeting, 2014 |
| [25] | Mercer DK, Iqbal M, Miller PGG, et al. Screening actinomycetes for extracellular peroxidase activity[J]. Applied and Environmental Microbiology, 1996, 62: 2186-2190 |
| [26] | Adav SS, Ng CS, Arulmani M, et al. Quantitative iTRAQ secretome analysis of cellulolytic Thermobifida fusca[J]. Journal of Proteome Research, 2010, 9: 3016-3024 |
| [27] | Mark A, Joseph NR, Elizabeth MH. Identification of DypB from Rhodococcus jostii RHA1 as a lignin peroxidase[J]. Biochemistry, 2011, 50: 5096-5107 |
| [28] | Asgher M, Shah SAH, Ali M, et al. Decolonization of some reactive textile yes by white rot fungi isolated in Pakistan[J]. World Journal of Microbiology and Biotechnology, 2006, 122: 89-96 |
| [29] | Gary W, Yitzhak H, Carlos G, et al. Lignin peroxidase-catalyzed polymerization and detoxification of toxic halogenated phenols[J]. Journal of Chemical Technology & Biotechnology, 2003, 78: 1239-1245 |
| [30] | Tien M, Kirk TK. Lignin degrading enzyme from the hymenomycete Phanetochaete chrysosporium burds[J]. Science, 1983, 221: 661-663 |
| [31] | Jing DB, Wang JH. Controlling the simultaneous production of laccase and lignin peroxidase from Streptomyces cinnamomensis by medium formulation[J]. Biotechnology for Biofuels, 2012, 5: 1-7 |
| [32] | Zhang H. New Advance of study on ligninolytic enzymes[J]. Tianjin Agricultural Sciences, 2006, 12: 8-12 (in Chinese) 张辉. 木质素降解酶系研究新进展[J]. 天津农业科学, 2006, 12: 8-12 |
| [33] | Eiji M, Yoshihiro K, Masao F. Genetic and biochemical investigations on bacterial catabolic pathways for lignin-derived aromatic compounds[J]. Bioscience, Biotechnology and Biochemistry, 2007, 71: 1-15 |
| [34] | Timothy DHB, Mark A, Elizabeth MH, et al. Pathways for degradation of lignin in bacteria and fungi[J]. Natural Product Reports, 2011, 28: 1883-1896 |
| [35] | Shigehiro K, Yoshimasa S. Structural and enzymatical comparison of lignostibene- α,β-dioxygenase isozymes, I, II, and III, from Pseudomonas paucimobilis TMY1009[J]. Bioscience, Biotechnology and Biochemistry, 1993, 57: 931-934 |
| [36] | Shigehiro K, Yoshimasa S. Cloning of a lignostibene- α,β-dioxygenase isozyme gene from Pseudomonas paucimobilis TMY1009[J]. Bioscience, Biotechnology and Biochemistry, 1995, 59: 1866-1868 |
| [37] | Paul DS, Elizabeth MH, Mark A, et al. Breaking down lignin to high-value chemicals: The conversion of lignocellulose to vanillin in a gene deletion mutant of Rhodococcus jostii RHA1[J]. ACS Chemical Biology, 2013, 8: 2151-2156 |
| [38] | Zhu DC, Tanabe SH, Yang C, et al. Bacterial community composition of South China sea sediments through pyrosequencing-based analysis of 16S rRNA genes[J]. PLoS One, 2013, 8: 1-9 |
| [39] | Shi Y, Chai LY, Tang CJ, et al. Characterization and genomic analysis of kraft lignin biodegradation by the beta-proteobacterium Cupriavidus basilensis B-8[J]. Biotechnology for Biofuels, 2013, 6: 1-14 |
| [40] | Cai YF, Jia ZJ. Progress in environmental transcriptomics based on next-generation high-throughput sequencing[J]. Biodiversity Science, 2013, 21: 401-410 (in Chinese) 蔡元锋, 贾仲君. 基于新一代高通量测序的环境微生物转录组学研究进展[J]. 生物多样性, 2013, 21: 401-410 |
| [41] | Shin S, Alex F, Prashanti I, et al. The firrst genome-level transcriptome of the wood-degrading fungus Phanerochaete chrysosporium grown on red oak[J]. Current Genetics, 2009, 55: 273-286 |
| [42] | Gong X, Zhao J, Guo BH. Progress of proteomics and its application in the research of pathogenic microorganisms[J]. Journal of Dairy Science and Technology, 2006, 3: 105-107 (in Chinese) 宫霞, 赵骏, 郭本恒. 蛋白质组学及其在病原微生物研究中的应用进展[J]. 乳业科学与技木, 2006, 3: 105-107 |
| [43] | Kristen MD, Deepak S, Rebecca V, et al. Evidence supporting dissimilatory and assimilatory lignin degradation in Enterobacter lignolyticus SCF1[J]. Frontiers in Microbiology, 2013, 4: 1-14 |
| [44] | Chen Y, Ding YF, Yang L, et al. Integrated omics study delineates the dynamics of lipid droplets in Rhodococcus opacus PD630[J]. Nucleic Acids Research, 2013: 1-13 |
| [45] | McLeod MP, Warren RL, Hsiao WWL, et al. The complete genome of Rhodococcus sp. RHA1 provides insights into a catabolic powerhouse[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103: 15582-15587 |
| [46] | Kellner H, Luis P, Zimdars B, et al. Diversity of bacterial laccase-like multi-copper oxidase genes in forest and grassland Cambisol soil samples[J]. Soil Biology and Biochemistry, 2008, 40: 638-648 |
| [47] | Cameron RS, Rahul S, David V, et al. Metagenomic scaffolds enable combinatorial lignin transformation[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111: 10143-10148 |
| [48] | Ram C, Ram NB. Bacterial degradation of synthetic and kraft ligninby axenic and mixed culture and their metabolic products[J]. Journal of Environmental Biology, 2013, 34: 991-999 |
| [49] | Zheng Y, Chai LY, Yang ZH, et al. Characterization of a newly isolated bacterium Pandoraea sp. B-6 capable of degrading kraft Lignin[J]. Journal of Central South University, 2013, 20: 757-763 |
| [50] | Niladevi KN, Prema P. Effect of inducers and process parameters on laccase production by Streptomyces psammoticus and its application in dye decolourization[J]. Bioresource Technology, 2008, 99: 4583-4589 |
 2015, Vol. 42
2015, Vol. 42