扩展功能
文章信息
- 王瑶, 许杨, 陈楠, 徐欣怡, 刘伟丰, 陶勇
- WANG Yao, XU Yang, CHEN Nan, XU Xin-Yi, LIU Wei-Feng, TAO Yong
- 在大肠杆菌中利用SCLM 系统进行高效率λ-Red 基因敲除/整合的新策略
- Novel efficient strategy for λ-Red-mediated gene knock-out/in in Escherichiacoli usingSCLM system
- 微生物学通报, 2015, 42(4): 699-711
- Microbiology China, 2015, 42(4): 699-711
- 10.13344/j.microbiol.china.140339
-
文章历史
- 收稿日期: 2014-04-22
- 接受日期: 2014-06-03
- 优先数字出版日期(www.cnki.net): 2014-12-26
2. 中国科学院微生物研究所 北京 100101
2. Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101, China
大肠杆菌是生命科学研究中一类重要的模式微生物,同时也是生物工程领域最常用的宿主菌。在大肠杆菌生理研究及代谢工程改造过程中,常常需要对染色体上相关靶点序列进行敲除、突变、替换或外源序列插入等遗传操作,以使大肠杆菌可以获得对生产、研究有利的稳定性状[1, 2, 3, 4]。例如,在利用大肠杆菌生产化合物时,通过对代谢通路中相关基因的敲除可以阻断大肠杆菌的代谢旁路、减弱毒副作用,以提高目标产物的产量或质量;通过基因插入将染色体上基因的启动子替换为强启动子,从而强化基因表达水平以增强大肠杆菌的某种代谢能力。
目前,大肠杆菌染色体上基因敲除、整合等操作绝大多数是通过λ-Red同源重组方法实现的[5, 6]。完整的λ-Red重组方法2000年由Wanner等建立[7]。该方法通过将两侧含有同源序列的线性抗性基因片段转入细胞,使片段在λ噬菌体重组酶Redα、Redβ和Redγ的作用下与染色体序列发生双交换[8, 9, 10],从而实现了对基因的敲除。在对外源基因片段进行染色体上的插入整合操作时,则需要将外源基因拼接于同源序列与抗性基因之间,然后在λ-Red重组酶的作用下插入到染色体相关位点。相比于基因敲除,基因整合能够赋予大肠杆菌更多新的性状,在大肠杆菌生理研究和代谢工程改造中成为越来越重要的研究手段[11]。特别是近年来随着耐药性、重组表达、多态性等大肠杆菌生理机制研究的广泛开展,以及合成生物学中对基因表达微调控要求的提高,蛋白表达跟踪、启动子替换等手段必须通过染色体上的基因整合操作才能实现[12, 13]。
然而由于缺乏技术步骤统一、方便高效的操作系统,λ-Red重组在实际操作尤其是在基因整合操作时受到了很大限制[14]。目前λ-Red重组体系普遍采用了pKD4-pKD46-pCP20的3质粒系统:其中包括用于扩增打靶DNA片段的模板质粒pKD4 (或pKD3、pKD13)、含有λ-Red重组酶的pKD46以及辅助质粒pCP20[7, 15]。该系统采用FRT位点作为抗性基因的消除标记,抗性基因两侧连有相同的FRT位点,该位点可以在pCP20质粒Flp酶的作用下发生重组而消除抗性基因,以进行下一轮基因敲除。但由于抗性消除后染色体被敲除基因座位处留有完整的FRT“痕”,当敲除或整合基因位点数量达到5-6个时,会造成染色体不稳定从而无法实现基因的连续敲除[16]。另一方面,这套系统最大的问题是无法有效实现基因整合。基因整合需要将目的基因序列与抗性基因拼接,由于pKD4无法进行直接的构建操作,基因整合只能采用如下策略:(1) 尝试改造模板pKD4用于构建,但pKD4拷贝数极低,并只能在特殊的BW25141菌株中复制,因此很难用传统的构建方式实现。(2) 一些人试图在普通质粒上进行拼接,但往往在重组后存在大量假阳性,后续处理繁琐。(3) 交错PCR等不依赖质粒的拼接方式,但效率很低,很难获得高浓度的目的基因片段。以上问题导致在进行外源基因整合时,没有统一的技术步骤,许多方法难以重复。使大肠杆菌基因整合操作很难在普通实验室推广。
随着代谢工程等研究的发展与实际生产的需要,亟需建立一种具有统一技术步骤、快捷、高效的λ-Red操作系统,以有利于相关研究手段进一步应用。本文报道了一种高效的基因整合与敲除方法,该方法以λ-Red重组技术为基础,通过采用Cre/LoxP的抗性消除策略替代Flp/FRT策略,从而有利于实现多基因连续稳定敲除与整合;通过引入多克隆位点并对模板质粒复制起始位点进行改造,实现了基因构建与高效筛选的兼容。从而建立了一个方便快捷、易于推广、100%阳性筛选率且具有统一技术步骤的λ-Red操作系统。
1 材料与方法 1.1 材料 1.1.1 菌株和质粒: 大肠杆菌菌株(Escherichia coli) BW25113、T1由本实验室保存,相关质粒见表 1。| 质粒 Plasmids | 质粒特征 Characteristics | 来源及参考文献 References or sources | ||||
| pKD46 | Ampr;λ-Red recombinase under araBAD promoter;ts origin | 本实验室保存[7] | ||||
| pKD4 | Ampr;FRT-Kanr-FRT;oriR6k origin | 本实验室保存[7] | ||||
| pUC19 | Ampr;ColE1 origin | 本实验室保存 | ||||
| pSB1s | Strr;araBAD promoter;pSC101 origin | 本实验室保存 | ||||
| pGAPZαA-Cre | Zeocinr;Cre recombinase | 本实验室保存 | ||||
| pS95s-glk | Strr;P119 promoter-E. coli glk fragment | 本实验室保存 | ||||
| pS95s-ispDF | Strr;P119 promoter-A. tumefaciens ispDF fragment | 本实验室保存 | ||||
| pS95s-acs | Strr;P119 promoter-E. coli acs fragment | 本实验室保存 | ||||
| pULM | Lox66-Kanr-Lox71;MCS;ColE1 origin | 本研究 | ||||
| pSCre | Strr;Cre recombinase under araBAD promoter;pSC101ts origin | 本研究 | ||||
| pCLM | Lox66-Kanr-Lox71;MCS;pSC101 origin | 本研究 | ||||
| pSLM | Lox66-Kanr-Lox71;MCS;pSC101G93R origin | 本研究 | ||||
| pSLM-glk | Kanr;P119 promoter-E. coli glk fragment;pSC101G93R origin | 本研究 | ||||
| pSLM-acs | Kanr;P119 promoter-E. coli acs fragment;pSC101G93R origin | 本研究 | ||||
| pSLM-ispDF | Kanr;P119 promoter-E. coli ispDF fragment;pSC101G93R origin | 本研究 | ||||
本研究中所涉及到的引物见表 2,引物位置如图 1所示。
| 引物 Primers | 序列 Sequences (5′→3′) | 用途 Purpose | |||
| 构建用引物 Constructing primers | |||||
| pSC101-F | AGACTCGGCGCGCCGGTTGGCGCGCAGACCCGCCAT | Amplification of pSC101 origin | |||
| pSC101-R | AGACTCGCGGCCGCGGGTTTTGCTGCCCGCAAACGGG | ||||
| glk-F | AGACTCGCGGCCGCATGCAAAGTATGCATTAGTCG | Amplification of glk gene | |||
| glk-R | AGACTCGCTAGCTTACAGAATGTGACCTAAGG | ||||
| cre-F | AGACTCCCATGGGATCCAATTTACTGACCGT | Amplification of cre gene | |||
| cre-R | AGACTCCTCGAGCTAATCGCCATCTTCCAG | ||||
| acs-F | AGACTCGCGGCCGCATGAGCCAAATTCACAAACA | Amplification of acs gene | |||
| acs-R | AGACTCGCTAGCTTACGATGGCTCGCGATAG | ||||
| IspDF-F | AGACTCGCGGCCGCATGAAATTCGGCATCGTCAT | Amplification of IspDF gene | |||
| IspDF-R | AGACTCGCTAGCTCATAGCGGTCTGCCTTGGT | ||||
| pSC101C-F | AAAGGCTTTCGGATTTTCCAGTGGAC | pSC101 origin point mutation of copy number (G93→R) | |||
| pSC101C-R | GAAAATCCGA AAGCCTTTAACCAAAG | ||||
| pSC101T-F | ACCAATACGTTCAGATGATGAACAT | pSC101 origin point mutation of temperature sensitive (A56→V) | |||
| pSC101T-R | ATGTTCATCATCTGAACGTATTGGT | ||||
| 打靶片段扩增引物 Amplification primers of targeting fragment | |||||
| pgi-P1 | TACAATCTTCCAAAGTCACAATTCTCAAAATCAGAAGAGTATTGCTAATGATTCCGGGGATCCGTCGACC | Amplification of pgi gene knock-out fragment | |||
| pgi-P2 | GCCTTATCCGGCCTACATATCGACGATGATTAACCGCGCCACGCTTTATATGTAGGCTGGAGCTGCTTCG | ||||
| ptsG-P3 | CCCCCCTTGCCACGCGTGAGAACGTAAAAAAAGCACCCATACTCAGGAGCACTCTCAATTGCATGCCACAGCTAACACC | Amplification of glk gene knock-in fragment | |||
| ptsG-P2 | CATCTGGCTGCCTTAGTCTCCCCAACGTCTTACGGATTAGTGGTTACGGATGTACTCGACGGATCCCTGCAGACTA | ||||
| poxB-P1 | GATGAACTAAACTTGTTACCGTTATCACATTCAGGAGATGGAGAACCATGATTCCGGGGATCCGTCGACC | Amplification of poxB gene knock-out fragment | |||
| poxB-P2 | CGTAAATCAATCATGGCATGTCCTTATTATGACGGGAAATGCCACCCTTTATGTAGGCTGGAGCTGCTTCG | ||||
| galR-P1 | TCCGTAACACTGAAAGAATGTAAGCGTTTACCCACTAAGGTATTTTATGATTCCGGGGATCCGTCGACC | Amplification of galR gene knock-out fragment | |||
| galR-P2 | GTCGCCAGACCATCGAAGAATTACTGGCGCTGGAATTGCTTTAACTGCGGATGTAGGCTGGAGCTGCTTCG | ||||
| yliE-P1 | TTGCAGCGGCACAGCGTTCAGATAGTTATTTTGTTAAATGTATTAACATGATTCCGGGGATCCGTCGACC | Amplification of yliE gene knock-out fragment | |||
| yliE-P2 | TCAGCAGACTGACTGTAAGTACGAACTTATTGATTCTGGACATACGTAAAATGTAGGCTGGAGCTGCTTCG | ||||
| poxB-P3 | TCAGATGAACTAAACTTGTTACCGTTATCACATTCAGGAGATGGAGAACCGCATGCCACAGCTAACACC | Amplification of ispDF and acs gene knock-in fragment | |||
| poxB-P2 | CGTAAATCAATCATGGCATGTCCTTATTATGACGGGAAATGCCACCCTTTGACGGATCCCTGCAGACTA | ||||
| 鉴定引物 Identification primers | |||||
| pgi-A1 | GCCATCAGATCCTTGGCGGCAAG | Identification of recombinants of pgi gene knock-out | |||
| pgi-A2 | ATGAAAAACATCAATCCAACGC | ||||
| K1 (Kan+-F) | TGGCTACCCGTGATATTGCTGAAGA | Identification of recombinants of gene knock-out/knock-in | |||
| ptsG-A1 | TGGATCTCTTCACCCGCTACCAGG | Identification of recombinants of glk gene knock-in | |||
| ptsG-A2 | ATGTTTAAGAATGCATTTGCTAAC | ||||
| poxB-A2 | CCGCGTTCGCAGTGACTGAG | Identification of recombinants of poxB gene knock-out | |||
| galR-A2 | TCGTTCTCTGGAACACGCGC | Identification of recombinants of galR gene knock-out | |||
| yliE-A2 | AGCTGTGCGCAAACCAACCA | Identification of recombinants of yliE gene knock-out | |||
Note: The underline primers were the restriction enzyme cutting site; pSC101-F was AscⅠrestriction enzyme cutting site; pSC101-R, IspDF-F, acs-F and glk-F were NotⅠrestriction enzyme cutting site; glk-R, IspDF-R, acs-R were NheⅠrestriction enzyme cutting site; cre-F were NcoⅠrestriction enzyme cutting site; cre-R was XhoⅠrestriction enzyme cutting site; The bold part of the primers were homologous arms.

|
|
图 1
基因敲除/整合及其卡纳抗性基因去除设计示意图
Figure 1
Schemes for gene knock-out/knock-in and excision of kanamycin resistance gene
注:A:基因敲除过程;B:基因整合过程. Note: A: Procedures for gene knock-out; B: Procedures for gene knock-in. |
以pKD4质粒为模板扩增卡那霉素抗性基因,同时将多克隆位点和Lox位点序列设计在引物中,以pUC19质粒为模板扩增ColE1复制起始位点片段,以pSB1s质粒(本实验室构建,待发表)为模板扩增复制子pSC101,利用引物通过NotⅠ和AscⅠ限制性酶切位点连接,所获得的质粒pULM (复制子为ColE1)与pCLM (复制子为pSC101)即为含有Lox66/Lox71位点及两个多克隆位点的敲除/整合模板质粒。以质粒pCLM为基础,利用引物pSC101C-F与pSC101C-R将起始位点的甘氨酸突变为缬氨酸,该位点能增加质粒的拷贝数[17],从而获得了新的敲除/整合模板质粒pSLM。将glk基因转录子连接至模板质粒pSLM的多克隆位点位置,构建的模板质粒pSLM-glk便于基因的整合。将Cre重组酶基因片段通过NcoⅠ和XhoⅠ连接到链霉素抗性、含有阿拉伯糖启动子和pSC101复制起始位点的表达载体pSB1s上,利用引物pSC101T-F与pSC101T-R将pSC101复制起始位点中的丙氨酸位点突变为缬氨酸,该位点突变能将质粒改造成温度敏感型[18],从而获得了辅助质粒pSCre用于抗性基因的消除。分子克隆操作采用分子克隆实验指南进行操作[19],转化T1感受态细胞,构建载体通过测序鉴定。
1.4 打靶片段的制备以模板质粒(pULM、pCLM、pSLM、pSLM-glk、pSLM-acs、pSLM-ispDF)为模板,利用打靶片段扩增引物扩增含有LoxP位点的卡那霉素抗性片段(或包括待插入基因片段),PCR产物分别通过PCR产物纯化试剂盒或凝胶回收试剂盒进行纯化(必要时纯化后以DpnⅠ限制性内切酶37 °C处理6 h以去除模板质粒,并再次通过PCR产物纯化试剂盒回收打靶片段)。
1.5 Red基因的诱导表达及电转感受态细胞的制备质粒pKD46利用CaCl2法转化进入宿主菌BW25113,于含有氨苄青霉素的LB培养基中30 °C培养过夜,次日按1:100接种至100 mL LB培养基,30 °C培养至OD600为0.2,加入L-阿拉伯糖至终浓度30 mmol/L,诱导1 h (OD600不超过0.6),使Redα、Redβ和Redγ三种蛋白表达。冰上预冷30 min,然后4 000 r/min、4 °C离心10 min,用预冷的无菌水洗涤1次,然后用预冷的10%甘油洗涤3次,浓缩成500 μL感受态细胞,取50 μL感受态用于电转化。
1.6 电转化将处理过的打靶DNA片段与感受态细胞混匀,转入预冷的0.1 cm的电击杯,然后进行电击,电击条件为电压1.8 kV,电击时间5 ms,电击完成后立即加入预热的LB培养基,30 °C复苏2 h,涂布卡那霉素+氨苄青霉素抗性平板,30 °C培养过夜。
1.7 基因敲除菌株鉴定挑取平板上的重组子于LB液体培养基 (Amp++Kan+),30 °C培养,利用鉴定引物PCR扩增方法鉴定是否整合,挑选阳性克隆。将阳性菌种置于LB液体培养基(Kan+) 42 °C培养,以去除质粒pKD46,然后划线LB平板(Kan+),对所得的单菌落进行氨苄青霉素敏感型检测,对氨苄青霉素敏感的菌种即为去除了质粒pKD46的菌种。
1.8 卡那霉素抗性基因的去除将质粒pSCre转化以上获得的卡那霉素抗性的菌种,涂布LB平板(Str+),30 °C培养过夜,表达的Cre酶识别LoxP位点将卡那霉素抗性去除。从平板挑选克隆于LB液体培养基(Str+) 30 °C培养,利用0.2% L-阿拉伯糖进行诱导Cre酶表达,通过鉴定引物进行PCR扩增鉴定抗性基因是否去除,将阳性菌种置于42 °C培养,以去除质粒pSCre,然后划线无抗LB平板,对所得的单菌落进行卡那霉素敏感型检测及PCR性状验证。
2 结果与分析 2.1 Cre-LoxP-MCS (CLM)系统的构建和检测为了构建一种有利于基因整合操作,并能够实现多基因的连续敲除或整合的模板质粒,在模板质粒pULM中引入如下功能单元:(1) Lox66/Lox71序列作为抗性标记基因的消除位点。Lox66和Lox71在Cre重组酶的作用下发生重组并消除抗性基因。形成的Lox72序列很难被Cre酶进一步作用,从而能够在后续的多基因连续敲除或整合时保持染色体的稳定[20]。(2) 在Lox66及Lox71两个位点的外侧分别引入两个多克隆位点MCS1和MCS2,从而可以通过限制酶连接或其他方法方便地实现基因拼接(图 2)。

|
| 图 2 模板质粒pULM及多克隆位点示意图 Figure 2 Map of template plasmid pULM and cloning regions |
以质粒pULM为模板对pgi基因(6-磷酸果糖异构酶)进行敲除。打靶片段采用琼脂糖凝胶电泳纯化和PCR产物纯化两种纯化方式,并经DpnⅠ酶处理。结果显示,经琼脂糖凝胶电泳与PCR产物纯化的DNA片段均有约103/50 μL感受态细胞的克隆,分别随机挑选20个克隆,用鉴定引物pgi-A2与K1对pgi基因敲除菌种进行鉴定。结果表明:经PCR产物纯化的DNA片段全部为假阳性(阳性率为0, 图 3A),而经琼脂糖凝胶电泳回收的DNA片段,有2个克隆的基因扩增条带大小为600 bp,与实际大小一致,为成功敲除的阳性克隆(图 3B)。经提取质粒鉴定,假阳性均为模板质粒转入引起。而通过延长DpnⅠ的处理时间,能够一定程度上消除假阳性(琼脂糖凝胶电泳纯化后处理超过16 h,16阳 性/20克隆,阳性率为80%)。

|
|
图 3
菌落PCR对pgi基因敲除菌种的鉴定
Figure 3
PCR identification of recombinants of pgi gene knock-out
注:A:打靶片段通过PCR产物纯化试剂盒纯化;B:打靶片段通过琼脂糖凝胶电泳纯化. M:DNA marker;1:阳性对照;2-11:随机挑选的重组子. Note: A: DNA fragment used for knocking out the pgi gene was purified by cycle-pure kit; B: DNA fragment used for knocking out the pgi gene was purified by gel extraction kit. M: DNA marker; 1: Positive control; 2-11: Recombinants. |
将pSCre质粒转入pKD46消除的敲除菌株用于消除抗性基因,利用引物pgi-A1与pgi-A2对抗性基因消除菌种进行验证。由于含有卡那霉素抗性基因,因此扩增条带大小为2 024 bp (图 4,泳道1),消除卡那霉素抗性基因后,条带大小为600 bp (图 4,泳道2-11),条带大小与实际一致,表明卡那霉素抗性基因已被Cre酶的作用去除。

|
|
图 4
菌落PCR对pgi基因敲除菌种的卡那霉素抗性基因去除后的鉴定
Figure 4
PCR identification of recombinants of pgi gene knock-out without kanamycin resistance
注:M:DNA marker;1:阴性对照;2-11:去除卡那抗性的重组子. Note: M: DNA marker; 1: Recombinant with kanamycin resistance; 2-11: Recombinants without kanamycin resistance. |
基于质粒pULM和pSCre的Cre-LoxP-MCS系统能够成功实现大肠杆菌基因的敲除和抗性消除,同时构建了多克隆位点也便于基因整合的分子操作。但操作过程中存在较多的假阳性,还无法实现高效率的敲除/整合。为了解决模板质粒造成的假阳性问题,构建了质粒pCLM。将1 ng的pCLM质粒分别电转化BW25113和BW25113 (pKD46),取1/10体积的复苏细胞涂布具有相应抗性的平板。结果表明,pULM质粒转化BW25113 (pKD46)的转化子数(Kan++Amp+平板)约为104/ng DNA (图 5A),pCLM质粒转化BW25113的转化子数量约为5×103/ng DNA (Kan+平板)(图 5B),而转化BW25113 (pKD46)菌株时无转化子(Kan++Amp+平板)(图 5C)。以上结果证明由于质粒的不相容性,采用同样pSC101衍生的复制起始位点的策略能够有效消除模板质粒转入BW25113 (pKD46)。

|
|
图 5
pCLM质粒与pULM质粒转化比较
Figure 5
Comparison of transformants of plasmid pCLM and pULM
注:A:pULM转化BW25113 (pKD46);B:pCLM转化BW25113;C:pCLM转化BW25113 (pKD46). Note: A: Colonies for BW25113 (pKD46) transformed by pULM; B: Colonies for BW25113 transformed by pCLM; C: Colonies for BW25113 (pKD46) transformed by pCLM. |
质粒pCLM所采用的pSC101复制起始位点为低拷贝(5 copies/cell),在采用常规酶切连接操作时,往往需要制备较多的质粒,造成操作的繁琐,点突变改造的质粒pSLM提高了拷贝数便于分子生物学操作。突变后获得质粒pSLM并通过测序验证(图 6)。采用相同菌体量分别提取相关质粒pULM、pCLM和pSLM,凝胶电泳结果表明pSLM的浓度与pULM基因一致(图 7,泳道1与泳道3),证明pSLM质粒具有高拷贝特性从而有利于基因操作。此外将pSLM质粒电转化BW25113 (pKD46)时,转化能力与pCLM质粒相似,0.1 ng的DNA仍然无转化子,证明pSLM仍然保持与pKD46不相容特性。

|
| 图 6 模板质粒pSLM示意图 Figure 6 Map of template plasmid pSLM |

|
|
图 7
模板质粒pULM、pCLM和pSLM浓度的分析
Figure 7
Agarose gel electrophoresis identification of template plasmid pULM, pCLM and pSLM
注:M:DNA marker:1:模板质粒pULM;2:模板质粒pCLM;3:模板质粒pSLM. Note: M: DNA marker; 1: Template plasmid pULM; 2: Template plasmid pCLM; 3: Template plasmid pSLM. |
以pSLM为模板,敲除pgi基因,经PCR产物纯化与琼脂糖凝胶电泳的DNA片段均长有约5×102个克隆,分别随机挑选20个克隆,用鉴定引物对pgi基因敲除菌种进行鉴定。结果表明,分别有18个和20个阳性克隆,阳性率分别为90%和100% (图 8)。对PCR产物纯化中2个假阳性克隆进行鉴定时,未发现pSLM特征序列,推测为痕量DNA污染造成。琼脂糖凝胶电泳回收具有DNA纯化作用,因此后续实验均采用该方法进行DNA 回收。

|
|
图 8
菌落PCR对pgi基因敲除菌种的鉴定
Figure 8
PCR identification of recombinants of pgi gene knock-out
注:A:打靶片段通过PCR产物纯化试剂盒纯化;B:打靶片段通过琼脂糖凝胶电泳纯化. M:DNA marker;1:阳性对照;2-11:随机挑选的重组子. Note: A: DNA fragment used for knocking out the pgi gene was purified by cycle-pure kit; B: DNA fragment used for knocking out the pgi gene was purified by gel extraction kit. M: DNA marker; 1: Positive control; 2-11: Recombinants. |
进一步,选择丙酮酸氧化酶poxB (1 719 bp)、乳糖代谢调控因子galR (1 032 bp)、磷酸化调控因子yliE (2 349 bp)进行基因敲除操作。采用pSLM模板质粒,分别对poxB、galR、yliE基因进行扩增,然后对其进行敲除,采用验证引物进行验证,poxB (图 9A)、galR (图 9B)、yliE (图 9C)敲除后大小与阳性对照大小一致。与敲除pgi基因的结果类似,在约5×102个克隆中分别挑取20个克隆进行验证,3个基因均被成功敲除,阳性率均为100%。以上结果表明,以pSLM模板质粒为基础,能够实现高效率的基因敲除。

|
|
图 9
菌落PCR对poxB、galR、yliE基因敲除菌种的鉴定
Figure 9
PCR identification of recombinants of poxB, galR and yliE gene knock-out
注:A:poxB基因敲除;B:galR基因敲除;C:yliE基因敲除. M:DNA marker;1:阳性对照;2:随机挑选的重组子. Note: A: poxB gene knocking out; B: galR gene knocking out; C: yliE gene knocking out. M: DNA marker; 1: Positive control; 2: Recombinants. |
以pSLM质粒为基础构建连接有大肠杆菌葡萄糖激酶glk基因的模板质粒,并将glk基因整合到大肠杆菌染色体的ptsG位点上。获得50个左右的克隆,随机挑选8个克隆,采用引物K1与ptsG-A2进行鉴定,扩增条带大小为600 bp (图 10,泳道1-8),证明glk基因成功整合至ptsG基因位置,阳性率为100%,并进一步采用pSCre成功消除了卡那霉素抗性基因(图 11,泳道2-9)。

|
|
图 10
菌落PCR对glk基因整合菌种的鉴定
Figure 10
PCR identification of recombinants of glk gene knock-out
注:M:DNA marker;1-8:随机挑选的重组子. Note: M: DNA marker; 1-8: Recombinants. |

|
|
图 11
菌落PCR对glk基因整合菌种的卡那霉素抗性基因去除后的鉴定
Figure 11
PCR identification of recombinants of glk gene knock-in without kanamycin resistance
注:M:DNA marker;1:阴性对照;2-9:去除卡那抗性的重组子. Note: M: DNA marker; 1: Recombinant with kanamycin resistance; 2-9: Recombinants without kanamycin resistance. |
为了进一步验证系统在整合操作时适应的片段种类和大小范围,选择大肠杆菌乙酰辅酶A合成酶acs基因与农杆菌萜类合成代谢ispDF基因进行基因整合操作。acs转录子与卡那抗性基因大小为 4 113 bp,ispDF基因与卡那抗性基因大小为3 548 bp。与glk基因整合结果类似,获得50个左右的克隆,随机挑选8个克隆,采用引物poxB-A2与K1进行鉴定,扩增条带大小为600 bp (图 12,泳道2),证明基因成功整合至poxB基因位置,阳性率为100%。以上结果表明,以pSLM模板质粒为基础,能够实现高效率的基因整合。

|
|
图 12
菌落PCR对acs,ispDF基因整合菌种的鉴定
Figure 12
PCR identification of recombinants of acs and ispDF gene knock-in
注:A:acs基因整合;B:ispDF基因整合. M:DNA marker;1-2:随机挑选的重组子. Note: A: acs gene knocking in; B: ispDF gene knocking in. M: DNA marker; 1-2: Recombinants. |
通过本研究的优化,建立了一套基于SCLM系统的大肠杆菌λ-Red基因敲除整合策略(图 13)。在该策略中,通过在模板质粒中引入2个多克隆位点,能够方便实现待整合基因与抗性标记基因的拼接,便于制备打靶DNA片段。通过对复制起始位点的改造,节省了整个DNA制备和筛选的时间,并实现了克隆筛选的100%阳性率(表 3)。
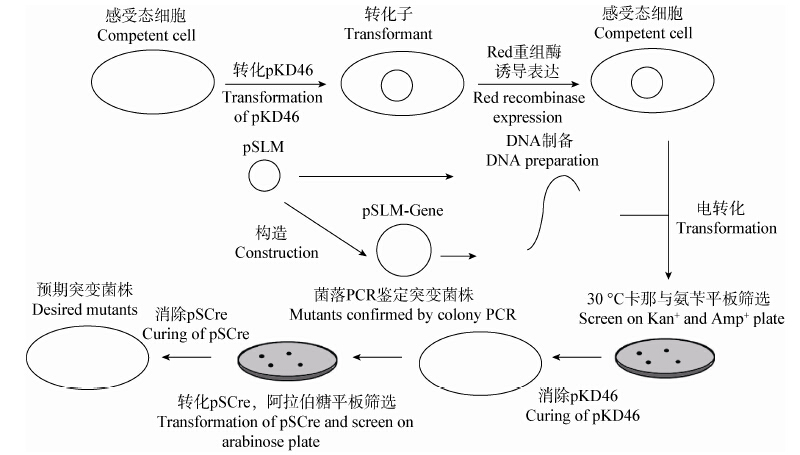
|
| 图 13 本文的基因敲除/整合系统示意图 Figure 13 Schemes for the gene knock-out/knock-in system developed in this study |
| 项目 Item | 模板质粒 Template plasmid | |||||
| pULM | pSLM | |||||
| 处理过程 Method of treatment | PCR产物纯化 | + | - | - | + | - |
| 琼脂糖凝胶电泳纯化 | - | + | + | - | + | |
| Dpn I处理 | + | + | 处理16 h | - | - | |
| 阳性率 Positive ratio (%) | 0 | 20 | 80 | 90 | 100 | |
Note: +: the process was adopted; −: the process was not adopted.
Wanner等[7]建立的λ-Red同源重组体系由于操作简单、成功率高,是大肠杆菌染色体基因敲除整合的首选方式。λ-Red同源重组体系目前普遍采用Wanner等[7]构建的pKD4 (包括衍生的pKD3、pKD13)为模板质粒。该模板质粒采用不含pir蛋白的R6k为复制起始位点,只能在特殊的宿主菌BW25141中才能复制,可以最大限度地消除同源重组的假阳性。但pKD4没有多克隆位点,基因整合操作需要通过质粒外拼接或对质粒进行改造才能实现。然而由于低拷贝特性且不能在常规菌株中进行构建,pKD4衍生质粒无法同时具备低阳性率和方便改造两种特性,从而限制了该方法在条件有限的常规实验室中进行推广。近年来针对肠杆菌λ-Red重组体系,人们开展了一些体系优化和改造工作,以试图解决操作繁琐等问题。2013年,Chan等[11]将分别表达λ-Red重组酶和Cre重组酶的两种质粒整合在一起,创建了单一辅助质粒,节省了基因连续敲除操作的时间。但在该系统中模板质粒不含有多克隆位点,并采用了常规p15A复制起始位点,因此不利于基因整合操作,也没针对阳性率进行改进。而本研究的SCLM系统利用pSC101复制子,减少了DpnⅠ酶处理等繁琐的DNA片段制备过程,成功解决了阳性率和改造不能同时进行的问题。采用多克隆位点适合大多数分子生物学实验室进行,也可以通过Gibson等[21]方法实现快速片段拼接(表 4)。
| 模板质粒 Template plasmid | 抗性消除位点 (多基因敲除是否稳定) Site of eliminating resistance marker (Stability of multi-gene knock-out/knock-in) | 复制起始位点 (是否有消除假阳性功能) Origin of replication (Reduce false positive rate) | 质粒拷贝数 Plasmid copy number | 多克隆位点 Multiple cloning sites |
| pKD4[7] | FRT/FRT (不稳定) | R6k (有) | 低 | 无 |
| pECLox2272k[20] | Lox66/Lox71 (稳定) | p15A (无) | 中等 | 无 |
| pSLM (This study) | Lox66/Lox71 (稳定) | pSC101 (G93R) (有) | 高 | 有 |
综上所述,通过对λ-Red重组体系的改造和优化,建立了一套简单、高效率的大肠杆菌λ-Red基因敲除、整合系统。利用该系统进行基因整合时,可以快捷地实现整合片段与抗性基因间的拼接。同时由于该方法阳性率高,减少了不必要的DNA片段处理过程,从而能够快速、高效地进行基因的连续敲除和整合,适合在普通分子生物学实验室中推广。
| [1] | Bailey JE. Toward a science of metabolic engineering[J]. Science, 1991, 252(5013): 1668-1674 |
| [2] | Stephanopoulos G, Vallino JJ. Network rigidity and metabolic engineering in metabolite overproduction[J]. Science, 1991, 252(5013): 1675-1681 |
| [3] | Li Y, Cao ZA. Microbial metabolic engineering: gateway to develop blueprints for cell factories[J]. Journal of Chemical Industry and Engineering (China), 2004, 55(10): 1573-1580 (in Chinese) 李寅, 曹竹安. 微生物代谢工程: 绘制细胞工厂的蓝图[J]. 化工学报, 2004, 55(10): 1573-1580 |
| [4] | Lee JW, Na D, Park JM, et al. Systems metabolic engineering of microorganisms for natural and non-natural chemicals[J]. Nature Chemical Biology, 2012, 8(6): 536-546 |
| [5] | Tomoya B, Takeshi A, Miki H, et al. Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection[J]. Molecular System Biology, 2006, 2: 1-11 |
| [6] | Huruma NT, Bruno MG. Use of lambda Red-mediated recombineering and Cre/Lox for generation of markerless chromosomal deletions in avian pathogenic Escherichia coli[J]. Federation of European Microbiological Societies Microbiology Letters, 2011, 325(2): 140-147 |
| [7] | Datsenko KA, Wanner BL. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products[J]. Proceedings of the National Academy of Sciences of the United States of America, 2000, 97(12): 6640-6645 |
| [8] | Murphy KC. Lambda Gam protein inhibits the helicase and chi O’Stimulated recombination activities of Escherichia coli RecBCD enzyme[J]. Journal of Bacteriology, 1991, 173(18): 5808-5821 |
| [9] | Poteete AR. What makes the bacteriophage lambda Red system useful for genetic engineering: molecular mechanism and biological function[J]. Federation of European Microbiological Societies Microbiology Letters, 2001, 201(1): 9-14 |
| [10] | David IF, Donald LC. Bacteriophage lambda: alive and well and still doing its thing[J]. Current Opinion in Microbiology, 2001, 4(2): 201-207 |
| [11] | Chan WS, Sang YL. Rapid one-step inactivation of single or multiple genes in Escherichia coli[J]. Biotechnology Journal, 2013, 8: 776-784 |
| [12] | Thakker C, Martinez I, San KY, et al. Succinate production in Escherichia coli[J]. Biotechnology Journal, 2012, 7(2): 213-224 |
| [13] | Park JH, Oh JE, Lee KH, et al. Rational design of Escherichia coli for L-isoleucine production[J]. American Chemical Society Synthetic Biology, 2012, 1(11): 532-540 |
| [14] | Hu FX, Ding R, Cui ZH, et al. Approaches and strategies of gene scarless knockout in the Escherichia coli genome[J]. Letters in Biotechnology, 2013, 24(4): 552-557 (in Chinese) 胡逢雪, 丁锐, 崔震海, 等. 大肠杆菌基因无痕敲除技术及策略[J]. 生物技术通讯, 2013, 24(4): 552-557 |
| [15] | Doublet B, Douard G, Targant H, et al. Antibiotic marker modifications of λ Red and FLP helper plasmids, pKD46 and pCP20, for inactivation of chromosomal genes using PCR products in multidrug-resistant strains[J]. Journal of Microbiological Methods, 2008, 75(2): 359-361 |
| [16] | Mahesh M, Hend AA, Zdeno L. Gene replacement techniques for Escherichia coli genome modification[J]. Folia Microbiologica, 2011, 56(3): 253-263 |
| [17] | James P, Gregory JP. New pSC101-derivative cloning vectors with elevated copy numbers[J]. Plasmid, 2008, 59: 193-201 |
| [18] | Tamotsu H, Mutsuo S. Mutations to temperature sensitivity in R plasmid pSC101[J]. Journal of Bacteriology, 1977, 31(2): 405-412 |
| [19] | Sambrook J, Russell DW. Molecular Cloning: A Laboratory Manual[M]. Translated by Huang PT. 3rd Edition. Beijing: Science Press, 2002: 68-70 (in Chinese) 萨姆布鲁克J, 拉塞尔DW. 分子克隆实验指南[M]. 黄培堂, 译. 第3版. 北京: 科学出版社, 2002: 68-70 |
| [20] | Nobuaki S, Hiroshi N, Yota T, et al. New multiple-deletion method for the Corynebacterium glutamicum genome, using a mutant Lox sequence[J]. Applied and Environmental Microbiology, 2005, 71(12): 8472-8480 |
| [21] | Merryman C, Gibson DG, Methods and applications for assembling large DNA constructs[J]. Metabolic Engineering, 2012, 14(3): 196-204 |
 2015, Vol. 42
2015, Vol. 42




