扩展功能
文章信息
- 冯娟, 李荷
- FENG Juan, LI He
- 源于红树林土壤宏基因组文库的新型磷脂酶A1 基因的筛选、克隆表达及酶学性质
- Cloning, expression and characterization of a novel phospholipase A1 gene from the metagenomic library of mangrove soil
- 微生物学通报, 2015, 42(3): 489-496
- Microbiology China, 2015, 42(3): 489-496
- 10.13344/j.microbiol.china.140542
-
文章历史
- 收稿日期: 2014-07-11
- 接受日期: 2014-07-30
- 优先数字出版日期(www.cnki.net): 2014-09-18
1998年,Handelsman等[1]首次提出“宏基因组”的概念,即特定生态环境中所有生物遗传物质的总和,目前宏基因组学已经被广泛用于挖掘新型生物催化剂中。宏基因组的产生克服了传统微生物分离培养的难题,扩大了微生物资源的利用。利用宏基因组学技术已经筛选到多种新型生物催化剂[2, 3, 4, 5],使得宏基因组成为微生物活性物质筛选的新途径[6]。
磷脂酶来源不同,其与磷脂的作用方式也不同,根据其作用位点的差异(图1)可以将磷脂酶分为磷脂酶A1、磷脂酶A2、磷脂酶C和磷脂酶D四种,其中磷脂酶A1和磷脂酶A2能应用于植物油脱胶,它们能特异性的水解磷脂的1位或2位脂肪酸链[7],其中磷脂酶A2必须以钙离子为激活剂,且来源有限,价格昂贵。因此,磷脂酶A1日益受到研究者的关注,对磷 脂酶A1应用于植物油脱胶的研究越来越深入[8, 9, 10, 11]。传统的油脂精炼由于脱胶不完全导致精炼损失大,对设备影响也较大,且易造成成品油酸败和回色现象[7],而利用磷脂酶A1的酶法脱胶反应条件温和,大大节约化学物质的消耗量,几乎不产生废水,在工业应用中具有潜在优势,但是磷脂酶A1在天然材料中含量低以及其理化性质的缺陷无法完全满足生产的要求,因此获得高产量且适合工业生产应用的磷脂酶迫在眉睫。目前对于磷脂酶的研究,主要是通过筛选性能优良的磷脂酶产生菌,但是环境中的微生物大部分是不可培养的,大大限制了微生物磷脂酶资源的利用。
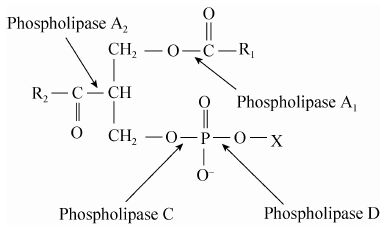
|
| 图1 不同磷酸酶作用的位点 Figure.1 Hydrolysis sites of different phospholipase |
本研究通过宏基因组学的方法,利用功能筛选方法以三丁酸甘油脂为底物,从红树林这一特殊生境中筛选新型酯酶,而磷脂酶是酯酶的一种,也可以三丁酸甘油脂为底物,所以同时也可用于磷脂酶的筛选。本研究中筛选到的磷脂酶能水解三丁酸甘油脂且不需钙离子作激活剂,所以为磷脂酶A1类。对筛选到的磷脂酶A1进行酶学性质分析,为其工业化应用奠定基础。
1 材料与方法 1.1 材料土样采自广东珠海琪澳岛沿海红树林,采集深度10-20 cm的表层土样,于-20 °C保存。Escherichia coli DH5α和原核表达载体pET-32a(+)由本实验室保存。载体pUC118/BamH I (BAP)、BamHⅠ、Hind Ⅲ、EcoRⅠ、PstⅠ、Prime STARTM Max Premix、Protein marker及DNA marker购自TaKaRa公司;氨苄青霉素(Amp)、X-gal (5-溴-4- 氯-3-吲哚-α-D-半乳糖苷)、IPTG (异丙基-β-D-硫代吡喃半乳糖苷)购自Amresco公司;Plasmid Mini Kit、DNA Gel Extraction Kit购自OMGEA公司;三磷酸甘油酯(Tributyrin)购自阿拉丁;p-nitrophenyl acetate (C2)、p-Nitrophenyl butyrate (C4)、p-Nitrophenyl caproate (C6)、p-Nitrophenyl octanoate (C8)、p-Nitrophenyl caprate (C10)、p-Nitrophenyl laurate (C12)、p-Nitrophenyl myristate (C14)、p-Nitrophenyl palmitate (C16)、乙腈购自Sigma公司。
1.2 方法 1.2.1 提取环境样品宏基因组
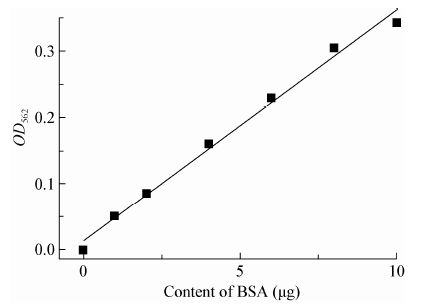
|
| 图2 蛋白质标准曲线 Figure.2 The standard curve of protein |
获得高纯度、高浓度的基因组DNA是构建宏基因组文库的前提。本研究土壤基因组DNA提取结果如图3所示,提取的宏基因组DNA主要集中在23 kb以上,且较少降解,满足构建宏基因组文库的需要。
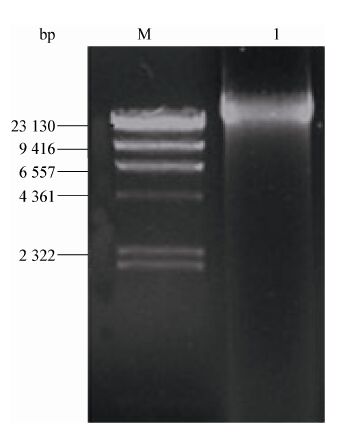
|
| 图3 土壤基因组DNA的提取电泳图 Figure.3 Electrophoresis identification of soil genomic DNA Note: M: λDNA/Hind III DNA marker; 1: soil genomic DNA. |
不完全酶切后的基因组片段与pUC118载体相连后,采用电击方法转化入E. coli DH5α超级感受态中,蓝白斑平板显示转化情况良好,计算该文库约含50 000个阳性克隆子,文库含量较多,随机挑取20个阳性克隆子提质粒、BamH I酶切验证,电泳检测插入片段的大小及多样性。插入片段平均大小约3.5 kb,且文库基因片段的多样性较好,文库质量符合后期筛选要求。
2.3 磷脂酶基因的筛选以1%三丁酸甘油酯为底物和罗丹明B为背景染料配制磷脂酶筛选培养基,对宏基因组文库进 初筛,筛选出一株有明显透明圈的克隆,初步确定其有水解活性。后期通过提质粒、酶切验证后送Invitrogen公司测序。
2.4 磷脂酶基因phop1413的序列分析经NCBI中的ORF Finder进分析,插入片段中含有一个全长1 413 bp的磷脂酶基因,该基因命名为phop1413,已将其序列提交到GenBank获得登录号为KF767097。该基因可编码一个由470个氨基酸残基组成的、预计分子量为51.7 kD的磷脂酶蛋白。经NCBI的BLAST软件分析Phop1413与已报道的磷脂酶Pseudomonas (WP 018928790.1),序列相似性最大为42% ,所以我们将其归为磷脂酶类。而采用MEGA软件对Phop1413与已知报道的酯水解酶类做进化关系分析,构建系统发育树(图4),结果显示Phop1413属于fAMILY Ⅵ家族。
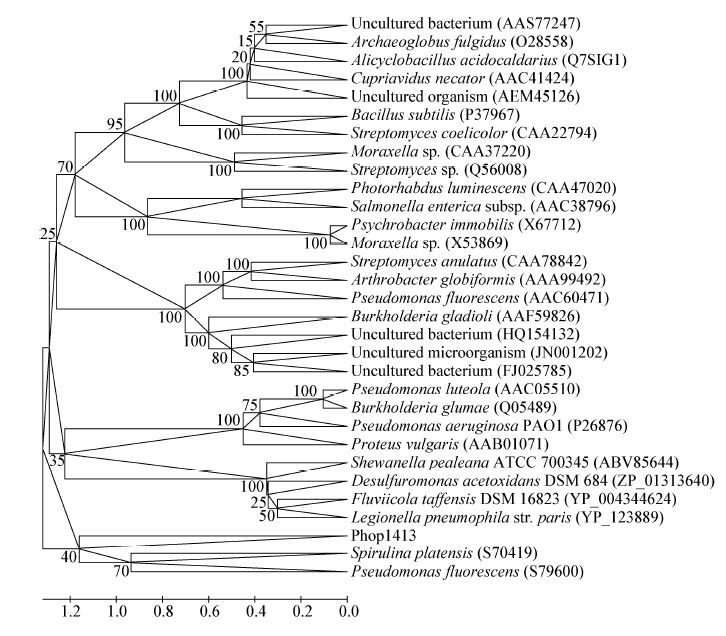
|
| 图4 phop1413与亲缘性较近蛋白的系统进化树 Figure.4 Unrooted neighbor-joining phylogenetic analysis of Phop1413 |
以重组质粒pUC118-phop1413为模板,以phop1 545-Fw/phop1 545-Rv为引物扩增,得到一条明显的特异性DNA条带(图5),片段大小在1 400 bp左右,与预期相符。
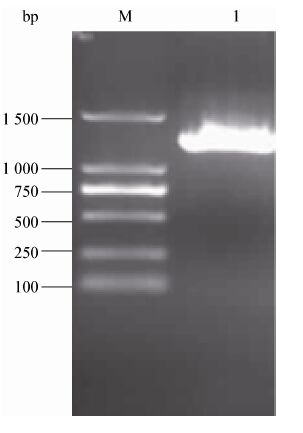
|
| 图5 基因phop1413的PCR扩增产物 Figure.5 Amplified products of gene phop1413 Note: M: DL2000 DNA marker; 1: gene phop1413. |
将重组质粒pET-32a(+)-phop1413转化至E. coli BL21感受态细胞,IPTG诱导后30 °C表达16 h,超声裂解菌体,SDS-PAGE电泳分析表达产物,如图6A所示。在65 kD处有明显的蛋白条带,与理论预测大小相符,说明phop1413基因正确表达。纯化后的SDS-PAGE电泳检测结果如图6B所示,为单一条带,分子量大小与预测相符。可见,该重组蛋白表达量较高,采用BCA法测得 蛋白表达量高达220 mg/L。
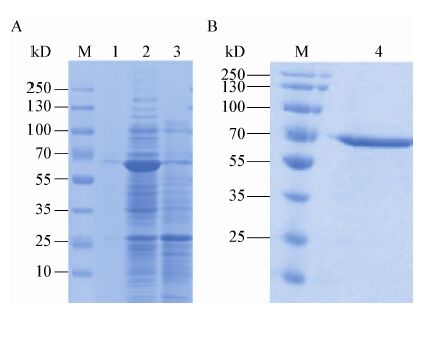
|
|
图6
E. coli BL21(pET-32a(+)-phop1413)表达(A)及纯化(B)结果的SDS-PAGE鉴定
Figure.6
SDS-PAGE of gene expression in E. coli BL21 (pET-32a(+)-phop1413)
注:M:标准量分子蛋白;1:诱导液上清蛋白;2:超声破碎后上清蛋白;3:阴性对照[E. coli BL21(pET-32a(+)]的破碎液;4:纯化的Phop1413. Note: M: Protein MW marker; 1: centrifugal supernatant of fermentation; 2: the cells lysis solution of E. coli BL21 (pET-32a(+)-phop1413); 3: the cells lysis solution of E. coli BL21(pET-32a(+)); 4: purified recombinant Phop1413. |
如表1所示,Phop1413对C2-C16的对硝基苯酚酯有不同的水解活性,其中对对硝基苯酚己酸酯(C6)活性最高,计算其比酶活为124 U/mg。该酶对C2-C16底物都具有水解活性,表明该重组酶水解底物范围较广,由于对C14仍具有较高活性,推测该磷脂酶也具有部分脂肪酶活性,更适合用于油脂精炼。
| 底物 Substrates | 相对酶活 Relative activity (%) |
| C2 | 71.2±3.6 |
| C4 | 55.3±2.9 |
| C6 | 100.0±4.3 |
| C8 | 15.4±1.3 |
| C10 | 33.3±2.7 |
| C12 | 41.6±2.4 |
| C14 | 74.3±3.7 |
| C16 | 39.1±2.2 |
从图7中可以看出,随着温度的升高,酶活性逐渐上升,且在54 °C时达到最大值,因此该重组蛋白的最适温度为54 °C,且温度在40-65 °C时,相对酶活性仍保持50%以上,说明该重组蛋白较耐高温。在40、45、50、60 °C温育不同时间,重组酶相对活力呈现下降趋势,50 °C温育1.5 h后残余酶活为44%,45 °C温育2.5 h后仍残余45.3%,表明该重组酶有较好的热稳定性。

|
| 图7 温度对重组酯酶Phop1413活性(A)及稳定性的影响(B) Figure.7 Effect of temperature on activity (A) and stability (B) of Phop1413 |
如图8所示,当pH小于5.5时,Phop1413的酶活性随着pH值的升高而增加;pH达到7.8时,酶活性最高;之后随着pH值的升高,酶活性逐渐降低,当pH为4.0或10.0时该酶几乎没有活性,pH为6.0时,温育2.5 h后仍残存50%以上酶活, pH为8.0时,温育1 h后,酶活仅剩16%,说明该重组酶对酸的耐受性比对碱的耐受性好。

|
| 图8 pH对重组酶Phop1413活性(A)及稳定性的影响(B) Figure.8 Effect of pH on activity (A) and stability (B) of Phop1413 |
新开发的脱胶工艺,主要是采用酸脱胶、化学添加剂络和法脱胶、酶法脱胶及超滤脱胶等方 法[14]。酶法脱胶广泛应用于各种植物油,与其他脱胶方法相比,该法脱胶条件温和,营养成分不被破坏,而且节省能源,且由于酶专一性强,副反应少,产品容易回收[15],目前酶法脱胶技术已经引起了世界油脂工业界的高度重视。但是脱胶工业生产过程的高温、高压、高酸度以及重金属离子超标等造成的极端环境,使得野生的磷脂酶很难满足工业生产的需要[16]。
长期生活在特定环境中的微生物具有特有的酶学特性,红树林是自然分布于热带、亚热带海岸的潮间带的木本植物群落,处于海洋与陆地的动态交界面,该地区的微生物由于长期处于湿热环境,环境中的土著菌及其酶类具有耐热等特质。本研究中的新型磷脂酶Phop1413的最适反应温度为54 °C,这比大多数已报道的磷脂酶最适温度都要高。如Gomes Heleno等[17]所研究的磷脂酶最适温度为35-45 °C,Mo等[18]研究的磷脂酶C最适温度为45 °C;该酶最适反应pH为7.8,与大多数已报道的磷脂酶相差不大;此外该酶较其他已报道的磷脂酶有更好的热稳定性以及在弱酸环境中稳定性更好的特性。但由于工业化油脂精炼生产中的极端环境,下一步我们将对该重组蛋白进行定向进化分子改造,以期获得对温度和pH耐受性更好的酶;其次通过固定化技术获 得性质更稳定的磷脂酶,从而为工业化应用奠定基础。
| [1] | Handelsman J, Rondon MR, Brady SF, et al. Molecularbiological access to the chemistry of unknown soil microbes: anew frontier for natural products[J]. Chemistry & Biolology,1998, 5(10): 245-249 |
| [2] | Jiang CJ, Chen G, Huang J, et al. A novel beta-glucosidase withlipolytic activity from a soilmetagenome[J]. FoliaMicrobiologica (Praha), 2011, 56(6): 563-570 |
| [3] | Ko KC, Rim SO, Han Y, et al. Dentification and characterizationof a novel cold-adapted esterase from a metagenomic library ofmountain soil[J]. Journal of Industrial Microbiology andBiotechnology, 2012, 39(5): 681-689 |
| [4] | Jimenez DJ, Montana JS, Alvarez D, et al. A novel cold activeesterase derived from Colombian high Andean forest soilmetagenome[J]. World Journal of Microbiology andBiotechnology, 2012, 28(1): 361-370 |
| [5] | Tang XX, Yi ZW, Li N, et al. Bioactivity screening offermentation crude extract from deep sea metagenomic libraryclones[J]. China Biotechnology, 2011, 31(6): 58-63 (in Chinese)汤熙翔, 易志伟, 李宁, 等. 深海宏基因组文库克隆子发酵产物的生物活性筛选[J]. 中国生物工程杂志, 2011, 31(6):58-63 |
| [6] | Yan B, Hong K, Xu Y, et al. Metagenome cloning—a newapproach for novel microbial bioactive compounds discovery[J].Microbiology China, 2005, 32(1): 113-117 (in Chinese)阎冰, 洪葵, 许云, 等. 宏基因组克隆—微生物活性物质筛选的新途径[J]. 微生物学通报, 2005, 32(1): 113-117 |
| [7] | Li QS, Yang JG, Yang B, et al. Vegetable oil degumming bydifferent phospholipases[J]. China Oils and Fats, 2004, 29(1):19-22 (in Chinese)李秋生, 杨继国, 杨博, 等. 不同磷脂酶用于植物油脱胶的研究[J]. 中国油脂, 2004, 29(1): 19-22 |
| [8] | Loeffler F, Plainer H, Sproessler B. Vegetable oil enzymaticdegumming process by means of Aspergillus phospholipase: US,6001640[P]. 1999 |
| [9] | Clausen IG, Patkar SA, Borch K, et al. Method for reducingphosphorus content of edible oil: US, 6103505[P]. 2000 |
| [10] | Hasida M, Tsutsumi N, Halkier T, et al. Acid phospholipase,production and method using thereof: US, 6127137[P]. 2000 |
| [11] | Roy SK, Rao BVSK, Prasad RBN. Enzymatic degumming ofrice bran oil[J]. Journal of the American Oil Chemists Society,2002, 79: 845-846 |
| [12] | Zhou J, Bruns MA, Tiedje JM. DNA recovery from soils ofdiverse composition[J]. Applied and Environment Microbiology,1996, 62(2): 316-322 |
| [13] | Sambrook J, Russell DW. Molecular Cloning: A LaboratoryManual[M]. Translated by Huang PT. 3rd Edition. Beijing:Science Press, 2005: 98-102 (in Chinese)萨姆布鲁克J, 拉塞尔DW. 分子克隆实验指南[M]. 黄培堂,等译. 第3版. 北京: 科学出版社, 2005: 98-102 |
| [14] | Zhao ZG, Liu XL, Liu RF. The tecnology of oil degummingprocess[J]. Cereals Oils, 2004(1): 3-8 (in Chinese)赵国志, 刘喜亮, 刘锐锋. 油脂脱胶技术[J]. 粮食与油脂,2004(1): 3-8 |
| [15] | Wang YY, Luan X, Wei CP, et al. Application of enzymetechnology in oil processing industry[J]. China Oils and Fats,2010, 35(7): 8-11 (in Chinese)王瑛瑶, 栾霞, 魏翠平, 等. 酶技术在油脂加工业中的应用[J].中国油脂, 2010, 35(7): 8-11 |
| [16] | Liang L, Chang M, Liu RJ, et al. Research progress inphospholipase[J]. Science and Technology of Food Industry,2013, 34(4): 393-396 (in Chinese)梁丽, 常明, 刘睿杰, 等. 磷脂酶研究进展[J]. 食品工业科技,2013, 34(4): 393-396 |
| [17] | Gomes Heleno MA, Baldasso PA, Ponce-Soto LA, et al.Biochemical characterization and pharmacological properties ofnew basic PLA2 BrTX-I isolated from Bothrops roedingeri(Roedinger’s Lancehead) mertens, 1942, snake venom[J].BioMed Research International, 2013: 591470 |
| [18] | Mo S, Kim JH, Cho KW. Enzymatic properties of anextracellular phospholipase C purified from a marineStreptomycete[J]. Bioscience Biotechnology and Biochemistry,2009, 73(9): 2136-2137 |
 2015, Vol. 42
2015, Vol. 42




