扩展功能
文章信息
- 耿丹丹, 卢培利, 李微薇, 张代钧
- 硝态氮还原型厌氧甲烷氧化的研究进展及展望
- Nitrate/nitrite-dependent anaerobic methane oxidation: review on status and perspectives
- 微生物学通报, 2015, 42(2): 364-373
- Microbiology China, 2015, 42(2): 364-373
- 10.13344/j.microbiol.china.140441
-
文章历史
- 收稿日期: 2014-05-26
- 接受日期: 2014-08-13
- 优先数字出版日期(www.cnki.net): 2014-09-11
2. 重庆大学 煤矿灾害动力学与控制国家重点实验室 重庆 400044
2. State Key Laboratory of Coal Mine Disaster Dynamics and Control,Chongqing University,Chongqing 400044,China
甲烷的温室效应是等摩尔CO2的20−30倍[1]。全球甲烷年产生量大约为300 Tg[2],其中90%在进入大气前可能被微生物厌氧氧化(Anaerobic methane oxidation,AMO),减缓了其对气候变暖的贡献[3, 4]。以硫酸盐为电子受体的AMO (S ulphate-dependent anaerobic methane oxidation,SAMO)于2003年得到证实[5],其对海洋中甲烷氧化的贡献在75%以上[6]。由于淡水缺氧环境中的电子受体主要是硝态氮,且从热力学和生物化学角度,硝态氮比硫酸盐更适合作为甲烷氧化的电子受体[7, 8, 9, 10, 11, 12, 13],硝酸盐型AMO (nitrat e dependent AMO,N-damo)和亚硝酸盐型AMO (nitrite dependent AMO,n-damo),统称硝态氮型AMO (Nx-damo)更具存在基础[14]。其化学方程式表述如下:
|
5CH4+8NO3−+8H+→5CO2+4N2+14H2O ΔG0′=−765 kJ/mol CH4 |
(1) |
|
3CH4+8NO3−+8H+→3CO2+4N2+10H2O ΔG0′=−928 kJ/mol CH4 |
(2) |
2006年,Nx-damo过程被首次证实。但Nx-damo微生物的世代周期更长(几周到100 d以上)[15],富集时间一般在8-16个月[16, 17, 18, 19],且富集达到一定细胞密度后便停止[20],限制了研究的广泛开展。目前,关于Nx-damo微生物的生理特性、代谢机理、自然分布及作用、影响富集的因素以及工程应用潜力等问题尚不清楚。然而,推进该领域的研究有利于对C、N元素地球化学循环的认识,理解自然生态系统甲烷排放及其对全球气候变暖的贡献[21],也能够为废水生物脱氮新技术的开发、解决碳源不足问题提供新途径[22]。本文从功能微生物富集及其影响因素、生理特性、代谢机理等方面对Nx-damo的最新进展进行了梳理;评估了其应用于废水处理的潜力和优势;对未来的研究进行了展望,以期推动该领域更广泛的研究并为其提供有价值的参考。
1 Nx-damo功能微生物及其富集与分布 1.1 Nx-damo功能微生物及其富集研究者从20世纪70年代开始了富集Nx-damo微生物的尝试[23, 24, 25, 26],但直到2006年和2013年才分获成功[17, 27]。2006年,Raghoebarsing等以运河底泥为接种物、 以CH4/NO3−/NO2−为基质成功富集N-damo功能微生物,同位素标记证实古菌和细菌都参与了反应[17];16S rRNA的系统发育表明该细菌归属于非培养的、只能通过环境基因序列定义的新亚门——NC10门细菌,古菌在系统发育中的归类介于甲烷丝状菌(Methanosaeta)和ANME-II之 间[28]。根据细菌和古菌16S rRNA序列设计的FISH探针检测到了这2类功能微生物的存在,其细胞数量比为8:1,且古菌处于细菌的包围之中。细菌呈细杆状、1 μm长、0.3 μm宽、DNA聚集在细胞的中心;古菌呈球形、聚集成3−6 μm的圆形;对富集物的MALDI-TOF分析只发现了来自产甲烷菌、分子质量为905 Da的辅因子F430,没有发现与甲烷营养型古菌ANMEⅠ相关的分子质量为951 Da的辅因子F430[28]。随后继续富集15个月后,古菌消失,但反应活性并未降低[28]。以富营养化的沟渠沉积物接种,同样富集到NC10门细菌,细胞呈(0.8−1.0) μm×(0.3−0.5) μm的杆状,以单细胞或者聚合体的形式存在[18< /span>]。这类细菌被命名为“Candidatus Methylomirabilis oxyfera”,一种嗜温的(富集温度25−30 °C、pH 7.0−8.0)、生长缓慢(倍增时间1−2周)的革兰氏阴性杆状菌,长度0.8−1.1 μm、直径 0.25−0.50 μm;并获得了其全基因组和pmoA蛋白序列的系统发育(图 1);基于GC含量和优势度的鉴定归类分析表明,富集物的焦磷酸测序所得的宏基因组数据有60%与M. oxyfera相关[29]。它代表了甲烷氧化菌中一种新类群,与疣微菌门一起,是目前发现唯一的非变形杆菌的甲烷氧化菌[18]。2009年,Hu等以淡水湖沉积物、消化污泥、回流污泥的混合物为接种物,以CH4/NO3−为基质在不同温度下富集得到Nx-damo微生物[19]。2014年,Kampman等最新报道以污水处理厂污泥为唯一接种物,以CH4/NO2−为基质,在20 °C时也能成功富集细菌M. oxyfera[30]。

|
| 图 1 “M. oxyfera” pmoA蛋白系统发育[29] Figure 1 Phylogeny of “M. oxyfera”pmoA protein sequences[29] 注:邻接树显示出富集培养物“Twente”和“Ooij” (粗体)相对于其他pmoA 和amoA序列的位置. 距离树通过Dayhoff矩阵计算,bootstrapping重复值为100. 通过邻接法,最小进化法,最大简约法和最大似然进化法这4种方法来构建进化树. Bootstrapping结果总结在进化树上,实心圆代表在所有这4种方法中得到大于70%支持的分支点. 定标线条代表每100个氨基酸中有50个氨基酸改变. Note: Neighbour-joining tree showing the position of enrichment cultures “Twente” and “Ooij” (in bold) relative to other pmoA and amoA sequences. The distance tree was computed with the Dayhoff matrix-based method,and bootstrapping of 100 replicates was performed with in the neighbour-joining,minimum-evolution,maximum-parsimony and maximum-likelihood evolutionary methods. Bootstrapping results are summarized on the tree,with filled circles representing branch points at which all four methods give greater than 70% support. The scale bar represents 50 amino-acid changes per 100 amino acids. |
与上述结果不同,Haroon等最近报道以CH4/NO3−/NH4+为基质,富集到了AMO古菌ANME-2d和Anammox细菌的混培物,前者以CH4为电子供体、以NO3−为电子受体驱动N-damo过程厌氧氧化甲烷并将硝酸盐还原为亚硝酸盐,后者利用NH4+和生成的NO2−发生Anammox过程将NO2−还原为N2。N-damo与anammox耦合的发现,初步揭示了古菌在Nx-damo过程中的作用[27]。该古菌被命名为Methanoperedens nitroreducens,归属于Methanoperedens科,是一种直径1−3 μm的不规则球菌,通常以类似八叠球菌的族群方式存在,中温(22−35 °C)、pH 7.0−8.0条件下生长[27]。其形态如图 2所示。
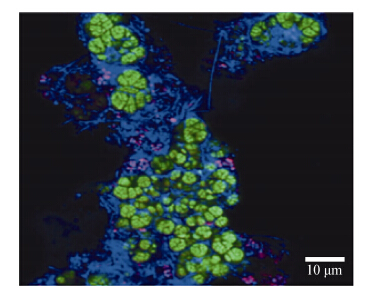
|
| 图 2 原位荧光杂交下反应器微生物组成显微照片[27] Figure 2 Fluorescence in situ hybridization micrograph of the bioreactor community[27] 注:高密度团体“M. nitroreducens”占主导数量(绿色),少量为Kuenenia细胞(红紫色)和其他细菌(蓝色). Note: Showing the dominant “M. nitroreducens” population in large,dense clusters (green),smaller flanking Kuenenia cells (magenta) and other bacteria (blue). |
目前公开报道的Nx-damo功能微生物实验室富集研究(表 1),主要以M. oxyfera驱动的n-damo为主,尚未有针对富集影响因素和条件优化的专门研究。这里通过不同研究者富集条件和结果的对比,梳理出可能的影响因素,以期为以后的富集提供参考。
| 接种物 Inoculum | 氮素供给 N feeding | 温度 Temperature (°C) | 富集时间 Enriching time (d) | 微生物组成 Composition (%) | 消耗速率 Conversion rate (mg NOx−-N/ (L·d)) | 文献 Reference | |
| 细菌 Bacteria | 古菌 Archaea | ||||||
| Canal sediments | NO3−/NO2− | 25 | 480 | 80 | 10 | NO2−: 14.98 | [17] |
| Ditch sediments | NO3−/NO2− | 30 | 270 | 70 | 0 | NO2−: 30.10 | [18] |
| Mixed inoculum | NO3− | 35 | 312 | 30 | 40 | NO3−: 28.00 | [19] |
| NO3− | 22 | 260 | 15 | 0 | NO3−: 0.91 | [19] | |
| Canal sediments | NO3−/NO2− | 30 | 270 | 70 | 0 | NO2−: 9.80 | [28] |
| Wastewater sludge | NO2− | 20±1 | 421 | 60-70 | 0 | NO2−: 36.00 | [30] |
| Wastewater sludge | NO2− | 20−23 | 315 | 60-70 | 0 | NO2−: 15.40 | [36] |
| Ditch sediments | NO2− | 30 | 730 | 70-80 | 0 | NO2−: 33.50,37.80 | [43] |
现有用于M. oxyfera细菌多样性和分布检测的分子生物学方法正在逐步增多和完善。16S rRNA和pmoA基因分子生物标记被用于确认NC10门细菌有关M. oxyfera细菌的多样性检测。各种PCR技术也被用来确定M. oxyfera类似细菌的丰度。
根据实验室富集的n-damo功能微生物设计出的探针或引物促进了对自然环境中M. oxyfera细菌基因水平的直接检测,DGGE、实时PCR检测发现淡水深层湖泊底泥浅层[45, 49]和内陆浅水湖泊底 泥[39, 50]中均含有相当数量的n-damo微生物,是最适合这种微生物生存的环境;最近研究表明受农业径流污染的淡水河流底泥含有更高多样性的M. oxyfera细菌[39],氨和有机物含量等环境因素也会影响M. oxyfera细菌的分布,M. oxyfera菌可以适应动态环境条件,长期施氮肥的稻田土也有n-damo菌的分布[37]。通过同位素标记在含高浓度硝酸盐地下水渗透的泥炭土中检测到了n-damo过程,PCR定量表明NC10细菌细胞数量最高点所在深度与甲烷、NO3−过渡区一致。16S rRNA和pmoA基因系统发育分析发现其功能微生物与M. oxyfera的同一性低于96%和90%,推测可能是另一种不同于M. oxyfera的NC10门细菌,实验室以此泥炭土接种也成功富集了该细菌[38]。同时在多个污水处理厂污泥中也直接检测到了M. oxyfera细菌。ANME-2d古菌的发现也展示了此类功能微生物潜在的多样性。
研究者分别以运河底泥[17]、沟渠沉积物[18]、污水处理厂污泥[36]、湖泊底泥[19]等为接种物成功富集M. oxyfera细菌间接表明了其分布情况,通过接种物与富集培养物中NC10细菌丰度的对比,发现成功富集的是NC10细菌中的一个亚种,该亚种占主导地位的菌群有更高的n-damo活性[36],而这种选择性物种富集的过程会造成生物多样性的减少。
由于方法的限制,这些微生物的环境分布和重要性还不能被完全理解。n-damo过程是NC10门细菌的一种共性,还是只有M. oxyfera细菌才能驱动,当前研究尚不清楚。所有这些AMO都由目前发现的三类微生物驱动的可能性极小。与Nx-damo类似的S-damo就至少有ANME-1、ANME-2和ANME-3三类古菌参与[51]。因此,随着工作的开展,发现新的Nx-damo功能微生物是完全有可能的。
2 Nx-damo微生物菌群的作用机理富集难度限制了对Nx-damo过程机理的研究,从最初认为在古菌和细菌协同作用下的反向产甲烷机理,到其后的细菌通过胞内产氧机制独自好氧氧化甲烷和还原亚硝酸盐,直到新近提出的古菌通过反向产甲烷机理独自完成甲烷氧化和硝酸盐至亚硝酸盐的还原。
2.1 细菌M. oxyfera胞内产氧好氧氧化甲烷 机理研究认为M. oxyfera细菌能够独立完成甲烷厌氧氧化和亚硝酸盐还原。该细菌的蛋白质组学和转录组学使我们对甲烷和NO2−转换的中心路径进行推测,全基因组显示出其明显的好氧甲烷氧化途 径[20];随后的同位素标记实验证实其将反硝化产生的2个NO分子通过歧化反应生成氮气和氧气,其中3/4的O2用于氧化甲烷(图 3);进一步的检测发现M. oxyfera细菌细胞中存在还原硝酸盐、亚硝酸盐、NO的功能基因,但没有发现编码N2O还原酶的基因[29, 52]。Ettwig等认为,在没有氧气和光照的情况下,M. oxyfera细菌可以将NO分解为N2和O2,然后用生成的O2通过甲烷单加氧酶的催化氧化甲烷获取能量[52]。细菌内部有一种隐秘的需氧菌,它通过周围的NO2−来获取氧气,而不是从大气中获得[47]。因此,可以认为n-damo过程本质是甲烷好氧氧化。然而,这一胞内产氧机理仍面临诸多挑战,包括没有检测到催化NO歧化反应的酶[52]、外加2%或8%的O2降低了M. oxyfera细菌的活性及硝酸盐和亚硝酸盐还原酶基因表达等[48]。一种可能的解释是M. oxyfera是一个缓慢生长和代谢的微生物,电子传递系统能力很低,过剩O2不能使电子完全转移[48]。
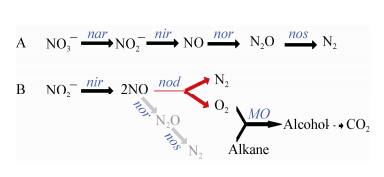
|
| 图 3 公认的反硝化途径(A)和推测的NO歧化反应产N2和O2途径(B)[52] Figure 3 Pathways of canonical denitrification (A) and proposed N2 and O2 production by NO dismutation (B)[52] 注:nar:硝酸盐还原酶;nir:亚硝酸盐还原酶;nor:NO还原酶;nos:N2O还原酶;nod:NO歧化酶;MO:单加氧酶. Note: nar: nitrate reductase; nir: nitritereductase; nor: nitric oxide reductase; nos: nitrous oxide reductase; nod: nitric oxide dismutase; MO: monooxygenase. |
Haroon等在氨与硝酸盐共存条件下富集得到了古菌ANME-2d与anammox细菌的共生物,FISH和16S rRNA分析揭示其中古菌占78%;研究认为古菌驱动了AMO同时将硝酸盐还原为亚硝酸盐,并将其命名为M. nitroreducens[27]。宏基因组和单细胞基因组揭示M. nitroreducens基因组中包含完全的产甲烷途径所需的mcrABCDG和mer基因。 完整的还原性乙酰辅酶A途径及其合成酶的存在表明M. nitroreducens可能像对ANME-1预计的那样能产生乙酸[53, 54]。宏转录显示甲烷氧化为二氧化碳所需的全部基因均高度表达;C13同位素标记实验证实投加的甲烷转化为CO2。在古菌中没有发现除硝酸盐还原酶以外的反硝化酶。这些实验证实了M. nitroreducens的反向产甲烷机理及其对硝酸盐的部分还原(图 4)。进一步的同位素标记实验和分子生物学检测也证实了其中的Anammox细菌的 协同作用[27]。在氨存在的情况下,anammox菌和M. oxyfera细菌竞争NO2−,前者的竞争优势限制了后者的生 长[27],因此无法得知古菌能否与M. oxyfera菌协同形成稳定的N-damo过程,或者古菌是否能够独立完成AMO驱动的硝酸盐完全还原。
废水生物脱氮缺乏碳源是普遍难题,投加有机物可能带来经济负担和二次污染。同时,实验证实大量甲烷溶解在厌氧生物处理出水中[14, 55]导致了温室气体排放[56, 57, 58]和低效能源回收,已经成为当前废水厌氧生物处理技术广泛应用的障碍[14]。尽管相对于传统的硝化-反硝化工艺,Anammox具有更低的能耗、更少的碳源需求和更低的污泥产量等优 势[56,59-61],但是亚硝化的控制一直是个难题[57]。Nx-damo耦联了硝态氮还原与甲烷氧化,基于这一原理的废水处理技术,能够为生物脱氮提供经济、清洁的碳源;同时能够有效利用溶解性甲烷产生更少的N2O[29],减少废水处理过程温室气体排放,并且减少90%污泥处理成本[16]。
就目前的研究进展来看(表 2),Anammox细菌比M. oxyfera细菌更容易富集,在氨存在的条件下,能够与M. nitroreducens古菌协同,更快地去除后者还原硝酸盐的产物亚硝酸盐;而后者能够去除Ananmox过程产生的硝酸盐,实现甲烷厌氧氧化、硝酸盐还原和Anammox的耦合,达到M. nitroreducens古菌的更快富集和获取更高的脱氮速率。在没有氨的条件下,M. oxyfera细菌能够独自完成甲烷厌氧氧化和亚硝酸盐还原,或者替代Anammox与M. nitroreducens古菌协同完成甲烷厌氧氧化和硝酸盐还原,只是速率更慢。Anammox细菌与M. oxyfera细菌已经被发现可以在自然生态系统共存[21, 37],在实验室中共培养[42, 44, 58]。因此,M. nitroreducens古菌、M. oxyfera细菌和Anammox细菌的协同,能够处理任何三氮比例的废水。Kampman等应用N-damo处理污泥消化UASB反应器出水,亚硝酸盐最大消耗速率达到 37.8 mg N/(L·d)[43];但可能由于污泥流失原因,反应速度无法继续提高,反而下降;认为反硝化速率需要提高10倍以上才能考虑工程应用。但是,Luesken等在SBR反应器内利用N-damo和Anammox耦合达到了100 mg N/(L·d)的亚硝酸盐转化速率[42];本文作者参与的研究更是在中空纤维膜反应器内获得了250 mg N/(L·d)的总氮去除速率和190 mg N/(L·d)的硝酸盐转化速率[44]。这些结果正逐步显示出Nx-damo应用于废水处理的可行性。
| N素供应 N feeding | 反应器类型 Reactor types | 微生物组成 Composition (%) | 速率 rate (mg NOx−-N/(L·d)) | 速率 rate (mg NH4+-N/(L·d)) | 文献 Reference | ||
| N-damo细菌Bacteria | N-damo古菌Archaea | Anammox细菌Bacteria | |||||
| NO3−/NH4+ | SBR | 0 | 78 | 3 | NO3−: 13.39 | 10.11 | [27] |
| NO2−/NH4+ | SBR | 50 | 0 | 50 | NO2−: 100 | 58.90 | [42] |
| NO3−/NH4+ | Membrane biofilm reactor | 20−30 | 20−30 | 20−30 | NO3−: 190 | 60.00 | [44] |
| NO3−/NH4+ | SBR | 有 | 0 | 有 | NO2−: 175 | 117.60 | [58] |
C、N循环是最基本的地球化学循环,甲烷对全球变暖的贡献可能达到20%[29],各类地球生态系统中甲烷的产生、转化机理及其排放规律是最重要的科学问题;废水中有机物不足限制硝酸盐反硝化是普遍面临的问题;废水厌氧处理过程甲烷的排放与控制也已经受到重视。因此,对于硝酸盐型厌氧甲烷氧化的研究具有深远的科学意义和明显的应用前景。然而,目前该领域的研究尚处于婴儿期,需要更广泛和更深入的研究。
2012年以前,只有“NC10”门细菌中的M. oxyfera被确认为n-damo的功能微生物。2012年另一类“NC10”门细菌和2013年具有N-damo功能的ANME-2d古菌的发现展示了此类功能微生物潜在的多样性。Nx-damo功能微生物的多样性、生理特性与代谢及更广泛的分布还需进一步研究证实。
当前的研究主要集中在n-damo细菌、n-damo古菌和Anammox细菌的混合体系。古菌主导n-damo将硝酸盐还原为亚硝酸盐同时氧化甲烷、细菌主导n-damo将生成的亚硝酸盐还原为N2同时氧化甲烷的演化路径仍需进一步调查。它们对甲烷的竞争、可能引起的亚硝酸盐积累等对这一共生体系的形成、稳定性的影响值得研究。尽管不排除ANME-2d或其他未知古菌具有独自完成硝酸盐全部反硝化的可能,但在ANME-2d中未检测到除硝酸盐还原外的其他反硝化还原酶[27]。相反,在M. oxyfera细菌中存在硝酸盐还原基因[29]。因此,开展NO3−/CH4条件下的相关研究,可能形成关于N-damo功能微生物及其代谢特性和途径、微生物之间的协同或竞争关系等科学问题的新知识。
尽管将M. oxyfera和Anammox细菌耦合获得了100−200 mg/(L·d)的脱氮速率,但Anammox的脱氮贡献达到77%[42, 58]。在中空纤维膜反应器内获得250 mg/(L·d)的脱氮速率中也有一半来源于Anammox[44]。Anammox发生的前提是有氨,对于含硝酸盐而不含氨的地下水、地表水和污水处理厂出水的处理,关键是要提高Nx-damo自身的效能。因此,利用生物膜反应器或者有助于污泥颗粒化的EGSB反应器、辅以优化的运行参数,有可能开发出基于Nx-damo原理的废水处理新技术。
| [1] | Strous M,jetten MSM. Anaerobic oxidation of methane and ammonium[J]. Annual Review of Microbiology,2004,58: 99-117 |
| [2] | Knittel K,Boetius A. Anaerobic oxidation of methane: progress with an unknown process[J]. Annual Review of Microbiology,2009,63: 311-334 |
| [3] | Gupta V,Smemo KA,Yavitt JB,et al. Stable isotopes reveal widespread anaerobic methane oxidation across latitude and peatland type[J]. Environmental Science & Technology,2013,47(15): 8273-8279 |
| [4] | Reeburgh SW. Oceanic methane biogeochemistry[J]. Chemical Reviews,2007,107(2): 486-513 |
| [5] | Devol AH,Ahmed SI. Are high rates of sulphate reduction associated with anaerobic methane oxidation[J]. Nature,1981,291: 407-408 |
| [6] | Reeburgh WS. “Soft spots” in the global methane budget[J]. Microbial Growth on C1 Compounds,1996: 335-342 |
| [7] | Krüger M,Meyerdierks A,Glöckner FO,et al. A conspicuous nickel protein in microbial mats that oxidize methane anaerobically[J]. Nature,2003,426: 878-881 |
| [8] | Hallam SJ,Putnam N,Preston CM. Reverse methanogenesis: testing the hypothesis with environmental genomics[J]. Science,2004,305(5689): 1457-1462 |
| [9] | Caldwell SL,Laidler JR,Brewer EA,et al. Anaerobic oxidation of methane: mechanisms,bioenergetics,and the ecology of associated microorganisms[J]. Environmental Science & Technology,2008,42(18): 6791-6799 |
| [10] | Thauer RK,Shima S. Methane as fuel for anaerobic microorganisms[J]. Annals of the New York Academy of Sciences,2008,1125: 158-170 |
| [11] | Strous M,Jetten MSM. Anaerobic oxidation of methane and ammonium[J]. Annual Review of Microbiology,2004,58: 99-117 |
| [12] | Shen LD,Hu BL,Zheng P. Progress in study on microorganisms responsible for anaerobic oxidation of methane[J]. Acta pedologica Sinica ,2011,48(3): 619-628 (in Chinese) 沈李东,胡宝兰,郑平. 甲烷厌氧氧化微生物的研究进展[J]. 土壤学报,2011,48(3): 619-628 |
| [13] | Zhang MZ,Li L,Liu JX . The parth way and methanotroph of anaerobic methane oxidation driven by nitrate or sulfate[J]. Microbiology China,2012,39(5): 702-710 (in Chinese) 张梦竹,李琳,刘俊新. 硝酸盐和硫酸盐厌氧氧化甲烷途径及氧化菌群[J]. 微生物学通报,2012,39(5): 702-710 |
| [14] | Liu ZH,Yin H,Dang Z,et al. Dissolved methane: a hurdle for anaerobic treatment of municipal wastewater[J]. Environmental Science & Technology,2014,48(2): 889-890 |
| [15] | Kruger M,Wolters H,Gehre M,et al. Tracing the slow growth of anaerobic methane-oxidizing communities by (15)N-labelling techniques[J]. Microbial ecology,2008,63(3): 401-411 |
| [16] | Shen LD,He ZF,Zhu Q,et al. Microbiology,ecology,and application of the nitrite-dependent anaerobic methane oxidation process[J]. Frontiers in Microbiology,2012,3: 269-273 |
| [17] | Raghoebarsing AA,Pol A,van de Pas-Schoonen KT,et al. A microbial consortium couples anaerobic methane oxidation to denitrification[J]. Nature,2006,440: 918-921 |
| [18] | Ettwig KF,Alen Tv,Pas-Schoonen KTvd. Enrichment and molecular detection of denitrifying methanotrophic bacteria of the NC10 phylum[J]. Applied and Environmental Microbiology,2009,75(11): 3656-3662 |
| [19] | Hu S,Zeng RJ,Burow LC,et al. Enrichment of denitrifying anaerobic methane oxidizing microorganisms[J]. Environmental Microbiology Reports,2009,1(5): 377-384 |
| [20] | Wu ML,Ettwig KF,Jetten MS,et al. A new intra-aerobic metabolism in the nitrite-dependent anaerobic methane- oxidizing bacterium Candidatus ‘Methylomirabilis oxyfera’[J]. Biochemical Society transactions,2011,39(1): 243-248 |
| [21] | Zhu G,Jetten MSM,Kuschk P. Potential roles of anaerobic ammonium and methane oxidation in the nitrogen cycle of wetland ecosystems[J]. Applied Microbiology and Biotechnology,2010,86(4): 1043-1055 |
| [22] | Modina O,Fukushi K,Yamamotoc K. Denitrification with methane as external carbon source[J]. Water Research,2007,41(12): 2726-2738 |
| [23] | Mason I. Methane as a carbon source in biological denitrification[J]. Journal Water Pollution Control Federation,1977,49(5): 855-857 |
| [24] | Islas-Limaa S,Thalassoa F,Gómez-Hernandezb J. Evidence of anoxic methane oxidation coupled to denitrification[J]. Water Research,2004,38(1): 13-16 |
| [25] | Eisentraeger A,Klag P,Vansbotter B. Denitrification of groundwater with methane as sole hydrogen donor[J]. Water Research,2001,35(9): 2261-2267 |
| [26] | Waki M,Suzuki K,Osada T,et al. Microbiological activities contributing to nitrogen removal with methane: effects of methyl fluoride and tungstate[J]. Bioresource Technology,2004,94(3): 339-343 |
| [27] | Haroon MF,Hu S,Shi Y,et al. Anaerobic oxidation of methane coupled to nitrate reduction in a novel archaeal lineage[J]. Nature,2013,500(7464): 567-570 |
| [28] | Ettwig KF,Shima S,Pas-Schoonen KTVD. Denitrifying bacteria anaerobically oxidize methane in the absence of Archaea[J]. Environmental Microbiology,2008,10(11): 3164-3173 |
| [29] | Ettwig KF,Butler MK,Le Paslier D,et al. Nitrite-driven anaerobic methane oxidation by oxygenic bacteria[J]. Nature,2010,464(7288): 543-548 |
| [30] | Kampman C,Temmink H,Hendrickx TLG,et al. Enrichment of denitrifying methanotrophic bacteria from municipal wastewater sludge in a membrane bioreactor at 20 °C[J]. Journal of Hazardous Materials,2014,274: 428-435 |
| [31] | Hu S,Zeng RJ,Keller J,et al. Effect of nitrate and nitrite on the selection of microorganisms in the denitrifying anaerobic methane oxidation process[J]. Environmental Microbiology Reports,2011,3(3): 315-319 |
| [32] | He Z,Cai C,Geng S,et al. Mdodeling a nitrite-dependent anaerobic methane oxidation process: parameters identification and model evaluation[J]. Bioresource Technology,2013,147: 315-320 |
| [33] | Duan Z,Mao S. A thermodynamic model for calculating methane solubility,density and gas phase composition of methane-bearing aqueous fluids from 273 to 523K and from 1 to 2 000 bar[J]. Geochimica et Cosmochimica Acta,2006,70(13): 3369-3386 |
| [34] | Serra MCC,Pessoa FLP,Palavra AMF. Solubility of methane in water and in a medium for the cultivation of methanotrophs bacteria[J]. The Journal of Chemical Thermodynamics,2006,38(12): 1629-1633 |
| [35] | He ZF,Cai C,Shen LD,et al. Establishing and verifying of model for methane mass transfer in DAMO process[J]. Journal of Chemical Industry and Engineering (China),2012,63(6): 1836-1841 (in Chinese) 何崭飞,琛蔡,沈李东,等. DAMO过程中甲烷传质模型的建立与验证[J]. 化工学报,2012,63(6): 1836-1841 |
| [36] | Luesken FA,van Alen TA,van der Biezen E,et al. Diversity and enrichment of nitrite-dependent anaerobic methane oxidizing bacteria from wastewater sludge[J]. Applied Microbiology and Biotechnology,2011,92(4): 845-854 |
| [37] | Wang Y,Zhu G,Harhangi HR,et al. Co-occurrence and distribution of nitrite-dependent anaerobic ammonium and methane-oxidizing bacteria in a paddy soil[J]. FEMS Microbiology Letters,2012,336(2): 79-88 |
| [38] | Zhu B,Dijk Gv,Fritz C. Anaerobic oxidization of methane in a minerotrophic peatland: enrichment of nitrite-dependent methane-oxidizing bacteria[J]. Applied and Environmental Microbiology,2012,78(24): 8657-8665 |
| [39] | Shen LD,Liu S,Zhu Q,et al. Distribution and diversity of nitrite-dependent anaerobic methane-oxidising bacteria in the sediments of the qiantang river[J]. Microbial Ecology,2014,67(2): 341-349 |
| [40] | Zhang Y,Henriet JP,Bursens J,et al. Stimulation of in vitro anaerobic oxidation of methane rate in a continuous high-pressure bioreactor[J]. Bioresource Technology,2010,101(9): 3132-3138 |
| [41] | Rasigraf O,Vogt C,Richnow HH,et al. Carbon and hydrogen isotope fractionation during nitrite-dependent anaerobic methane oxidation by Methylomirabilis oxyfera[J]. Geochimica et Cosmochimica Acta,2012,89: 256-264 |
| [42] | Luesken FA,Sanchez J,van Alen TA,et al. Simultaneous nitrite-dependent anaerobic methane and ammonium oxidation processes[J]. Applied Microbiology and Biotechnology,2011,77(19): 6802-6807 |
| [43] | Kampman C,Hendrickx TL,Luesken FA,et al. Enrichment of denitrifying methanotrophic bacteria for application after direct low-temperature anaerobic sewage treatment[J]. Journal of Hazardous Materials,2012,227-228: 164-171 |
| [44] | Shi Y,Hu S,Lou J,et al. Nitrogen removal from wastewater by coupling anammox and methane-dependent denitrification in a membrane biofilm reactor[J]. Environmental Science & Technology,2013,47(20): 11577-11583 |
| [45] | Kojima H,Tsutsumi M,Ishikawa K,et al. Distribution of putative denitrifying methane oxidizing bacteria in sediment of a freshwater lake,Lake Biwa[J]. Systematic and Applied Microbiology,2012,35(4): 233-238 |
| [46] | Thauer RK,Shima S. Biogeochemistry: Methane and microbes[J]. Nature,2006,440: 878-879 |
| [47] | Oremland RS. NO connection with methane[J]. Nature,2010,464(7288): 500-501 |
| [48] | Luesken FA,Wu ML,Op den Camp HJ,et al. Effect of oxygen on the anaerobic methanotroph ‘Candidatus Methylomirabilis oxyfera’: kinetic and transcriptional analysis[J]. Environmental Microbiology,2012,14(4): 1024-1034 |
| [49] | Deutzmann JS,Schink B. Anaerobic oxidation of methane in sediments of Lake Constance,an oligotrophic freshwater lake[J]. Applied and Environment Microbiology,2011,77(13): 4429-4436 |
| [50] | Zhu Q,Shen LD,Hu BL,et al. Molecular detection of denitrifying anaerobic methane oxidizing bacteria in the sediment of West Lake,Hangzhou[J]. Acta Scientiae Circumstantiae,2013,33(5): 1321-1325 (in Chinese) 朱群,沈李东,胡宝兰,等. 西湖底泥中的反硝化型甲烷厌氧氧化菌的分子生物学检测[J]. 环境科学学报,2013,33(5): 1321-1325 |
| [51] | Knittel K,Lösekann T,Boetius A,et al. Diversity and distribution of methanotrophic archaea at cold seeps[J]. Applied and Environment Microbiology,2005,71(1): 467-479 |
| [52] | Ettwig KF,Speth DR,Reimann J,et al. Bacterial oxygen production in the dark[J]. Frontiers in Microbiology,2012,3: 1-8 |
| [53] | Meyerdierks A,Kube M,Kostadinov I,et al. Metagenome and mRNA expression analyses of anaerobic methanotrophic archaea of the ANME-1 group[J]. Environmental Microbiology,2010,12(2): 422-439 |
| [54] | Hallam SJ,Girguis PR,Preston CM,et al. Identification of methyl coenzyme M reductase A (mcrA) genes associated with methane-oxidizing archaea[J]. Applied and Environmental Microbiology,2003,69(9): 5483-5491 |
| [55] | Smith AL,Skerlos SJ,Raskin L. Psychrophilic anaerobic membrane bioreactor treatment of domestic wastewater[J]. Water Research,2013,47(4): 1655-1665 |
| [56] | Li A,Sun G,Xu M. Recent patents on anammox process[J]. Recent Patents on Engineering,2008,2(3): 189-194 |
| [57] | Than K,Ajit PA. Novel microbial nitrogen removal processes[J]. Biotechnology Advances,2004,22(7): 519-532 |
| [58] | Zhu B,Sanchez J,van Alen TA,et al. Combined anaerobic ammonium and methane oxidation for nitrogen and methane removal[J]. Biochemical Society Transactions,2011,39(6): 1822-1825 |
| [59] | Abma WR,Driessen W,Haarhuis R,et al. Upgrading of sewage treatment plant by sustainable and cost-effective separate treatment of industrial wastewater[J]. Water Science and Technology,2010,61(7): 1715-1722 |
| [60] | Van der Star WR,Abma WR,Blommers D,et al. Startup of reactors for anoxic ammonium oxidation: experiences from the first full-scale anammox reactor in Rotterdam[J]. Water Research,2007,41(18): 4149-4163 |
| [61] | Adriano J,David S,Jack E,et al. Full-scale nitrogen removal from digester liquid with partial nitritation and anammox in one SBR[J]. Environmental Science & Technology,2009,43(14): 5301-5306 |
 2015, Vol. 42
2015, Vol. 42





