扩展功能
文章信息
- 王松, 梁宏彰, 贾春凤, 张柏林
- 干酪乳杆菌12A碳源代谢的比较基因组学分析
- Comparative genomics analysis of carbon metabolism of Lactobacillus casei 12A
- 微生物学通报, 2015, 42(2): 315-324
- Microbiology China, 2015, 42(2): 315-324
- 10.13344/j.microbiol.china.140441
-
文章历史
- 收稿日期: 2014-10-08
- 接受日期: 2014-11-21
- 优先数字出版日期(www.cnki.net): 2014-11-21
2. 东北农业大学 乳品科学教育部重点实验室 黑龙江 哈尔滨 150030
2. Key laboratory of Dairy Science, Ministry of Education, Northeast Agricultural University, Harbin, Heilongjiang 150030, China
干酪乳杆菌(Lactobacillus casei)属于乳杆菌属(Lactobacillus),是革兰氏阳性菌,不产芽孢。它是对营养非常挑剔,对发酵条件要求严格,以生产乳酸为主要代谢终产物的细菌 [1]。干酪乳杆菌作为益生菌通常被用来生产发酵类食品。然而,近年来的研究发现它具有一个巨大潜力——可以生产有广泛用途的生物燃料及其副产物 [2,3,4]。干酪乳杆菌起到的效用是具有菌株依赖性的,因为菌株间的基因组序列存在显著差异。对17株干酪乳杆菌基因组的比较分析发现,这些菌株平均具有2 800个同源基因(±151),其中1 715个基因是所有菌株共有的;有4 220个非保留直系同源基因,每个菌株中有平均119个独特区基因 [5]。基因组水平上的巨大差异导致干酪乳杆菌在工业应用上具有专一性。因此,详细比较菌株的基因组和表型水平的特定差异是必要的。DNA遗传功能的揭示和测序技术的推广使得人们能够从遗传物质本身对生物体代谢特性及调控机制进行研究。最近有研究利用比较基因组学方法分析干酪乳杆菌的物种多样性和基因组的进化,在生态多样性方面提供了很多有价值的见 解 [5,6]。然而,基因组学技术还没有被广泛应用于干酪乳杆菌的代谢分析中。目前,只有8株干酪乳杆菌的全基因组序列被公开,分别为ATCC 334 [7]、Zhang [8]、BL23 [9]、LCW2 [10]、BD-II [11]、W56 [12]、LOCK 919 [13]和12A。本文采用比较基因组学对干酪乳杆菌12A与7株已公布全基因组序列的干酪乳杆菌的碳水化合物代谢相关转运蛋白、代谢途径及产酶情况进行比较分析,初步确定菌株12A的可代谢碳源及代谢终产物;并验证比较基因组学技术可以用来预测菌株的生长代谢表现。
1 材料与方法 1.1 比较基因组学分析软件及方法CGView Server是环形基因组可视化在线比较工具(http://stothard.afns.ualberta.ca/cgview_server/ index.html)。BioCyc为生物模型数据库(http://www.biocyc.org/),收录了3 000多物种的代谢途径-基因组数据库Pathway/Genome database (PGDB)数据;MetaCyc为代谢数据库(http://metacyc.org/),收录了来自37 000多篇出版物的2 100多条代谢途径。Pathway Tools是基于BioCyc和MetaCyc两大数据库的生物模型构建软件,能够以物种的全基因组序列和注释信息,自动生成该物种的PGDB,即全基因组水平的代谢途径信息。
干酪乳杆菌12A分离自秸秆青储,它的全基因组测序已经完成,其基因组序列及注释信息可在NCBI (http://www.ncbi.nlm.nih.gov) GenBank中获得(GenBank登录号为CP006690.1)。(1) 干酪乳杆菌12A环形全基因组图谱的绘制:在NCBI中获得干酪乳杆菌12A和Zhang的全部CDS序列,应用BLAST软件进行序列对比后,利用CGView Server在线软件绘制菌株12A环形全基因组图谱。(2) 碳水化合物代谢相关转运蛋白和代谢途经信息:Pathway Tools的Pathologic选项中选择干酪乳杆菌12A基因组序列及注释文件,通过与代谢数据库数据比较自动生成其PGDB,在Refine选项中得到较为准确的PGDB,在Pathways中获取各种碳水化合物代谢相关转运蛋白和代谢途经信息。(3) 碳水化合物代谢相关途径信息的可靠性:Biocyc中收录了其他7种已测序干酪乳杆菌ATCC 334、Zhang、BL23、LCW2、BD-II、W56、LOCK 919的PGDB,在Comparative Analysis选项中选取该7株菌,同时得到它们的代谢途径和转运蛋白所有信息,并以此为参照,确定菌株12A的碳水化合物代谢相关途径信息的可靠性;结合参考文献中的相关信息在Universal Protein Database (UniProt)及GenBank中检索未全基因组测序干酪乳杆菌的碳水化合物代谢中关键酶及基因,以确定分析结果的普遍性
细胞内碳源代谢通路的判定标准:(1) 具有相关转运蛋白,能够将碳水化合物转运到细胞内代谢利用;(2) 具有完整代谢途径;(3) 能够合成相关酶催化代谢碳源,产生并利用中间产物,生成终产物,完成整个代谢途径。
1.2 细胞培养试验操作方法将MRS培养基中碳源以外的其他组分配制好,混合均匀后分装。分别加入同浓度单种碳源,作为唯一碳源替换原始配方中的葡萄糖,调节pH值到6.0。将活化好的干酪乳杆菌12A用PBS缓冲液清洗3次,以浓度10 8 cfu/ml分别加入以上培养基。在37 °C条件下培养24 h,观察菌株在不同碳源环境中的生长情况。
1.3 发酵试验的试剂与方法50 mmol/L葡萄糖和70 mmol/L果糖作为碳源加入到MRS培养基中,调节pH值至6.0。按 10 8 cfu/ml浓度接入菌株12A,做3个技术重复。37 °C条件下培养48 h,在4、8、12、20、24、30、36和48 h分别取样,?20 °C冷冻保存。采用高效液相色谱(HPLC)法检测葡萄糖和果糖的利用及有机酸和醇的产出情况。使用Waters 2695 (Alliance系统)高效液相色谱仪,Bio-Rad Aminex HPX-87H有机酸柱,以5 mmol/L的H 2SO 4为流动相,在柱温30 °C,流速0.6 mL/min条件下,用Waters 2414示差折光检测器检测样品。标准品葡萄糖、果糖、乳酸、乙酸和乙醇,均购自美国Sigma公司。
2 结果与分析 2.1 细胞内碳源分解代谢能力分析从GenBank中获得干酪乳杆菌12A全基因组序列及注释信息,绘制出全基因组图谱见 图 1。并通过Pathway Tools工具对该菌的代谢途径进行构建,获得以其全基因组信息为基础的转运能力和代谢途径信息。从 表 1中可以看出,菌株12A 和W56的转运蛋白明显多于其他菌株,且12A的转运蛋白种类与W56相似,说明菌株12A与W56可能具有代谢更多碳水化合物的能力。6-磷酸-β-D-葡糖 基-(1,4)-D-葡萄糖和甘露醇-1-磷酸可以被7个菌株转运到细胞内,但菌株12A没有相关转运蛋白。在碳水化合物降解途径分析中发现,惟独菌株12A没有2-脱氧-α-D-核糖-1-磷酸降解、蔗糖降解I和磷酸戊糖途径(氧化分支) 3个途径;另外,8个菌株都具有Leloir途径、糖原降解I、糖原降解II、蜜二糖降解、海藻糖降解I (低渗透性)、(R)-乙偶姻生物合成II、糖酵解I、异型乳酸发酵、混合酸发酵、丙酮酸发酵丁酸甲酯、丙酮酸发酵乙醇I、丙酮酸发酵乳酸等途径( 表 2),说明这几种途径在干酪乳杆菌中具有普遍性。
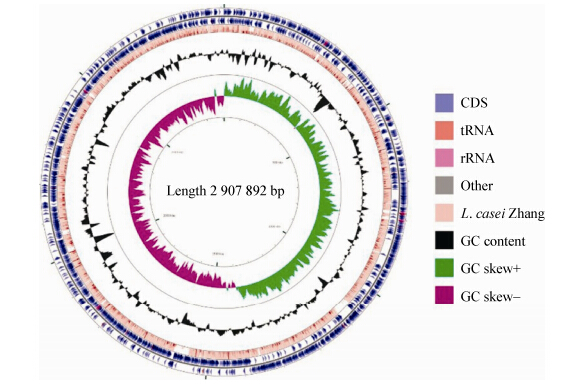
|
| 图 1 干酪乳杆菌12A的全基因组图谱 Figure 1 Genome atlas of L. casei 12A 注:图谱的内圈分别代表 G+C 含量和 GC 正负偏斜. Note: Inner circles in atlas, represent G+C% content and GC skew, respectively. |
| 注: -:无此转运蛋白. Note: -: No transporter. | |||||||||
| 转运蛋白 Transporters | 干酪乳杆菌 L. casei | ||||||||
| 12A | W56 | BD-II | BL23 | LC2W | LOCK919 | Zhang | ATCC 334 | ||
| hexose | D-glucose | ||||||||
| D-glucose-6-phosphate | |||||||||
| D-glucopyranose-6-phosphate | |||||||||
| α-D-glucose-6-phosphate | |||||||||
| β-D-glucose-6-phosphate | |||||||||
| D-gluconate | |||||||||
| D-glucosamine-6-phosphate | |||||||||
| D-glucosaminate-6-phosphate | - | - | - | - | - | - | |||
| Aldehydo-D-glucose-6-phosphate | - | - | - | - | - | - | |||
| 6-Phospho-β-D-glucosy-l-(1,4)-D-glucose | - | ||||||||
| fructose | |||||||||
| fructose-1-phosphate | - | - | |||||||
| D-fructofuranose-1-phosphate | - | ||||||||
| D-fructopyranose-1-phosphate | - | - | - | - | - | ||||
| α-D-fructofuranose-1-phosphate | - | - | - | - | - | - | |||
| α-D-fructopyranose-1-phosphate | - | - | - | - | - | - | |||
| β-D-fructopyranose-1-phosphate | - | - | - | - | - | - | |||
| keto-D-fructose-1-phosphate | - | - | - | - | - | - | |||
| D-galactose-6-phosphate | - | - | |||||||
| β-D-galactose | |||||||||
| β-D-galactose-6-phosphate | - | - | - | - | - | - | |||
| Aldehydo-D-galactose-6-phosphate | - | - | - | - | - | - | - | ||
| galactitol-1-phosphate | - | - | - | - | - | - | |||
| sorbose | |||||||||
| L-sorbose-1-phosphate | - | - | - | - | - | - | |||
| L-sorbofuranose-1-phosphate | - | - | - | - | - | ||||
| L-sorbopyranose-1-phosphate | - | - | - | - | - | ||||
| α-L-sorbofuranose-1-phosphate | - | - | - | - | - | - | |||
| α-L-sorbopyranose-1-phosphate | - | - | - | - | - | - | |||
| β-L-sorbofuranose-1-phosphate | - | - | - | - | - | - | |||
| β-L-sorbopyranose-1-phosphate | - | - | - | - | - | - | |||
| D-mannose | |||||||||
| D-mannose-6-phosphate | |||||||||
| D-mannopyranose-6-phosphate | |||||||||
| α-D-mannose-6-phosphate | - | - | |||||||
| β-D-mannose-6-phosphate | - | - | - | - | - | ||||
| Aldehydo-D-mannose-6-phosphate | - | - | - | - | - | - | |||
| Mannitol-1-phosphate | - | ||||||||
| pentose | D-xylose | ||||||||
| α-D-xylopyranose | - | - | - | - | - | ||||
| α-L-arabinopyranose | - | - | - | - | - | ||||
| D-ribose | |||||||||
| β-D-ribopyranose | - | - | |||||||
| disaccharide | maltose | ||||||||
| Maltose-6-phosphate | - | - | - | - | |||||
| Mannitol-1-phosphate | - | - | - | - | - | - | |||
| lactose | - | - | - | - | |||||
| lactose-6-phosphate | - | ||||||||
| β-lactose-6-phosphate | - | - | - | - | - | - | |||
| trehalose | - | - | - | ||||||
| trehalose-6-phosphate | |||||||||
| D-trehalose-6-phosphate | |||||||||
| α,β-trehalose-6-phosphate | - | - | - | - | - | - | |||
| Chitobiose-6-phosphate | - | - | - | - | - | - | |||
| α-chitobiose-6-phosphate | - | - | - | - | - | - | |||
| β-chitobiose-6-phosphate | - | - | - | - | - | - | |||
| cellobiose | |||||||||
| D-cellobiose-6-phosphate | |||||||||
| α-D-cellobiose-6-phosphate | - | - | - | - | - | - | |||
| sucrose | |||||||||
| sucrose-6-phosphate | |||||||||
| melibiose | |||||||||
| polysaccharide | lichenin | ||||||||
| 注:+:有此途径. Note: +: have pathway. | ||||||||
| 途径 Pathways | 干酪乳杆菌 L. casei | |||||||
| 12A | W56 | BD-II | BL23 | LC2W | LOCK919 | Zhang | ATCC 334 | |
| (1,4)-β-xylan degradation | + | + | + | |||||
| 2′-deoxy-α-D-ribose-1-phosphate degradation | + | + | + | + | + | + | + | |
| 2-O-α-mannosyl-D-glycerate degradation | + | + | + | + | ||||
| Chitin degradation II | + | + | + | |||||
| D-mannose degradation | + | + | + | + | + | + | ||
| Entner-Doudoroff pathway I | + | + | + | + | ||||
| fructose degradation | + | + | + | + | ||||
| galactose degradation I (Leloir pathway) | + | + | + | + | + | + | + | + |
| galactose degradation III | + | + | ||||||
| gellan degradation | + | |||||||
| glucose and glucose-1-phosphate degradation | + | + | + | |||||
| glycogen degradation I | + | + | + | + | + | + | + | + |
| glycogen degradation II | + | + | + | + | + | + | + | + |
| L-sorbose degradation | + | + | ||||||
| lactose and galactose degradation I | + | + | + | + | + | + | + | |
| lactose degradation III | + | + | + | + | + | + | ||
| maltose degradation | + | + | + | + | + | |||
| melibiose degradation | + | + | + | + | + | + | + | + |
| ribose degradation | + | + | + | + | + | |||
| stachyose degradation | + | |||||||
| sucrose degradation I | + | + | + | + | + | + | + | |
| trehalose degradation I (low osmolarity) | + | + | + | + | + | + | + | + |
| trehalose degradation IV | + | + | ||||||
| xylose degradation I | + | + | + | |||||
| (R)-acetoin biosynthesis II | + | + | + | + | + | + | + | + |
| 2-oxoglutarate decarboxylation to succinyl-CoA | + | + | + | + | + | |||
| 2-oxoisovalerate decarboxylation to isobutanoyl-CoA | + | + | + | + | ||||
| Entner-Doudoroff pathway I | + | + | + | + | ||||
| glutamate degradation V (via hydroxyglutarate) | + | + | ||||||
| glycerol-3-phosphate shuttle | + | + | + | |||||
| glycolysis I | + | + | + | + | + | + | + | + |
| glycolysis III (glucokinase) | + | + | + | + | ||||
| glycolysis VI (metazoan) | + | + | + | |||||
| heterolactic fermentation | + | + | + | + | + | + | + | + |
| mixed acid fermentation | + | + | + | + | + | + | + | + |
| NADH to cytochrome bd oxidase electron transfer | + | |||||||
| pentose phosphate pathway (non-oxidative branch) | + | + | + | + | + | + | + | |
| pentose phosphate pathway (oxidative branch) | + | + | + | + | + | + | + | |
| pentose phosphate pathway (partial) | + | + | ||||||
| pyruvate decarboxylation to acetyl CoA | + | + | + | + | + | + | ||
| pyruvate fermentation to acetate II | + | + | + | |||||
| pyruvate fermentation to butanoate | + | + | + | + | + | + | + | |
| pyruvate fermentation to ethanol I | + | + | + | + | + | + | + | + |
| pyruvate fermentation to ethanol II | + | + | ||||||
| pyruvate fermentation to ethanol III | + | + | ||||||
| pyruvate fermentation to lactate | + | + | + | + | + | + | + | + |
| pyruvate oxidation pathway | + | + | + | + | + | + | ||
| Respiration (anaerobic) | + | + | + | + | + | |||
| TCA cycle I (prokaryotic) | + | + | + | + | ||||
结合转运蛋白的种类及代谢途径的完整性,判断干酪乳杆菌12A能够在细胞内代谢葡萄糖、乳糖、半乳糖、蔗糖、海藻糖、核糖、果糖、甘露糖、麦芽糖。除了麦芽糖同时具有磷酸转移酶系统(PTS转运系统)和ATP结合盒(ABC转运系统);乳糖可通过β半乳糖苷酶转运系统转运到胞内;其他糖都可经PTS系统转运到胞内进行代谢。
Biocyc和KEGG代谢途径相关数据库收集并公布了7株已全基因测序干酪乳杆菌的代谢信息,对其糖转运系统和糖分解途径的信息进行整理与分析,发现蔗糖、葡萄糖、果糖、麦芽糖、甘露糖、纤维二糖、海藻糖、地衣多糖和动物多糖等磷酸葡萄糖转移酶系统(PTS转运系统),葡萄糖、麦芽糖、蜜二糖、纤维二糖通透酶系统以及核糖、木糖、阿拉伯糖ABC转运体系,都存在于大多数干酪乳杆菌。表明干酪乳杆菌具有一定的蔗糖、麦芽糖、纤维二糖、蜜二糖、葡萄糖、果糖、甘露糖以及木糖转运能力;并且该7株干酪乳杆菌中都具有糖酵解途径和戊糖磷酸途径、Leloir途径、蜜二糖、核糖及蔗糖分解途径,绝大部分具有乳糖、果糖分解途径以及部分具有麦芽糖、木糖、甘露糖、山梨糖、几丁质降解途径。
比较分析干酪乳杆菌12A的转运蛋白、代谢途径及其酶的合成。结果显示,拥有完整代谢机制的糖为D-葡萄糖、D-果糖、D-半乳糖、D-麦芽糖、D-甘露糖、D-核糖、乳糖、蔗糖、海藻糖。通过细胞培养试验发现,干酪乳杆菌12A在这9种碳水化合物中都可以生长,说明9种碳水化合物都可以被其代谢利用(表3)。
| 注: +:在此条件下可生长. Note: +: Growth in the presence of the carbohydrate. | ||
| 碳水化合物 Carbohydrate | 细胞培养 In vivo | 模型 Model |
| D-glucose | + | + |
| D-fructose | + | + |
| D-galactose | + | + |
| D-maltose | + | + |
| D-mannose | + | + |
| D-ribose | + | + |
| lactose | + | + |
| sucrose | + | + |
| trehalose | + | + |
干酪乳杆菌12A可经异型乳酸发酵途径降解葡萄糖和果糖生成乙醇及乳酸,也可经混合酸发酵途径中糖酵解产生的磷酸烯醇式丙酮酸生成乙醇、乳酸和醋酸等产物。深入分析干酪乳杆菌12A的基因组注释,我们得到这两个途径中参与发酵生成乙醇的酶相关信息。 图2为干酪乳杆菌12A代谢葡萄糖和果糖生产乙醇及其副产物的总途径,菌株12A在合成乙酰辅酶A后可经乙醛脱氢酶(E.C. 1.2.1.10)和醇脱氢酶(E.C. 1.1.1.1)催化合成乙醇。验证试验中,将适量的葡萄糖和果糖同时加入MRS培养基,分时段取样,利用高效液相色谱法测定碳源利用和产物产出情况。分析结果如 图 3所示,培养12 h后,干酪乳杆菌12A开始生成乙醇和乳酸;大约30 h培养基的葡萄糖和果糖全部被消耗殆尽,同时乙醇和乳酸的产出达到最高量。这组数据,证实了干酪乳杆菌12A可以利用葡萄糖和果糖发酵生成有机酸和乙醇这一代谢通路。
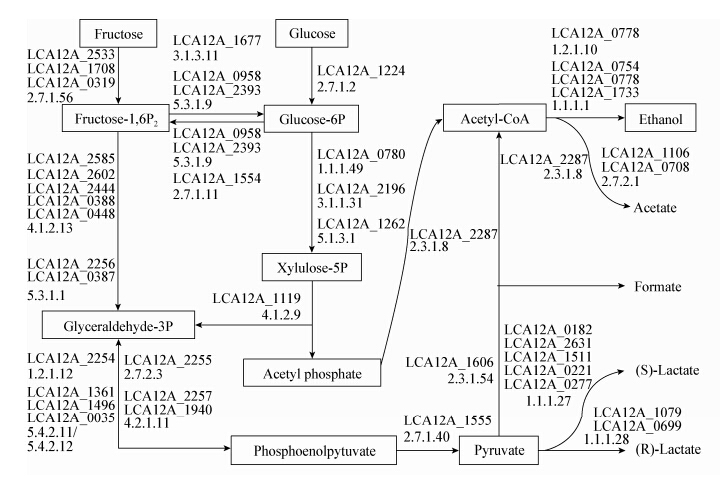
|
| 图 2 干酪乳杆菌12A生成乙醇的途径 Figure 2 The ethanol production pathways of L. casei 12A 注:箭头上标示:酶 EC 编号及其位点标签. Note: Labels on the arrows: Enzyme commission number and locus tag. |

|
| 图 3 干酪乳杆菌12A利用葡萄糖和果糖生成乳酸、乙酸和乙醇 Figure 3 Lactic acid, acetic acid and ethanol production of L. casei 12A using glucose and fructose |
通过基因组学比较分析发现,干酪乳杆菌12A不具有某些碳水化合物的转运蛋白,但可以通过β-半乳糖苷酶或者β-葡萄糖苷酶的催化将纤维二糖、龙胆二糖、松三糖、麦芽三糖、麦芽四糖、水苏糖、葡聚糖、N-乙酰-D-半乳糖胺、半乳糖苷、生氰苷、东莨菪苷、葡萄糖苷、百脉根苷等寡糖和多糖水解成半乳糖和葡萄糖。
3 讨论本研究揭示了干酪乳杆菌12A是生产乙醇的优势菌株之一。可利用碳源的分析结果为今后对菌株12A发酵底物的研究提供理论依据。通过分析代谢生成乙醇的途径及参与代谢的酶,可以推断出提高乙醇收率和生产速率的潜在手段。同时,本研究也验证了比较基因组学分析可以将干酪乳杆菌的基因结构与碳源代谢能力之间建立紧密的联系。
对非冗余蛋白数据库(UniProt)中糖分解相关的酶类进行检索发现,大量未全基因组测序的干酪乳杆菌中同样存在多种糖代谢相关酶类,如α-6-磷酸葡萄糖苷酶、β-6-磷酸葡萄糖甘酶、β-6-磷酸半乳糖苷酶、蔗糖-6-磷酸水解酶、葡萄糖激酶、半乳糖激酶、葡萄糖-6-磷酸异构酶、6-磷酸果糖激酶、果糖二磷酸醛缩酶、半乳糖-1磷酸尿甙转移酶、UDP-4-葡萄糖差向异构酶、α-甘露糖酶、甘露糖-6-磷酸异构酶、阿拉伯糖-5-磷酸异构酶、麦芽糖磷酸化酶、海藻糖磷酸化酶、海藻糖-6-磷酸水解酶、木糖异构酶、山梨糖-1-磷酸还原酶、α-L-岩藻糖苷酶、L-岩藻糖异构酶、木糖异构酶等。这些充分证明了干酪乳杆菌具有能够代谢多种碳源的潜能。
菌株利用碳水化合物的能力可预言菌株对不同生态位的适应性 [5]。据代谢模型分析可以看出,12A与W56代谢水平相似,运输底物明显多于另外6株菌。这可能与菌株的生长环境相关,干酪乳杆菌ATCC 334和LC2W是乳制品中分离出来的 [7,10],Zhang和BD-Ⅱ来自马奶酒 [8,11],由于乳制品环境中的碳源种类没有植物环境中复杂,菌株在乳制品中的进化可能引起菌株的基因衰变,使得菌株的碳源代谢水平有所下降;而12A分离自玉米青储,植物环境下碳源种类复杂得多,因此它能够代谢碳源的种类比较丰富;W56分离自人类粪便 [12],有研究证实分离自植物材料和人体胃肠道的干酪乳杆菌具有利用C5糖和某些C5、C6糖醇的能力,对比分离自奶酪的菌株更具有普遍性 [14]。其原因为水平基因平移是干酪乳杆菌基因进化的一种主导因素 [6,7]。一些研究结果证实专用于奶酪的干酪乳杆菌的进化是伴有参与碳水化合物利用和转录调控的基因衰变的 [6]。
干酪乳杆菌12A可水解多种寡糖和多糖,且又不具有转运它们的蛋白,说明菌株12A可以在细胞外将这些寡糖及多糖水解。根据分析发现,菌株12A可依靠β-半乳糖苷酶或β-葡萄糖苷酶水解寡糖及多糖成为半乳糖或葡萄糖,而半乳糖和葡萄糖是可以转运到细胞内利用的。这在一定程度上,说明12A可以将这两种酶分泌到细胞外。有研究报道,乳糖酶根据不同来源可分为胞内酶和胞外酶,如乳酸酵母、黑曲霉、米曲霉、米根霉等菌种生产的乳糖酶均为胞外酶,脆壁克鲁维酵母和大部分细菌生产胞内乳糖酶[15]。Coulon等[16]研究发现植物乳杆菌、干酪乳杆菌、德氏乳杆菌均可以生产β-葡萄糖苷酶,其中有的是胞外酶,有的为胞内酶。
乳酸菌具有发酵生产工业乙醇的潜力。在乳酸菌碳水化合物代谢相关酶的作用下,通常其糖代谢终产物倾向于转化为乳酸;然而一些乳酸菌具有异型发酵能力,可以使丙酮酸向乙醇及其他副产物转化。另外,乳酸菌具有一定的耐酸性,可以在较低pH值条件下生长,并且很多菌株能够耐受乙醇 [17,18,19,20,21]。在碳水化合物代谢上,乳酸菌具有一定的优势。许多乳酸菌能够发酵从水解木质纤维素生物质中释放出来的葡萄糖和木糖等糖类;然而,不同于细菌,常见的酿酒酵母只能利用葡萄糖而不能直接利用木糖 [22]。木糖占木质纤维素总糖质量10%?40%,如果能够对其充分利用,理论上至少可将乙醇产量提高25% [23,24]。London等 [25]的研究证明,干酪乳杆菌能够在戊糖磷酸途径中通过中间产物核酮糖-5-磷酸产生木酮糖-5-磷酸,木酮糖-5-磷酸与丙酮酸经丙酮酸激酶作用形成的乙酰磷酸,乙酰磷酸再经转乙酰作用和脱氢作用转化为乙醇。乳酸菌的这些特点都有助于开发新的微生物用于发酵生产乙醇。
| [1] | Axelsson L. Lactic acid bacteria: clasification and physiology[a]//Salminen S, von Wright A. Lactic Acid Bacteria Microbiological and Funtional Aspects[M]. 3rd ed. New York: Marcel Dekker Inc, 2004: 1-66 |
| [2] | Stiles ME, Holzapfel WH. Lactic acid bacteria of foods and their current taxonomy[J]. International Journal of Food Microbiology, 1997, 36(1): 1-29 |
| [3] | Goldin BR, Gorbach LS. Probiotics for humans[a]//Fuller R. Probiotics: the Scientific Basis[M]. New York: Chapman and Hall, 1992: 355-372 |
| [4] | MayraMakinen A. Industrial use and production of lactic acid bacteria[a]//Salminen S, Wright A, Lactic Acid Bacteria—Microbiology and Functional Aspects[M]. New York: Marcel Dekker Inc, 1998: 73-102 |
| [5] | Broadbent JR, Neeno-Eckwall EC, Stahl B, et al. Analysis of the Lactobacillus casei supra-genome and its influence in species evolution and lifestyle adaptation[J]. BMC Genomics, 2012, 13: 533 |
| [6] | Cai H, Thompson R, Budinich MF, et al. Genome sequence and comparative genome analysis of Lactobacillus casei: insights into their niche-associated evolution[J]. Genome Biology and Evoluton, 2009, 1: 239-257 |
| [7] | Makarova K, Slesarev A, Wolf Y, et al. Comparative genomics of the lactic acid bacteria[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(42): 15611-15616 |
| [8] | Zhang W, Yu D, Sun Z, et al. Complete genome sequence of Lactobacillus casei Zhang, a new probiotic strain isolated from traditional homemade koumiss in Inner Mongolia, China[J]. Journal of Bacteriology, 2010, 192(19): 5268-5269 |
| [9] | Maze A, Boel G, Zuniga M, et al. Complete genome sequence of the probiotic Lactobacillus casei strain BL23[J]. Journal of Bacteriology, 2010, 192(10): 2647-2648 |
| [10] | Chen C, Ai L, Zhou F, et al. Complete genome sequence of the probiotic bacterium Lactobacillus casei LC2W[J]. Journal of Bacteriology, 2011, 193(13): 3419-3420 |
| [11] | Ai L, Chen C, Zhou F, et al. Complete genome sequence of the probiotic strain Lactobacillus casei BD-II[J]. Journal of Bacteriology, 2011, 193(12): 3160-3161 |
| [12] | Hochwind K, Weinmaier T, Schmid M, et al. Draft genome sequence of Lactobacillus casei W56[J]. Journal of Bacteriology, 2012, 194(23): 6638 |
| [13] | Anna KB, Tamara AP, Jacek B, et al. Complete genome sequence of the probiotic strain Lactobacillus casei (formerly Lactobacillus paracasei) LOCK919[J]. Genome Announcements, 2013, 1(5): e00758-13 |
| [14] | Cai H, Rodriguez BT, Zhang W, et al. Genotypic and phenotypic characterization of Lactobacillus casei strains isolated from different ecological niches suggests frequent recombination and niche specificity[J]. Microbiology, 2007, 153(8): 2655-2665 |
| [15] | Gao XR, Ma L, Ye H. Research advancement of β-galactosidase[J]. Biotechnology Bulletin, 2005(3): 18-20 (in Chinese)高秀容, 马力, 叶华. β-半乳糖苷酶的研究进展[J]. 生物技术通报, 2005(3): 18-20 |
| [16] | Coulon S, Chemardin P, Gueguen Y, et al. Purification and characterization of an intracellular β-glucosidase from Lactobacillus casei ATCC 393[J]. Applied Biochemistry and Biotechnology, 1998, 74(2): 105-114 |
| [17] | Britz TJ, Tracey RP. The combination effect of pH, SO2, ethanol and temperature on the growth of Leuconostoc oenos[J]. Journal of Bacteriology, 1990, 68(1): 23-31 |
| [18] | Davis CR, Wibowo D, Fleet GH, et al. Properties of wine lactic acid bacteria: their potential enological significance[J]. American Journal of Enology and Viticulture, 1988, 39(2): 137-142 |
| [19] | Edwards CG, Jensen KA. Occurrence and characterization of lactic acid bacteria from Washington State wines: Pediococcus spp.[J]. American Journal of Enology and Viticulture, 1992, 43(3): 233-238 |
| [20] | G-Alegría E, López I, Ruiz JI, et al. High tolerance of wild Lactobacillus plantarum and Oenococcus oeni strains to lyophilisation and stress environmental conditions of acid pH and ethanol[J]. FEMS Microbiology Letters, 2004, 230(1): 53-61 |
| [21] | Gold RS, Meagher MM, Hutkins R, et al. Ethanol tolerance and carbohydrate metabolism in Lactobacilli[J]. Journal of Industrial Microbiology & Biotechnology, 1992, 10(1): 45-54 |
| [22] | Chen C. The study of ethanol co-fermentation with glucose and xylose[D]. Beijing: Master’s Thesis of Beijing University of Chemical Technology, 2009 (in Chinese)陈承. 葡萄糖木糖混合发酵乙醇的研究[D]. 北京: 北京化工大学硕士学位论文, 2009 |
| [23] | Kim TH, Taylor F, Hicks KB. Bioethanol production from barley hull using SAA (soaking in aqueous ammonia) pretreatment[J]. Bioresource Technology, 2008, 99(13): 5694-5702 |
| [24] | Buaban B, Inoue H, et al. Bioethanol production from ball milled bagasse using an on-site produced fungal enzyme cocktail and xylose-fermenting Pichia stipitis[J]. Journal of Bioscience and Bioengineering, 2010, 110(1): 18-25 |
| [25] | London J, Chace NM. Pentitol metabolism in Lactobacillus casei[J]. Journal of Bacteriology, 1979, 140(3): 949-954 |
 2015, Vol. 42
2015, Vol. 42




