扩展功能
文章信息
- 林强, 陈洲琴, 胡育龙, 洪文荣
- 庆大霉素生物合成基因genA的功能
- Function of genA in gentamicin biosynthesis gene cluster
- 微生物学通报, 2015, 42(2): 300-306
- Microbiology China, 2015, 42(2): 300-306
- 10.13344/j.microbiol.china.140440
-
文章历史
- 收稿日期: 2014-05-27
- 接受日期: 2014-09-18
- 优先数字出版日期(www.cnki.net): 2014-09-18
庆大霉素是一类广谱氨基糖苷类抗生素,对大部分革兰氏阳性和阴性病原菌有较强的抑制和杀灭作用[1]。根据其化学结构的差异,庆大霉素又可分为A族、B族、C族、X族。其中C族由于具有良好抗菌活性,已广泛应用于临床;A、B、X族抗菌活性相对较弱,虽未得到较好利用,但其潜在的抗原虫、抗病毒、抗肿瘤功能,受到药物学家的广泛关注。
庆大霉素产生菌是绛红色小单孢菌和棘孢小单孢菌。其生物合成途径研究已经进行了几十年。但是由于小单孢菌细胞壁特殊,且缺乏相应的分子遗传学操作手段,在很长的时间里,庆大霉素的生物合成研究进展缓慢。随着分子生物学和生物信息学技术的快速发展,从理论上对庆大霉素生物合成进行研究成为了可能。2004年之后,不同菌株的庆大霉素生物合成基因簇陆续被公布(GenBank登录号:AY524043,AJ628149,AJ575934,JQ975418),并预测了其中各个基因的功能。2009年,Kudo等[2]借助生物信息学软件,结合相关实验报道,进一步从理论上阐述了庆大霉素生物合成过程及各个基因的关系。2012年,Hong等[3]通过同源重组敲除gntK基因,阐明其为编码绛红糖胺C6′位的甲基化酶基因。2013年,Kim等[4]对gntK基因进行体外表征,进一步证实了这一结论。因此,利用基因工程手段,对庆大霉素生物合成基因进行研究是可行的。理论推测genA基因是一个关键的保守基因,GenA是一个疏水性蛋白,其氨基酸序列存在甲基转移结构域,可能参与修饰庆大霉素的生物合成,但是尚未被生物学实验所证实。
本研究借助已有的小单孢菌接合转移体系,在阐明gntK基因功能的基础上,利用分子遗传学操作手段对genA基因进行框内失活研究。通过分析genA缺失工程菌的次级代谢产物是否发生变化,进而推测genA基因的功能位点,为阐明庆大霉素生物合成途径提供科学依据。
1 材料与方法 1.1 材料 1.1.1 质粒和菌株:克隆载体pMD19-T购自TaKaRa公司。Escherichia coli Top10、Escherichia coli ET12567/pUZ8002、温敏性质粒pKC1139为本实验室保存。岀发菌Micromonospora purpurea G1008为实验室选育和保藏。 1.1.2 培养基及抗生素:绛红色小单孢菌G1008固体培养基(MM)、种子培养基(SM)及发酵培养基(FM)参见文献[3]。LB细菌培养基和接合转移使用的MS固体培养基参见文献[5]。本研究中使用的抗生素及其终浓度分别为:氨苄青霉素100 mg/L、安普霉素50 mg/L、氯霉素25 mg/L、卡那霉素25 mg/L、萘啶酮酸25 mg/L。 1.1.3 主要试剂:Taq DNA聚合酶、限制性内切酶和T4 DNA连接酶购自TaKaRa公司;溶菌酶、RNase A酶、Proteinase K和DNA凝胶回收试剂盒购自生工生物工程(上海)有限公司;其他常规试剂见文献[5]。 1.2 方法 1.2.1 引物设计:根据已公布的庆大霉素生物合成基因簇序列(GenBank登录号JQ975418),通过生物学软件Vector NTI 11.5设计靶基因序列上下游同源交换臂引物P1/P2和P3/P4,以及双交换鉴定引物P5/P6和P7/P8。P1:以绛红小单孢菌G1008的基因组DNA为模板,通过PCR扩增获得同源交换臂B1 (1 771 bp)和B2 (1 945bp)。将扩增的两条交换臂分别克隆到pMD19-T载体,得到阳性克隆子,提取质粒,分别命名为质粒pAB101和质粒pAB102。
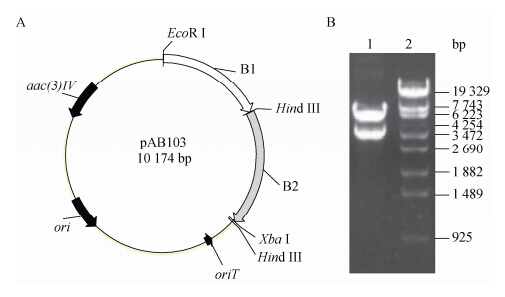
质粒pAB101用 (EcoRI/HindIII) 双酶切,电泳回收B1片段;质粒pAB102用(Hind III/Xba I)双酶切,电泳回收B2片段;温敏性质粒pKC1139用(EcoR I/Xba I)双酶切,并电泳检测分离,回收长片段。将回收的3个片段经酶连,转化大肠杆菌Top10感受态细胞,筛选阳性克隆子,获得穿梭质粒pAB103 (图 1A)。理论上穿梭质粒,用(EcoR I/XbaI)双酶切可以得到3 728和6446 bp的线性片段,结果与理论预测相一致,初步证明穿梭质粒pAB103构建正确(图 1B)。
|
| 图 1 穿梭质粒 pAB103 酶切鉴定图 Figure 1 Verification of the shuttle plasmid pAB103 by endonuclease digestion Note: 1: pAB103 (EcoR I/Xba I); 2: marker (λ-EcoT14). |
将穿梭质粒pAB103转入E. coli ET12567(pUZ8002)感受态中,得到供体菌E. coli ET12567(pUZ8002,pAB103)。然后,通过接合转移方法,将供体菌中的穿梭质粒pAB103导入绛红小单孢菌G1008。供体菌与受体菌细胞数量比按大约1:1混合。37 °C培养20 h后,用1 mL的安普霉素和萘啶酸(终浓度分别为50 mg/L和25 mg/L)水溶液覆盖,37°C继续培养7 d长出接合子。由于质粒pAB103是温度敏感型质粒,培养温度高于34 °C质粒就不能在绛红色小单孢菌中进行复制,只有该质粒整合到G1008基因组中才能表达安普霉素抗性,因此长出来的接合子是单交换菌株。随机挑取其中一株命名为绛红小单孢菌GA1。
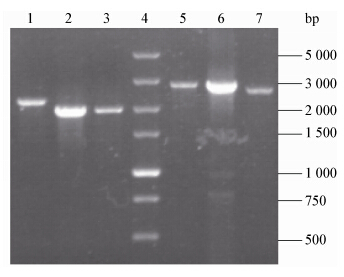
为进一步验证单交换菌株的正确性,根据同源重组原理(图 2),以GA1基因组DNA为模板,分别用引物P5/P6和P7/P8进行PCR,引物P5/P6扩增得到1 948 bp片段,与亲株相比片段减小225 bp,引物P7/P8扩增得到2 720 bp片段,与亲株条带大小一致,电泳检测结果见图 3,初步推断质粒pAB103已经整合到绛红小单孢菌G1008的基因组上,并且是同源交换臂B1上发生一次重组。

|
| 图 2 质粒 pAB103 与染色体 DNA 同源重组示意图 Figure 2 Homologous recombination between the plasmid pAB103 and chromosome DNA |
|
| 图 3 PCR 鉴定重组突变菌 Figure 3 The PCR verification of the recombinant mutant Note: 1: G1008 (P5/P6); 2: GA1 (P5/P6); 3: GA1048 (P5/P6);4: DL5000 bp marker; 5: G1008 (P7/P8); 6: GA1 (P7/P8); 7: GA1048 (P7/P8). |
由于单交换菌株GA1基因组上存在重复序列,不稳定,容易发生二次重组,产生回复突变菌株或是genA框内缺失工程菌。因此,进一步筛选双交换工程菌时,将GA1在斜面培养基上松弛培养3代,然后分离单菌落,将单菌落影印至添加安普霉素的抗性平板和不添加抗生素的普通平板上,培养后,取其中一株在抗性板上不长的安普霉素敏感菌株,提取其基因组DNA,再分别用引物P5/P6和P7/P8进行PCR验证,引物P5/P6扩增得到 1 948 bp片段,引物P7/P8扩增得到2 495 bp片段,与亲株相比片段均减小225 bp,初步确定该菌株是目标突变菌,命名为GA1048,电泳检测见图 3,经测序比对,证明GA1048确实为基因genA框内缺失工程菌。
2.3 GA1048 工程菌生长状态考察工程菌GA1048经斜面培养基连续培养3代,与出发菌株G1008相比,其菌落形态、菌丝体生长、生长周期没有明显改变(图 4),说明genA的敲除对菌株的表型没有明显的影响,遗传性状稳定。

|
| 图 4 G1008 和 GA1048 生长状态 Figure 4 The growth state of the G1008 andGA1048 注:A:第4天; B: 第6天; C:第8天. Note: A: The fourth day; B: The sixth day; C: The eighthday. |
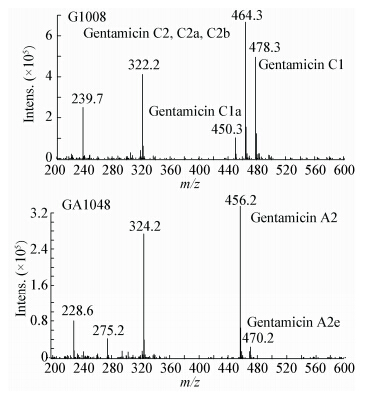
绛红小单孢菌G1008和GA1048工程菌同时按照1.2.3中的方法进行摇瓶发酵,提取代谢产物,并进行质谱分析(图 5)。
|
| 图 5 G1008 和 GA1048 次级代谢产物质谱分析图 Figure 5 MS analysis of metabolites from the G1008 and GA1048 |
结果表明,工程菌GA1048与出发菌G1008的代谢产物有明显差异。工程菌GA1048的主要离子峰为456[M+H]+,与庆大霉素A2同质,不再产生庆大霉素C1、庆大霉素C1a、庆大霉素C2、C2a和C2b。此外,工程菌GA1048的次要离子峰为470[M+H] +,根据Kim等[10]推测,该组分为庆大霉素A2的衍生物庆大霉素A2e(图 5)。
2.5 genA基因回补实验以绛红小单孢菌G1008的基因组DNA为模板,通过P1/P4引物PCR扩增获得3 941 bp片段,将其克隆到pMD19-T载体,得到阳性克隆子,提取其质粒,并命名为质粒pAB104。质粒pAB104和质粒pKC1139分别用(EcoR I/Xba I)双酶切,并电泳回收大片段。回收的两个片段经酶连,转化大肠杆菌Top10感受态细胞,筛选阳性克隆子,获得genA基因回补质粒pAB105,并将其转入E. coliET12567(pUZ8002)。
然后,通过接合转移方法,将供体菌中的回补质粒pAB105导入到绛红小单孢菌GA1048。筛选获得一株pAB105质粒整合到GA1048染色体上的重组菌株,命名为绛红小单孢菌GH1。该重组菌株存在同源片段,不稳定,容易发生二次重组。通过影印点板,再经安普抗性和PCR扩增筛选获得工程菌GH110。GH110工程菌同样按照1.2.3中的方法进行摇瓶发酵,提取代谢产物,并进行TLC分析(图 6)。

|
| 图 6 G1008 和 GH110 次级代谢产物薄层层析图 Figure 6 TLC analysis of metabolites from the G1008 and GH110 |
结果表明,工程菌GH110重新获得产庆大霉素C族组分的能力,与绛红小单孢菌G1008相比没有明显差异。
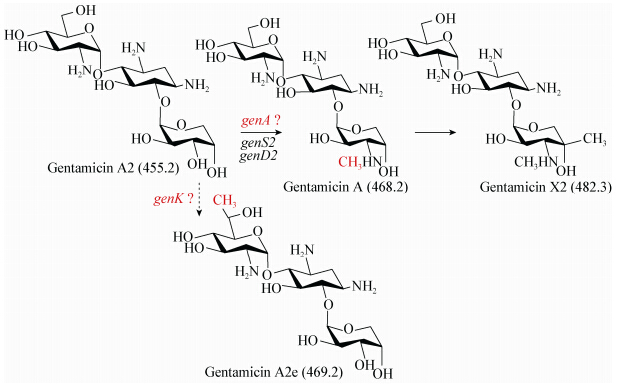
3 讨论庆大霉素A2是庆大霉素生物合成过程中的第一个拟三糖中间体,随后经生物化学修饰成为庆大霉素A,再进一步形成庆大霉素X2 (图 7)。从庆大霉素A2到庆大霉素A这一转化,可能涉及3个反应,分别是C3″位的脱氢作用以及氨化和氨甲基化。通过基因genA框内缺失,阻断了其在绛红小单孢菌G1008中的表达,得到工程菌GA1048,代谢产物发生显著变化。质谱分析表明,工程菌代谢产物主要积累庆大霉素A2和A2e,证明了genA参与加洛糖胺C-3″位的修饰作用。此外,在绛红小单孢菌G1008上缺失genA基因积累庆大霉素A2和A2e,这一结果进一步证明了Kim等[10]对甲基转移酶GntK专一性不高的推测。
|
| 图 7 基因genA催化庆大霉素生物合成途径位点 Figure 7 The functional sites of the genA in gentamicin biosynthetic pathway |
本研究通过Swiss-model等生物信息学软件分析,推测基因genS2和genD2参与从庆大霉素A2到庆大霉素A这一转化。GenS2存在氨基转移结构域,是一个氨基转移酶,而GenD2与NADH依赖的脱氢酶同属一个蛋白家族,是一个脱氢酶。在理论预测的基础上,本实验组对这两个基因进行失活研究,结果代谢产物也是积累在庆大霉素A2。实验结果与理论预测相符,证明了基因genD2[11]和基因genS2[12]分别参与加洛糖胺C-3″位的脱氢作用和氨化反应。此外,通过VectorNTI 11.5软件,将基因genA的ORF翻译成氨基酸序列,并在NCBI上进行保守结构域分析,结果表明GenA与转录调节蛋白ExsB以及核苷生物合成蛋白QueC具有较高的同源性。同时,在GenA的前60个氨基酸存在多功能结构域,该结构域与烟酰胺腺嘌呤二核苷酸合成酶、天冬酰胺合成酶以及琥珀酸精氨酸合成酶密切相关,也与tRNA甲基转移酶结构域有关,参与某些tRNA的甲基化修饰。因此,可以推测GenA是一个调节蛋白,或者是一个甲基化酶,参与加洛糖胺C-3″位的氨甲基化。
| [1] | KumarCG, Himabindu M, Jetty A. Microbial biosynthesis and applications ofgentamicin: a critical appraisal[J]. Critical Reviews in Biotechnology, 2008,28(3): 173-212 |
| [2] | KudoF, Eguchi T. Biosynthetic genes for aminoglycoside antibiotics[J]. The Journalof Antibiotics, 2009, 62: 471-481 |
| [3] | HongWR, Yan LB. Identification of gntK, a gene required for the methylationof purpurosamine C-6′ in gentamicin biosynthesis[J]. The Journal of General andApplied Microbiology, 2012, 58(5): 349-356 |
| [4] | KimHJ, McCarty RM, Ogasawara Y, et al. GenK-catalyzed C-6′ methylation in thebiosynthesis of gentamicin: isolation and characterization of acobalamin-dependent radical SAM enzyme[J]. Journal of the American ChemicalSociety, 2013, 135(22): 8093-8096 |
| [5] | TobiasK, Bibb M, Mark JB, et al. Practical Streptomyces Genetics[M]. Norwich: TheJohn Innes Foundation, 2000 |
| [6] | ZhuBY, Hong WR, Yan SD, et al. Construction of DNA homologous recombination systemin Streptomyces tenebrarius[J]. Biotechnology bulletin, 2011, 4: 162-166 (in Chinese)朱碧银, 洪文荣, 严绍德, 等. 黑暗链霉菌DNA同源重组系统的构建[J]. 生物技术通报, 2011, 4: 162-166 |
| [7] | HongWR, Yan SD. Engineering Streptomyces tenebrarius to synthesize singlecomponent of carbamoyl tobramycin[J]. Letters in Applied Microbiology, 2012,55(1): 33-39 |
| [8] | BiermanM, Logan R, Obrien K, et al. Plasmid cloning vectors for the conjugal transferof DNA from Escherichia coli to Streptomyces spp.[J]. Gene, 1992,116(1): 43-49 |
| [9] | YanLB, Hong WR, Fang ZK,et al. Construction of the conjugal transfersystem for Micromonospora purpurea G1008[J]. Chinese Journal ofAntibiotics, 2011, 12, 36(12): 899-904 (in Chinese)严凌斌, 洪文荣, 方志锴, 等. 绛红色小单孢菌G1008接合转移体系的构建[J]. 中国抗生素杂志, 2011, 36(12): 899-904 |
| [10] | KimJY, Suh JW, Kang SH, et al. Gene inactivation study of gntE reveals itsrole in the first step of pseudotrisaccharide modifications in gentamicinbiosynthesis[J]. Biochemical and Biophysical Research Communications, 2008,372(4): 730-734 |
| [11] | QueXQ, Chen ZB, Hong WR. Research on genD2 in gentamicin biosynthesis genecluster[J]. Journal of China Pharmaceutical University, 2014, 45(2): 237-241 (inChinese) 阙新桥, 陈泽斌, 洪文荣. 庆大霉素生物合成基因genD2的研究[J]. 中国药科大学学报, 2014, 45(2): 237-241 |
| [12] | ZhangSZ. Study on the aminotransferase gene of gentamicin biosynthesis[D]. Fuzhou:Master’s Thesis of fuzhou university, 2013 (in Chinese) 张书祖. 庆大霉素生物合成氨基转移酶基因的研究[D]. 福州: 福州大学硕士学位论文, 2013 |
 2015, Vol. 42
2015, Vol. 42




