扩展功能
文章信息
- 沈清清, 刘芳, 陈红惠
- 维氏气单胞菌 MreB 蛋白的结构分析及其系统发育学意义
- Structural analysis and phylogenetic implication of MreB protein in Aeromonas veronii
- 微生物学通报, 2015, 42(2): 272-279
- Microbiology China, 2015, 42(2): 272-279
- 10.13344/j.microbiol.china.140770
-
文章历史
- 收稿日期: 2014-10-10
- 接受日期: 2014-12-03
- 优先数字出版日期(www.cnki.net): 2014-12-05
2. 文山学院 化学与工程学院 云南 文山 663000
2. College of Chemistry and Engineering, Wenshan University, Wenshan, Yunnan 663000, China
长期以来人们认为细胞骨架蛋白是真核细胞所特有的成分,原核细胞中不存在该类蛋白,但近二十年来,人们在原核生物中陆续发现了在结构和功能上与3种真核骨架蛋白(肌动蛋白、微管蛋白和中间丝)相类似的蛋白,MreB就是其中之一,它与真核生物肌动蛋白丝类似性较高,具有决定细胞形态、调控染色体分离、维持生物活性和调控细胞壁合成等方面的功能,目前在许多细菌如肺炎克雷伯菌(Klebsiella pneumoniae)、解淀粉芽孢杆菌(Bacillus amyloliquefaciens)和大肠埃希氏菌(Escherichia coli)等菌[1]中均发现了MreB蛋白的 存在。
维氏气单胞菌(Aeromonas veronii)为气单胞菌属革兰氏阳性杆菌,是一种人-鱼共患致病菌,危害性强,能引发人类腹泻、脑膜炎和败血症等疾病,因对该菌预防和治疗难度较大,至今仍缺乏有效的防治手段,目前一些西方国家已将其作为食品安全的检疫项目[2, 3, 4, 5, 6, 7, 8, 9, 10, 11]。2012年Hadi 等[12]报道了维氏气单胞菌中也存在MreB蛋白,且该蛋白对维氏气单胞菌细胞形态建成与维持具有关键性作用,一旦MreB蛋白结构发生解体,维氏气单胞菌的生存与活性将受到不可逆转的扼制。这一发现对维氏气单胞菌的防治提供了新的启示:如果能阻碍MreB蛋白的表达或使其蛋白结构解体势必抑制其正常生长,因此对维氏气单胞菌MreB蛋白化学结构详细信息的解析将有助于寻找阻碍蛋白表达或降解蛋白的方法与途径,以及为相关防治药物设计提供重要参考。但目前MreB蛋白的三维晶体结构尚未获得,因此本研究利用生物信息学手段对MreB蛋白的一级、二级结构进行研究与分析,通过同源建模的原理对该蛋白的三级结构进行预测,旨在为实验研究提供一定的结构基础;另外,近年来蛋白组学研究的发展表明氨基酸序列比DNA序列更为保守,能为物种的进化提供更为可靠的信息,因此对不同物种MreB蛋白同源性进行比较并分析维氏气单胞菌与其他几种微生物的进化关系,以期为MreB蛋白在系统发育学研究中的应用奠定理论基础和参考价值。
1 分析材料分析材料全部来源于美国生物信息中心(National Center for Biotechnologyinformation) Protein数据库;MreB (Aeromonas veronii bv.sobria)登录号:AFF60301。
2 方法 2.1 MreB蛋白的结构分析 2.1.1 蛋白一级结构分析:利用瑞士ExPASy网站上的生物信息学软件ProtParam分析MreB蛋白的氨基酸总数、分子量、等电点和亲水性等参数;利用在线工具PSORT预测蛋白的亚细胞定位。 2.1.2 蛋白二级结构分析:将序列提交到欧洲分子生物学实验室提供的PredictProtein服务器[13]预测MreB的二级结构,分析蛋白的局部结构元件。 2.1.3 蛋白三级结构预测与分析:将序列提交至瑞士生物信息学研究所(SIB)建立的蛋白质分析专家系统ExPASy,利用SWISS-MODEL服务器对MreB蛋白进行同源建模,获得该蛋白的三级结构和相关参数;再利用加利福尼亚大学洛杉矶分校(UCLA)在线工具Structural Analysis and Verification Server对该蛋白的三级结构的可信度与合理性进行评估(PROCHECK)。 2.2 氨基酸序列比对与系统发育分析将序列输入NCBI的网页中,从GenBank中Blast出与维氏气单胞菌MreB蛋白相似性在53%以上、覆盖率在95%以上的蛋白,选择不同物种来源的MreB蛋白,使用clustalX 2.0进行比对,之后再利用蛋白及核酸分析工具ESpript 3.0美化与分析多重比对序列图。根据比对结果用MEGA 6构建系统进化树。Bootstrap分析中使用1 000次重复计算NJ树的支持率。
3 结果与分析 3.1 MreB 氨基酸组成及理化性质分析维氏气单胞菌MreB蛋白由346个氨基酸组成,含19种氨基酸,其中甘氨酸(Gly)含量最高为10.7%,其次是缬氨酸(Val)含量为9.8%和丙氨酸(Ala)含量为9.2%,不含色氨酸(Trp)、吡咯赖氨酸(Pyl)和硒氨酸(Sec)。酸性氨基酸残基总数为45,碱性氨基酸总数为35;分子量为37.06kD,理论等电点PI为5.24;分子式为C1628H2646N456O502S13,原子总数是5 245;其疏水性平均值(GRAVY)为0.013。PSORT的分析结果表明在维氏气单胞菌中MreB蛋白主要定位于细胞膜上,其概率值为11.9%。
3.2 MreB二级结构预测与分析利用SOPMA服务器预测MreB蛋白的二级结构,结果如图 1所示,MreB二级结构主要由α-螺旋(Alpha helix)、扩展长链(Extended strand)和无规则卷曲(Random coil) 3种结构构成,其中α-螺旋占34.39%,扩展长链占19.08%,无规则卷曲占46.53%,表明α-螺旋和无规则卷曲是MreB蛋白的主要成分。
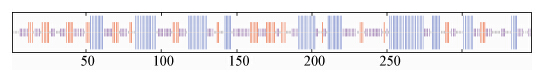
|
| 图 1 MreB二级结构分析 Figure 1 Secondary structure analysis of the MreB protein 注:横轴表示氨基酸位置;蓝色:α-螺旋;红色:扩展长链. Note: Horizontal axis indicates amino acid site. Blue: alpha helix; red: extendedstrand. |
经过计算筛选,SWISS-MODEL为预测MreB蛋白的三级结构选择了3个模板,PDB id分别为2wus、4cze和1jcg,3个模板预测的模型评估结果显示4cze蛋白为最佳模板如图 2A所示。预测出的三维模型如图 2B所示,该模型含有12个α-螺旋结构,由128个氨基酸组成,占36%;5个β-折叠结构,由98个氨基酸残基组成,占28%;另外还含有8个β-发夹结构,22个β-转角和4个γ-转角。将预测模型与模板4cze图形进行叠合处理,如图 2C所示,两者折叠模式一致,核心结构差别不大,仅有少量环区构象存在细微差异,序列同一性为48%,覆盖率为93%,覆盖范围为10-333;PROCHECK检测结果如图 3所示,100%的氨基酸残基落在允许区,其中95.2%落在最适区,4.0%落在附加区,0.7%落在宽容区。以上结果表明通过SWISS-MODEL构建的MreB蛋白三维结构符合立体化学规则,有较高的合理性与可靠性,属高质量模型(落于最适区的氨基酸残基数>90%)。
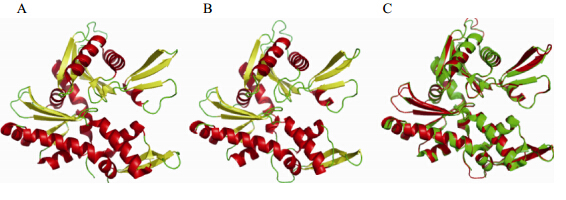
|
| 图 2 MreB蛋白的三维结构 Figure 2 Three-dimensional structure of the MreB protein 注:A:模板;B:预测模型;C:A (绿)与B (红)的叠合比较图形. Note: A: Thetemplate; B: The prediction model; C: The superposition of the two model, A (green)and B (red). |
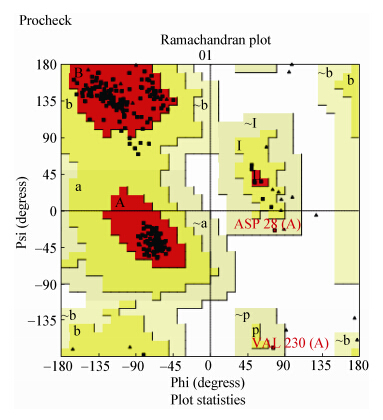
|
| 图 3 3MreB蛋白氨基酸残基二面角分布图 Figure 3 Ramachandran plot of the MreB protein |
NCBI数据库中Blast搜索结果显示维氏气单胞菌MreB蛋白具有高度保守性,属于Sugar kinase/HSP70/actin超家族。将7个物种即Aeromonas veronii (登录号:AFF60301.1)、Aeromonas hydrophila (登录号:WP_017778502.1)、Oceanimonas smirnovii (登录号:WP_019935063.1)、Glaciecola pallidula(登录号:WP_006012812.1)、Haemophilus influenza (登录号:P44474.1)、Caulobacter vibrioides (登录号:4CZE_A)、Escherichia coli (登录号:P0A9X4.1)的MreB蛋白与另外3个物种即Geobacillus sp. (登录号:C5D4U1.1)、Bacillus pumilus (登录号:A8FFD2.1)和Bos Taurus (登录号:2QW9_A)的热休克(HSP70)蛋白氨基酸序列进行比对,结果如图 4所示,MreB蛋白主要含有核酸粘附位点、Mg2+粘附位点和RodZ相关位点,这些位点的氨基酸残基均具有高度的保守性。氨基酸序列比对结果显示细菌的MreB同源性较高,与同一属的细菌相似性达96%以上,与其他细菌相似性达56%−93%之间,另外与同属一个超家族的HSP70蛋白含有25%以上的同源序列。将图 5所列出的7种细菌即AFF60301.1到P0A9X4.1的MreB蛋白的三级结构进行叠合比对,发现几种MreB蛋白三级结构也极为相似,仅少数表面环区构象存在细微差异。

|
| 图 4 蛋白氨基酸序列比对 Figure 4 Amino acid residue sequence alignment of proteins 注:AFF60301.1到P0A9X4.1为MreB蛋白,C5D4U1.1到2QW9_A为热休克(HSP70)蛋白,完全保守的残基用红色阴影表示,核酸粘附位点用浅蓝色阴影表示,Mg2+粘附位点用深蓝色阴影表示,RodZ相关位点用黄色阴影表示. Note: AFF60301.1 to P0A9X4.1 are MreB protein. C5D4U1.1 to 2QW9_A are heat shock protein (HSP70) protein. Amino acids completely conserved are shaded in red, nucleotide binding site are shaded in lightblue, Mg2+ binding site are shaded in deep blue, RodZ interactionsite are shaded in yellow. |
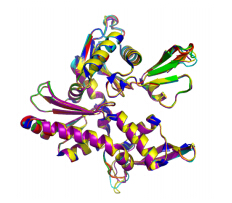
|
| 图 5 MreB蛋白晶体结构、同源建模结构的叠合比较 Figure 5 Superposition of crystalstrucutures and homolog models of 7 MreB proteins 注:不同的分子用不同颜色表示. Note: Different structures are drawn with different colors. |
将维氏气单胞菌MreB蛋白氨基酸序列键入GenBank数据库中进行检索,将检索结果中分属于发光杆菌属(Photobacterium sp.)、邻单胞菌属(Plesiomonas sp.)、埃希氏菌属(Escherichia sp.)、弧菌属(Vibrio sp.)、噬琼胶菌属(Agarivorans sp.)、铁单胞菌属(Ferrimonassp.)、甲苯单胞菌属(Tolumonas sp.)、气单胞菌属(Aeromonas sp.)共14个菌种的MreB蛋白序列构建系统发育树,进行系统进化分析,结果如图 6所示,整棵进化树明显分为两支,弧菌科中的发光杆菌属、邻单胞菌属和弧
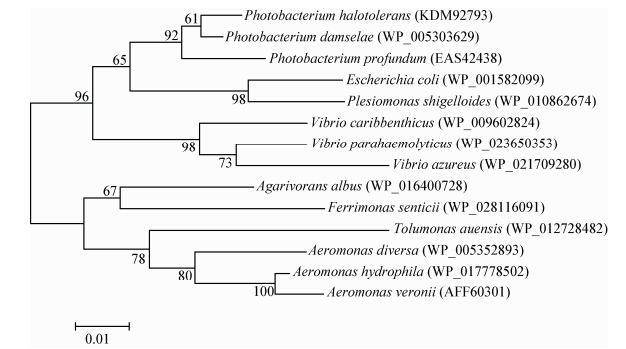
|
| 图 6 MreB蛋白系统发育树分析 Figure 6 Analysis of phylogenetic tree of the MreB protein |
菌属以及肠杆菌科中的埃希氏菌属同属于第一分支,而噬琼胶菌属、铁单胞菌属、甲苯单胞菌属、气单胞菌属同属于第二分支,其中维氏气单胞菌与同属的另外两个菌种即Aeromonas diversa和Aeromonas hydrophila亲缘关系最近。整棵树明显将传统分类法下同一科系的菌种聚集在同一分支下,同时也将不同科系的菌种区分聚类。表明MreB蛋白进化上保守性较高,变异度适宜,能够较好的用于研究含MreB蛋白细菌的分子系统发育与进化关系,可以在科属级水平上将细菌进行归类。
4 讨论分析蛋白的结构是后基因组计划中一个重要的研究内容,研究蛋白的结构能帮助人类认识蛋白的功能和生物学意义,对生物学、医学和药学领域的发展具有举足轻重的作用[14, 15]。尽管近年来X射线晶体衍射和核磁共振技术(NMR)有了很大进步,但目前蛋白质空间结构的测定速度进展仍较缓慢,大部分蛋白的晶体结构尚未解析,因此应用计算机技术预测蛋白高级结构并利用生物信息学分析蛋白性质已成为一种快速且具较高可信度的解析蛋白的方法[16]。目前国内已有一些学者开始应用ProtParam、SWISS-MODEL、Swiss Pdb-Viewer、Protscale和HNN等在线工具或软件对蛋白进行分析与结构预测[17, 18, 19, 20, 21],均获得了详实可靠的蛋白分析数据,大幅提高了蛋白的解析效率。本研究通过一系列蛋白分析软件也获得了MreB蛋白一、二级结构相关理化性质与组成的详细信息;另外,由于蛋白数据库中筛出的MreB蛋白晶体模板相似性较高(50%以上)[22, 23, 24, 25],因此保证了建模后的模型质量,PROCHECK评估分数高,模型符合质量要求,表明预测出的三维模型具有较高可信度。值得注意的是,由于蛋白结构性质和作用途径极其复杂,软件分析与预测结果并不能作为最为确切的结论,但无疑本研究结果为进一步实验验证提供了参考与指导,具有重要的意义与价值。
细菌为原核生物,缺乏复杂的表型结构,因此过去利用细菌表型特征对细菌进行分类鉴定与进化研究的传统方法存在较大偏差,直到20世纪70年代之后,随着核酸测序技术的发展,人们开始利用16S rDNA作为分子标记对细菌进行分子鉴定与系统发育研究,使得细菌相关研究取得了较大进展,到目前16S rDNA分子标记技术已成为研究菌种鉴定和分类的一种成熟的技术手段[26, 27]。然而20世纪90年代,人们发现基因除了可发生纵向转移外也存在水平转移的现象,并且在不同物种或同一物种之间都可发生水平转移,如果仍仅依靠16S rDNA作为唯一的分子鉴定指标已明显缺乏说服力与可靠性[28, 29],因此把16SrDNA与其他的分子指标结合进行系统学研究才是客观且可信度更高的研究方法。肌动蛋白作为真核生物中广泛存在的一类蛋白,因具有高度保守性,一直被科研人员作为研究生物进化的常用工具,人们在细菌中发现细胞骨架蛋白类似物之后也逐渐认识到这些蛋白同样也属氨基酸序列与结构高度保守的蛋白质[28, 30, 31, 32, 33, 34, 35],具备作为分子指标的特性,本文对几种细菌MreB蛋白氨基酸序列比对及系统发育学分析研究的结果,也充分证实了MreB是一种保守性较高的蛋白质,可在科属级水平上将细菌进行明确的分类 鉴定。
5 结论通过一系列的蛋白分析软件对维氏气单胞菌MreB蛋白进行了研究,根据蛋白一级结构的氨基酸组成及理化性质进行了分析,确定其为酸性蛋白;预测了该蛋白的二级结构,表明α-螺旋和无规则卷曲结构是其主要结构;同源建模预测的三级结构评估分数较高,能基本反映出MreB蛋白真实的空间构象。通过对几个物种中存在的MreB蛋白的氨基酸序列进行比对及系统进化分析后,结果表明MreB蛋白进化上具有较高的保守性,适合应用于细菌科属级水平的分子系统发育与进化关系研究。
| [1] | De Maayer P, Chan WY,Venter SN, et al. Genome sequence of pantoea ananatis LMG20103, the causativeagent of eucalyptus blight and dieback[J]. Journal of Bacteriology, 2010,192(11): 2936-2937 |
| [2] | Hickman-Brenner FW,Macdonald KL, Steigerwalt AG, et al. Aeromonas veronii, a newornithine decarboxylase-positive species that may cause diarrhea[J]. Journal ofClinical Microbiology, 1987, 25(8): 900-906 |
| [3] | Zhang LX, Zhang L, Du BF.One aeromonas veronii was isolated from sputum of patients with severepneumonia[J]. Chinese journalof nosocomiology, 2008, 18(5):680 (in Chinese) 张丽霞, 张立, 杜佰风. 重症肺炎患者痰中培养出维氏气单胞1例[J]. 中华医院感染学杂志, 2008, 18(5): 680 |
| [4] | Joseph SW, Carnahan AM,Brayton PR, et al. Aeromonas jandaei and Aeromonas veroniidual infection of a human wound following aquatic exposure[J]. Journal ofClinical Microbiology, 1991, 29(3): 565-569 |
| [5] | Wu TL, Dan XF, Meng QF,et al. Advances in Aeromonas veronii[J]. Chinese journal of veterinarydrug, 2011, 45(7): 41-44 (inChinese)吴同垒, 单晓枫, 孟庆峰, 等. 维氏气单胞菌研究进展[J]. 中国兽药杂志, 2011, 45(7): 41-44 |
| [6] | Zhou G, Xie J, Zhou QL, etal. The pathogenicity and drug-resistance of Aeromonas veronii in differenceecological nichefrom Megalobrama amblycephala culture pond[J]. Jiangsuagricultural sciences, 2012, 40(5): 192-196 (inChinese)周光, 谢骏, 周群兰, 等. 团头鲂养殖池塘维氏气单胞菌的致病性耐药性及在不同生态位间的差异[J]. 江苏农业科学, 2012, 40(5): 192-196 |
| [7] | Gong Q, Gao SQ, San XF, etal. Isolation and identification of pathogenic Aeromonas veronii from Cyprinuscarpio[J]. Chinese journal of preventive veterinary medicine,2010, 32(12): 981-983 (in Chinese)龚倩, 高淑琴, 单晓枫, 等. 框镜鲤致病性维氏气单胞菌的分离鉴定[J]. 中国预防兽医学报, 2010, 32(12): 981-983 |
| [8] | Wang ZH. Development andapplication of Loop-Mediated isothermal amplification for detection of Aeromonasveronii[D]. Ya’an: master’s thesis of Sichuan agricultural university, 2013 (in Chinese)王子浩. 致病性维氏气单胞菌LAMP方法的建立与应用[D]. 雅安: 四川农业大学硕士学位论文, 2013 |
| [9] | Huang WM, Wang L, Ji GZ, etal. Identification and drug sensitivity of bacterial Aeromonas veronii andA. media isolated from Chinese sucker Myxocyprinus asiaticus[J].Fisheries science, 2013, 32(4):210-214 (in Chinese)黄文明, 王利, 冀国桢, 等. 胭脂鱼维氏气单胞菌和中间气单胞菌的鉴定及药物敏感性[J]. 水产科学, 2013, 32(4): 210-214 |
| [10] | Xu XL, Shao P, Cui KK, etal. Identification of bacterial pathogens isolated from the diseased Xiphophorushellerii[J]. Freshwater fisheries,2014, 44(1): 66-72 (in Chinese) 徐晓丽, 邵蓬, 崔宽宽, 等. 剑尾鱼烂鳃、烂尾病病菌的分离鉴定[J]. 淡水渔业, 2014, 44(1): 66-72 |
| [11] | Zhao M, Wang KY, Wang J, etal. Tetracycline antibiotics resistance and its genetic determinants in Aeromonasveronii isolated from channel catfish[J]. Acta hydrobiologica sinica,2014, 38(2): 386-392 (in Chinese)赵敏, 汪开毓, 王均, 等. 斑点叉尾鮰源维氏气单胞菌对四环素类抗生素的耐药性及耐药基因的检测[J]. 水生生物学报, 2014, 38(2): 386-392 |
| [12] | Hadi N, Yang Q, BarnettTC, et al. Bundle-forming pilus locus of Aeromonas veronii bv.sobria[J]. Infection and Immunity, 2012, 80(4): 1351-1360 |
| [13] | Rost B, Yachdav G, Liu J.The PredictProtein server[J]. Nucleic Acids Research, 2004, 32(Web Server):W321-W326 |
| [14] | Berman HM, Westbrook J, Feng Z, et al. The protein data bank[J]. Nucleic Acids Research,1999, 28(1): 235-242 |
| [15] | Brenner SE, Levitt M. Expectations from structural genomics[J]. ProteinScience, 2000, 9(1): 197-200 |
| [16] | Peng QS, Hu M. Approachesfor 3D protein structure similarity comparison―a survey[J]. Journal of computer-aided design & computer graphics, 2006,18(10): 1465-1471 (in Chinese)彭群生, 胡敏. 蛋白质三维结构相似性比较方法综述[J]. 计算机辅助设计与图形学学报, 2006, 18(10): 1465-1471 |
| [17] | Lu ZC, Wang BP, Li ZX. Structureanalysis and homology modeling of Cra c 8 protein from Crangon crangon[J].Food science, 2012, 33(9): 177-181 (in Chinese) 陆宗超, 王邦平, 李振兴. 褐虾过敏原Cra c 8蛋白的结构分析和同源建模[J]. 食品科学, 2012, 33(9): 177-181 |
| [18] | Yuan XH, Wang YC, Qu ZY, etal. Novel rapid molecular modeling method based on evolutional tree for humanadenovirus hexon proteins family[J]. Chemical journalof chinese universities, 2011, 32(8): 1838-1844 (inChinese) 袁晓辉, 王迎晨, 曲章义, 等. 进化树指导的腺病毒六邻体家族蛋白的快速建模[J]. 高等学校化学学报, 2011, 32(8): 1838-1844 |
| [19] | Wang SJ, Lin HP, Li SK.Structure analysis and homology modeling of NOS protein from Mud Crab Scyllaparamamosain[J]. Journal of shantouuniversity (Natural ScienceEdition) , 2013, 28(2): 39-44 (in Chinese)王世佳, 林浩鹏, 李升康. 拟穴青蟹(Scylla paramamosain)NOS蛋白结构分析及同源建模[J]. 汕头大学学报: 自然科学版, 2013, 28(2): 39-44 |
| [20] | Zhang XM, Li PL. Quorumsensing in class II bacteriocin-producing lacticacid bacteria and itsapplication-A review[J]. Acta microbiologicasinica, 2011, 51(9): 1152-1157 (inChinese)张香美, 李平兰. 产Ⅱ类细菌素乳酸菌群体感应及其应用[J]. 微生物学报, 2011, 51(9): 1152-1157 |
| [21] | Zhang Y, Mao CQ.Sequence analysis and structure modeling study of acyl-ACP desaturase from Flavobacteriumpsychrophilum[J]. Journal of Biology, 2014, 31(4): 50-54 (in Chinese) 张远, 茆灿泉. 嗜冷黄杆菌酰基-Acp去饱和酶(AAD)的序列分析和结构建模[J]. 生物学杂志, 2014, 31(4): 50-54 |
| [22] | van den Ent F, zoré T,Bharat TA, et al. Bacterial actin MreB forms antiparallel double filaments[J].ELife, 2014, 3: e02634 |
| [23] | van den Ent F, JohnsonCM, Persons L, et al. Bacterial actin MreB assembles in complex with cell shapeprotein RodZ[J]. The EMBO Journal, 2010, 29(6): 1081-1090 |
| [24] | Favini-Stabile S, Contreras-Martel C, Thielens N, et al. MreB and MurG as scaffoldsfor the cytoplasmic steps of peptidoglycan biosynthesis[J]. Environmental Microbiology,2013, 15(12): 3218-3228 |
| [25] | Roeben A, Kofler C, NagyI, et al. Crystal structure of an archaeal actin homolog[J]. Journal of Molecular Biology,2006, 358(1): 145-156 |
| [26] | Brown JR, Doolittle WF.Archaea and the prokaryote-to- eukaryote transition[J]. Microbiology andMolecular Biology Reviews, 1997, 61(4): 456-502 |
| [27] | Woese CR. Bacterial evolution[J]. Microbiological Reviews, 1987, 51(2): 221-271 |
| [28] | Zhang B, Dong XZ. Partialsequence homology of FtsZ in phylogenetics analysis of lactic acid bacteria[J].Acta microbiologica sinica, 2005, 45(5): 661-664 (inChinese) 张斌, 东秀珠. FtsZ蛋白同源性分析在乳酸菌系统学研究中的应用[J]. 微生物学报, 2005, 45(5): 661-664 |
| [29] | Shen QQ, Hu ZY. Research inhorizontal gene transfer from eukaryote to prokaryote[J]. Guizhou agricultural sciences, 2014, 42(10): 51-55 (in Chinese) 沈清清, 胡展玉. 基因从真核生物向原核生物水平转移的研究进展[J]. 贵州农业科学, 2014, 42(10): 51-55 |
| [30] | Wang X, Lutkenhaus J. FtsZ ring: the eubacterial divisionapparatusconserved in archaebacteria[J]. Molecular Microbiology, 1996, 21(2): 313-319 |
| [31] | Zeaiter Z, Liang Z,Raoult D. Genetic classification and differentiation of bartonella speciesbased on comparison of partial ftsZ gene sequences[J]. Journal of ClinicalMicrobiology, 2002, 40(10): 3641-3647 |
| [32] | Figge RM, Divakaruni AV, Gober JW. MreB, the cell shape-determining bacterial actinhomologue, co-ordinates cell wall morphogenesis in Caulobacter crescentus[J]. Molecular Microbiology,2004, 51(5): 1321-1332 |
| [33] | Chen XY, Wang ZP, YangLY. The structure and function of prokaryotic cytoskeleton[J]. Chinese journal of cell biology,2006, 28(5): 699-703 (in Chinese)陈晓燕, 汪志平, 杨灵勇. 原核细胞骨架蛋白的结构与功能[J]. 细胞生物学杂志, 2006, 28(5): 699-703 |
| [34] | Egelman EH. Actin’s prokaryotic homologs[J]. Current Opinion in Structural Biology, 2003,13(2): 244-248 |
| [35] | Usmanova A, Astier C,Mejean C, et al. Coevolution of actin and associated proteins: analpha-actinin-like protein in a cyanobacterium (Spirulina platensis)[J].Comparative Biochemistry and Physiology Part B:Biochemistry and Molecular Biology, 1998, 120(4): 693-700 |
 2015, Vol. 42
2015, Vol. 42




