扩展功能
文章信息
- 庞建, 刘占英, 郝敏, 兰辉, 吴涛
- PANG Jian, LIU Zhan-Ying, HAO Min, LAN Hui, WU Tao
- 革兰氏阳性细菌基因组DNA 提取方法的比较及优化
- Comparison and optimization of methods for genomic DNA extraction from Gram positive bacteria
- 微生物学通报, 2015, 42(12): 2482-2486
- Microbiology China, 2015, 42(12): 2482-2486
- 10.13344/j.microbiol.china.150133
-
文章历史
- 收稿日期: 2015-02-10
- 接受日期: 2015-04-07
- 优先数字出版日期(www.cnki.net): 2015-05-08
2. 内蒙古工业大学 煤炭转化与循环经济研究所 内蒙古 呼和浩特 010051
2. Institute of Coal Conversion & Cyclic Economy,Inner Mongolia University of Technology, Hohhot, Inner Mongolia 010051,China
随着分子生物学的不断发展,细菌基因组DNA的提取已成为一种常规的实验手段,但作为分子生物学研究的第一步,高效稳定的细菌基因组DNA提取方法对后续分子生物学的研究有着重要的意义。革兰氏阳性细菌细胞壁是由一层厚且致密的肽聚糖和磷壁酸组成,而肽聚糖的肽链通过5个甘氨酸互相交联着,不易裂解,而目前通行的分子生物学手册中,提取基因组总DNA均以革兰氏阴性菌大肠杆菌为例,这些方法在高效提取革兰氏阳性细菌基因组DNA方面有一定的局限性[1]。作为分子生物学研究的基础,细菌样品中基因组DNA的浓度、纯度、片段大小及一致性,对后续PCR和克隆的效果会产生较大影响,更制约着Q-PCR的准确性[2]。张涛涛等[3]的研究也表明DNA提取方法对基因组DNA的质量、得率以及后续PCR扩增效果影响显著,因此,选用适宜的DNA提取方法对研究至关重要。为保证革兰氏阳性菌分子生物学实验顺利开展,不但要考虑DNA提取得率,还要尽可能地减少DNA的降解[4],并保证不同批次DNA提取效率和质量的一致。
1 材料与方法 1.1 实验菌株嗜热梭菌(Clostridium thermocellum)和热解糖厌氧杆菌(Thermoanaerobacterium thermosaccharolyticum)均为革兰氏阳性细菌,由达特茅斯学院(Dartmouth College)的Lynd教授赠予。
1.2 主要试剂和仪器蛋白酶K、溶菌酶、琼脂糖、SDS均购自Sigma公司。
凝胶成像系统Bio-Rad Gel Doc 2000型,美国伯乐公司;NanoDrop ND1000微量紫外分光光度计,基因有限公司;恒温摇床,上海天呈实验仪器制造有限公司;LDZX-40Ⅱ型立式自动电热压力蒸汽灭菌器,上海申安医疗器械厂。
1.3 实验方法 1.3.1 培养基和培养条件:实验采用改良的MTC培养基(g/L)[5]:丙磺酸100 (pH 7.9);A液(酵母粉50);B液(柠檬酸钾50、柠檬酸31.25、Na2SO4 25、KH2PO4 25、NaHCO3 62.5);C液(尿素250、NH4Cl 75);D液(MgCl2·6H2O 50、CaCl2·2H2O 10、FeCl2·4H2O 5、L-半胱氨酸盐酸盐50);E液(盐酸吡哆胺1、对氨基苯甲酸0.2、生物素0.1、维生素B12 0.1、维生素B1 0.2);F液(MnCl2·4H2O 0.025、CoCl2·6H2O 0.025、ZnCl2 0.01、CuCl2·2H2O 0.002 5、H3BO3 0.002 5、Na2MoO4·2H2O 0.002 5、NiCl2·6H2O 0.002 5)。将储液按比例加入装有5 g/L微晶纤维素(或纤维二糖)为底物的西林瓶中,除氧后分别接种5% (体积比)C. thermocellum、或T. thermosaccharolyticum,于55 °C、180 r/min培养至对数生长期。
1.3.2 几种细菌基因组DNA提取方法比较:选取对数期的C. thermocellum菌种,用水煮法[1]、传统法[6]、CTAB-SDS法[7]、SDS-碱裂解法、酚氯法[8]和SDS-酶裂解法[9]提取基因组DNA。SDS-碱裂解法提取的基因组DNA产率较高,但纯度较低,为提高其提取纯度,对该法进行了改良,主要改良之处:在加入碱裂解液Ⅰ的同时加入溶菌酶和RNase A酶,改良SDS-碱裂解法的具体提取步骤为:取菌液1.5 mL,-4 °C、12 000 r/min离心2 min,弃上清液,沉淀依次加入100 μL碱裂解液Ⅰ (50 mmol/L葡萄糖,25 mmol/L pH 8.0 Tris-Cl,10 mmol/L pH 8.0 EDTA)、溶菌酶和RNase A酶,用漩涡振荡仪振荡1 min。加入200 μL现配的碱裂解液Ⅱ (0.2 mol/L NaOH溶液,1% SDS),快速温柔地颠倒5次,冰浴20 min。加入150 μL预冷的碱裂解液Ⅲ (5 mol/L醋酸钾60.0 mL,冰乙酸11.5 mL,灭菌蒸馏水28.5 mL),反复颠倒5次,冰浴5 min后-4 °C、12 000 r/min离心5 min取上清液450 μL,加入等体积的酚/氯仿/异戊醇混匀,离心,取上清液450 μL,加入2倍体积的冰冻无水乙醇,混合均匀,室温沉淀DNA 10 min后离心,干燥沉淀。沉淀加入1 mL 70%的乙醇,颠倒混匀后离心,干燥沉淀。用50 μL含有RNase A酶(10 mg/L)的TE溶液重悬,-20 °C保存。 1.3.3 基因组DNA产率和纯度及完整性检测:将提取所得样液DNA按一定倍数稀释后,用紫外分光光度计测定OD260/OD280值和OD260/OD230值,按照1个OD260值相当于50 mg/L和稀释倍数来换算DNA的浓度,并计算DNA的产率,用OD260/OD280的比值表示DNA样品的纯度。取10 μL基因组DNA提取原液,用0.8%琼脂糖凝胶,电泳(1×TAE,5 V/cm) 40 min,再用核酸染料染色,紫外透射仪观察和拍照,检测DNA条带的亮度及基因组DNA完整性。 1.3.4 统计分析:所有的数据通过3次平行实验收集得到,利用SAS软件进行方差分析,肩注小写字母表示差异的显著性,肩注大写字母表示差异的极显著性,显著性由a、b、c……的顺序依次减弱,小写字母不同表示差异显著(P<0.05),大写字母不同表示差异极显著(p<0.01),除非特别说明。 2 结果与分析 2.1 6种提取方法的DNA产率、纯度和完整性6种提取方法中,有4种能够提取出C. thermocellum基因组DNA,但提取效果有明显差异,见表 1。其中SDS-碱裂解法的产率最高,平均达268.27 mg/L,酚氯法最低,仅为93.68 mg/L;SDS-碱裂解法的OD260/OD280平均值在1.7以下,提取的DNA纯度较低,可能是残留的蛋白质较多。酚氯法的OD260/OD280平均值为1.77,提取的DNA蛋白质去除效果较好,但OD260/OD230值为0.97,DNA盐分去除效果最差,传统法和SDS-酶裂解法提取所得DNA纯度及产量均较低。综上,对SDS-碱裂解法进行改良,在保持其产率的条件下,提高纯度。
| 提取方法 Method of extraction | OD260/OD280 ( 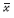 ± s) ± s) | OD260/OD230 ( 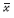 ± s) ± s) | 产率 productivity (mg/L) |
| 传统法 Traditional method | 1.48±0.04Dd | 1.10±0.03Cc | 230.85±3.13Bb |
| SDS-碱裂解法 SDS-baselysis method | 1.64±0.03Bb | 1.65±0.01Aa | 268.27±7.39Aa |
| 酚氯法 Phenol-chloroform method | 1.77±0.07Aa | 0.97±0.01Dd | 93.68±1.87Dd |
| SDS-酶裂解法 SDS-enzyme lysis method | 1.58±0.01Cc | 1.49±0.02Bb | 135.93±9.72Cc |
Note: The values in the table are:
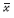 ± s. A-D:
± s. A-D: 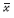 ± s within the columns with uppercase superscripts is different. a-d:
± s within the columns with uppercase superscripts is different. a-d: 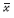 ± s within the columns of with lower-case superscripts is significantly different.
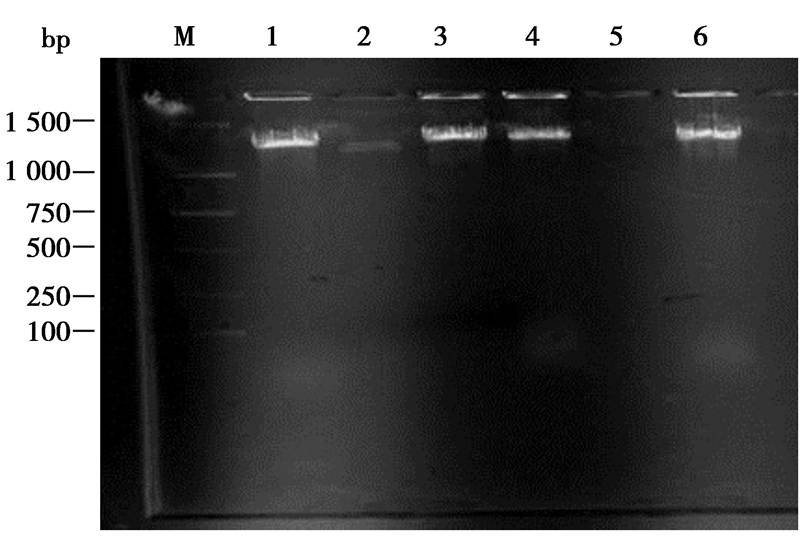
± s within the columns of with lower-case superscripts is significantly different. DNA样液琼脂糖凝胶电泳分析图谱,见图 1。从图 1中可以看出,4种提取方法均有基因组DNA条带。虽加样量均为10 μL,但DNA条带的亮度明显不同,SDS-碱裂解法和传统法的条带亮度较酚氯法和SDS-酶裂解法明显亮,即提取所得产率较高,这也说明前2种方法产率高,与紫外检测结果相符。
|
|
图 1
不同方法提取C. thermocellum基因组DNA的电泳图谱
Figure 1
The electrophoretogram of C. thermocellum genomic DNA with different extraction methods
注:M:Marker;1:传统法;2:CTAB-SDS法;3:SDS-碱裂解法;4:酚氯法;5:水煮法;6:SDS-酶裂解法. Note: M: Marker; 1: Traditional method; 2: CTAB-SDS method; 3: SDS-baselysis method; 4: Phenol-chloroform method; 5: The water boiling; 6: SDS-enzyme lysis method. |
改良SDS-碱裂解法提取C. thermocellum和T. thermosaccharolyticum基因组DNA产率见表 2。其中C. thermocellum基因组DNA产率为418.23 mg/L,较未改良前产率提高近2倍,且2株菌的OD260/OD280值和OD260/OD230值均在1.8-2.0之间,说明提取所得基因组DNA中蛋白质和盐分去除效果均较好,有利于后续实验的进行。
| 菌种 Strain | OD260/OD280 ( 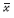 ± s) ± s) | OD260/OD230 ( 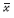 ± s) ± s) | 产率 productivity (mg/L) |
| C. thermocellum | 1.89±0.01 | 1.93±0.03 | 418.23±18.45 |
| T. thermosaccharolyticum | 1.90±0.02 | 1.87±0.02 | 397.51±12.76 |
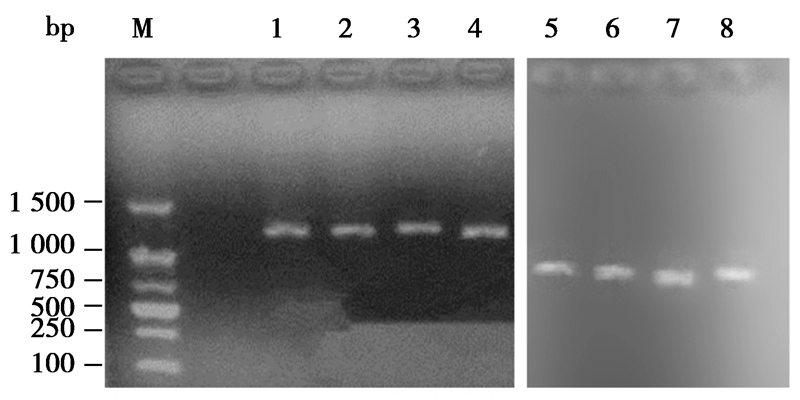
DNA样液琼脂糖凝胶电泳分析图谱,见图 2。从图 2中可以看出,改良SDS-碱裂解法提取所得基因组DNA条带完整性较好,杂质去除效果好。基因组DNA条带的亮度一致,说明提取所得基因组DNA产率基本一致,稳定性好,与紫外检测结果相符。
|
|
图 2
改良SDS-碱裂解法电泳检测图
Figure 2
The electrophoretogram of genomic DNA with improved SDS alkaline lysis method
注:M:Marker;1-4:改良SDS-碱裂解法所提C. thermocellum基因组DNA;5-8:改良SDS-碱裂解法所提T. thermosaccharolyticum基因组DNA.
Note: M: Marker; 1-4: Genomic DNA of C. thermocellum with improved SDS alkaline lysis method; 5-8: Genomic DNA of T. thermosaccharolyticum with improved SDS alkaline lysis method. |
实验通过对比目前常见的6种提取方法,对提取所得C. thermocellum的基因组DNA质量和得率进行比较后,对SDS-碱裂解法进行了改良,发现改良后的SDS-碱裂解法提取所得C. thermocellum和T. thermosaccharolyticum的DNA纯度和完整性都较高,且产率、纯度和稳定性明显比未改良前有所提高,这说明本实验在加入碱裂解液Ⅰ的同时加入溶菌酶和RNase A酶的改进是成功的,SDS不仅可以破坏细胞膜,并且可以促进溶菌酶的乳化作用[10],二者结合,促进了细胞的裂解,进而优化了基因组DNA的提取步骤,也为其他革兰氏阳性菌总DNA的提取提供参考。
3 结论通过比较水煮法、传统法、CTAB-SDS法、SDS-碱裂解法、酚氯法和SDS-酶裂解法对革兰氏阳性细菌(Clostridium thermocellum)基因组DNA的提取效率和产率发现,SDS-碱裂解法提取所得基因组DNA产率最高,为268.27 mg/L,但OD260/OD280值为1.64且OD260/OD230值为1.65,说明蛋白质和其他影响后续检测的杂质去除情况较差,为适应基因组DNA的提取需要,也为后续实验的进行打下良好的基础,因此对其进行改良。改良后的SDS-碱裂解法提取所得C. thermocellum和T. thermosaccharolyticum基因组DNA产率分别为418.23 mg/L和397.51 mg/L、OD260/OD280值分别为1.89和1.90,且OD260/OD230值分别为1.93和1.87,说明改良SDS-碱裂解法提取所得2株不同革兰氏阳性菌的产率稳定,且纯度较高,有利于后续实验的进行。此外,本法所用试剂价格便宜、性质稳定,适用于一般实验室的提取需求。
| [1] | Hu XH, Peng HM, Liu X, et al.Methods of DNA extraction from bacteria for PCR and real-time PCR[J]. Journal of Chongqing Medical University, 2008, 33(2):155-158(in Chinese)胡晓红,彭惠民,刘昕,等.PCR及Real-time PCR评价细菌 DNA提取方法[J].重庆医科大学学报, 2008, 33(2):155-158 |
| [2] | Hao M, Liu ZY, Yang TS, et al.Overview of bacterial genomic DNA extraction method[J]. Bulletin of Biology, 2014, 49(3):4-6 (in Chinese)郝敏,刘占英,杨天舒,等.细菌基因组DNA提取方法概述 [J].生物学通报, 2014, 49(3):4-6 |
| [3] | Zhang TT, Wang L, Gong P, et al.Optimization of genomic DNA extraction method of Staphylococcus aures[J]. Food Research and Development, 2014, 35(3):8-10(in Chinese)张涛涛,王兰,龚频,等.金黄色葡萄球菌基因组DNA提取方法的优化[J].食品研究与开发, 2014, 35(3):8-10 |
| [4] | Chen H, Rangasamy M, Tan SY, et al.Evaluation of five methods for total DNA extraction from western corn rootworm beetles[J]. PLoS One, 2010, 5(8):e11963 |
| [5] | Özkan M, Yllmaz EI, Lynd LR, et al.Cloning and expression of the Clostridium thermocellum L-lactate dehydrogenase gene in Escherichia coli and enzyme characterization[J]. Canadian Journal of Microbiology, 2004, 50(10):845-851 |
| [6] | Dong YL.Optimization of bacterial DNA extraction method[J]. China High-Tech Enterprises, 2012(1):41-43(in Chinese)董艳磊.细菌DNA提取方法的优化[J].中国高新技术企业, 2012(1):41-43 |
| [7] | Guo HY, Yu C, Guo WS, et al.Comparative study of microbial genome DNA extraction methods from the gastrointestinal tract of murine embryos[J]. Biotechnology, 2012, 21(4):37-40(in Chinese)郭海勇,于超,郭伟生,等.小鼠胚胎胃肠道细菌基因组 DNA提取方法比较[J].生物技术, 2012, 21(4):37-40 |
| [8] | Shen DX, Feng ZC, Du J.Comparison of bacterial DNA extraction method[J]. Central Plains Medical Journal, 2004, 31(10):20-22(in Chinese)沈德新,封志纯,杜江.细菌DNA提取方法比较[J].中原医刊, 2004, 31(10):20-22 |
| [9] | Li R, Chen DY, Chen P, et al.DNA extraction of Green-covering Guanyin Tuqu[J]. China Brewing, 2010(11):127-128(in Chinese)李睿,陈道玉,陈平,等.绿衣观音土曲细菌DNA的提取[J].中国酿造, 2010(11):127-128 |
| [10] | Yang WJ, Liu HZ, Chen JY, et al.Effect of SDS on emulsification of aqueous lysozyme[J]. Engineering Chemistry &Metallurgy, 1996, 17(3):254-258(in Chinese)杨为进,刘会洲,陈家镛,等.SDS对溶菌酶溶液乳化作用的影响研究[J].化工冶金, 1996, 17(3):254-258 |
 2015, Vol. 42
2015, Vol. 42