扩展功能
文章信息
- 刘福芮, 钟志军, 周紫峣, 彭广能, 杨平, 王亚萍, 廖莉
- LIU Fu-Rui, ZHONG Zhi-Jun, ZHOU Zi-Yao, PENG Guang-Neng, YANG Ping, WANG Ya-Ping, LIAO Li
- 细菌 sRNA 对环境胁迫的响应机制
- The mechanism of small RNAs response to environmental stresses in bacteria
- 微生物学通报, 2015, 42(10): 2012-2018
- Microbiology China, 2015, 42(10): 2012-2018
- 10.13344/j.microbiol.china.140973
-
文章历史
- 收稿日期: 2014-12-02
- 接受日期: 2015-03-17
- 优先数字出版日期(www.cnki.net): 2015-03-24
2. 四川省雅安市名山区农业局 四川 雅安 625100
2. Mingshan District Agricultural Bureau,Ya’an, Sichuan 625100, China
在自然环境中,细菌常常暴露在恶劣的环境下,给细菌的生存和繁殖带来巨大的挑战和压力。为了适应环境的各种变化,细菌自身进化出了一系列精密的机制来感受环境变化,并通过调整基因表达和蛋白质活性来适应这些变化。非编码RNA (non-coding RNA,ncRNA)就是其中一个非常重要的调节机制。研究发现,许多较为复杂的反应机制,例如基因沉默、基因转录、DNA标记、DNA去甲基化、RNA干扰等,都与非编码RNA的功能有关[1]。我们实验室发现大熊猫肠道中部分芽孢杆菌具有良好的益生作用[2, 3]。然而,肠道环境对于细菌来说却是十分严酷的,大多数肠道中的细菌转录活性均被抑制[4]。在进一步对大熊猫源芽孢杆菌在模拟肠道环境下的转录水平研究中发现,他们适应高纤维压力的能力与非编码RNA的调控有着紧密联系[5]。
一般来说,RNA可分为两个类别:可以翻译成蛋白质的RNA (如tRNA,mRNA)和各种虽然未翻译成蛋白质,但参与细胞调节,具有丰富生物学功能的RNA,即非编码RNA。它们包含高密度的终止密码子,但不包含大的开放阅读框(Open reading frames,ORF),因而这些调控RNA也常常被称为小RNA (small RNAs,sRNAs)。
1 细菌sRNA的概念及特征 1.1 细菌sRNA的概念细菌sRNA是一类长度大约在40−400个核苷酸,在细胞中被转录但是不编码蛋白质的一类非编码RNA分子。随着现代生物信息学手段和技术的发展,现已发现了超过150多种细菌sRNA[6, 7],其中大肠杆菌中已经通过实验证实的sRNA就有约八十种[8]。在其他细菌中也发现一些sRNA,如枯草芽孢杆菌(Bacillus subtilis)[9]、霍乱弧菌(Vibrio cholerae)[10]、布鲁氏菌(Brucella abortus)[11]、嗜肺军团菌(Legionella pneumophila)[12]、单核细胞增多性李斯特菌(Listeria monocytogenes)[13]、铜绿假单胞菌(Pseudomonas aeruginosa)[14]、鼠伤寒沙门氏菌(Salmonella typhimurium)[15]、假结核耶尔森氏菌(Yersinia pseudotuberculosis)[16]等。
1.2 细菌sRNA的类型基于基因组位置不同,sRNA可以被分为顺式编码sRNA (cis-encoded sRNA)和反式编码sRNA (trans-encoded sRNA)。顺式编码sRNA存在于蛋白质编码基因的内部,主要通过与其DNA互补的靶mRNA进行严格的匹配结合,对其进行表达抑制或基因沉默。反式编码sRNA存在于蛋白质编码基因的间隙,由于它们的靶mRNA位于远端,因此只有部分互补[17],且它们之间的碱基配对往往不严格,细菌中以这类反式编码sRNA为主。
基于发挥生物学功能形式不同,细菌sRNA可分为3种类型:(1) 功能sRNA,目前发现该类型的sRNA主要包括具有酶催化活性的sRNA[18]和转移信使tmRNA (transfer-messenger RNA)[19];(2) 蛋白质结合sRNA,如RNA CsrB和CsrC能够特异性地与CsrA蛋白结合,调控CsrA蛋白的活性;(3) 调控sRNA,这类细菌sRNA与靶mRNA配对结合,通过影响其翻译效率和稳定性来发挥作用。目前发现的细菌sRNA绝大多数都属于第3类型。
2 细菌sRNA的作用机制细菌sRNA以反式编码sRNA为主,它们的主要作用方式是通过不严格的碱基互补配对与靶mRNA结合,抑制或促进靶mRNA的翻译,加速或减缓靶mRNA的降解,并且这一类调控sRNA大部分都依赖于与RNA伴侣Hfq蛋白的结合而发挥作用。Hfq是最早在大肠杆菌中发现的一种大小为102个氨基酸的RNA结合蛋白,是RNA噬菌体Qβ (RNA phage Qβ)在大肠杆菌中复制的宿主因素[20]。Hfq优先与细菌sRNA单链上位于转录终止子上游富含AU的部位紧密结合[21],不仅促进了sRNA的稳定性,还对sRNA具有保护作用。同时Hfq还与sRNA的靶mRNA的5′端靠近核糖体结合位点(Ribosome bind site,RBS)和起始密码子的位置结合,增加sRNA和靶mRNA之间双重构造的比例,抑制了靶mRNA的翻译起始。随后参与靶mRNA降解的核酸酶E (RNase E)特异性地作用于富含AU的单链RNA,降解靶mRNA的同时也降解与之结合的sRNA[22, 23](图 1)。还有些细菌sRNA结合在靶mRNA的3′端,起到稳定靶mRNA的作用。

|
| 图 1 反式编码sRNA与靶mRNA碱基配对结合,抑制靶mRNA翻译,加速靶mRNA降解 Figure 1 A class of trans-encoded sRNAs imperfectly base-pair with target mRNAs in order to repress translation and speed up the degradation |
这一类调控sRNA中,还有些可以结合在靶mRNA的茎环结构上,使茎环结构打开,从而促进靶mRNA翻译[24](图 2)。这些对靶mRNA的上调作用为原核生物所特有,在真核生物中尚未发现。

|
| 图 2 反式编码sRNA结合在靶mRNA的茎环结构上,使茎环结构打开,促进靶mRNA翻译 Figure 2 A class of trans-encoded sRNAs bind to the stem structure on target mRNAs, relieve the occlusion of the RBS, and allow translation initiation |
细菌中另外还有一类蛋白质结合反式编码sRNA,他们不是通过与靶mRNA的碱基配对发挥作用,而是通过直接与靶蛋白结合来调节靶蛋白的生物活性[25](图 3)。

|
| 图 3 反式编码sRNA与靶蛋白结合调节靶蛋白活性 Figure 3 A class of trans-encoded sRNAs act by binding to the target protein to regulate their activities |
除此之外,例如MtlS、SymR、GadY等顺势编码sRNA,可以直接与靶mRNA进行严格的碱基配对,这种结合通常引导一个负调控,比如RNA降解、抑制翻译或转录终止,从而抑制(图 4)或者激活(图 5)蛋白质的表达。
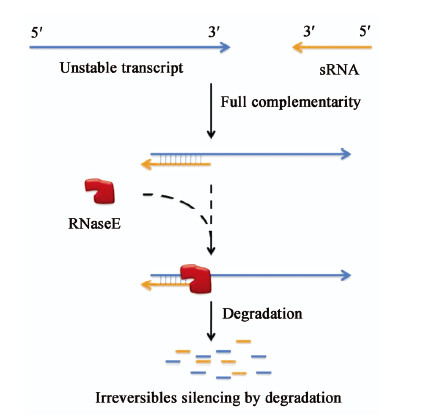
|
| 图 4 顺式编码sRNA与靶mRNA严格碱基配对后被核酸酶E降解 Figure 4 A class of cis-encoded sRNAs share perfect complementarity with the target, and both of them are degraded by an RNaseE |

|
| 图 5 顺式编码sRNA与靶mRNA形成RNA双链结构,靶mRNA不被核酸酶E降解 Figure 5 A class of cis-encoded sRNAs form duplex structure with mRNA, increase the stability of mRNA, and protect them from degradation |
在细菌的温度、营养、外膜蛋白、氧化应激、pH、铁等环境变化时,sRNA均起到了重要的调节作用。大部分sRNA通过连接双组分信号转导系统和调节蛋白,具有传递环境胁迫信号和调节应激响应的作用。
3.1 细菌sRNA对温度压力的响应通常细菌依靠信号转导系统的反馈回路对温度波动产生反应,但这种响应方式通常有一个迟滞期。相反,基于RNA的温度波动反馈机制可通过调控RNA的结构改变,使RNA温度感应计外露,使反应更为迅速[26]。
在大肠杆菌中有一种RNA温度感应器DsrA,它是一种反式编码sRNA。低温时,DsrA可以大量表达,诱导RpoS基因调控细菌RNA聚合酶的σ因子[27]。在大肠杆菌中,DsrA主要有两种形式,一种是全长转录形式(Full-length transcript,F form),一种是被截短的转录形式(Ttruncated transcript,T form),这两种形式的比例会随着温度的变化而存在差异。当温度较低时,DsrA以F形式为主[28]。DsrA的F形式的5′端区域可以借助Hfq蛋白与RpoS mRNA的前导序列碱基配对,导致5′端的非编码区结构改变,激活mRNA的翻译,促进σ因子的表达(图 2)。
3.2 细菌sRNA对铁离子压力的响应铁离子是细胞生长代谢、氧气运输、电子传递等生命活动不可缺少的重要物质。然而,细胞内过高的铁离子水平则会导致活性氧(Reactive oxygen species,ROS)形成而对细胞产生危害。
Fur蛋白(Ferric uptake regulator)是铁平衡调节的传感器及调节器,RyhB是环境中铁离子浓度下降时受Fur蛋白调控表达的一种sRNA。在正常铁环境下,Fur蛋白与Fe2+结合形成Fur-Fe2+复合体,抑制RyhB基因转录表达。当铁浓度下降时,Fur-Fe2+复合体失去铁而构象改变,不能阻遏RyhB基因转录,于是RyhB得以表达。RyhB可以促进6种铁结合蛋白mRNA被核酸酶E降解,包括编码琥珀酸脱氢酶的sdhCDAB、编码顺乌头酸酶的acnA、编码延胡索酸酶的fumA、编码铁蛋白的bfr和ftnA,以及编码超氧化物歧化酶(Superoxide dismutase,SOD)的sodB等[29],抑制其表达,降低细菌对铁的需求量。
3.3 细菌sRNA对pH压力的响应肠道细菌能否在胃中强酸性环境下生存,对于其是否能够在肠道定殖十分关键。在一些革兰氏阳性和阴性细菌中,谷氨酸基耐酸系统(GAD)在高H+浓度环境下保护细胞起到关键作用[30]。在大肠杆菌中,酸应力主要受GadY调控,它也是Hfq依赖性sRNA。GadY最初被报道可以与gadX的3′-UTR区结合,形成RNA双链结构,保护mRNA不被核酶E降解[31](图 5)。随后GadY被报道可在pH较低时,调节GadY操纵子,导致转录调节蛋白GadX的积累,并对mRNA的稳定和下游耐酸性基因的表达起到正调控作用[32]。
原核生物可利用多种方法来提高细胞内的pH值,包括由氨基酸脱氨酶和糖发酵等途径增加酸的生产[33]。其中,AlxmRNA的5′-UTR已经被报道可作为对pH值敏感的RNA元件,激活细菌在碱性条件下生产的Alx蛋白质(推定的转运蛋白)。在极端碱性条件下,RNA聚合酶与PRE区域短暂结合,暂停信号可促使上游的AlxmRNA的替代结构合成[34]。
最近也有研究发现,RyhB的同系物RyhB-1和RyhB-2对鼠伤寒沙门氏菌的耐酸性也起积极作用[15]。
3.4 细菌sRNA对外膜蛋白的响应革兰氏阴性细菌的外膜(Outer membrane,OM),由肽聚糖层和内层膜形成。研究已经证实,外膜是由Hfq依赖性反式编码调控sRNA,MicA所调控的[35](图 1)。MicA是一个17个碱基长度的sRNA,存在于ompAmRNA的5′-UTR中。OM可以防止许多有毒分子进入细胞,并确保细菌生存在恶劣的环境中。孔蛋白起到帮助穿过OM营养物质的吸收和废物排泄的作用。此外,外膜蛋白也可作为酶黏附细菌表面蛋白,赋予细菌毒力[36]。
3.5 细菌sRNA对氧化应激的响应OxyR和SoxR是主要的研究较为透彻的两种调节细菌对氧化应激适应性反应的信号转导蛋白,现也发现大肠杆菌中的另一种sRNA,OxyS,可在过氧化氢(H2O2)的诱导下进行转录。OxyS sRNA可以在Hfq蛋白的辅助下,抑制编码转录激活剂的fhlA基因和编码RNA聚合酶σS亚基的ropS基因的转录起始[37](图 1),并且σS被证实会阻碍细菌中氧化应激反应[38]。
最近的研究发现,鼠伤寒沙门氏菌中的RyhB-1和RyhB-2在氧化应激反应中也扮演着重要的角色[39]。
3.6 细菌sRNA对代谢/营养压力的响应微生物经常会处于营养匮乏的环境,6-磷酸葡萄糖(Glucose-6-phosphate,G6P)的堆积以及不可代谢的葡萄糖类似物的出现,均会导致细菌生长停滞,甚至死亡[40]。在大肠杆菌中,葡萄糖和α-甲基葡萄糖苷(α-methyl-glucoside,αMG)被磷酸转移酶系统(Phosphotransferase system,PTS)转运到细胞中的同时,也被氧化磷酸化。PtsG转运蛋白是PTS的主要葡萄糖转运蛋白之一,在磷酸葡萄糖缺乏时,一个与Hfq相关的非编码RNA (SgrS)下调PtsG基因转录后水平,防止新的PtsG蛋白和G6P或αMG合成[41, 42, 43, 44]。这个过程的实现依赖于SgrS与PtsG mRNA特定的碱基配对,抑制PtsG mRNA的翻译,随后被核酶E降解(图 1)。
CsrA基因是碳储存系统(Carbon storage regulatory,CSR)中最重要的基因,CsrA蛋白能促进糖酵解的同时抑制糖异生途径。研究表明,在碳源缺乏的情况下,CsrA基因会明显下调[45]。CsrA活性是间接通过一个双组分体系(BarA/UvrY)所介导的,其中BarA是传感器蛋白而UvrY是同源响应调节蛋白。UvrY蛋白可以激活两个sRNA,即CsrB和CsrC,这2个sRNA分子作为CsrA蛋白的拮抗物,与CsrA蛋白质结合,从而减少CsrA与目标mRNA结合的机会,以此来降低CsrA的活性[46] (图 3)。
RsaE是另一个主要调节碳源通路的sRNA,在金黄色葡萄球菌及芽孢杆菌中被发现,其结构为两个茎环,中间被一段长度为17 nt的单链区域分隔开。基于RNA-RNA相互作用的研究发现,由于RsaE的特殊结构,它可以防止同一个操纵子操纵的两个靶mRNA形成核糖体起始复合物[47]。RsaE也可以通过下调TCA循环和嘌呤生物合成的关键酶来发挥其调控作用,对金黄色葡萄球菌适应营养剥夺条件起到重要作用[48]。
4 小结与展望基于目前对sRNA的研究,学者们已经提出了大量关于sRNA如何被细菌利用以调控细菌适应各种环境压力的观点。比如小RNA温度感应器、顺/反式编码sRNA及其他的一些调控sRNA,通过在交替构象之间切换、与靶mRNA碱基配对调节mRNA转录活性和稳定性、选择性的降解异形核酸、与调控蛋白结合并控制蛋白活性等方式,来调控环境胁迫下的响应。我们也通过实验发现sRNA在细菌对高纤维环境压力的适应中起到了非常重要的调节作用[5],帮助我们解释大熊猫消化系统中不含纤维素酶却能消化高纤维食物的原理。
值得一提的是,sRNA在调节压力响应中较蛋白转录调控因子有着显著的优势。细菌sRNA较小,通常不需要翻译成蛋白质或多肽,因此其合成和周转所需要的能量和时间就更少。此外,许多sRNA在转录后水平起作用,确保了对环境压力信号的迅速反应。现阶段对大肠杆菌中的sRNA调控机制研究较为广泛和透彻,对其他细菌,如肠道益生菌枯草芽孢杆菌sRNA的广泛研究,将扩展sRNA多样性,为细菌调控提供了一个新的补充调控层面,即目标基因如何被调控。
总的来说,随着现代高通量技术的发展和生物信息学的进步,越来越多的参与细菌环境胁迫下调控的sRNA将浮出水面。未来的主要挑战在于如何更好地了解细菌中sRNA具体的生物合成和作用机制以及它与生物起源、致病机理的联系。以上相关发现将为sRNA作为提高有益菌抗逆性的靶点提供参考依据,同时也为全新的诊断标记及药物靶点提供重要的参考依据。
| [1] | Costa FF. Non-coding RNAs: new players in eukaryotic biology[J]. Gene, 2005, 357(2): 83-94 |
| [2] | Zhou XX, He TM, Peng GN, et al. Isolation, indengtification and resistance analysis of 7 bacillus strains from the intestinal tract of Giant Panda[J]. Veterinary Science in China, 2013, 43(11): 115-1121 (in Chinese) 周潇潇, 何廷美, 彭广能, 等. 大熊猫肠道芽孢杆菌的分离鉴定及其抗逆性研究[J]. 中国兽医科学, 2013, 43(11): 1115-1121 |
| [3] | Zhou Z, Zhou X, Zhong Z, et al. Investigation of antibacterial activity of Bacillus spp. isolated from the feces of Giant Panda and characterization of their antimicrobial gene distributions[J]. World Journal of Microbiology and Biotechnology, 2014, 30(12): 3129-3136 |
| [4] | Franzosa EA, Morgan XC, Segata N, et al. Relating the metatranscriptome and metagenome of the human gut[J]. Proceedings of the National Academy of Sciences, 2014, 111(22): E2329-E2338 |
| [5] | Zhou Z, Zhou X, Li J, et al. Transcriptional regulation and adaptation to a high-fiber environment in Bacillus subtilis HH2 isolated from feces of the Giant Panda[J]. PLoS One, 2015, 10(2): e0116935 |
| [6] | Abu-Qatouseh LF, Chinni SV, Seggewiß J, et al. Identification of differentially expressed small non-protein-coding RNAs in Staphylococcus aureus displaying both the normal and the small-colony variant phenotype[J]. Journal of Molecular Medicine, 2010, 88(6): 565-575 |
| [7] | Raabe CA, Hoe CH, Randau G, et al. The rocks and shallows of deep RNA sequencing: Examples in the Vibrio cholerae RNome[J]. RNA, 2011, 17(7): 1357-1366 |
| [8] | Waters LS, Storz G. Regulatory RNAs in bacteria[J]. Cell, 2009, 136(4): 615-628 |
| [9] | Condon C, Bechhofer DH. Regulated RNA stability in the Gram positives[J]. Current Opinion in Microbiology, 2011, 14(2): 148-154 |
| [10] | Vincent HA, Henderson CA, Ragan TJ, et al. Characterization of Vibrio cholerae Hfq provides novel insights into the role of the Hfq C-terminal region[J]. Journal of Molecular Biology, 2012, 420(1): 56-69 |
| [11] | Caswell CC, Gaines JM, Roop RM. The RNA chaperone Hfq independently coordinates expression of the VirB type IV secretion system and the LuxR-type regulator BabR in Brucellaabortus 2308[J]. Journal of Bacteriology, 2012, 194(1): 3-14 |
| [12] | Tiaden A, Spirig T, Weber SS, et al. The Legionella pneumophila response regulator LqsR promotes host cell interactions as an element of the virulence regulatory network controlled by RpoS and LetA[J]. Cellular Microbiology, 2007, 9(12): 2903-2920 |
| [13] | Mraheil MA, Billion A, Mohamed W, et al. The intracellular sRNA transcriptome of Listeria monocytogenes during growth in macrophages[J]. Nucleic Acids Research, 2011, 39(10): 4235-4248 |
| [14] | Morris ER, Hall G, Li C, et al. Structural rearrangement in an RsmA/CsrA ortholog of Pseudomonas aeruginosa creates a dimeric RNA-binding protein, RsmN[J]. Structure, 2013, 21(9): 1659-1671 |
| [15] | Kim JN, Kwon YM. Genetic and phenotypic characterization of the RyhB regulon in Salmonella Typhimurium[J]. Microbiological Research, 2013, 168(1): 41-49 |
| [16] | Lu P, Zhang Y, Hu Y, et al. A cis-encoded sRNA controls the expression of fabH2 in Yersinia[J]. FEBS Letters, 2014, 588(10): 1961-1966 |
| [17] | Richards GR, Vanderpool CK. Molecular call and response: the physiology of bacterial small RNAs[J]. Biochimicaet Biophysica Acta (BBA)-Gene Regulatory Mechanisms, 2011, 1809(10): 525-531 |
| [18] | Hao Y, Xu L, Shi H. Theoretical analysis of catalytic-sRNA-mediated gene silencing[J]. Journal of Molecular Biology, 2011, 406(1): 195-204 |
| [19] | Mikulík K, Palečková P, Felsberg J, et al. SsrA genes of streptomycetes and association of proteins to the tmRNA during development and cellular differentiation[J]. Proteomics, 2008, 8(7): 1429-1441 |
| [20] | Franze de Fernandez M, Eoyang L, August J. Factor fraction required for the synthesis of bacteriophage Qbeta-RNA[J]. Nature, 1968, 219(5154): 588-590 |
| [21] | Faner M, Feig A. Identifying and characterizing Hfq-RNA interactions[J]. Methods, 2013, 63(2):144-159 |
| [22] | Mackie GA. RNase E: at the interface of bacterial RNA processing and decay[J]. Nature Reviews Microbiology, 2012, 11(1): 45-57 |
| [23] | Saramago M, Bárria C, dos Santos RF, et al. The role of RNases in the regulation of small RNAs[J]. Current Opinion in Microbiology, 2014, 18(4): 105-115 |
| [24] | Repoila F, Majdalani N, Gottesman S. Small non-coding RNAs, co-ordinators of adaptation processes in Escherichia coli: the RpoSparadigm[J]. Molecular Microbiology, 2003, 48(4): 855-861 |
| [25] | Majdalani N, Vanderpool CK, Gottesman S. Bacterial small RNA regulators[J]. Critical Reviews in Biochemistry and Molecular Biology, 2005, 40(2): 93-113 |
| [26] | Hoe CH, Raabe CA, Rozhdestvensky TS, et al. Bacterial sRNAs: regulation in stress[J]. International Journal of Medical Microbiology, 2013, 303(5): 217-229 |
| [27] | Dudin O, Lacour S, Geiselmann J. Expression dynamics of RpoS/Crl-dependent genes in Escherichia coli[J]. Research in Microbiology, 2013, 164(8): 838-847 |
| [28] | Cayrol B, Hwang W, Busi F, et al. Tracking bacterial riboregulation by DsrA noncoding RNA[J]. Biophysical Journal, 2012, 102(3): 646a |
| [29] | Massé E, Gottesman S. A small RNA regulates the expression of genes involved in iron metabolism in Escherichia coli[J]. Proceedings of the National Academy of Sciences, 2002, 99(7): 4620-4625 |
| [30] | Bearson S, Bearson B, Foster JW. Acid stress responses in enterobacteria[J]. FEMS Microbiology Letters, 1997, 147(2): 173-180 |
| [31] | Opdyke JA, Kang JG, Storz G. GadY, a small-RNA regulator of acid response genes in Escherichia coli[J]. Journal of Bacteriology, 2004, 186(20): 6698-6705 |
| [32] | Opdyke JA, Fozo EM, Hemm MR, et al. RNase Ⅲ participates in GadY-dependent cleavage of the gadX-gadWmRNA[J]. Journal of Molecular Biology, 2011, 406(1): 29-43 |
| [33] | Padan E, Bibi E, Ito M, et al. Alkaline pH homeostasis in bacteria: new insights[J]. Biochimica et Biophysica Acta (BBA)-Biomembranes, 2005, 1717(2): 67-88 |
| [34] | Nechooshtan G, Elgrably-Weiss M, Sheaffer A, et al. A pH-responsive riboregulator[J]. Genes & Development, 2009, 23(22): 2650-2662 |
| [35] | Vogel J, Papenfort K. Small non-coding RNAs and the bacterial outer membrane[J]. Current Opinion in Microbiology, 2006, 9(6): 605-611 |
| [36] | Nikaido H. Molecular basis of bacterial outer membrane permeability revisited[J]. Microbiology and Molecular Biology Reviews, 2003, 67(4): 593-656 |
| [37] | Updegrove TB, Wartell RM. The influence of Escherichia coli Hfq mutations on RNA binding and sRNA· mRNA duplex formation in rpoS ribo regulation[J]. Biochimica et Biophysica Acta (BBA)-Gene Regulatory Mechanisms, 2011, 1809(10): 532-540 |
| [38] | Barth E, Gora KV, Gebendorfer KM, et al. Interplay of cellular cAMP levels, σS activity and oxidative stress resistance in Escherichia coli[J]. Microbiology, 2009, 155(5): 1680-1689 |
| [39] | Calderón IL, Morales EH, Collao B, et al. Role of Salmonella Typhimurium small RNAs RyhB-1 and RyhB-2 in the oxidative stress response[J]. Research in Microbiology, 2014, 165(1): 30-40 |
| [40] | Polakof S, Mommsen TP, Soengas JL. Glucosensing and glucose homeostasis: from fish to mammals[J]. Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology, 2011, 160(4): 123-149 |
| [41] | Kawamoto H, Koide Y, Morita T, et al. Base-pairing requirement for RNA silencing by a bacterial small RNA and acceleration of duplex formation by Hfq[J]. Molecular Microbiology, 2006, 61(4): 1013-1022 |
| [42] | Vanderpool CK, Gottesman S. Involvement of a novel transcriptional activator and small RNA in post-transcriptional regulation of the glucose phosphoenolpyruvate phosphotransferase system[J]. Molecular Microbiology, 2004, 54(4): 1076-1089 |
| [43] | Papenfort K, Sun Y, Miyakoshi M, et al. Regulation of glucose homeostasis by small RNA mediated activation of sugar phosphatase mRNA[J]. Cell, 2013, 153(2): 426 |
| [44] | Vogel J, Luisi BF. Hfq and its constellation of RNA[J]. Nature Reviews Microbiology, 2011, 9(8): 578-589 |
| [45] | Wang X, Dubey AK, Suzuki K, et al. CsrA post-transcriptionally represses pgaABCD, responsible for synthesis of a biofilm polysaccharide adhesin of Escherichia coli[J]. Molecular Microbiology, 2005, 56(6): 1648-1663 |
| [46] | Pernestig AK, Georgellis D, Romeo T, et al. The Escherichia coli BarA-UvrY two-component system is needed for efficient switching between glycolytic and gluconeogenic carbon sources[J]. Journal of Bacteriology, 2003, 185(3): 843-853 |
| [47] | Lioliou E, Sharma CM, Altuvia Y, et al. In vivo mapping of RNA-RNA interactions in Staphylococcus aureus using the endoribonuclease Ⅲ[J]. Methods, 2013, 63(2): 135-143 |
| [48] | Bohn C, Rigoulay C, Chabelskaya S, et al. Experimental discovery of small RNAs in Staphylococcus aureus reveals a riboregulator of central metabolism[J]. Nucleic Acids Research, 2010, 38(19): 6620-6636 |
 2015, Vol. 42
2015, Vol. 42




