扩展功能
文章信息
- 沈辉, 万夕和, 何培民, 黎慧, 乔毅, 蒋葛
- SHEN Hui, WAN Xi-He, HE Pei-Min, LI Hui, QIAO Yi, JIANG Ge
- 脊尾白虾肠道微生物菌群结构
- Bacterial community structure in the intestine ofExopalaemon carinicauda Holehuis
- 微生物学通报, 2015, 42(10): 1922-1928
- Microbiology China, 2015, 42(10): 1922-1928
- 10.13344/j.microbiol.china.140959
-
文章历史
- 收稿日期: 2014-11-28
- 接受日期: 2015-03-04
- 优先数字出版日期(www.cnki.net): 2015-06-26
2. 上海海洋大学 水产与生命学院 上海 201306
2. Collegeof Fisheries and Life Science, Shanghai Ocean University, Shanghai 201306, China
脊尾白虾又称小白虾,是中国特有的经济虾类之一[1]。由于味道鲜美、养殖效益较高,近年来脊尾白虾养殖规模迅速扩大。随着养殖密度及规模的增大,脊尾白虾的病害问题也日益增多[2, 3, 4]。微生态制剂进行虾类疾病的防控研究受到广泛关注。微生态制剂的施用可以改善养殖生态环境,抑制病原微生物,提高水生经济动物的免疫力,从而减少养殖过程中疾病的发生[5, 6]。微生态制剂的研究与开发是基于动物肠道土著细菌群落结构的背景资料。
迄今为止,关于脊尾白虾肠道微生物菌群结构的研究还未见报道。传统的微生物培养技术无法获得细菌群落结构的全部信息[7],为更加全面地了解脊尾白虾肠道的菌群结构,本文通过建立16S rRNA基因克隆文库,利用PCR-RFLP技术对脊尾白虾的肠道微生物菌群组成进行研究,以期为脊尾白虾的病害防治及微生态制剂的研究开发提供基础资料。
1 材料与方法 1.1 样品采集20尾脊尾白虾样品(3−4 g/尾)于2013年9月取自江苏省启东黄海滩涂开发有限公司,冷藏带回实验室。70%酒精擦拭样品体表,用灭菌后的镊子和剪刀取出脊尾白虾的肠道,接种针取出肠道内容物,并用灭菌生理盐水简单冲洗肠道表面,随后将肠道置于无菌管中−80 °C保存备用。
1.2 样品总DNA提取取5条解冻的肠道样品混合,4 °C、10 000×g离心10 min,弃上清[8]。称取100 mg样品置于灭菌后的EP管研杵搅匀,应用天根海洋动物组织DNA提取试剂盒(DP324)进行基因组总DNA的提取,获得的DNA通过琼脂糖凝胶电泳(1.0%)检测后−20 °C保存。
1.3 细菌16s rRNA基因PCR扩增以提取的基因组DNA作为模板进行16s rRNA基因PCR扩增,引物为27F (5′-AGAGTTTGATCCT GGCTCAG-3′)和1492R (5′-GGTTACCTTGTTAC GACTT-3′),由大连宝生物公司合成。PCR反应体系为(25 μl):10×PCR缓冲液(Mg2+ plus) 2.5 μl,dNTPs mixture (各2.5 mmol/L) 2 μl,引物(10 mmol/L)各1 μl,模板DNA 1 μl,rTaq酶(5 U/μL) 0.20 μl,灭菌双蒸水补足至25 μl。PCR反应条件:94 °C 5 min;94 °C 1 min,55 °C 45 s,72 °C 1 min,35个循环;72 °C 10 min。PCR扩增产物经1%的琼脂糖凝胶电泳后,切下目的条带,用胶回收试剂盒进行回收纯化。
1.4 PCR产物的克隆文库构建及RFLP将回收的PCR产物按照大连宝生物公司PMD19-T载体试剂盒(D102A)说明进行克隆。平板上所有阳性克隆提取质粒后用引物RV-M/M13-47进行PCR扩增。扩增后的产物稀释1 000倍后作为模板进行巢式PCR (通用引物27F和1492R),扩增产物约为1 500 bp。将巢式PCR的产物用MspⅠ和Hae Ⅲ限制性内切酶37 °C消化3 h,消化产物以3%琼脂糖凝胶电泳检测,分析克隆子RFLP图谱。
1.5 DNA序列测序及分析挑选筛选出的差异克隆子送至上海生工生物工程技术服务有限公司进行测序,所获序列通过NCBI进行比对找出相关同源序列,取GenBank中与比对序列具有90%以上同源性的前3条序列,用软件MEGA 5.1的邻近相接法(Neighbor-Joining method)构建系统树,自举值(Bootstrap)设定为1 000。以Coverage (C)评估所构建文库的库容,所获得的信息可以体现脊尾白虾肠道微生物的多样性。计算公式如下:
C=[1−(n1/N)]×100%
式中N为所分析的文库库容,即分析的克隆数;n1为克隆文库中不重复序列的克隆数。
2 结果与分析 2.1 酶切结果统计及分析对脊尾白虾肠道总DNA的16s rRNA基因PCR扩增产物克隆后获得128个阳性克隆,所有阳性克隆菌落进行质粒提取,用M13-47和RV-M引物PCR扩增,电泳后扩增产物约为1.7 kb,共获得阳性克隆为114个,阳性率为81.3%。利用两种限制性内切酶Msp I和Hae Ⅲ酶切,3%琼脂糖电泳后进行图谱分析(图 1)。结果表明,脊尾白虾肠道中共存在11种细菌RFLP谱型。以Coverage (C)计算,所构建的16S rRNA克隆文库覆盖率达到90.4%,表明该文库可较完整地反映脊尾白虾肠道环境的细菌多样性状况。
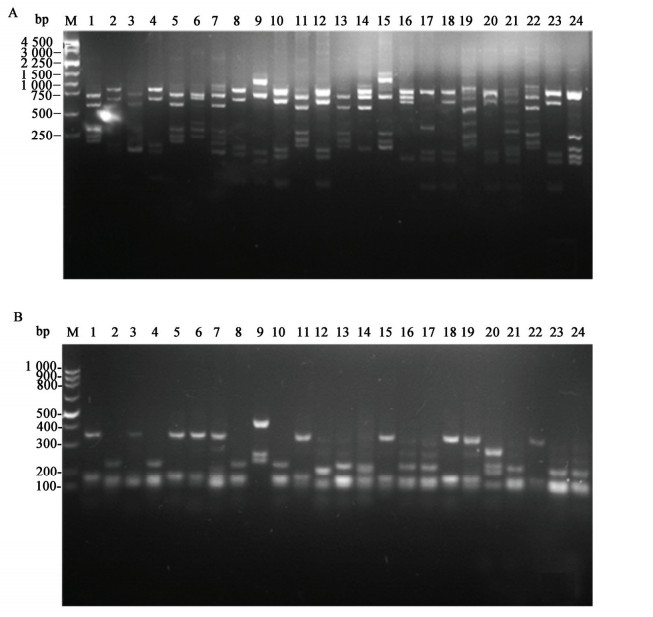
|
| 图 1 Agrose gel electrophoresis restriction fragment patterns of PCR products of positive clones Figure 1 脊尾白虾肠道文库阳性克隆PCR产物RFLP电泳图谱 Note:A:MspⅠ;B:Hae Ⅲ;M:100 bp DNA ladder (Takara). |
通过酶切分析,发现共有11种代表克隆子类型,测序后将代表克隆子的序列上传至GenBank(表 1),获得登录号为KM403082−KM403092。同源比对发现,脊尾白虾肠道细菌优势菌为变形杆菌,其中α-变形杆菌纲4条,γ-变形杆菌纲占5条,其他2条为厚壁菌。11个序列中有8个与已知细菌具有同源性,分别是假单胞菌属(Pseudomonas)占17.5%、肠杆菌属(Enterobater)占21.1%、冰冻小杆菌属(Frigoribacterium)占8.8%、褐杆菌属(Phaeobacter)占5.3%、弧菌属(Vibrio)占10.5%、赤杆菌属(Erythrobacter)占9.6%、气单胞菌属(Aeromonas)占4.4%、葡萄球菌属(Staphylococcus)占2.6%,未培养细菌占19.3%。
| 差异代表序列 Representative sequence |
克隆子 Clone |
最相似序列 Closest relative sequence |
相似性 Identity (%) |
克隆数 clones |
类群 Phylum |
| 1 | SH-01 | Pseudomonas aeruginosa (KJ806426.1) | 99 | 20 | γ |
| 2 | SH-21 | Enterobacter sp. (JX941543.1) | 99 | 24 | γ |
| 3 | SH-45 | Uncultured Enterobacter sp. (JF703615.1) | 99 | 4 | γ |
| 4 | SH-49 | Uncultured bacterium clone (JF747814.1) | 90 | 7 | α |
| 5 | SH-56 | Frigoribacterium sp. (EU584512.1) | 99 | 10 | F |
| 6 | SH-66 | Phaeobacter daeponensis (NR044026.1) | 99 | 6 | α |
| 7 | SH-72 | Vibrio harveyi (FJ154796.1) | 100 | 12 | γ |
| 8 | SH-84 | Uncultured alpha proteobacterium (AB491819.1) | 96 | 11 | α |
| 9 | SH-95 | Erythrobacter sp. (DQ480144.1) | 99 | 12 | α |
| 10 | SH-107 | Aeromonas hydrophila (JX978427.1) | 99 | 5 | γ |
| 11 | SH-112 | Staphylococcus hominis (HG941662.1) | 99 | 3 | F |
系统进化分析结果表明,克隆子归属于两个细菌门类:变形细菌门(Proteobacteria,即γ-变形细菌纲和α-变形细菌纲)和厚壁细菌门(图 2)。γ-变形细菌纲为优势类群,包含5个差异序列,系统发育分析归为4个属,分别为假单胞菌属、肠杆菌属、弧菌属和气单胞菌属。α-变形杆菌纲中,SH-45和SH-84都属于未培养的细菌序列,两者的克隆子数量占α-变形杆菌纲的60%,表明α-变形杆菌纲中大多为未培养菌株。厚壁细菌门包含2个差异序列,系统发育归为2个属,属于冰冻小杆菌属及葡萄球菌属。
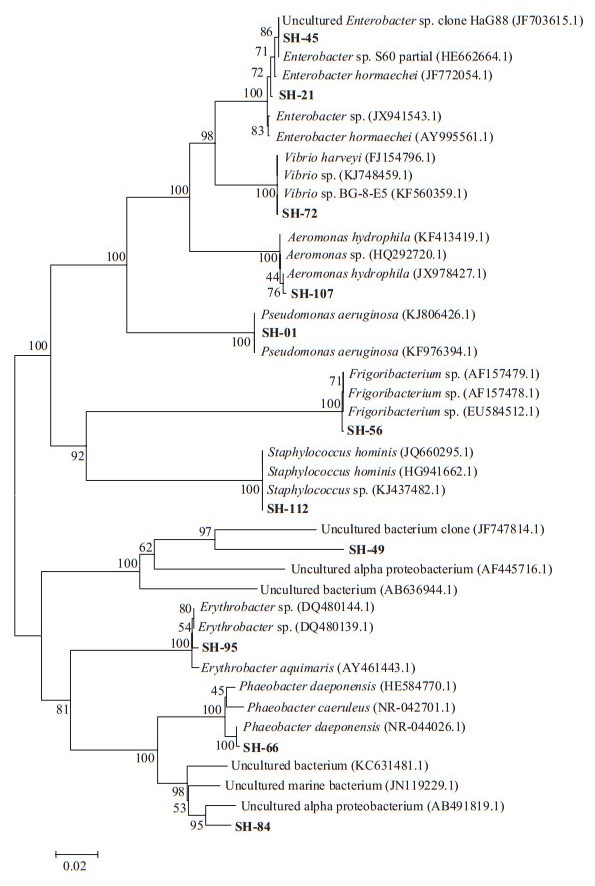
|
|
图 2
脊尾白虾肠道细菌系统发育树
Figure 2
Unrooted tree based on 16S rRNA gene sequences showing the phylogenetic position of bacteria fromExopalaemon carinicauda Holehuis
注:分离的菌株号用加粗表示,采用ClustalW两两比对,用MEGA 5.0软件按照邻接法聚类(选择Bootstrap检验值≥50%,1 000次重复)进行系统发育树的构建. Note:The number of bacteria strains isolated in this study are indicated in bold.Multiple Sequence alignment was performed using ClusterW.Phylogenetic tree was constructed with MEGA version 5.0 using a Neighbor-Joining algorithm,plus the Jukes-Cantor distance estimation method with bootstrap analyses for 1 000 replicates was performed. |
目前,对南美白对虾肠道微生物群落结构组成的报道较多。研究者们利用不同的分子生物学方法对南美白对虾的肠道微生物组成进行研究分析,得出的研究结论也较为一致。研究表明,南美白对虾肠道微生物群落的主要优势菌为变形细菌门和厚壁细菌门,包括假单胞菌属、肠道细菌属和弧菌属等[8, 9, 10]。据研究报道,在养殖池塘环境中最主要的细菌类群一般为α-变形杆菌和γ-变形杆菌(Gamma-proteobacteria),其中水生动物肠道优势微生物群落为γ-变形杆菌[11-12]。由此表明,水生动物的肠道微生物群落结构常随着水体环境中的微生物群落结构而变化,虾类肠道的微生物菌群结构很大程度上受到养殖环境微生物的影响[13, 14]。罗鹏等[9]在对凡纳滨对虾咸淡水养殖系统内的细菌群落组成研究中,分析认为虾肠道内的细菌群落遵循从水体到虾肠道内的演替规律。同样,王春忠等[15]研究了长毛虾养殖环境中的水体、底泥的微生物群落结构与虾肠道微生物群落结构的相关性后,发现肠道内的优势菌(厚壁细菌、梭杆菌和γ-变形杆菌)主要从养殖池塘的水体中演替过来。在本研究中,脊尾白虾肠道细菌的优势细菌也属于γ-变形杆菌纲的假单胞菌属和肠杆菌属,表明脊尾白虾的养殖环境对其肠道微生物群落组成影响较大,也验证了微生物从水体到虾肠道的演替规律。
脊尾白虾在自然条件下是多种病原的宿主[2, 3, 4],在不同的环境理化条件下,多种病原因子如弧菌与病毒的共同作用毒力加倍下会引起对虾的大批量死亡[16, 17]。在本研究中,脊尾白虾的肠道内具有一定数量的条件性致病细菌,如哈维氏弧菌和嗜水气单胞菌,表明肠道内存在细菌毒力叠加的可能性,具有一定的发病风险。然而,在脊尾白虾的肠道中同时发现了一定比例的褐杆菌属细菌,该细菌分布在海水和海洋环境中,是一类革兰氏阴性的卵杆形菌,能产生具有细菌抗性的化合物[18]和扩散性的褐色色素[19]。董秀娟等[20]对一株海洋细菌Phaeobacter DL2的抑菌作用进行了研究,发现该菌株具有广泛的抑菌谱,且对致病性弧菌表现出较强的抑菌作用。初步推论,该细菌的寄生(10.5%)可使脊尾白虾肠道内具有较高的抑菌活性。因此,在随后的研究中,可以采用可培养方法对脊尾白虾肠道内的海洋细菌进行分离筛选,进一步对该菌株的生理特征和抑菌活性研究,可为脊尾白虾肠道细菌结构的调整及微生态制剂的研究提供基础资料。另外,本研究在脊尾白虾肠道中还发现了一定数量的绿脓杆菌和葡萄球菌,这两种细菌广泛存在于水、土壤、空气以及动物肠道中,对人、哺乳动物及禽类具有较强的毒力,是典型的条件致病性细菌[21]。由此提示,脊尾白虾的养殖环境可能受到了一定程度的人为因素影响,人们应注意生食脊尾白虾的安全性。
众多研究表明,在海洋环境中,目前已培养的细菌只占不到总细菌数的1%,在海洋环境中还存在许多未被培养过的新细菌物种。利用传统技术对微生物菌群培养时,不可避免地会造成菌株的富集或衰减,研究结果不能真实反映原微生物菌群的结构[22]。国内外学者利用PCR-16S rRNA为基础的方法对不同水生动物的肠道微生物多样性进行了研究,从分子角度更全面地了解了肠道微生物菌群的结构组成及影响因素[8, 9, 10, 11, 12]。本文通过RFLP技术对脊尾白虾的肠道微生物菌群结构进行了研究,结果发现了环境中常见的假单胞菌属、弧菌属和气单胞菌属细菌,以及动物肠道中的肠杆菌属和葡萄球菌属细菌,还发现了环境中较难分离培养的冰冻小杆菌属、褐杆菌属和赤杆菌属细菌,另外未能培养细菌占19.3%,表明分子生物学方法更能全面地反映复杂环境中微生物菌群结构的多样性,可以用来进行复杂环境中微生物菌群结构的研究。
| [1] | Li MY. An approach to the reproduction and growth of shrimp Exopelaemon carinicanda cultured in earthen ponds with reference to its maximum sustaining yield on catch rotation[J]. Journal of Fisheries of China, 1994, 18(2): 85-92 (in Chinese)李明云.池养脊尾白虾的繁殖、生长及其最大持续轮捕量的初步探讨[J].水产学报, 1994, 18(2): 85-92 |
| [2] | Shen H, Wan XH, Wang LB, et al. Study on experimental infection of Exopalaemon carinicauda Holehuis with white spot syndrome virus[J]. Marine Sciences, 2013, 37(5): 55-60 (in Chinese)沈辉, 万夕和, 王李宝, 等.白斑综合征病毒对脊尾白虾的致病性研究[J].海洋科学, 2013, 37(5): 55-60 |
| [3] | Zhang WW, Wang GS, Shi H, et al. Preliminary study on one pathogenic bacterium―Vibrio harveyi associated with red body disease in cultured Exopalaemon carinicauda[J]. Journal of Zhejiang Ocean University (Natural Science Edition), 2014, 33(1): 65-71 (in Chinese)张文文, 王庚申, 施慧, 等.一种引起脊尾白虾红体病病原菌的初步研究[J].浙江海洋学院学报:自然科学版, 2014, 33(1): 65-71 |
| [4] | Xu WJ, Xie JJ, Shi H, et al. Infection of Hematodinium sp. in farmed ridgetail white prawn Exopalaemon carinicauda[J]. Oceanologia Et Limnologia Sinica, 2010, 41(3): 396-402 (in Chinese)许文军, 谢建军, 施慧, 等.池塘养殖脊尾白虾(Exopalaemon carinicauda)感染血卵涡鞭虫的研究[J].海洋与湖沼, 2010, 41(3): 396-402 |
| [5] | Devaraja TN, Yusoff FM, Shariff M. Changes in bacterial populations and shrimp production in ponds treated with commercial microbial products[J]. Aquaculture, 2002, 206(3): 245-256 |
| [6] | Verscuere L, Rombaut G, Sorgeloos P, et al. Probiotic bacteria as biological control agents in aquaculture[J]. Microbiology and Molecular Biology Reviews, 2000, 64(4): 655-661 |
| [7] | Pace NR. A molecular view of microbial diversity and the biosphere[J]. Science, 1997, 276: 734-740 |
| [8] | Li YH, Chai PC, Hu XG, et al. Analysis of intestinal microecology of Litopenaeus vannamei in industrial aquaculture by RFLP and DGGE techniques[J]. Progress in Fishery Sciences, 2014, 35(2): 83-89 (in Chinese)李玉宏, 柴鹏程, 胡修贵, 等.应用 RFLP和 DGGE技术分析工厂化养殖凡纳滨对虾肠道微生物群落特征[J].渔业科学进展, 2014, 35(2): 83-89 |
| [9] | Luo P, Hu CQ, Xie ZY, et al. PCR-DGGE analysis of bacterial community composition in brackish water Litopenaeus vannamei culture system[J]. Journal of Tropical Oceanography, 2006, 25(2): 49-53 (in Chinese)罗鹏, 胡超群, 谢珍玉, 等.凡纳滨对虾咸淡水养殖系统内细菌群落组成的 PCR-DGGE分析[J].热带海洋学报, 2006, 25(2): 49-53 |
| [10] | Li K, Zheng TL, Tian Y, et al. Bacterial community structure in intestine of the white shrimp, Litopenaeus vannamei[J]. Acta Microbiologica Sinica, 2007, 47(4): 649-653 (in Chinese)李可, 郑天凌, 田蕴, 等.南美白对虾肠道微生物群落的分子分析[J].微生物学报, 2007, 47(4): 649-653 |
| [11] | Oxley AP, Shipton W, Owens L, et al. Bacterial flora from the gut of the wild and cultured banana prawn, Penaeus merguiensis[J]. Journal of Applied Microbiology, 2002, 93(2): 214-223 |
| [12] | Liu H, Wang L, Liu M, et al. The intestinal microbial diversity in Chinese shrimp (Fenneropenaeus chinensis) as determined by PCR-DGGE and clone library analyses[J]. Aquaculture, 2011, 317(4): 32-36 |
| [13] | Wang F, Yang JF, Chen JG, et al. Comparison of the bacterial community structure in the crab seawater cultured and the outside environment by PCR-DGGE fingerprint technique: Portunus trituberculatus and Scylla serrata[J]. Journal of Marine Sciences, 2010, 28(4): 59-64 |
| [14] | Han S, Liu Y, Zhou Z, et al. Analysis of bacterial diversity in the intestine of grass carp (Ctenopharyngodon idellus) based on 16S rRNA gene sequences[J]. Aquaculture Research, 2010, 42(1): 47-56 |
| [15] | Wang CZ, Lin GR, Yan T, et al. Microbial community in the shrimp (Penaeus penicillatus) intestine and its culture environment[J]. Journal of Fisheries of China, 2014, 38(5): 706-712 (in Chinese)王春忠, 林国荣, 严涛, 等.长毛对虾海水养殖环境以及虾肠道微生物群落结构研究[J].水产学报, 2014, 38(5): 706-712 |
| [16] | Alapide-Tendencia EV, Dureza LA. Isolation of Vibrio spp. from penaeus monodon (fabricius) with red disease syndrome[J]. Aquaculture, 1997, 154(2): 107-114 |
| [17] | Lee KK, Yang TI, Liu PC, et al. Dual challenges of infectious pancreatic necrosis virus and Vibrio carchariae in the grouper, Epinephelus sp.[J]. Virus Research, 1999, 63(1/2): 131-134 |
| [18] | Martens T, Heidorn T, Pukall R, et al. Reclassification of Roseobacter gallaeciensis RuizPonte et al. 1998 as Phaeobacter gallaeciensis gen. nov., comb. nov., description of Phaeobacter inhibens sp. nov., reclassification of Ruegeria algicola (Lafay et al. 1995) Uchino et al. 1999 as Marinovum algicola gen. nov., comb. nov., and emended descriptions of the genera Roseobacter, Ruegeria and Leisingera[J]. International Journal of Systematic and Evolutionary Microbiology, 2006, 56(Pt6): 1293-1304 |
| [19] | Brinkhoff T, Bach G, Heidorn T, et al. Antibiotic product ion by a Roseobacter clade-affiliat ed species from the German Wadden Sea and its antagonistic effects on indigenous isolates[J]. Applied and Environmental Microbiology, 2004, 70: 2560-2565 |
| [20] | Dong XJ, Li J, Zhang XH, et al. Identification and inhibitory activity to pathogenic Vibrio sp. of a marine bacterium Phaeobacter DL2[J]. Journal of Fishery Sciences of China, 2007, 14(6): 996-1003 (in Chinese)董秀娟, 李筠, 张晓华, 等.海洋细菌 Phaeobacter DL2的鉴定及其对致病弧菌抑制作用[J].中国水产科学, 2007, 14(6): 996-1003 |
| [21] | Lu CP. Veterinary Microbiology[M]. 3rd Edition. Beijing: China Agriculture Press, 2001: 4 (in Chinese)陆承平.兽医微生物学[M].第3版.北京:中国农业出版社, 2001: 4 |
| [22] | Burgmann H, Pesaro M, Widmer F. A strategy for optimizing quality and quantity of DNA extracted from soil[J]. Journal of Microbiological Methods, 2001, 45(1): 7-20 |
 2015, Vol. 42
2015, Vol. 42




