扩展功能
文章信息
- 程甜, 郝志强, 魏强, 李广林
- CHENG Tian, HAO Zhi-Qiang, WEI Qiang, LI Guang-Lin
- 细菌萜类合成酶的生物信息学分析
- Analysis of bacterial terpenoid synthase by bioinformatics
- 微生物学通报, 2015, 42(10): 1877-1887
- Microbiology China, 2015, 42(10): 1877-1887
- 10.13344/j.microbiol.china.140983
-
文章历史
- 收稿日期: 2014-12-06
- 接受日期: 2015-03-20
- 优先数字出版日期(www.cnki.net): 2015-04-10
萜类化合物(Terpenoid)是自然界中结构最为多样、种类最为丰富的天然产物[1]。萜类合成酶(Terpenoid synthase,TPS)是萜类化合物合成的关键酶。根据异戊二烯(isoprene,C5)单元数的不同,萜类化合物可分为单萜(monoterpene,C10)、倍半萜(sesquiterpene,C15)、二萜(diterpene,C20)、三萜(triterpene,C30)和多萜等。在生物体内,萜类化合物可通过两条不同的途径合成,即甲羟戊酸(Mevalonate,MVA)途径与甲基赤藓糖磷酸(2-C-methyl-D-erythritol-4-phosphate,MEP)途径。除了植物可同时利用MVA和MEP途径外,大部分生物体只能利用两条途径中的一种[2]。细菌主要利用MEP途径进行萜类产物的合成[3],即首先合成异戊烯基焦磷酸(Isopentenylallyl diphosphate,IPP)前体和二甲基丙烯基焦磷酸(Dimethylallyl diphosphate,DMAPP);然后IPP和DMAPP在聚异戊二烯焦磷酸合成酶(Polyisoprenyl diphosphate synthase)的作用下缩合成长度不同的非环化前体,即香叶基焦磷酸(geranyl diphosphate,GPP)、法尼基焦磷酸(farnesyl diphosphate,FPP)、香叶基香叶基焦磷酸(geranylgeranyl diphosphate,GGPP);最后这些前体在TPS的催化下环化重排形成萜类化合物。
萜类化合物具有重要的生理、生态和药用价值,它们作为激素、味道与气味成分以及抗生素被广泛应用。长期以来人们在各种物种中不断探究TPS的功能。在植物中,TPS已经在拟南芥(Arabidopsis thaliana)[4]、水稻(Oryza sativa)[5]、杨树(Populus trichocarpa)[5]、葡萄(Vitis vinifera)[5]和番茄(Solanum lycopersicum)[6]等物种中被报道。在真菌中,TPS已经在娄地青霉(Penicillium roqueforti)[7]、土曲霉(Aspergillus terreus)[8]和葡萄孢菌(Botrytis cinerea)[9]等物种中被发现。以前人们认为TPS只存在于植物和真菌中[10],但随着人们对细菌气味与挥发性物质代谢的探索[11,12,13],细菌TPS的研究逐渐引起人们的重视。Dairi等[14]于2001年首次在Kitasatospora griseola MF730-N6中发现了二萜合成酶。目前发现的细菌TPS主要集中在革兰氏阳性菌如链霉菌属的细菌以及其他一些放线菌中[15],如从链霉菌属Streptomyces lasaliensis NRRL 3382[16]与Streptomyces coelicolor A3(2)[17]分别发现能够合成2-methylisoborneol的基因tpc和sco7700;从Streptomyces coelicolor A3(2)[18]发现能够合成epi-isozizaene的基因sco5222;从Streptomyces melanosporofaciens MI614-43F2[19]中发现了1个二萜合成酶基因簇,由cotB1、cotB2、cotB3、cotB4组成。
目前对于TPS的研究主要集中在植物和真菌中,而对细菌TPS的研究尚少。随着越来越多的细菌基因组被测序,单靠传统的实验方法去鉴定细菌TPS的功能,已经远远落后于数据增长的速度,迫切需要开发和利用新的手段对细菌TPS进行系统研究。建立在已经被大量测序的细菌基因组基础上,本文利用生物信息学方法,对细菌TPS在全基因组范围内进行了识别、分类和功能分析,这为系统理解细菌TPS的物种分布、分类、功能和进化奠定了很好的基础。
1 材料与方法 1.1 数据来源Pfam数据库中[20]标号为PF03936的结构域是TPS蛋白的特征结构域。首先从Pfam数据库中(http://pfam.xfam.org/)下载此结构域包含的信息,然后从NCBI (National Central for Biotechnology Information,http://www.ncbi.nlm.nih.gov/genome/?term=bacteria)下载4 705个完成基因组测序的细菌的蛋白质序列(共44 322 195条)构建本地细菌蛋白质组数据库。
1.2 细菌TPS的识别利用基于隐马尔科夫模型的HMMER2.3.2[21]程序,以PF03936为检索模型,搜索本地细菌蛋白质组数据库中含有TPS结构域的候选序列,E-value设为<10−5[22, 23]。
1.3 多序列比对与分子系统进化树的构建利用MAFFT 7.130b[24]中的L-INS-i方法对搜集到的细菌TPS序列进行多序列比对分析。比对结果用MEGA 6.0[25]软件构建细菌TPS蛋白的分子系统进化树。建树方法为最大似然法(Maximum likelihood)。具体设置参数Test of Phylogeny:Bootstrap method;Replications:500;Model:Jones-Taylor-Thornton (JTT) model;Rates among Sites:4;Gamma Distributed (G);Gaps:Complete deletion。其余参数为默认。
1.4 Motifs的分析利用MEME[26] (http://meme.nbcr.net/meme/)对TPS蛋白的保守基序(Motifs)进行了图示,具体参数设置如下:The occurrences of a single motif are distributed among the sequences:Zero or one per sequence;Minimum width (≥2):6;Maximum width (≤300):50;Maximum number of motifs to find:20。其余参数为默认。
1.5 点突变影响的分析为了研究各个点突变对整条蛋白序列功能的影响,选择代表性的细菌TPS序列,使用PredictProtein (http://ppopen.rostl-ab.org/)对其进行点突变分析,此预测基于SNAP2,在分析点突变对功能的影响时准确性能达到82%[27]。
2 结果与分析 2.1 细菌萜类合成酶的全基因组范围识别和分布为了系统探索细菌TPS,以TPS的隐马尔科夫模型HMM (Hidden Markov models) PF03936为查找模型,利用HMMER软件搜索本地自建的细菌蛋白质数据库,共得到具有统计学显著性的1 423条匹配序列(E-value<10−5),即为预测的细菌TPS。
分析表明,这1 423条TPS来自553个细菌基因组中,它们主要分布于细菌8个门中(图 1A),在放线菌门(Actinobacteria)的405个基因组中发现了1 206条TPS,占总数的84.751%,在变形菌门(Proteobacteria)的95个基因组中发现了141条,在蓝藻门(Cyanobacteria)的21个基因组中发现了29条,在拟杆菌门(Bacteroidetes)的16个基因组中发现了27条,在厚壁菌门(Firmicutes)的8个基因组中发现了9条,在绿弯菌门(Chloroflexi)的5个基因组中发现了8条,在酸杆菌门(Acidobacteria)的2个基因组中发现了2条,在衣原体门(Chlamydiae)的1个基因组中发现了1条,它们所占TPS总数的比例依次为9.909%、2.038%、1.897%、0.632%、0.562%、0.141%和0.07%。研究结果显示,细菌TPS主要集中在放线菌中,这与所报道的结果是一致的[15]。与前人[10]的研究结果相比,厚壁菌门(Firmicutes)和酸杆菌门(Acidobacteria)中的细菌TPS是我们基于预测模型发现的。对放线菌门(Actinobacteria) TPS的物种分布进一步分析表明,链霉菌科(Streptomycetaceae)所含序列数目最多(图 1B),达到902条之多,占放线菌门TPS总数的74.793%,其余科所含序列数目均在100条以内。
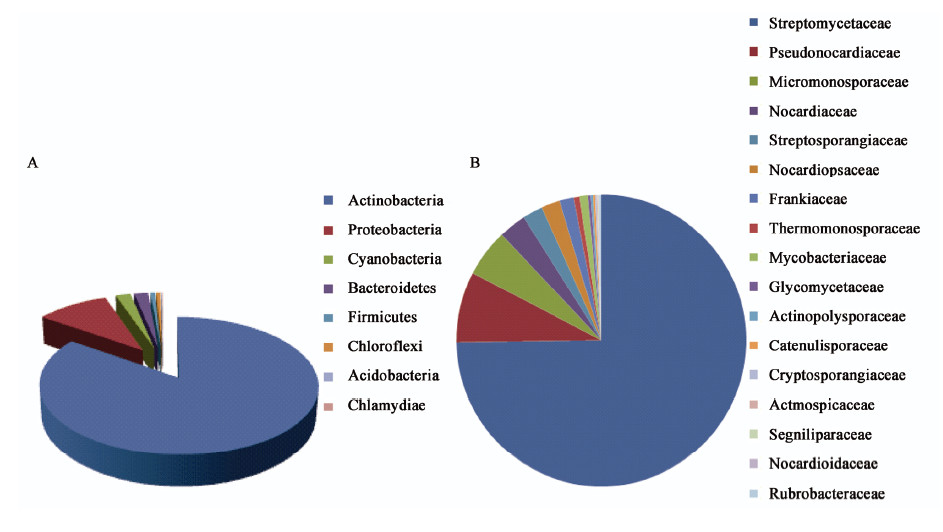
|
|
图 1
细菌萜类合成酶的分布情况
Figure 1
Distribution of terpenoid synthase in bacteria
注:A:在各个门中的分布情况;B:在放线菌门各个科中的分布情况. Note: A: Distribution for each phylum; B: Distribution for each family of Actinobacteria. |
通常单种细菌含有的TPS数在10个以内(表 1),但我们的研究发现一些细菌含有更多数目的TPS,它们的物种分布是:棒状链霉菌(Streptomyces clavuligerus ATCC 27064,拷贝数为26个)、砂栖海水小单胞菌(Salinispora arenicola CNS-205,拷贝数为27个),分别属于放线菌门(Actinobacteria)的链霉菌科(Streptomycetaceae)和小单胞菌科(Micromonosporaceae)。这些编码多拷贝的TPS基因可能是由细菌TPS基因倍增或者基因水平转移引起的,它们在细菌TPS的产物多样性中扮演着重要角色,其潜在的分子机制还需要进一步研究。
| 拷贝数 Number of copy | 物种数 Number of species | |
| 注:拷贝数为单个细菌含有的TPS数目;物种数为含有该拷贝数的细菌数目. Note: Copy numbers mean the numbers of TPS in signal bacteria; Species numbers mean the numbers of bacteria with this copy number. | ||
| 1 | 199 | |
| 2 | 128 | |
| 3 | 94 | |
| 4 | 74 | |
| 5 | 26 | |
| 6 | 13 | |
| 7 | 10 | |
| 8 | 4 | |
| 9 | 3 | |
| 26 | 1 | |
| 27 | 1 | |
为了研究细菌TPS的进化关系,根据细菌萜类合成酶在各个门类中的分布情况选取109个典型细菌TPS以及6个已被实验证实的二萜合成酶进行蛋白序列分析。首先利用多序列比对软件MAFFT对细菌TPS蛋白序列进行比对,然后利用MEGA软件中的最大似然法构建细菌TPS蛋白序列的分子进化树。根据进化树的拓扑结构,将细菌分为4大类(图 2)。第1类(Group Ⅰ)包含41个序列,分布在放线菌门(Actinobacteria)、变形菌门(Proteobacteria)、蓝藻门(Cyanobacteria)中;第2类(Group Ⅱ)包含33个序列,分布在放线菌门(Actinobacteria)、变形菌门(Proteobacteria)、蓝藻门(Cyanobacteria)、绿弯菌门(Chloroflexi)、拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)、酸杆菌门(Acidobacteria)、衣原体门(Chlamydiae)中;第3类(Group Ⅲ)包含19个序列,分布在放线菌门(Actinobacteria)和变形菌门(Proteobacteria)中;第4类(Group Ⅳ)包含9个序列分布在放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)中;未能分类的包含13个序列,分布在放线菌门(Actinobacteria)和酸杆菌门(Acidobacteria)中。其中放线菌门(Actinobacteria)的TPS分布最广,在各大类中都存在,Group Ⅱ所含细菌种类最多,包含了预测到的TPS所在的所有门类。绿弯菌门(Chloroflexi)的TPS仅在Group Ⅱ中出现,这一方面暗示这些TPS具有功能特异性,另一方面可能是由绿弯菌门发现的包含TPS的细菌种类较少造成的(仅有5种细菌),但对其进一步的探索需要实验验证。
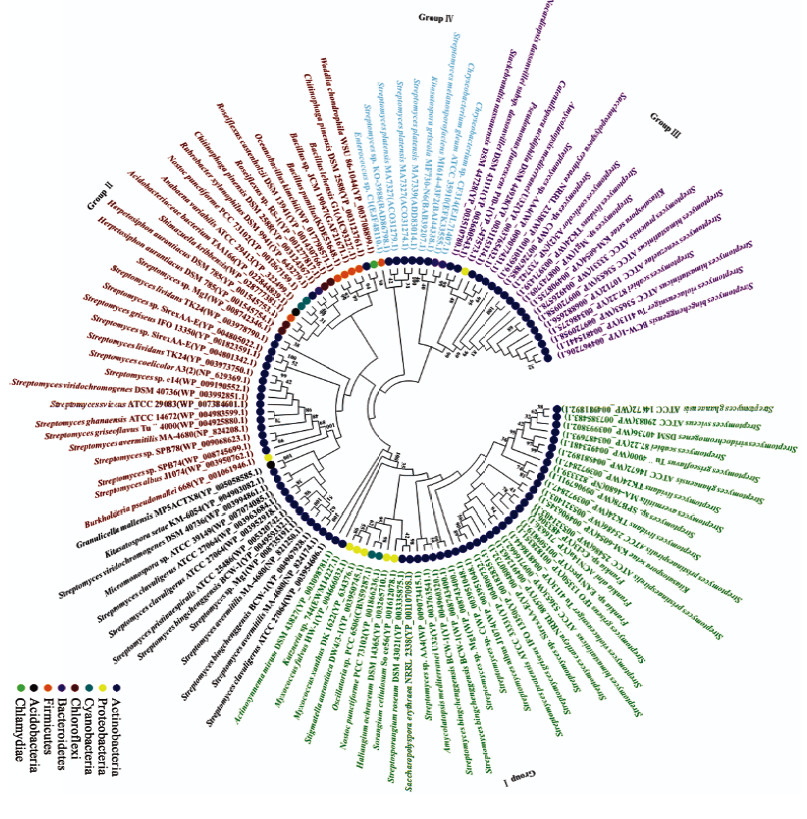
|
|
图 2
115个典型细菌TPS蛋白序列的系统进化树
Figure 2
Phylogenetic tree of 115 typically bacterial TPS protein sequences
注:括号中的序号表示GenBank蛋白登录号,分支上的数字表示Bootstrap值(<30的隐藏). 进化树中不同颜色的序列名称代表不同类别的TPS:绿色代表Group Ⅰ,红褐色代表Group Ⅱ,紫色代表Group Ⅲ,水绿色代表Group Ⅳ,黑色为无法分类的TPS序列. Note: The numbers in parenthesis represent GenBank accession numbers, the numbers on each branch indicate the bootstrap values (hide if <30). Sequences names with different colors in the tree represent different groups of TPS: green represent Group Ⅰ, reddish brown represent Group Ⅱ, purple represent Group Ⅲ, aqua represent Group Ⅳ, black represent sequences that can’t be classified. |
建立在细菌TPS分类的基础上(图 2),可以在每个类别中根据已知功能的细菌TPS去预测未知TPS的功能。因为在Group III中,已知的TPS功能主要为单萜合成酶(monoterpene synthase),所以我们推测Group III中的未知TPS也可能为单萜合成酶。同样的道理,我们可以推测Group I和Group II中未知功能的TPS主要为倍半萜合成酶(sesquiterpene synthase)。Group Ⅳ包含了6条被实验证实的二萜合成酶(diterpene synthase)及3条本研究预测的TPS,推测此类可能为二萜合成酶。
2.3 细菌萜类合成酶序列的特征虽然植物、真菌和细菌中TPS的氨基酸序列差异很大,但是所有的TPS序列都具有2个高度保守的金属离子结合基序:第一个基序为-DDxx[D/E]-或-DDxxx[D/E]-,此基序富含酸性氨基酸,在细菌TPS中经常位于N末端下游80−120个氨基酸处,本研究发现此基序在Group Ⅱ中为-DDxx[D/E]-,而在Group Ⅰ与Group Ⅲ中为-DDxxx[D/E]-;第二个基序为-xxNDxxSxxxE-,通常位于第一基序下游140±5 aa处,这与以前报道的结果是一致的[28]。
值得注意的是我们发现-DDxx[D/E]-或-DDxxx[D/E]-基序的氨基酸组成在不同类别的细菌TPS中具有很大差异(图 3,红色方框标记):Group Ⅰ与Group Ⅱ的上游基序富含酸性氨基酸并含有高比例的芳香族氨基酸,这个保守基序又分为-FxFDDHFLE- (图 3A)和-FFxxDDxxD- (图 3B)两种情况。Group Ⅲ的上游基序为-xVDDxxx[D/E]- (图 3C),虽然同样富含酸性氨基酸,但芳香族氨基酸的含量要低于Group Ⅰ与Group Ⅱ。相对而言,下游的保守基序-xxNDxxSxxxE-在这3个类群中都很保守(图 3,蓝色方框标记)。虽然Group Ⅳ的6条蛋白序列已被实验证实是二萜合成酶[29] (diterpene synthase),但序列比对结果表明-DDxx[D/E]-和-xxNDxxSxxxE-的保守性较低。

|
|
图 3
细菌TPS在各个类群的序列比对结果
Figure 3
Alignment of amino acid sequences of bacterial TPS in each group
注:A:Group Ⅰ的序列比对结果;B:Group Ⅱ的序列比对结果;C:Group Ⅲ的序列比对结果;D:Group Ⅳ的序列比对结果. 红色方框中为-DDxx[D/E]-或-DDxxx[D/E]-基序,蓝色方框中为-xxNDxxSxxxE-基序. Note: A: Alignment result in Group Ⅰ; B: Alignment result in Group Ⅱ; C: Alignment result in Group Ⅲ; D: Alignment result in Group Ⅳ. The sequence in red box is -DDxx[D/E]- or -DDxxx[D/E]- motif, the sequence in blue box is -xxNDxxSxxxE- motif. |
为了进一步分析细菌TPS序列在各个类别之间的差异性,利用MEME软件对TPS序列的Motifs进行了系统分析,并得到20个基序(图 4)。研究发现,这20个基序在不同类群细菌TPS中的分布差异很大。Group Ⅰ所含基序数目最多,为17−19个。Group Ⅱ和Group Ⅲ所含基序数目分别为5−7个和4−5个。Group Ⅳ所含基序数目最少,为1−3个。分析发现Group Ⅰ、Group Ⅱ、Group Ⅲ所共有的Motifs为Motif 2和Motif 5 (图 5A);Group Ⅰ特有的Motifs为Motif 15和Motif 19 (图 5B);Group Ⅲ所特有的Motif为Motif 18 (图 5C),Group Ⅱ和Group Ⅳ所含的Motifs均在其他类群中出现过,无特异Motifs。

|
|
图 4
典型细菌TPS的Motifs
Figure 4
The motifs of typical bacterial TPS
注:左半部分为TPS的分子进化关系;中间为TPS motifs组成结构的Combined P-value;右半部分为20个Motifs在TPS中的分布. Note:Left part illustrates the molecular phylogenetic relationships among TPS. Middle part indicates combined |

|
|
图 5
细菌TPS在各类群中共有及特有的Motifs
Figure 5
Common and group-specific motifs of bacteria TPS in each group
注:A:Group Ⅰ、Group Ⅱ、Group Ⅲ共有的基序(Motifs);B:Group Ⅰ特有的基序(Motifs);C:Group Ⅲ特有的基序(Motif). Note: A: In common motifs of Group Ⅰ, Group Ⅱ and Group Ⅲ; B: Motifs specific to Group Ⅰ; C: Motif specific to Group Ⅲ. |
萜类产物是由其TPS决定的,而TPS的重要氨基酸位点的点突变通常可导致TPS功能(主要指TPS的酶活和TPS的萜类产物)的改变。为了识别和理解细菌TPS中重要氨基酸位点突变对TPS功能的影响,选取Group Ⅲ中的一条序列(登录号为NP_733742.1)进行点突变分析(图 6)。由于-xxNDxxSxxxE-这个基序在不同细菌TPS之间是非常保守的,并且D的保守性最强,所以无论D被替换成20个氨基酸中的哪一个,都会对TPS的酶活产生严重的影响;而D后第6位的E替换为D时,预测结果为白色,证明此处E可与D互换,但当E替换为除D外的其他氨基酸时,则会对TPS的酶活产生严重的影响。除-xxNDxxSxxxE-外,其他位置的氨基酸突变也会对细菌TPS的功能产生

|
|
图 6
分析点突变对NP_733742.1的功能影响
Figure 6
Analysis of effect of point mutations of NP_733742.1
注:深红色(>50):替换后对功能影响很大;绿色和白色(<50):替换后对功能比较小;黑色:野生型. Note: Deep red (>50): have great influence on function if replaced; Green and White (<50): have relatively small influence on function if replaced; Black: Wild type. |
很大影响(详见图 6中红色箭头位置所示的氨基酸位点E、A、G、A、N和L),由于这些氨基酸位点在不同细菌之间是不保守的,所以这些氨基酸位点的突变可能导致萜类产物的改变。细菌TPS的重要氨基酸位点的识别,为今后定向改变萜类合成酶的功能,利用代谢工程方法改变目标萜类产物提供了理论基础。同时,我们的点突变方法也可以应用到其他细菌TPS的功能分析中。
3 结论鉴于TPS具有重要的生理、生态和药用价值,长期以来人们一直对不同的物种进行TPS的识别和功能研究,并在拟南芥、水稻、杨树、葡萄、番茄、土曲霉、娄地青霉、葡萄孢菌等许多物种中进行了较为系统的研究。但相对于植物和真菌TPS的识别和功能研究,细菌TPS的系统研究尚少,本研究通过对细菌TPS的系统分析,在厚壁菌门(Firmicutes)和酸杆菌门(Acidobacteria) 2个门中发现了新的TPS,这为今后利用这些新发现TPS进行功能分析和揭示不同细菌门类之间TPS的进化关系打下了很好的基础。
不同物种TPS的功能探究一直是TPS研究领域的热点,为了系统探究细菌TPS的特征和功能,本文利用MAFFT、MEGA、MEME、PredictProtein等生物信息学方法,从不同角度对细菌TPS进行了预测和分析,通过分子进化、Motifs及点突变影响分析,我们对细菌TPS功能差异的可能原因进行了多层次研究(不同类别之间的差异、Motifs差异和TPS中不同位点氨基酸的差异),这为今后深入研究细菌TPS的功能提供了理论支撑和数据来源。
| [1] | Daum M, Herrmann S, Wilkinson B, et al. Genes and enzymes involved in bacterial isoprenoid biosynthesis[J]. Current Opinion in Chemical Biology, 2009, 13(2): 180-188 |
| [2] | Vranová E, Coman D, Gruissem W. Network analysis of the MVA and MEP pathways for isoprenoid synthesis[J]. Annual Review of Plant Biology, 2013, 64(4): 665-700 |
| [3] | Rohmer M, Knani M, Simonin P, et al. Isoprenoid biosynthesis in bacteria: a novel pathway for the early steps leading to isopentenyl diphosphate[J]. The Biochemical Journal, 1993, 295(Pt 2): 517-524 |
| [4] | Aubourg S, Lecharny A, Bohlmann J. Genomic analysis of the terpenoid synthase (AtTPS) gene family of Arabidopsis thaliana[J]. Molecular Genetics and Genomics, 2002, 267(6): 730-745 |
| [5] | Chen F, Tholl D, Bohlmann J, et al. The family of terpene synthases in plants: a mid-size family of genes for specialized metabolism that is highly diversified throughout the kingdom[J]. The Plant Journal, 2011, 66(1): 212-229 |
| [6] | Falara V, Akhtar TA, Nguyen TT, et al. The tomato terpene synthase gene family[J]. Plant Physiology, 2011, 157(2): 770-789 |
| [7] | Kimura M, Tokai T, Takahashi-Ando N, et al. Molecular and genetic studies of Fusarium trichothecene biosynthesis: pathways, genes, and evolution[J]. Bioscience, Biotechnology, and Biochemistry, 2007, 71(9): 2105-2123 |
| [8] | Brase S, Encinas A, Keck J, et al. Chemistry and biology of mycotoxins and related fungal metabolites[J]. Chemical Reviews, 2009, 109(9): 3903-3990 |
| [9] | Pinedo C, Wang CM, Pradier JM, et al. Sesquiterpene synthase from the botrydial biosynthetic gene cluster of the phytopathogen Botrytis cinerea[J]. ACS Chemical Biology, 2008, 3(12): 791-801 |
| [10] | Yamada Y, Cane DE, Ikeda H. Diversity and analysis of bacterial terpene synthases[J]. Methods in Enzymology, 2012, 515: 123-162 |
| [11] | Berthelot M, André G. Sur l’odeur proper de la terre[J]. Comptes Rendus de l’Académie des Sciences, 1891, 112: 598-599 |
| [12] | Gerber NN. A volatile metabolite of actinomycetes, 2-methylisoborneol[J]. The Journal of Antibiotics, 1969, 22(10): 508-509 |
| [13] | Bentley R, Meganathan R. Geosmin and methylisoborneol biosynthesis in streptomycetes: Evidence for an isoprenoid pathway and its absence in non-differentiating isolates[J]. FEBS Letters, 1981, 125(2): 220-222 |
| [14] | Dairi T, Hamano Y, Kuzuyama T, et al. Eubacterial diterpene cyclase genes essential for production of the isoprenoid antibiotic terpentecin[J]. Journal of Bacteriology, 2001, 183(20): 6085-6094 |
| [15] | Jáchymová J, Votruba J, Víden I, et al. Identification of Streptomyces odor spectrum[J]. Folia Microbiologica, 2002, 47(1): 37-41 |
| [16] | Komatsu M, Tsuda M, Omura S, et al. Identification and functional analysis of genes controlling biosynthesis of 2-methylisoborneol[J]. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(21): 7422-7427 |
| [17] | Wang CM, Cane DE. Biochemistry and molecular genetics of the biosynthesis of the earthy odorant methylisoborneol in Streptomyces coelicolor[J]. Journal of the American Chemical Society, 2008, 130(28): 8908-8909 |
| [18] | Lin X, Hopson R, Cane DE. Genome mining in Streptomyces coelicolor: molecular cloning and characterization of a new sesquiterpene synthase[J]. Journal of the American Chemical Society, 2006, 128(18): 6022-6023 |
| [19] | Kim SY, Zhao P, Igarashi M, et al. Cloning and heterologous expression of the cyclooctatin biosynthetic gene cluster afford a diterpene cyclase and two p450 hydroxylases[J]. Chemistry & Biology, 2009, 16(7): 736-743 |
| [20] | Finn RD, Bateman A, Clements J, et al. Pfam: the protein families database[J]. Nucleic Acids Research, 2014, 42(D1): D222-D230 |
| [21] | Eddy SR. Hidden Markov models[J]. Current Opinion in Structural Biology, 1996, 6(3): 361-365 |
| [22] | Reis MS, Takita MA, Palmieri DA, et al. Bioinformatics for the Citrus EST Project (CitEST)[J]. Genetics and Molecular Biology, 2007, 30(3): 1024-1029 |
| [23] | Li G, Köllner TG, Yin Y, et al. Nonseed plant Selaginella moellendorffi has both seed plant and microbial types of terpene synthases[J]. Proceedings of the National Academy of Sciencesof the United States of America, 2012, 109(36): 14711-14715 |
| [24] | Katoh K, Standley DM. MAFFT multiple sequence alignment software version 7: improvements in performance and usability[J]. Molecular Biology and Evolution, 2013, 30(4): 772-780 |
| [25] | Tamura K, Stecher G, Peterson D, et al. MEGA 6: molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology and Evolution, 2013, 30(12): 2725-2729 |
| [26] | Bailey TL, Boden M, Buske FA, et al. MEME SUITE: tools for motif discovery and searching[J]. Nucleic Acids Research, 2009, 37(Web Server issue): W202-W208 |
| [27] | Yachdav G, Kloppmann E, Kajan L, et al. PredictProtein—an open resource for online prediction of protein structural and functional features[J]. Nucleic Acids Research, 2014, 42(Web Server issue): W337-W343 |
| [28] | Felicetti B, Cane DE. Aristolochene synthase: mechanistic analysis of active site residues by site-directed mutagenesis[J]. Journal of the American Chemical Society, 2004, 126(23): 7212-7221 |
| [29] | Smanski MJ, Yu Z, Casper J, et al. Dedicated ent-kaurene and ent-atiserene synthases for platensimycin and platencin biosynthesis[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(33): 13498-13503 |
 2015, Vol. 42
2015, Vol. 42




