中国科学院微生物研究所、中国微生物学会主办
文章信息
- 董雅君, 崔世修, 刘延峰, 李江华, 堵国成, 吕雪芹, 刘龙
- DONG Yajun, CUI Shixiu, LIU Yanfeng, LI Jianghua, DU Guocheng, LÜ Xueqin, LIU Long
- 功能膜微域在七烯甲萘醌合成过程中的作用解析
- Functional analysis of functional membrane microdomains in the biosynthesis of menaquinone-7
- 生物工程学报, 2023, 39(6): 2215-2230
- Chinese Journal of Biotechnology, 2023, 39(6): 2215-2230
- 10.13345/j.cjb.230111
-
文章历史
- Received: February 16, 2023
- Accepted: March 31, 2023
- Published: April 4, 2023
2. 江南大学 未来食品中心, 江苏 无锡 214122
2. Science Center for Future Foods, Jiangnan University, Wuxi 214122, Jiangsu, China
七烯甲萘醌(menaquione-7, MK-7)是维生素K2的一种亚型,由2-甲基-(1, 4)-萘醌核和C3位侧链上7个异戊二烯基团构成[1](图 1A),主要贮存在细胞膜中,在维持骨骼[2]、心脑血管健康[3]等方面具有良好的作用。目前MK-7主要通过发酵食品提取、化学合成和微生物发酵3种方法进行制备,其中,通过微生物发酵法对于实现MK-7的高效合成具有重要意义[4]。在枯草芽孢杆菌(Bacillus subtilis)中存在完整的MK-7合成途径,可以分为糖酵解-磷酸戊糖、分支酸和甲基-4-磷酸赤藓糖醇(methylerythritol-4-phosphate, MEP),以及典型的甲萘醌合成4个模块[5]。其中,骨架结构(1, 4)-2-羟基-2-萘甲酸(1, 4-dihydroxy-2- naphtooic acid, DHNA)由甲萘醌模块负责合成,以分支酸(chorismic acid, CHA)为底物,经由(1, 4)-二羟基-2-萘酰辅酶A水解酶[(1, 4)-dihydroxy-2-naphthoyl-CoA hydrolase, YuxO]、(1, 4)-二羟基-2-萘酰辅酶A合酶[(1, 4)-dihydroxy-2-naphthoyl-CoA synthase, MenB]等7种不同的酶参与催化[6];侧链结构七异戊二烯基焦磷酸(heptaprenyl diphosphate, HDP)由MEP途径负责合成,以1-脱氧-d-木糖-5磷酸(1-deoxyxylulose- 5-phosphate, DXP)为底物,经由七异戊二烯基焦磷酸合酶(heptaprenyl diphosphate synthase component Ι/II, HepS/HepT)、法呢基焦磷酸合酶(farnesyl diphosphate synthase, IspA)和异戊烯基焦磷酸异构酶(isopentenyl diphosphate isomerase, Fni)等11种不同的酶参与催化[7];随后HDP在1, 4-二羟基-2-萘甲酸辛二烯酯转移酶[(1, 4)-dihydroxy-2-naphthoate heptaprenyltransferase, MenA]的催化下转移到骨架结构的C3位[8],最终通过去甲基甲萘醌甲基转移酶(demethylmenaquinone methyltransferase, MenH)形成MK-7[9](图 1B)。Ma等[11]在枯草芽孢杆菌168中增加部分限速酶的表达量并进行不同组合,最终使MK-7产量达到50 mg/L。Cui等[12]通过增强前体供应和构建Phr60-Rap60-Spo0A群体响应调控系统,使碳流量更多进入MK-7合成途径,最终成功获取一株高产MK-7的菌株,摇瓶水平MK-7产量达到360 mg/L。
区室化可以将代谢途径中亚细胞定位不同的酶聚集于一个区域,以增加底物或酶的局部浓度,实现更快的反应速率和更高的生产率[13]。在真核生物中,可以通过脂筏[14]、线粒体[15]和内质网[16]等实现区室化。例如,Deloache等[17]将紫色杆菌素前体(prodeoxyviolacein, PDV)合成的途径酶VioB和VioE与标准过氧化物酶体基质蛋白1型信号肽(peroxisome target signal type 1, PTS1)标签融合,使酶在过氧化物酶体中表达,显著提高了PDV产量;Avalos等[18]在酵母线粒体中表达Ehrlich途径的相关酶,提高了局部酶的浓度,显著提高了异丁醇的产量。在细菌中,功能膜微域(functional membrane microdomains, FMMs)等结构域的存在亦可使质膜区室化。由脚手架蛋白和聚异戊二烯类物质构成的FMMs参与多种不同类型的细胞生命活动[19]。其中,脚手架蛋白是FMMs的标记蛋白,可以募集蛋白、脂质等物质至FMMs中,并促进这些物质间的相互作用和寡聚化[20-21];聚异戊二烯化合物的存在使FMMs具有高度致密性和疏水性,有助于相关蛋白在FMMs中集聚[22]。Lv等[23]将N-乙酰氨基葡萄糖(N-acetylglucosamine, GlcNAc)合成所需的途径酶氨基葡萄糖-6-磷酸乙酰转移酶(GlcNAc-6-phosphate N-acetyltransferase, GNA1)、GlcN-6-磷酸合酶(GlcN-6-phosphate synthase, Glms)、磷酸葡萄糖异构酶(phosphoglucose isomerase, Pgi)和卤酸脱卤酶样磷酸酶(haloacid dehalogenase-like phosphatases, YqaB)与脚手架蛋白融合表达,使4种酶聚集在FMMs中,通过集聚效应和底物通道效应提高酶的催化效率,最终促进了GlcNAc的高效合成。
MK-7的合成同细胞膜有关,FMMs在MK-7的合成过程中可能存在不可或缺的作用。本研究以枯草芽孢杆菌为研究对象,探讨膜上聚异戊二烯化合物MK-7和FMMs之间的相关性,并通过FMMs实现MK-7合成途径中关键酶的区室化。首先,通过可视化观察MK-7和FMMs的分布特征,明确了二者的相关性;其次,通过分析FMMs的完整性破坏前后,细胞膜上MK-7含量和膜有序度的变化,明确MK-7是构成FMMs关键聚异戊二烯类化合物的一种;最后,通过FloA蛋白将MK-7合成途径中的关键酶锚定至FMMs中,获得了一株高产MK-7的菌株BS3AT,摇瓶水平MK-7产量达到300.3 mg/L,3 L发酵罐中MK-7产量达到464.2 mg/L。结果表明,在枯草芽孢杆菌中通过FMMs实现MK-7合成途径中关键酶的区室化,可以有效提高MK-7的产量,这为MK-7的高效合成提供了一种新的思路。
1 材料与方法 1.1 菌株和质粒本研究所使用的菌株、质粒如表 1所示。
| Name | Description | Reference |
| Strains | ||
| E. coli JM109 | For plasmid construction | Lab stock |
| BS168 | Bacillus subtilis 168 | Lab stock |
| BS168C | BS168 derivate, ΔfloA ΔfloT ΔyisP ΔsqhC | |
| BS3 | BS168 derivate, Pveg-kinA-ΔPAS-A ΔkinB ΔspoIIA Δspo0IIE, P43-menF P43-menB Phbs-menE P43-entC ΔdhbB Phbs-tkt P43-ppsA ΔptsG Phbs-aroGfbr: : lox72 P43-aroK Phbs-ispA P43-hepS/T Phbs-kdpG P43-dxr P43-dxs P43-fni Pmena-menA: : lacA Pmena-menA: : thrC Pmena-menA: : dacA | Lab stock |
| BS3C | BS3 derivate, ΔfloA ΔfloT ΔyisP ΔsqhC | This work |
| BS3FA | BS3 derivate, : : P43-floA (integrated site is between yvoA and nagBA) | This work |
| BS3FT | BS3 derivate, : : P43-floT (integrated site is between yvoA and nagBA) | This work |
| BS3AF | BS3 derivate, : : P43-floA-fni (integrated site is between yvoA and nagBA) | This work |
| BS3F | BS3 derivate, : : P43-fni (integrated site is between yvoA and nagBA) | This work |
| BS3AI | BS3 derivate, : : P43-floA-ispA (integrated site is between yvoA and nagBA) | This work |
| BS3I | BS3 derivate, : : P43-ispA (integrated site is between yvoA and nagBA) | This work |
| BS3AT | BS3 derivate, : : P43-floA-hepT (integrated site is between yvoA and nagBA) | This work |
| BS3T | BS3 derivate, : : P43-hepT (integrated site is between yvoA and nagBA) | This work |
| BS3AO | BS3 derivate, : : P43-floA-yuxO (integrated site is between yvoA and nagBA) | This work |
| BS3O | BS3 derivate, : : P43-yuxO (integrated site is between yvoA and nagBA) | This work |
| Plasmids | ||
| P7C6 | Pmd18-T containing lox71-Cmr-lox66 cassette | Lab stock |
| P7S6 | Pmd18-T containing lox71-Spec-lox66 cassette | Lab stock |
| P7Z6 | Pmd18-T containing lox71-Zeo-lox66 cassette | Lab stock |
| pP43NMK | Amp, Kan, E. coli-B. subtilis shuttle vector | Lab stock |
| pHT01 | Amp, Cm, E. coli-B. subtilis shuttle vector | Lab stock |
| pHT01-floA-eGFP | floA-eGFP | This work |
| pHT01-floT-eGFP | floT-eGFP | This work |
| pHT01-menH-eGFP | menH-eGFP | This work |
| pHT01-menA-eGFP | menA-eGFP | This work |
| pHT01-menB-eGFP | menB-eGFP | This work |
| pHT01-ispA-eGFP | ispA-eGFP | This work |
| pHT01-fni-eGFP | fni-eGFP | This work |
| pHT01-yuxO-eGFP | yuxO-eGFP | This work |
| pHT01-hepS-eGFP | hepS-eGFP | This work |
| pHT01-hepT-eGFP | hepT-eGFP | This work |
LB (Luria Broth)培养基:10 g/L蛋白胨,10 g/L NaCl,5 g/L酵母粉,121 ℃灭菌20 min。LB固体培养基中添加2%的琼脂粉。
发酵培养基:50 g/L葡萄糖、50 g/L甘油、50 g/L大豆蛋白胨、0.06 g/L磷酸二氢钾,115 ℃灭菌20 min。
实验所用抗生素:卡那霉素50 µg/L、博来霉素20 µg/L、壮观霉素100 g/L、氯霉素30 µg/L。
1.3 仪器和设备本研究中所使用的主要仪器:PCR扩增仪(伯乐有限公司)、核酸电泳仪(六一电泳仪厂)、超声波细胞破碎仪(伯乐有限公司)、分光光度计(赛默飞有限公司)、超速离心机(赛默飞有限公司)、激光共聚焦显微镜(徕卡显微系统公司)、荧光显微镜(尼康公司)、HPLC-1260 (安捷伦公司)、3 L迪必尔发酵罐(NBS有限公司)。
1.4 质粒和菌株构建通过一步克隆法构建本研究所用质粒,所用引物如表 2所示。以pHT01-floA-eGFP质粒构建为例:以BS168的基因组作为模板,以FloA-1F和FloA-1R为引物扩增得到floA基因片段,以eGFP-1F和eGFP-1F为引物扩增得到eGFP基因片段;以pHT01质粒为模板,以Z-1F和Z-1R为引物扩增得到载体基因片段。参照一步克隆说明书中的质粒构建方法,获得质粒pHT01-floA-eGFP。
| Primer name | Primer sequence (5′→3′) | Size (bp) |
| Z-1F | GGATGAACTATACAAATAATCTAGAGTCGACGTCCCCGG | 39 |
| Z-1R | GTGTACATTTCACCTCCTTTAAATTACTTTCATTATGAGTT | 41 |
| eGFP-1F | GGAAGCGGCGGTGGCGGCAGCATGGGTAAGGGAGAAGAACTTTTCACT | 48 |
| eGFP-1R | GTCGACTCTAGATTATTTGTATAGTTCATCCATGCCATGTGTAATCC | 47 |
| FloA-1F | TGAAATGTACACATGGATCCGTCAACACTTATGATTCTGG | 40 |
| FloA-1R | TACCCATGCTGCCGCCACCGCCGCTTCCTGATTTGCGGTCTTCATCCGAAGG | 52 |
| FloA-1F | AGGAGGTGAAATGTACACATGACAATGCCGATTATAATGATCATCGGA | 48 |
| FloA-1R | GCCACCGCCGCTTCCTTACTCTGATTTTTGGATCGTTTTGGCTTCT | 46 |
| MenH-1F | AGGAGGTGAAATGTACACATGCAGGACTCAAAAGAACAGCG | 41 |
| MenH-1R | GCTGCCGCCACCGCCGCTTCCTTTCCATCCGATATGCGTGGCAG | 44 |
| MenA-1F | GCCAGTGAATTCGAGCTCATCAAAAATTCCCTTCCCGTTTTTTCGA | 59 |
| MenA-1R | TTACCCATGCTGCCGCCACCGCCGCTTCCTCGGAAATAGCTGATCAATAATCCGATCGA | 59 |
| MenB-1F | AGGAGGTGAAATGTACACATGGCTGAATGGAAAACAAAACGGA | 43 |
| MenB-1R | GCCGCTTCCCGGAAAACGAGGGAACTGTCC | 43 |
| IspA-1F | AGGAGGTGAAATGTACACGTGACAAATAAATTAACGAGCTTTCTGGCG | 48 |
| IspA-1R | ACCGCCGCTTCCGTGATCTCTTGCCGCAATTAAATCAC | 48 |
| Fni-1F | AGGAGGTGAAATGTACACGTGACTCGAGCAGAACGAAAAAGAC | 43 |
| Fni-1R | GCCGCTTCCTCGCACACTATAGCTTGATGTATTGACC | 37 |
| YuxO-1F | GGAGGTGAAATGTACACATGGATATGAAGCACACATTGCTTGAAG | 45 |
| YuxO-1R | GCTGCCGCCACCGCCGCTTCCTTTCTTGATGACAGCCAGCGTGC | 45 |
| HepS-1F | GGAGGTGAAATGTACACTTGCAAGACATCTACGGAACTTTAGCC | 52 |
| HepS-1R | CCATGCTGCCGCCACCGCCGCTTCCCCCTTCTTCCACTTTCTGATGATAGGT | 52 |
| HepT-1F | AGGAGGTGAAATGTACACATGTTAAATATCATTCGTTTACTGGCGGAG | 48 |
| HepT-1R | CCGCCGCTTCCAAATTTTCTTTTACCGATATATTTTGCGATGGCT | 48 |
| QC-yisP-U-1F | TGATGCCTGATGCGAAAGTCTTTT | 53 |
| QC-yisP-U-1R | ATCCGCTCAAAACACCCGGCCTTGATTGAG | 53 |
| QC-yisP-S-1F | AGGCCGGGTGTTTTGAGCGGATAACAATTTCACACAGGAA | 53 |
| QC-yisP-S-1R | AGACTTGAAGAAGCATAACGCCAGGGTTTTCCCAGTC | 53 |
| QC-yisP-D-1F | GGAAAACCCTGGCGTTATGCTTCTTCAAGTCTAGTATGTTTTTTCCTAATTGT | 53 |
| QC-yisP-D-1R | ACTGGACAGAAAACAATTAGATCAGCTTTTAGA | 53 |
| QC-FloA-U-1F | CAGATCACATTTATATGGCGCCTGG | 58 |
| QC-FloA-U-1R | GTGTGAAATTGTTATCCGCTCATAACTTCTCCTCGTTTCTATTTAAATTTCTCTCACG | 58 |
| QC-FloA-S-1F | AGAAACGAGGAGAAGTTATGAGCGGATAACAATTTCACACAGGAA | 58 |
| QC-FloA-S-1R | CTCCCTTCTTAGAGAGATAACGCCAGGGTTTTCCCAGT | 58 |
| QC-FloA-D-1F | AAACCCTGGCGTTATCTCTCTAAGAAGGGAGAACGCTGATG | 58 |
| QC-FloA-D-1R | ACATTTAAATTTACTTCTTACCCGCCGAA | 58 |
| QC-FloT-U-1F | TTGGCTGCCTGAAATAAAATCATGCG | 58 |
| QC-FloT-U-1R | CCTGTGTGAAATTGTTATCCGCTCATCAAATTCCTCCTTTTATGTTAAATGGGTTCAT | 58 |
| QC-FloT-C-1F | AAAAGGAGGAATTTGATGAGCGGATAACAATTTCACACAGGAAAC | 58 |
| QC-FloT-C-1R | GTTCTGCCCTTTCCTAACGCCAGGGTTTTCCCAGTC | 58 |
| QC-FloT-D-1F | GAAAACCCTGGCGTTAGGAAAGGGCAGAACCGTATGG | 58 |
| QC-FloT-D-1R | GGTGTCAAAGCAGGTCTTACTACAGG | 58 |
| QC-sqhC-U-1F | GGATTTGTTTGACGAGCTGAAAAGC | 51 |
| QC-sqhC-U-1R | GTGTGAAATTGTTATCCGCTCATGAAACGTGAATCTTATCAAGCGGAG | 51 |
| QC-sqhC-C-1F | GCTTGATAAGATTCACGTTTCATGAGCGGATAACAATTTCACACAGGAAAC | 51 |
| QC-sqhC-C-1R | GAGTTGAGCGTGTAACGCCAGGGTTTTCCCAGTCA | 51 |
| QC-sqhC-D-1F | AAAACCCTGGCGTTACACGCTCAACTCCTTCAGAAGC | 51 |
| QC-sqhC-D-1R | CCGCTTTCAGCTCTTTATTTGGCAG | 51 |
| U-1F | AAACCGATTATTTTGAATGTAACGTATATACCAATGAA | 54 |
| U-1R | GTGTGAAATTGTTATCCGCTCATGAATATCAATAAACAATCGCCTATTCCGATT | 54 |
| C-1F | ATAGGCGATTGTTTATTGATATTCATGAGCGGATAACAATTTCACACAGGAAAC | 54 |
| C-1R | GGTAAGAGAGGAATGTACACTAACGCCAGGGTTTTCCCAGTC | 54 |
| P43-1F | GGAAAACCCTGGCGTTAGTGTACATTCCTCTCTTACCTATAATGGTACCG | 54 |
| P43-FloA-1R | ATAAGTGTTGACGGATCCATTGATAGGTGGTATGTTTTCGCT | 54 |
| FloA-1F | ACATACCACCTATCAATGGATCCGTCAACACTTATGATTCTG | 54 |
| FloA-1R | TTGAAAGGAACATGCTGACTTTGATTTGCGGTCTTCATCCGAAGG | 54 |
| FloA-D-1F | ACCGCAAATCAAAGTCAGCATGTTCCTTTCAAGGTCTTAAT | 54 |
| D-1R | GTTCGATCCTCAAAATGAACGAAGGC | 54 |
| P43-SPFH-1R | CGGCATTGTCATTGATAGGTGGTATGTTTTCGCTTGAACT | 45 |
| SPFH-1F | ACATACCACCTATCAATGACAATGCCGATTATAATGATCATCGGA | 45 |
| SPFH-1R | CATGCTGACTTTTTATCAGCCTCTGCTGTTGCGA | 45 |
| SPFH-D-1F | CAGCAGAGGCTGATAAAAAGTCAGCATGTTCCTTTCAAGGTC | 45 |
| FLoA-2R | GCTGCCGCCACCGCCGCTTCCTGATTTGCGGTCTTCATCCGAAGG | 58 |
| Fni-1F | GACCGCAAATCAGGAAGCGGCGGTGGCGGCAGCGTGACTCGAGCAGAACGAAAAAGAC | 58 |
| Fni-1R | CTTGAAAGGAACATGCTGACTTTTATCGCACACTATAGCTTGATGTATTGACC | 53 |
| Fni-D-1F | GCTATAGTGTGCGATAAAAGTCAGCATGTTCCTTTCAAGGTCT | 58 |
| IspA-1F | GCAAATCAGGAAGCGGCGGTGGCGGCAGCGTGACAAATAAATTAACGAGCTTTCTGGCG | 59 |
| IspA-1R | AACATGCTGACTTTTAGTGATCTCTTGCCGCAATTAAATCACA | 59 |
| IspA-D-1F | GCGGCAAGAGATCACTAAAAGTCAGCATGTTCCTTTCAAGGTC | 59 |
| HepT-1F | CGCAAATCAGGAAGCGGCGGTGGCGGCAGCATGTTAAATATCATTCGTTTACTGGCGGAG | 63 |
| HepT-1R | AGACCTTGAAAGGAACATGCTGACTTTTAAAATTTTCTTTTACCGATATATTTTGCGATGGCT | 63 |
| HepT-D-1F | CGGTAAAAGAAAATTTTAAAAGTCAGCATGTTCCTTTCAAGGTCTTAATGACGCGGCT | 63 |
| YuxO-1F | CCGCAAATCAGGAAGCGGCGGTGGCGGCAGCATGGATATGAAGCACACATTGCTTGAAG | 59 |
| YuxO-1R | AAGGAACATGCTGACTTTTATTTCTTGATGACAGCCAGCGTG | 59 |
| YuxO-D-1F | CATCAAGAAATAAAAGTCAGCATGTTCCTTTCAAGGTC | 59 |
| P43-Fni-1R | CTGCTCGAGTCACTGATAGGTGGTATGTTTTCGCTTGAACTTT | 43 |
| Fni-2F | ACATACCACCTATCAGTGACTCGAGCAGAACGAAAAAGAC | 43 |
| P43-IspA-1F | GCCAGAAAGCTCGTTAATTTATTTGTCACTGATAGGTGGTATGTTTTCGCTTGAACTTTTAAAT | 64 |
| IspA-2F | ACATACCACCTATCAGTGACAAATAAATTAACGAGCTTTCTGGC | 64 |
| P43-HepT-1R | CGCCAGTAAACGAATGATATTTAACATTGATAGGTGGTATGTTTTCGCTTGAACT | 60 |
| HepT-2F | ACATACCACCTATCAATGTTAAATATCATTCGTTTACTGGCGGA | 60 |
| P43-YuxO-1R | TGTGTGCTTCATATCCATTGATAGGTGGTATGTTTTCGCTTGAACT | 59 |
| YuxO-2F | ACATACCACCTATCAATGGATATGAAGCACACATTGCTTGA | 59 |
| The underlined sequences are the linker used in the experiment, and the linker used is the flexible linker. | ||
通过Cre/loxP系统构建本研究所用重组枯草芽孢杆菌,所用引物如表 2所示。以重组工程菌BS3FA的构建为例:以BS168的基因组作为模板,以U-1F和U-1R为引物扩增得到上游同源臂基因片段,以FloA-1F和FloA-1R为引物扩增得到floA基因片段,以D-1F和D-1R为引物扩增得到下游同源臂基因片段;以pP43NMK质粒为模板,以P43-1F和P43-1R为引物扩增得到P43启动子基因片段;以P7C6质粒为模板,以C-1F和C-1R引物对扩增得到lox71-zeo-lox66基因片段。通过重叠延伸PCR融合表达5个片段。通过化学转化的方法将构建好的表达框转入BS3中,通过菌落PCR验证获得重组工程菌BS3FA。
1.5 摇瓶发酵取保藏在甘油管中的重组枯草芽孢杆菌接种至LB固体培养基上进行活化,挑取单菌落接种至2 mL LB液体培养基中,在37 ℃、220 r/min培养条件下振荡培养8 h;取1 mL种子液接种至20 mL发酵培养基中于40 ℃、220 r/min振荡培养6 d。每24 h取样500 µL监测细胞生长情况,以及MK-7含量的检测。每组样品3个平行。
1.6 细胞组分的分离和提取发酵样品:按照上述摇瓶发酵方法,取发酵2 d后的发酵液15 mL用于细胞组分分离。
细胞外:将发酵样品在5 000 r/min离心10 min,分为“上清1”和“沉淀1”。“上清1”用作检测胞外MK-7含量。
全细胞:“沉淀1”用无菌水洗涤3次并用无菌水重悬后,用作检测全细胞MK-7产量。
细胞内:重悬后的“沉淀1”通过超声波细胞破碎仪进行破碎,在10 000 r/min离心40 min,分为“上清2”和“沉淀2”;“上清2”通过超速离心机在140 000 r/min下离心1 h,分为“上清3”和“沉淀3”,“上清3”用作检测细胞内MK-7含量。
细胞膜:“沉淀3”用无菌水重悬后,用作检测细胞膜上MK-7含量。
1.7 荧光显微镜观察eGFP样品制备:取保藏在甘油管中的重组枯草芽孢杆菌接种到LB固体培养基上进行活化,挑取单菌落接种到2 mL LB液体培养基中在37 ℃、220 r/min培养条件下振荡培养10 h;5 000 r/min、10 min离心收集菌体,用LB液体培养基清洗3遍,得到荧光观察样品。
罗丹明123样品制备:在发酵36 h后的摇瓶发酵液中加入20 µL的2 mmol/L罗丹明123母液,继续孵育1 h;5 000 r/min、10 min离心收集菌体,用LB液体培养基清洗3遍,得到荧光观察样品。
Di-4-ANEPPDHQ样品制备[23]:取保藏在甘油管中的重组枯草芽孢杆菌接种到LB固体培养基上进行活化,挑取单菌落接种到2 mL LB液体培养基中在37 ℃、220 r/min培养条件下振荡培养10 h;5 000 r/min、10 min离心收集菌体,弃上清,添加1 mL LB培养基(含有5 µmol/L di-4-ANNEPPDHQ),于冰上孵育10 min,离心弃上清,用预冷的LB培养基洗涤3次。
eGFP和罗丹明123荧光观察:通过使用尼康显微镜C-HGF (100×油镜)观察eGFP和罗丹明123,使用FITC滤光片,通过ImageJ软件进行图像处理和分析。
Di-4-ANEPPDHQ荧光观察:使用激光共聚焦显微镜Leica TCS SP8显微镜(60×油镜),样品采用488 nm的激发光波长,500−580 nm (有序相)和620−750 nm (无序相)的发射光波长,显微镜设置(偏移值,PMT电压和激光功率)均相同。通过均一化极性数值(generalized polarization value, GP value)来表示定量分析,GP图像根据Owen等[24]提供的ImageJ自定义宏生成。计算公式如下:
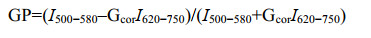
|
其中,I500−580和I620−750分别代表500−580 nm和620−750 nm通道中每个像素的荧光强度。Gcor为校准因子,计算公式如下:

|
GPexp是二甲基亚砜(dimethyl sulfoxide, DMSO)溶液在相同显微镜设置下中di-4-ANEPPDHQ的GP值,GPthe则是染料在DMSO中的参考值,一般认为GPthe=–0.85。处理后的荧光图像中每个像素强度乘GP值,则可以生成色相饱和度亮度的伪彩色GP图像。
1.8 MK-7的萃取和检测MK-7萃取:按照异丙醇和正己烷1:2 (体积比)的比例配制萃取液。将待测样品和萃取液按照1:4 (体积比)的比例混匀离心后,上层有机相检测MK-7含量。
MK-7检测:检测仪器为配备有紫外检测器的高性能液相色谱(high performance liquid chromatography, HPLC),色谱柱为C18-ODS色谱柱(5 µm, 250 mm×4.6 mm),流动相为90%的甲醇和10%的二氯甲烷(色谱级)。进样量为10 µL,流速为1 mL/min,柱温40 ℃,检测波长254 nm。MK-7校准曲线在1 mg/L至100 mg/L之间呈线性(R2=0.998)。
1.9 3 L发酵罐发酵3 L发酵罐所用培养基:50 g/L葡萄糖、50 g/L甘油、50 g/L大豆蛋白胨、0.06 g/L磷酸二氢钾,115 ℃灭菌20 min。
取保藏在甘油管中的重组枯草芽孢杆菌接种到LB固体培养基上进行活化,挑取一个单菌落接种到50 mL LB液体培养基中在37 ℃、220 r/min培养条件下振荡培养10 h;将种子液按照4% (体积分数)的接种量接种到含有1.5 L发酵培养基的3 L发酵罐中,培养6 d。
发酵罐条件设置:通气量为1.5 vvm,发酵温度为40 ℃,pH通过50% (体积分数)的氨水维持在7左右,搅拌转速根据溶氧进行调节,溶氧维持在50%,转速在400−600 r/min。
2 结果与分析 2.1 FMMs和MK-7的相关性分析为了明确FMMs与MK-7之间是否具有相关性,该研究对两者的亚细胞定位进行了分析。有研究指出,脚手架蛋白FloA和FloT构成FMMs的骨架结构,可以作为FMMs的标志蛋白[25]。首先,分别将脚手架蛋白FloA和FloT与eGFP进行融合表达,进行荧光显微镜观察。结果显示,FloA-eGFP和FloT-eGFP主要以不连续的点状结构分布于细胞膜上(图 2A、2B)。进一步使用MK-7的特异性染料罗丹明123染料对课题组前期构建的一株高产MK-7菌株BS3进行染色[26]。结果显示,MK-7也以不均匀的点状结构分布在细胞膜上(图 2C),推测FMMs和MK-7存在相关性。

|
| 图 2 FloA、FloT和MK-7的分布特征 Fig. 2 Characteristic of FloA, FloT and MK-7. A: Subcellular localization of FloA. B: Subcellular localization of FloT. C: Subcellular localization of MK-7. Bar=1 µm. A:FloA的亚细胞定位. B:FloT的亚细胞定位. C:MK-7的亚细胞定位. 标尺=1 µm |
| |
除脚手架蛋白外,聚异戊二烯化合物也参与维持FMMs的完整性。聚异戊二烯化合物的合成需要YisP[27](合成C30萜类化合物)和SqhC[28](聚异戊二烯环化酶)的参与。因此,为了探讨FMMs的完整性对MK-7合成的影响,该研究在BS3菌株中通过Cre/loxP基因编辑方法敲除了基因floA、floT、sqhC和yisP,获得菌株BS3C,检测BS3和BS3C菌株中不同组分的MK-7含量。结果发现,FMMs的完整性被破坏后,MK-7的总产量基本不变。其中,BS3C细胞内MK-7的含量增加了26.8%,细胞膜上MK-7的含量降低了24.8% (图 3B)。这表明在破坏FMMs的完整性后,细胞膜和细胞内MK-7的含量发生明显变化。推测可能原因是FMMs的完整性被破坏后,会影响MK-7进入细胞膜的过程,进而使MK-7在细胞内积累。

|
| 图 3 FMMs完整性破坏对MK-7的影响 Fig. 3 Effect of disruption of FMMs integrity on MK-7 content. A: Diagram of gene knockout expression box. B: Changes in MK-7 content in different fractions of strains BS3 and BS3C. A:基因敲除表达框示意图. B:菌株BS3和BS3C不同组分中的MK-7含量 |
| |
FMMs是一类高度有序的膜结构域,可以通过膜有序度来表征细胞膜上FMMs的变化情况[23]。为了明确FMMs的完整性和MK-7对细胞膜有序度的影响,同样在BS168菌株中通过敲除基因floA、floT、sqhC和yisP,获得菌株BS168C,使用di-4-ANEPPDHQ[29]染料对BS3、BS3C和BS168C进行染色,通过激光共聚焦显微镜采集双通道图像(500−580 nm和620−750 nm),并通过ImageJ进行GP值分析和图像转化。GP值可指示膜有序度,GP值越高,膜有序度越高。对获得的直方图进行拟合,获得膜有序度定量分析结果。结果发现,BS3的GP峰值为0.48,平均GP值为0.47±0.01;BS3C的GP峰值为0.43,平均GP值为0.43±0.01;BS168C的GP峰值为0.33,平均GP值为0.32±0.01 (图 4A–4C)。其中,BS3的膜有序度高于BS3C,表明FMMs完整性被破坏后影响了MK-7进入细胞膜的过程,导致了BS3C细胞膜上MK-7含量降低。BS3C的膜有序度明显高于BS168C,表明MK-7可以有效提高细胞膜有序度。上述结果表明MK-7是FMMs中的一类重要的聚异戊二烯类化合物。
|
| 图 4 BS3、BS3C和BS168C膜有序度的定量分析 Fig. 4 Quantitative analysis of membrane order in BS3, BS3C and BS168C. A: Confocal images of BS3, BS3C and BS168C strains from the green channel (500−580 nm) and red channel (620−750 nm), the merged images from both channels and the GP images. Bar=1 µm. B: GP histograms of BS3, BS3C and BS168C strains show the number of pixels distributed in each GP value. C: Average GP values of BS3, BS3C and BS168C. A:每一行从左到右依次为500−580 nm和620−750 nm波长下拍摄的图像、Merged处理后的图像和GP图像. 标尺=1 µm. B:BS3、BS3C和BS168C不同区域的GP值分布直方图. C:BS3、BS3C和BS168C的平均GP值 |
| |
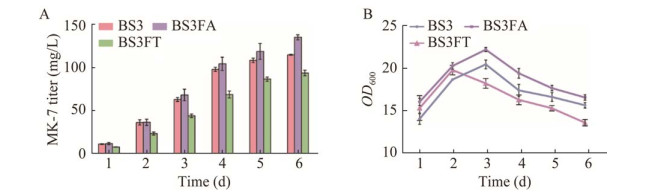
为了探究MK-7如何定位至细胞膜甚至FMMs中,该研究选择8个MK-7合成的关键酶:MenH、MenA、MenB、YuxO、HepS、HepT、Fni、IspA,拟对它们的亚细胞定位情况进行分析。其中,Fni、IspA、HepS和HepT参与MK-7侧链合成;MenB和YuxO参与MK-7骨架结构合成;MenA将侧链HDP整合到骨架DHNA中形成DMK,最后通过MenH甲基化形成MK-7。
分别将上述8个关键酶与eGFP融合表达,进行荧光显微镜观察。结果显示,MenH-eGFP、HepS-eGFP和MenB-eGFP均以不连续的点状结构分布在细胞膜上,和FMMs标记蛋白的亚细胞定位极为类似,推测MenH、HepS和MenB主要定位于FMMs中;MenA-eGFP蛋白则是典型的细胞膜定位蛋白;YuxO-eGFP、Fni-eGFP、HepT-eGFP和IspA-eGFP均匀地分布在细胞质中(图 5)。由上述结果可知,负责骨架结构和侧链合成的关键酶在细胞中存在一定的空间距离,可能会降低代谢反应速率,进而影响MK-7的合成。

|
| 图 5 MK-7合成途径关键酶的分布特征 Fig. 5 Subcellular localization of pathway enzymes. Bar=1 µm. 标尺=1 µm |
| |
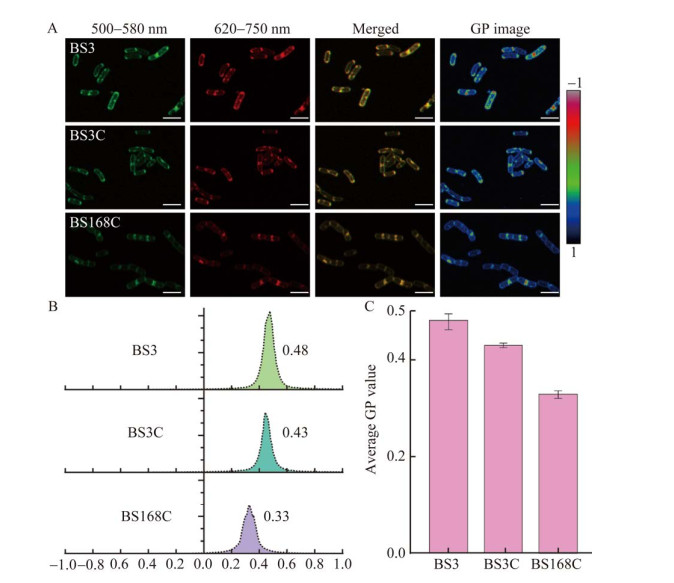
鉴于FMMs和MK-7的相关性,本研究尝试使用FMMs来进行MK-7合成途径关键酶的区室化。首先以BS3为出发菌株,分别构建过表达FloA、FloT的重组菌株BS3FA和BS3FT,验证过表达不同脚手架蛋白对MK-7产量的影响。结果发现,BS3FA的MK-7产量达到135.3 mg/L,与BS3相比提高了17.8% (图 6A),同时过表达FloA对细胞生长也有一定的促进作用。BS3FT的产量为94.3 mg/L,与BS3相比降低了18.0%,且2 d后,与其他菌株相比,OD600明显减小(图 6B),这可能是BS3FT菌株MK-7产量降低的主要原因。因此,选择FloA蛋白进行后续研究。
|
| 图 6 过表达脚手架蛋白对MK-7产量的影响 Fig. 6 Effect of overexpression scaffold proteins on MK-7 production. A: MK-7 titer of BS3, BS3FA and BS3FT. B: OD600 of BS3, BS3FA and BS3FT. A:BS3、BS3FA和BS3FT的MK-7产量. B:BS3、BS3FA和BS3FT的细胞生长情况 |
| |
接下来尝试通过使用FloA蛋白将MK-7合成途径关键酶锚定在FMMs中,以实现MK-7合成途径关键酶的区室化(图 7A)。以B3S为出发菌株,构建得到融合表达FloA-Fni、FloA-IspA、FloA-HepT和FloA-YuxO的重组菌株BS3AF、BS3AI、BS3AT和BS3AO。分别以过表达Fni、IspA、HepT和YuxO的重组菌株BS3F、BS3I、BS3T和BS3O作为对照,验证MK-7合成途径关键酶区室化对MK-7产量的影响。结果发现,除过表达IspA外,重组工程菌的产量与对照相比都得到了明显提高(图 7B–7I)。其中,BS3AT的MK-7产量最高,达到了300.3 mg/L,较对照菌株提高了73.4%。
|
| 图 7 MK-7合成途径的区室化 Fig. 7 Compartmentalization of MK-7 synthesis. A: Assembly of pathway enzymes via FloA. B: MK-7 titer of BS3AF and BS3F. C: MK-7 titer of BS3AI and BS3I. D: MK-7 titer of BS3AT and BS3T. E: MK-7 titer of BS3AO and BS3O. F: OD600 of BS3AF and BS3F. G: OD600 of BS3AI and BS3I. H: OD600 of BS3AT and BS3T. I: OD600 of BS3AO and BS3O. A:MK-7合成途径关键酶区室化示意图. B:BS3AF和BS3F的MK-7产量. C:BS3AI和BS3I的MK-7产量. D:BS3AT和BS3T的MK-7产量. E:BS3AO和BS3O的MK-7产量. F:BS3AF和BS3F的细胞生长情况. G:BS3AI和BS3I的细胞生长情况. H:BS3AT和BS3T的细胞生长情况. I:BS3AO和BS3O的细胞生长情况 |
| |
重组菌株BS3AT通过分批培养的方式在3 L中发酵罐中进行进一步的发酵验证。初始葡萄糖浓度为50 g/L,通过50%氨水维持pH在7左右。OD600在2 d后达到峰值,随着发酵时间的延长,MK-7的增长速率随着细胞裂解而减缓,MK-7产量在发酵6 d后达到了464.2 mg/L (图 8)。在3 L发酵罐和摇瓶中MK-7产量的变化整体趋势一致。该结论进一步证明了通过FMMs实现MK-7合成途径关键酶区室化的有效性。

|
| 图 8 BS3AT在3 L发酵罐中的发酵结果 Fig. 8 Fermentation of BS3AT in a 3 L bioreactor. A: MK-7 titer of BS3AT. B: Trend of glucose and OD600 of BS3AT. A:BS3AT的MK-7产量. B:BS3AT中葡萄糖和OD600的变化情况 |
| |
MK-7作为脂溶性维生素K2的重要亚型,在功能食品等领域有着广泛应用。因此,通过微生物发酵法实现MK-7的高效合成有着重要意义。在模式微生物枯草芽孢杆菌中,许多研究者通过增强前体供应、阻断副产物合成等方法对MK-7的代谢途径进行优化,期望实现MK-7的高效合成[30-31]。相比于其他改造策略,区室化可以使目标代谢通路的酶集中于一个区域,缩短途径酶之间的空间距离,加快代谢反应速率,进而实现目标产物的高效合成[32-34]。因此,通过细胞膜上特殊的结构域FMMs实现枯草芽孢杆菌中MK-7合成途径的区室化可以为MK-7的代谢改造提供一种新的思路。
本研究首先探讨了MK-7和FMMs的相关性。通过可视化分析发现,MK-7和FMMs均以不连续的点状结构分布于细胞膜上,初步推测二者存在相关性。其次FMMs的完整性被破坏导致了细胞膜上MK-7含量和膜有序度的下降。但是在FMMs的完整性被破坏后,MK-7仍可以有效提高膜有序度,证明MK-7是FMMs聚异戊二烯类的关键组分。在此基础上,尝试通过FMMs来实现MK-7的高效合成。
在MK-7合成过程中,关键酶MenH、HepS和MenB定位于FMMs中,Fni、IspA、HepT和YuxO游离在细胞质中,MenA则是典型的细胞膜定位。MK-7合成相关的关键酶在细胞中的空间位置相对较远,这可能会导致MK-7的合成效率较低。因此,接下来通过FloA蛋白分别融合表达关键酶Fni、IspA、HepT和YuxO,使得关键酶定位在FMMs中,缩短关键酶之间的空间距离,实现MK-7合成途径相关关键酶的区室化。通过FloA蛋白将MK-7合成途径关键酶HepT锚定到FMMs后,成功获得一株高产MK-7菌株BS3AT,在摇瓶水平MK-7产量达到300.3 mg/L,相比于对照菌株提高了73.4%,在3 L生物反应器中的产量为464.2 mg/L。
综上所述,基于FMMs的MK-7合成途径关键酶区室化,有效提高了MK-7的产量,为构建高效合成MK-7的微生物细胞工厂提供了新思路。
| [1] |
GRÖBER U, REICHRATH J, HOLICK MF, KISTERS K. Vitamin K: an old vitamin in a new perspective. Dermato-Endocrinology, 2014, 6(1): e968490. DOI:10.4161/19381972.2014.968490
|
| [2] |
BEULENS JWJ, BOOTH SL, van den HEUVEL EGHM, STOECKLIN E, BAKA A, VERMEER C. The role of menaquinones (vitamin K2) in human health. British Journal of Nutrition, 2013, 110(8): 1357-1368. DOI:10.1017/S0007114513001013
|
| [3] |
DALMEIJER GW, van den SCHOUW YT, MAGDELEYNS E, AHMED N, VERMEER C, BEULENS JWJ. The effect of menaquinone-7 supplementation on circulating species of matrix Gla protein. Atherosclerosis, 2012, 225(2): 397-402. DOI:10.1016/j.atherosclerosis.2012.09.019
|
| [4] |
PAINTER KL, HALL A, HA KP, EDWARDS AM. The electron transport chain sensitizes Staphylococcus aureus and Enterococcus faecalis to the oxidative burst. Infection and Immunity, 2017, 85(12): e00659-17.
|
| [5] |
YANG SM, CAO YX, SUN LM, LI CF, LIN X, CAI ZG, ZHANG GY, SONG H. Modular pathway engineering of Bacillus subtilis to promote de novo biosynthesis of menaquinone-7. ACS Synthetic Biology, 2019, 8(1): 70-81. DOI:10.1021/acssynbio.8b00258
|
| [6] |
MEGANATHAN R. Biosynthesis of menaquinone (vitamin K2) and ubiquinone (coenzyme Q): a perspective on enzymatic mechanisms. Vitamins & Hormones, 2001, 61: 173-218.
|
| [7] |
JOHNSTON JM, BULLOCH EM. Advances in menaquinone biosynthesis: sublocalisation and allosteric regulation. Current Opinion in Structural Biology, 2020, 65: 33-41. DOI:10.1016/j.sbi.2020.05.005
|
| [8] |
HIROTA Y, NAKAGAWA K, SAWADA N, OKUDA N, SUHARA Y, UCHINO Y, KIMOTO T, FUNAHASHI N, KAMAO MY, TSUGAWA N, OKANO T. Functional characterization of the vitamin K2 biosynthetic enzyme UBIAD1. Public Library of Science, 2015, 10(4): e0125737.
|
| [9] |
LIU Y, YANG ZM, XUE ZL, QIAN SH, WANG Z, HU LX, WANG J, ZHU H, DING XM, YU F. Influence of site-directed mutagenesis of UbiA, overexpression of dxr, menA and ubiE, and supplementation with precursors on menaquinone production in Elizabethkingia meningoseptica. Process Biochemistry, 2018, 68: 64-72. DOI:10.1016/j.procbio.2018.01.022
|
| [10] |
崔世修. 代谢工程改造枯草芽孢杆菌高效合成七烯甲萘醌[D]. 无锡: 江南大学博士学位论文, 2020. CUI SX. Metabolic engineering of Bacillus subtilis for efficient synthesis of menaquinone-7[D]. Wuxi: Doctoral Dissertation of Jiangnan University, 2020 (in Chinese). |
| [11] |
MA YW, MCCLURE DD, SOMERVILLE MV, PROSCHOGO NW, DEHGHANI F, KAVANAGH JM, COLEMAN NV. Metabolic engineering of the MEP pathway in Bacillus subtilis for increased biosynthesis of menaquinone-7. ACS Synthetic Biology, 2019, 8(7): 1620-1630. DOI:10.1021/acssynbio.9b00077
|
| [12] |
CUI SX, LV XQ, WU YK, LI JH, DU GC, LEDESMA-AMARO R, LIU L. Engineering a bifunctional Phr60-Rap60-Spo0A quorum-sensing molecular switch for dynamic fine-tuning of menaquinone-7 synthesis in Bacillus subtilis. ACS Synthetic Biology, 2019, 8(8): 1826-1837. DOI:10.1021/acssynbio.9b00140
|
| [13] |
FARHI M, MARHEVKA E, MASCI T, MARCOS E, EYAL Y, OVADIS M, ABELIOVICH H, VAINSTEIN A. Harnessing yeast subcellular compartments for the production of plant terpenoids. Metabolic Engineering, 2011, 13(5): 474-481. DOI:10.1016/j.ymben.2011.05.001
|
| [14] |
LAUDE AJ, PRIOR IA. Plasma membrane microdomains: organization, function and trafficking. Molecular Membrane Biology, 2004, 21(3): 193-205. DOI:10.1080/09687680410001700517
|
| [15] |
YEE DA, DENICOLA AB, BILLINGSLEY JM, CRESO JG, SUBRAHMANYAM V, TANG Y. Engineered mitochondrial production of monoterpenes in Saccharomyces cerevisiae. Metabolic Engineering, 2019, 55: 76-84. DOI:10.1016/j.ymben.2019.06.004
|
| [16] |
SHENG JY, STEVENS J, FENG XY. Pathway compartmentalization in peroxisome of Saccharomyces cerevisiae to produce versatile medium chain fatty alcohols. Scientific Reports, 2016, 6: 26884. DOI:10.1038/srep26884
|
| [17] |
DELOACHE WC, RUSS ZN, DUEBER JE. Towards repurposing the yeast peroxisome for compartmentalizing heterologous metabolic pathways. Nature Communications, 2016, 7: 11152. DOI:10.1038/ncomms11152
|
| [18] |
AVALOS JL, FINK GR, STEPHANOPOULOS G. Compartmentalization of metabolic pathways in yeast mitochondria improves the production of branched-chain alcohols. Nature Biotechnology, 2013, 31(4): 335-341. DOI:10.1038/nbt.2509
|
| [19] |
BRAMKAMP M, LOPEZ D. Exploring the existence of lipid rafts in bacteria. Microbiology and Molecular Biology Reviews, 2015, 79(1): 81-100. DOI:10.1128/MMBR.00036-14
|
| [20] |
PIKE LJ. Lipid rafts: bringing order to chaos. Journal of Lipid Research, 2003, 44(4): 655-667. DOI:10.1194/jlr.R200021-JLR200
|
| [21] |
OTTO GP, NICHOLS BJ. The roles of flotillin microdomains-endocytosis and beyond. Journal of Cell Science, 2011, 124(23): 3933-3940. DOI:10.1242/jcs.092015
|
| [22] |
STUERMER CAO. Reggie/flotillin and the targeted delivery of cargo. Journal of Neurochemistry, 2011, 116(5): 708-713. DOI:10.1111/j.1471-4159.2010.07007.x
|
| [23] |
LV XQ, ZHANG C, CUI SX, XU XH, WANG LL, LI JH, DU GC, CHEN J, LEDESMA-AMARO R, LIU L. Assembly of pathway enzymes by engineering functional membrane microdomain components for improved N-acetylglucosamine synthesis in Bacillus subtilis. Metabolic Engineering, 2020, 61: 96-105. DOI:10.1016/j.ymben.2020.05.011
|
| [24] |
OWEN DM, MAGENAU A, WILLIAMSON D, GAUS K. The lipid raft hypothesis revisited-new insights on raft composition and function from super-resolution fluorescence microscopy. BioEssays, 2012, 34(9): 739-747. DOI:10.1002/bies.201200044
|
| [25] |
LANGHORST MF, REUTER A, STUERMER CAO. Scaffolding microdomains and beyond: the function of reggie/flotillin proteins. Cellular and Molecular Life Sciences CMLS, 2005, 62(19/20): 2228-2240.
|
| [26] |
CHAZOTTE B. Labeling mitochondria with rhodamine 123. Cold Spring Harbor Protocols, 2011, 2011(7): pdb.prot5640.
|
| [27] |
FENG XX, HU YM, ZHENG YY, ZHU W, LI K, HUANG CH, KO TP, REN FF, CHAN HC, NEGA M, BOGUE S, LÓPEZ D, KOLTER R, GÖTZ F, GUO RT, OLDFIELD E. Structural and functional analysis of Bacillus subtilis YisP reveals a role of its product in biofilm production. Chemistry & Biology, 2014, 21(11): 1557-1563.
|
| [28] |
BOSAK T, LOSICK RM, PEARSON A. A polycyclic terpenoid that alleviates oxidative stress. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(18): 6725-6729. DOI:10.1073/pnas.0800199105
|
| [29] |
DINIC J, BIVERSTÅHL H, MÄLER L, PARMRYD I. Laurdan and di-4-ANEPPDHQ do not respond to membrane-inserted peptides and are good probes for lipid packing. Biochimica et Biophysica Acta (BBA)-Biomembranes, 2011, 1808(1): 298-306.
|
| [30] |
JIN L, LI LH, ZHANG WC, ZHANG RZ, XU Y. Heterologous expression of bovine lactoferrin C-lobe in Bacillus subtilis and comparison of its antibacterial activity with N-lobe. Systems Microbiology and Biomanufacturing, 2022, 2(2): 345-354. DOI:10.1007/s43393-021-00066-4
|
| [31] |
WANG Q, FU WL, LU RQ, PAN CL, YI GF, ZHANG X, RAO ZM. Characterization of Bacillus subtilis Ab03 for efficient ammonia nitrogen removal. Systems Microbiology and Biomanufacturing, 2022, 2(3): 580-588. DOI:10.1007/s43393-022-00088-6
|
| [32] |
ZHU Y, LIU JQ, PARK J, RAI P, ZHAI RG. Subcellular compartmentalization of NAD+ and its role in cancer: a sereNADe of metabolic melodies. Pharmacology & Therapeutics, 2019, 200: 27-41. DOI:10.12092/j.issn.1009-2501.2019.01.005
|
| [33] |
MA T, SHI B, YE ZL, LI XW, LIU M, CHEN Y, XIA J, NIELSEN J, DENG ZX, LIU TG. Lipid engineering combined with systematic metabolic engineering of Saccharomyces cerevisiae for high-yield production of lycopene. Metabolic Engineering, 2019, 52: 134-142. DOI:10.1016/j.ymben.2018.11.009
|
| [34] |
LV XM, WANG F, ZHOU PP, YE LD, XIE WP, XU HM, YU HW. Dual regulation of cytoplasmic and mitochondrial acetyl-CoA utilization for improved isoprene production in Saccharomyces cerevisiae. Nature Communications, 2016, 7: 12851. DOI:10.1038/ncomms12851
|
 2023, Vol. 39
2023, Vol. 39





