中国科学院微生物研究所、中国微生物学会主办
文章信息
- 王辰, 徐玉泉
- Wang Chen, Xu Yuquan
- 非核糖体肽合成酶工程改造研究进展
- Advances in engineering non-ribosomal peptide synthetase
- 生物工程学报, 2021, 37(6): 1845-1857
- Chinese Journal of Biotechnology, 2021, 37(6): 1845-1857
- 10.13345/j.cjb.200736
-
文章历史
- Received: November 20, 2020
- Accepted: March 3, 2021
- Published: April 30, 2021
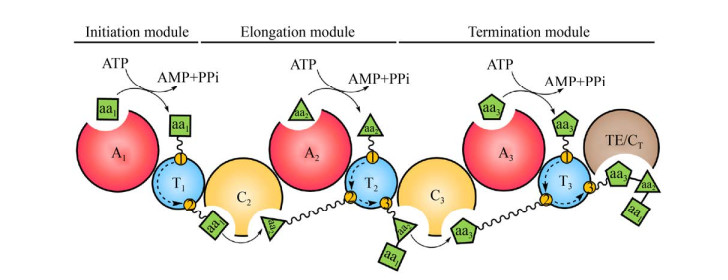
微生物来源的非核糖体肽(Nonribosomal peptide) 类天然产物是临床抗感染、抗肿瘤以及免疫抑制类药物的重要来源,如万古霉素、棘白菌素、达托霉素和环孢霉素等。非核糖体肽的核心骨架由模块化的非核糖体肽合成酶(Nonribosomal peptide synthetase,NRPS) 组装合成。一个典型的NRPS由多个模块构成,每个模块包含3个核心功能结构域,分别为:识别和活化氨基酸装配单元的腺苷化(Adenylation,A) 结构域;运载氨基酸和新生肽链的硫酯化(Thiolation,T) 结构域;以及催化肽键形成的缩合(Condensation,C) 结构域(图 1)。除了C-A-T三联结构域外,NRPS模块还可能选择性地包含一些修饰结构域,如甲基化(Methylation,MT) 结构域、氧化(Oxidation,Ox) 结构域、杂环化(Heterocyclization,Cy) 结构域、异构化(Epimerization,E) 结构域等。NRPS的C末端通常含有一个硫酯酶(Thioesterase,TE) 或末端缩合(Terminal condensation,CT) 结构域,负责终产物的释放。绝大多数NRPS遵循共线性装配规则,即NRPS模块的数目、种类和排列顺序与其产物结构中氨基酸组成单元的数目、种类和排列顺序一致。在生物合成过程中,NRPS按照固定的逻辑逐个行使各个模块的催化功能将氨基酸单体顺序装配成特定结构的非核糖体肽(图 1)。此外,一些NRPS还具有特殊的装配机制,产物合成过程中存在模块跳跃、迭代延伸和反式上载等现象。
|
| 图 1 NRPS的结构组成和生物合成机制示意图(氨基酸底物(aan) 经腺苷化结构域(An) 识别和活化后转移至硫酯化结构域(Tn) 的4′-磷酸泛酰巯基乙胺连接臂(波浪线) 上;随后两分子氨基酸在缩合结构域(Cn) 催化下形成肽键,经过多轮延伸后肽链在硫酯酶或末端缩合结构域(TE/CT) 催化下从NRPS上释放) Fig. 1 The structure of NRPS and the mechanism for catalyzing biosynthesis. The amino acid substrates (aan) are selected and activated by the adenylation domain (An) and then transferred onto the 4′-phosphopantetheine arm (wavy line) of the thiolation domain (Tn). The condensation domain (Cn) catalyzes the peptide bond formation between the two amino acid molecules. After several rounds of extension process, the mature peptide is released from the NRPS by the thioesterase or terminal condensation domain (TE/CT). |
| |
在漫长的进化过程中,微生物通过排列组合功能多样的结构域形成了成千上万种NRPS,从而造就了非核糖体肽天然产物丰富的结构和多样的功能。同时,NRPS模块化的结构特点为人工设计和工程改造非核糖体肽装配线、组合生物合成可用于药物筛选的新骨架非核糖体肽提供了可能。随着高通量基因测序、基因合成以及多基因装配等合成生物技术的快速发展,研究者在NRPS工程改造领域不断取得突破。本文主要针对不同类型的NRPS工程改造策略,如腺苷化结构域特异性调整、多结构域交换以及对接结构域改造等,对近年来该领域的一些重要研究进展进行综述。
1 腺苷化结构域特异性调整在制定NRPS工程改造策略时,研究者首先想到的是改造识别和活化氨基酸的腺苷化结构域。Marahiel团队通过异源交换芽孢杆菌表面活性素合成酶(Surfactin synthetase A,SrfA) 的腺苷化结构域,首次证明重组SrfA能够合成具有溶血活性的表面活性素类似物[1]。不过,这些重组酶与天然SrfA相比合成催化效率显著下降,说明引入异源腺苷化结构域可能影响NRPS的整体结构或者C-A、A-T结构域之间的相互作用[2]。为了减轻异源大片段交换引起的负面效应,研究者尝试仅对负责底物结合的亚结构域进行替换。虽然生化实验证明替换这些片段能够成功改变腺苷化结构域的特异性,但是重组腺苷化结构域能否和其他催化结构域协作合成完整的产物并未得到充分的验证[3-4]。随后,Marahiel和Townsend团队根据短杆菌肽S合成酶起始模块腺苷化结构域(A domain of gramicidin S synthetase A,GrsA-A) 的晶体结构[5] (图 2A) 预测了腺苷化结构域特异性识别氨基酸底物的10个位点(图 2B),并称之为“特异性代码” (Specificity-conferring code)[6-7]。人们普遍认为,调整“特异性代码”能够在改变腺苷化结构域选择性的同时最大程度地维持NRPS的结构完整性,从而避免替换整个结构域所引起的问题。Marahiel团队运用这一策略改造了GrsA-A以及SrfA模块1和模块5的腺苷化结构域(A1 and A5 domains of SrfA,SrfA-A1和SrfA-A5),体外生化实验显示重组腺苷化结构域的特异性发生了显著的松弛或改变[6-8]。达托霉素、钙依赖抗生素(Calcium-dependent antibiotic,CDA) 和A54145是链霉菌产生的一类环脂肽抗生素,三者的化学结构和生物合成基因簇十分相似。Mickfield团队对CDA合成酶A7和A10结构域进行氨基酸定点突变,成功改变了二者对底物的特异性,并在表达重组CDA合成酶的天蓝色链霉菌中检测到新型CDA类似物[9-10]。
与通过氨基酸序列比对选择性地替换“特异性代码”相比,基于“特异性代码”饱和突变的定向进化策略能够更大程度地探索腺苷化结构域催化功能的可塑性。在andrimid生物合成中,AdmK模块负责识别和装载缬氨酸(Val)。Evans等对AdmK腺苷化结构域的3个“特异性代码”色氨酸(Trp) 240、异亮氨酸(Ile) 265和甘氨酸(Gly) 291进行饱和突变,共构建1 404种重组AdmK。利用液相色谱-质谱联用仪(Liquid chromatography-tandem mass,LC-MS/MS) 筛选14 000多个转化子后发现4个突变株能够合成新型andrimid类似物,可以将andrimid的Val替换成丙氨酸(Ala)、亮氨酸(Leu)、Ile或苯丙氨酸(Phe) (Val→Ala/Leu/Ile/Phe)[11]。Hollfelder团队运用相似的策略对短杆菌酪肽合成酶起始模块腺苷化结构域(A domain of tyrocidine synthetase A,TycA-A) 进行定向进化,并采用三磷酸腺苷/焦磷酸(Adenosine triphosphate/pyrophosphate,ATP/PPi) 交换实验筛选到一个特异性显著变化的重组TycA-A (Phe→Ala)[12]。在另外一项研究中,Zhang等巧妙地将酵母表面展示技术运用于杆菌素合成酶芳香酸活化结构域(DhbE) 的特异性改造研究中。利用腺苷氨基磺酸探针搭配荧光激活细胞分选技术对上百万个酵母转化子进行高通量筛选,发现部分重组DhbE能够高效识别和活化非天然芳香酸[13]。该方法与LC-MS/MS和ATP/PPi交换方法相比,将筛选通量提高了3个数量级。最近,Tanabe团队根据DhbE和肠杆菌素合成酶芳香酸活化结构域EntE的晶体结构,对EntE进行理性设计,拓展其底物结合腔室的体积,从而容纳具有大体积官能团取代的芳香酸。通过构建4种EntE突变型,并利用孔雀绿比色分析法高通量测试重组EntE的腺苷化活性,发现将天冬酰胺(Asn235) 突变为Gly (Asn235Gly) 能够显著改变EntE的底物选择性,使其活化含有硝基、氰基以及炔基官能团取代的芳香酸。当重组EntE和芳香酸载体蛋白(Aryl acid carrier protein,ArCP) 共同孵育时能够将非天然芳香酸传递给ArCP[14]。
在化学生物学研究中,经常需要向生物活性小分子结构中引入炔基或叠氮官能团,随后借助点击化学反应进行生物正交实验。因此,合成含有炔基或叠氮官能团的非核糖体肽活性分子有助于新型药物靶点的快速发现。Hilvert团队将Trp239突变为丝氨酸(Ser),扩大了GrsA-A的底物结合腔室,使其能够识别和活化含有叠氮或炔基官能团的芳香氨基酸,如p-azido-L-Phe和O-propargyl-L-Tyr等[15]。在近期一项工作中,该团队综合运用定向进化、荧光激活细胞分选和点击化学探针技术高通量筛选上百万个转化子后,发现Ala236Val突变并缩短β13-β14之间的无规卷曲结构可将TycA-A (识别α-Phe) 改造成特异性活化β-Phe的腺苷化结构域(图 3),实现约40 000倍的α/β-Phe特异性转换。令人欣喜的是,由重组TycA-A活化的β-Phe能够被下游延伸模块利用,高产量合成自然界中较为罕见的含有β-氨基酸的非核糖体肽[16]。
上述研究充分验证了腺苷化结构域“特异性代码”在不同NRPS系统中的通用性,同时展示了腺苷化结构域特异性调整在NRPS工程改造研究中的应用潜力。但是,多数研究只是通过体外生化反应验证了重组腺苷化结构域底物选择性的变化[6, 8, 12-15],仅有部分研究在体内构建了含有重组腺苷化结构域的杂合NRPS,并获得了相应的产物。其中最具代表性的成果是针对短杆菌酪肽合成酶起始模块腺苷化结构域的改造,高产率合成了含有β-氨基酸的新型非核糖体肽[16]。另外两个成功案例是改造CDA合成酶延伸模块的腺苷化结构域,但引起的产物结构变化十分保守[9-10]。其他研究表明对NRPS延伸模块腺苷化结构域的特异性进行大幅度改造则会阻碍产物的合成[17-18]。这说明腺苷化结构域并不是非核糖体肽生物合成的唯一门控单元,其他结构域也会根据自身特异性调控氨基酸单体的顺利装配。
2 多结构域替换20世纪末,Walsh等发现短杆菌酪肽和肠杆菌素合成酶的缩合结构域对受体底物(下游硫酯化结构域连接的氨基酸) 的侧链结构和立体构型(D-或L-氨基酸) 具有明显的选择性,但是对供体底物(上游硫酯化结构域连接的氨基酸) 仅表现出立体构型选择性[19-20]。这说明除了腺苷化结构域外,NRPS的缩合结构域也具有严格的底物特异性。Walsh团队的发现很快得到了NRPS研究领域学者的广泛认可,对NRPS工程改造策略的制定产生了重要影响。研究者认为缩合结构域特异性是限制腺苷化结构域改造成功率的主要因素,由于缩合结构域无法处理侧链结构变化显著的受体底物,腺苷化结构域只能容忍小幅度的改造,使其上载侧链结构相似的氨基酸,否则会严重影响NRPS的合成效率。这就解答了为什么改造起始模块腺苷化结构域(缺少上游缩合结构域) 特异性能够获得有功能的杂合NRPS[16],但是改变延伸模块腺苷化结构域(具备上游缩合结构域) 特异性则会使NRPS失活[17-18]。为了避免缩合结构域和腺苷化结构域特异性之间的冲突,研究者开始尝试在维持同源C-A双结构域的前提下以C-A、C-A-T或T-C-A为交换单元进行非核糖体肽装配线改造。Marahiel团队将短杆菌酪肽合成酶的模块2与模块9或模块10组合,构建双模块人工亚单元C-A-TPro-C-A-T-TELeu和C-A-TPro-C-A-T-TEOrn (Pro:脯氨酸;Orn:鸟氨酸)。二者和起始模块共同孵育时能够合成与预测相符的三肽产物,说明NRPS能够在T-C链接处进行拆分,以C-A-T为交换单元进行重组可以获得有功能的杂合NRPS[21]。在一系列针对达托霉素合成酶的研究中,Baltz团队通过替换单个或多个C-A或C-A-T单元,在体内构建了40多个杂合NRPS,共检测到120余种新型达托霉素类似物。其中42种可被分离,部分类似物的抑菌活性和达托霉素相当[22-25]。这些工作为工程改造非核糖体肽装配线组合生物合成重要药物分子类似物提供了优秀范例。但不足的是,多数杂合NRPS的总体产量与野生型达托霉素合成酶相比出现不同程度的下降,另外一些杂合NRPS甚至完全丧失催化功能。
荧光嗜铁素是一类分离自荧光假单胞菌的铁载体类天然产物,其多肽骨架和荧光发色团由4个NRPS亚单元(PvdL、PvdI、PvdJ和PvdD) 协作组装完成。Ackerley团队通过替换腺苷化结构域对PvdD的两个苏氨酸(Thr) 装载模块进行了改造。结果显示这两个模块的缩合结构域均具有鲜明的受体特异性,无法处理异源腺苷化结构域提供的非Thr底物,如Ser、赖氨酸(Lys)、Gly等。随后,该团队尝试替换C-A单元来回避缩合结构域的受体特异性限制。在13组C-A替换实验中,有3个重组PvdD (搭载特异性装配Thr、Ser和Lys的C-A) 能够与其他亚单元协作,合成荧光嗜铁素及其结构类似物[17-18]。虽然替换C-A单元能够获得有功能的杂合荧光嗜铁素合成酶,但是整体成功率只有23%。另外,3个有功能的杂合NRPS与野生型荧光嗜铁素合成酶相比,合成效率有不同程度的下降。该团队怀疑异源硫酯化结构域和缩合结构域的不兼容可能会影响杂合NRPS的功能,但是以T-C-A为交换单元进行替换并不能复苏或显著提升杂合荧光嗜铁素合成酶的合成效率,从而否定了这一推测[26]。从改造达托霉素和荧光嗜铁素合成酶的实例中可以看出,同时替换C-A或C-A-T的确能够回避缩合结构域受体特异性引起的限制,生成有功能的杂合NRPS。但是该策略的整体成功率较低,不能稳定高效合成新骨架非核糖体肽。
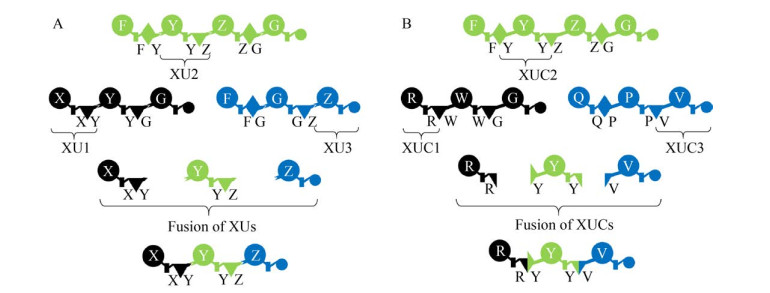
最近,Bode等提出C-A结构域之间的链接区(Linker region) 比A-T和T-C链接区长度更长并且更加具有柔性,因此更适合作为融合位点进行结构域交换[27]。据此,研究者在C-A链接区处拆分10余种NRPS,获得了一系列A-T-C交换单元(Exchange unit,XU)。通过替换或重组不同的XU构建杂合NRPS,合成了一系列新型非核糖体肽(图 4A)[27]。尽管一些研究建议在NRPS改造过程中保持完整的C-A及相应链接区[28],但是Bode团队通过系统性的实验证实了C-A链接区融合位点的有效性,并展示了A-T-C XU策略在构建杂合NRPS时的优势。但是,该策略严格受限于缩合结构域的受体特异性,选择下游XU时必须充分考虑上游缩合结构域的受体特异性。因此,想要任意组合20种常规氨基酸定制新型非核糖体肽需要鉴定和标准化800种A-T-C XU元件。这个特点在一定程度上限制了A-T-C交换策略的实用性。典型的NRPS缩合结构域是由N端和C端两个亚结构域构成的假二聚体,催化中心位于两个亚结构域的交界处[29-31]。在催化缩合反应过程中,N端的亚结构域负责结合供体底物(N-terminalsub-domain of C domain,CDsub),C端的亚结构域则负责结合受体底物(C-terminal sub-domain of C domain,CAsub)。Bode等认为,连接CDsub和CAsub的无规卷曲结构适合作为融合位点进行缩合结构域拆分和重组,并提出在A-T双结构域基础上加入上游CAsub和下游CDsub,形成由CAsub-A-T-CDsub构成的缩合交换单元(Exchange unit condensation domain,XUC)[32]。由于包含同源CAsub和腺苷化结构域,利用XUC构建杂合NRPS将不受缩合结构域受体特异性的限制,从而显著提升工程改造操作的灵活性(图 4B)。与A-T-C XU相比,XUC策略可以大幅度减少定制新型非核糖体肽所需的交换元件种类(80种)。研究者首先在模式系统GameXPeptide合成酶(GameXPeptide synthetase,GxpS) 中验证了XUC的有效性,随后利用10组来源不同的XUC构建了多种杂合NRPS,合成了一系列新型非核糖体肽。利用底物特异性松弛的XUC构建杂合NRPS,并饲喂含有特殊官能团的氨基酸底物可以合成含有非天然氨基酸的产物。最后,以9种来源和功能不同的XUC为元件,利用转化介导的重组克隆技术(Transformation-associated recombination) 对GxpS的1–2个特定位点进行随机替换,可以同步构建一系列杂合GxpS来合成结构多样的非核糖体肽文库,从而加速生物活性多肽的发现过程[32]。XUC策略解决了缩合结构域特异性引起的上、下游交换单元底物适配问题,为从头设计NRPS和定制新骨架非核糖体肽提供了更优方案。但是,硫酯酶结构域或末端缩合结构域的特异性可能会影响重组产物的释放效率和释放形式。挖掘和改造杂泛性强的硫酯酶结构域和末端缩合结构域能够进一步增强XUC策略的实用性。
NRPS工程改造的另一研究热点在于对接结构域(Docking domain,DD),也称为通讯结构域(Communication-mediating domain,COM domain) 的改造。原核生物非核糖体肽装配线通常由多个独立的NRPS亚单元构成,各个亚单元通过特异性的相互作用进行有序、高效的协作,完成终产物的正确组装。21世纪初期,Stachelhaus团队率先揭示了NRPS亚单元之间相互选择性识别、高效协作的结构基础[33]。研究者发现短杆菌酪肽合成酶亚单元(TycA、TycB和TycC) 的N和C末端具有由15–25个氨基酸组成的对接结构域(NDD和CDD;原文称之为通讯结构域)。通过NDD和CDD之间特异性的相互作用,伴侣亚单元(如TycA-TycB和TycB-TycC) 才能够准确地相互识别,实现氨基酸或新生肽链的顺利传递。破坏N/CDD的适配性会阻碍伴侣亚单元之间的通讯;相反,引入适配的N/CDD则能够重建非伴侣亚单元(如TycA-TycC) 之间的相互作用。生物信息学分析显示,与短杆菌酪肽合成酶对接结构域相似的N/CDD在细菌NRPS系统中广泛存在。而且,来源不同但氨基酸序列高度一致的N/CDD (如TycB-、GrsB-和SrfAC-NDD) 具有通用性,因此相应的异源亚单元(如TycA-GrsB和TycA-SrfAC) 不需要任何改造即可进行高效协作。随后,该团队通过氨基酸序列比对预测了这些N/CDD中维持特异性相互作用的关键氨基酸,并通过实验证明单位点氨基酸突变(如Lys9Glu) 即可改变TycB-NDD的选择性。另外,Stachelhaus团队还提出了通用对接系统(Universal communication system,UCS) 概念,即多个NRPS亚单元使用同一对N/CDD,使各个亚单元不受对接结构域选择性的约束进行随机配对,非特异性地合成链长和氨基酸序列各不相同的非核糖体肽[34]。上述关于对接结构域的开创性工作为工程改造NRPS提供了新的思路。研究者利用对接结构域删除/替换、关键氨基酸突变以及UCS等手段分别改造了表面活性素和制磷脂菌素装配线,并在相应的枯草芽孢杆菌突变株中检测到了链长改变的产物,进一步在体内验证了对接结构域改造策略的实用性[35-36]。
实际上,微生物在漫长的进化过程中已经发展了类似UCS的组合生物合成策略来丰富自身非核糖体肽的化学多样性。致病杆菌rhabdopeptide/xenortide (RXP) 合成酶Kj12ABC由两个单模块NRPS亚单元(Kj12A和Kj12B) 和一个独立行使末端缩合结构域功能的酶(Kj12C) 组成(图 5A)。在RXP合成过程中,Kj12B能够重复地行使催化功能,最终合成一系列链长为3–6个氨基酸的同系物[37]。在研究RXP合成酶的过程中,Bode和Wohnert团队发现Kj12A、Kj12B和Kj12C的N末端均具有约65个氨基酸、序列高度一致的对接结构域(图 5A)。结构生物学实验显示这些NDD由3个α-螺旋和两个β-折叠组成(α1-α2-β1-β2-α3),其中反向平行的β1和β2形成典型的发卡结构(图 5B)。另外,Kj12A和Kj12B还包含约20个氨基酸的CDD,其二级结构由一个α-螺旋和β-折叠(α4-β3) 构成。Kj12ABC-N/CDD的序列长度、氨基酸组成和二级结构与已报道的对接结构域相比存在明显差异,是一类新型的NRPS对接结构域[38]。Kj12A-、Kj12B-和Kj12C-NDD结构的高度相似性(图 5B) 赋予它们类似UCS的属性:3种NDD均能与Kj12B-CDD配对,因此Kj12B可以和包括自身在内的所有亚单元协作,从而使得Kj12B重复利用。随后,研究者巧妙地利用柔性甘氨酸-丝氨酸链接区串联NDD和CDD,促进二者进行相互作用,从而通过核磁共振波谱首次解析了N/CDD复合物的高分辨三维结构。由此可知Kj12C-NDD和Kj12B-CDD之间的相互作用主要依靠α2/β2与β3上氨基酸侧链之间的静电和疏水效应来维持(图 5C)。有目的性地替换这些氨基酸能够影响N/CDD之间的结合稳定性,从而调节Kj12ABC亚单元之间的相互作用,最终改变RXP终产物的链长[38]。通过BLAST搜索发现,Kj12ABC-N/CDD类似物广泛存在于假单胞菌、紫色杆菌、蓝细菌、粘细菌等原核微生物的NRPS和聚酮合酶/非核糖体肽合成酶系统中。最近,Bode和Wohnert团队再次合作,在另一种致病杆菌NRPS (PaxABC) 中发现了一类新型对接结构域。与Kj12ABC不同,PaxABC遵循共线性原则,3个亚单元按照固定的顺序进行相互作用,最终合成结构单一的致病杆菌抗菌肽(Peptide- antimicrobial-Xenorhabdus,PAX)。PaxB和PaxC的NDD以及PaxA和PaxB的CDD具有较低的序列一致性,说明两组N/CDD具有不同的结构和对接形式,从而提升相应NRPS亚单元之间的互作选择性。其中,PaxB-CDD和PaxC-NDD分别由一条连续的长α-螺旋和两个呈V型的短α-螺旋构成,二者的相互作用主要由氨基酸侧链之间的疏水作用维持[39]。
基于上述工作基础,Bode团队以不同来源的对接结构域为工具,对两种NRPS系统进行了工程改造。用源于XabABC和PaxABC的特异性N/CDD替换Kj12ABC的非特异性N/CDD,可以调节3个亚单元之间的相互作用,较为专一地合成特定结构的非核糖体肽[40]。Kegler等对致病杆菌xefoampeptide合成酶(Xefoampeptide synthetase, XfpS) 进行改造时,根据XfpS的模块构成将其划分为C-A-T-E (XfpS-1)、C-A-T (XfpS-2) 和C-A-T-TE (XfpS-3) 三部分。依照该片段划分方式对XfpS进行单位点和双位点拆分,并在相应“人工”亚单元N和(或) C末端引入对接结构域(图 6)。结果显示,在XfpS-1和XfpS-2的E-C结构域链接处拆分并引入对接结构域后,XfpS-1和XfpS23依然能够高效合成天然xefoampeptide。但是,在XfpS-2和XfpS-3的T-C结构域链接处拆分则会显著影响NRPS (XfpS-12和XfpS3) 的合成效率。完全拆分XfpS并引入两组对接结构域后,xefoampeptide产量会进一步下降(图 6)[41]。这两项工作充分展现了对接结构域在不同NRPS工程改造场景中的应用潜力:前者以限制NRPS的泛杂性为目的,通过对接结构域替换将迭代型Kj12ABC改造成共线性的NRPS,从而获取特定结构的非核糖体肽;后者以拆分大型多模块、单蛋白NRPS为目标,通过引入对接结构域促进“人工”亚单元之间的相互作用,使拆分后的NRPS系统仍然能够合成天然非核糖体肽。
上述前沿研究明确了对接结构域广泛存在于各种NRPS系统中,揭示了代表性N/CDD复合物的三维结构及相互作用方式,并初步探索了对接结构域改造在NRPS系统中的通用性。但是,目前已鉴定的NRPS对接结构域种类有限,且多数来源于芽孢杆菌和致病杆菌,它们能否有效应用于进化关系较远物种(如链霉菌、丝状真菌等) 的NRPS系统中还尚未可知。另外,绝大多数天然NRPS经过对接结构域改造后非核糖体肽产量会显著下降,这说明对接结构域的适配性并不是决定NRPS亚单元协作效率的唯一因素。因此,未来利用对接结构域拆分或重组非核糖体肽装配线时,应当考虑与N/CDD直接相连催化结构域的结构对亚单元相互作用的影响,谨慎选择N/CDD和该结构域的融合位点;同时,还应该考虑重组NRPS的下游亚单元(尤其是缩合结构域和硫酯酶结构域) 能否接受和处理上游亚单元提供的多肽中间体,必要时可以结合其他NRPS工程改造手段。总之,基于对接结构域的NRPS工程改造研究才刚刚起步,尚且存在许多关键问题亟待解决。但是,该方法无疑具有广阔的应用前景,是未来利用合成生物技术理性设计和构建新型非核糖体肽装配线的有效手段之一。
4 总结与展望青霉素的发现和应用开启了人类借助抗生素治疗感染性疾病的新时代,随即推动了头孢菌素、达托霉素、万古霉素等其他微生物源非核糖体肽类药物的发展。通过研究这些重要非核糖体肽的生物合成机制,研究者深入解析了NRPS的结构和功能,并尝试在大肠杆菌、酵母等模式微生物中重构以NRPS为核心的非核糖体肽生物合成途径,从而简化生产非核糖体肽药物所需的菌株发酵和产物纯化过程[42-45]。但是,这些工程菌株往往需要进一步优化来提高非核糖体肽药物的产量。另外,在明确NRPS模块化的结构特点和各个结构域的催化功能后,研究者能够有目的地改造NRPS,合成新型非核糖体肽用于药物筛选。
本文针对腺苷化结构域特异性调整、多结构域替换和对接结构域改造这3种NRPS工程改造的通用策略,在回顾一些经典文献的同时介绍近年来这些研究方向上取得的进展。值得一提的是,包括模块/结构域插入[46]、模块/结构域删除[47]和硫酯化结构域替换[48]在内的其他策略也被成功应用于NRPS工程改造研究中。但是,这些方法仅在个别NRPS系统中验证有效,由于缺乏通用性而没有在本文中详细介绍。经过20多年的发展,NRPS工程改造研究从最初的非理性逐渐走向理性设计和改造的方向,甚至开始尝试从头定制非核糖体肽装配线,而这些进步离不开NRPS结构生物学研究提供的指导。例如,GrsA-A晶体结构的解析[5]帮助研究者鉴定了腺苷化结构域的“特异性代码”,推进了腺苷化结构域特异性调整研究的开展;缩合结构域假二聚形式的发现[29-31]使拆分和重组CDsub-CAsub成为可能,促进了CAsub-A-T-CDsub缩合交换单元概念的产生;对接结构域复合结构的鉴定[38-39]揭示了N/CDD相互作用的分子基础,为对接结构域改造指明了方向。不可否认的是,这20多年的NRPS工程改造研究取得了令人欣喜的成绩,充分展现了其在组合生物合成非核糖体肽中的应用潜力。但是该领域尚有许多关键问题亟待解决,未来的研究工作应该重点关注:1) 如何确保杂合NRPS具备完整功能;2) 如何提升杂合NRPS的合成效率;3) 如何以理想的结构形式释放非核糖体肽终产物。随着结构生物学研究技术的持续进步,一些多结构域、多模块NRPS的高分辨三维结构先后被解析,揭示了NRPS功能结构域在非核糖体肽组装过程中是高度动态的,结构域之间的相互作用发生在频繁的构象变化过程中[49-53]。另外,一些酶-底物复合结构的解析真实地反映了多肽中间体在酶催化腔室内的结合方式以及酶在底物结合后发生的构象变化[53-54]。这些重要发现有助于研究者深入探索功能结构域之间的相互作用以及缩合结构域和硫酯酶结构域底物特异性的分子机制,为NRPS工程改造新策略的制定提供思路。
| [1] |
Stachelhaus T, Schneider A, Marahiel MA. Rational design of peptide antibiotics by targeted replacement of bacterial and fungal domains. Science, 1995, 269(5220): 69-72. DOI:10.1126/science.7604280
|
| [2] |
Schneider A, Stachelhaus T, Marahiel MA. Targeted alteration of the substrate specificity of peptide synthetases by rational module swapping. Mol Gen Genet, 1998, 257(3): 308-318. DOI:10.1007/s004380050652
|
| [3] |
Crüsemann M, Kohlhaas C, Piel J. Evolution-guided engineering of nonribosomal peptide synthetase adenylation domains. Chem Sci, 2013, 4(3): 1041-1045. DOI:10.1039/C2SC21722H
|
| [4] |
Kries H, Niquille DL, Hilvert D. A subdomain swap strategy for reengineering nonribosomal peptides. Chem Biol, 2015, 22(5): 640-648. DOI:10.1016/j.chembiol.2015.04.015
|
| [5] |
Conti E, Stachelhaus T, Marahiel MA, et al. Structural basis for the activation of phenylalanine in the non-ribosomal biosynthesis of gramicidin S. EMBO J, 1997, 16(14): 4174-4183. DOI:10.1093/emboj/16.14.4174
|
| [6] |
Stachelhaus T, Mootz HD, Marahiel MA. The specificity-conferring code of adenylation domains in nonribosomal peptide synthetases. Chem Biol, 1999, 6(8): 493-505. DOI:10.1016/S1074-5521(99)80082-9
|
| [7] |
Challis GL, Ravel J, Townsend CA. Predictive, structure-based model of amino acid recognition by nonribosomal peptide synthetase adenylation domains. Chem Biol, 2000, 7(3): 211-224. DOI:10.1016/S1074-5521(00)00091-0
|
| [8] |
Eppelmann K, Stachelhaus T, Marahiel MA. Exploitation of the selectivity-conferring code of nonribosomal peptide synthetases for the rational design of novel peptide antibiotics. Biochemistry, 2002, 41(30): 9718-9726. DOI:10.1021/bi0259406
|
| [9] |
Uguru GC, Milne C, Borg M, et al. Active-site modifications of adenylation domains lead to hydrolysis of upstream nonribosomal peptidyl thioester intermediates. J Am Chem Soc, 2004, 126(16): 5032-5033. DOI:10.1021/ja048778y
|
| [10] |
Thirlway J, Lewis R, Nunns L, et al. Introduction of a non-natural amino acid into a nonribosomal peptide antibiotic by modification of adenylation domain specificity. Angew Chem Int Ed, 2012, 51(29): 7181-7184. DOI:10.1002/anie.201202043
|
| [11] |
Evans BS, Chen YQ, Metcalf WW, et al. Directed evolution of the nonribosomal peptide synthetase Admk generates new andrimid derivatives in vivo. Chem Biol, 2011, 18(5): 601-607. DOI:10.1016/j.chembiol.2011.03.008
|
| [12] |
Villiers B, Hollfelder F. Directed evolution of a gatekeeper domain in nonribosomal peptide synthesis. Chem Biol, 2011, 18(10): 1290-1299. DOI:10.1016/j.chembiol.2011.06.014
|
| [13] |
Zhang KY, Nelson KM, Bhuripanyo K, et al. Engineering the substrate specificity of the DhbE adenylation domain by yeast cell surface display. Chem Biol, 2013, 20(1): 92-101. DOI:10.1016/j.chembiol.2012.10.020
|
| [14] |
Ishikawa F, Miyanaga A, Kitayama H, et al. An engineered aryl acid adenylation domain with an enlarged substrate binding pocket. Angew Chem Int Ed, 2019, 58(21): 6906-6910. DOI:10.1002/anie.201900318
|
| [15] |
Kries H, Wachtel R, Pabst A, et al. Reprogramming nonribosomal peptide synthetases for "clickable" amino acids. Angew Chem Int Ed, 2014, 53(38): 10105-10108. DOI:10.1002/anie.201405281
|
| [16] |
Niquille DL, Hansen DA, Mori T, et al. Nonribosomal biosynthesis of backbone-modified peptides. Nat Chem, 2018, 10(3): 282-287. DOI:10.1038/nchem.2891
|
| [17] |
Ackerley DF, Lamont LL. Characterization and genetic manipulation of peptide synthetases in Pseudomonas aeruginosa PAO1 in order to generate novel pyoverdines. Chem Biol, 2004, 11(7): 971-980. DOI:10.1016/j.chembiol.2004.04.014
|
| [18] |
Calcott MJ, Owen JG, Lamont IL, et al. Biosynthesis of novel pyoverdines by domain substitution in a nonribosomal peptide synthetase of Pseudomonas aeruginosa. Appl Environ Microbiol, 2014, 80(18): 5723-5731. DOI:10.1128/AEM.01453-14
|
| [19] |
Belshaw PJ, Walsh CT, Stachelhaus T. Aminoacyl-CoAs as probes of condensation domain selectivity in nonribosomal peptide synthesis. Science, 1999, 284(5413): 486-489. DOI:10.1126/science.284.5413.486
|
| [20] |
Ehmann DE, Trauger JW, Stachelhaus T, et al. Aminoacyl-SNACs as small-molecule substrates for the condensation domains of nonribosomal peptide synthetases. Chem Biol, 2000, 7(10): 765-772. DOI:10.1016/S1074-5521(00)00022-3
|
| [21] |
Mootz HD, Schwarzer D, Marahiel MA. Construction of hybrid peptide synthetases by module and domain fusions. Proc Natl Acad Sci USA, 2000, 97(11): 5848-5853. DOI:10.1073/pnas.100075897
|
| [22] |
Nguyen KT, Ritz D, Gu JQ, et al. Combinatorial biosynthesis of novel antibiotics related to daptomycin. Proc Natl Acad Sci USA, 2006, 103(46): 17462-17467. DOI:10.1073/pnas.0608589103
|
| [23] |
Miao V, Coëffet-Le Gal MF, Nguyen K, et al. Genetic engineering in Streptomyces roseosporus to produce hybrid lipopeptide antibiotics. Chem Biol, 2006, 13(3): 269-276. DOI:10.1016/j.chembiol.2005.12.012
|
| [24] |
Doekel S, Coëffet-Le Gal MF, Gu JQ, et al. Non-ribosomal peptide synthetase module fusions to produce derivatives of daptomycin in Streptomyces roseosporus. Microbiology, 2008, 154(9): 2872-2880. DOI:10.1099/mic.0.2008/020685-0
|
| [25] |
Nguyen KT, He XW, Alexander DC, et al. Genetically engineered lipopeptide antibiotics related to A54145 and daptomycin with improved properties. Antimicrob Agents Chemother, 2010, 54(4): 1404-1413. DOI:10.1128/AAC.01307-09
|
| [26] |
Calcott MJ, Ackerley DF. Portability of the thiolation domain in recombinant pyoverdine non-ribosomal peptide synthetases. BMC Microbiol, 2015, 15: 162. DOI:10.1186/s12866-015-0496-3
|
| [27] |
Bozhüyük KAJ, Fleischhacker F, Linck A, et al. De novo design and engineering of non-ribosomal peptide synthetases. Nat Chem, 2018, 10(3): 275-281. DOI:10.1038/nchem.2890
|
| [28] |
Strieker M, Tanović A, Marahiel MA. Nonribosomal peptide synthetases: structures and dynamics. Curr Opin Struct Biol, 2010, 20(2): 234-240. DOI:10.1016/j.sbi.2010.01.009
|
| [29] |
Tanovic A, Samel SA, Essen LO, et al. Crystal structure of the termination module of a nonribosomal peptide synthetase. Science, 2008, 321(5889): 659-663. DOI:10.1126/science.1159850
|
| [30] |
Keating TA, Marshall CG, Walsh CT, et al. The structure of VibH represents nonribosomal peptide synthetase condensation, cyclization and epimerization domains. Nat Struct Biol, 2002, 9(7): 522-526.
|
| [31] |
Samel SA, Schoenafinger G, Knappe TA, et al. Structural and functional insights into a peptide bond-forming bidomain from a nonribosomal peptide synthetase. Structure, 2007, 15(7): 781-792. DOI:10.1016/j.str.2007.05.008
|
| [32] |
Bozhüyük KAJ, Linck A, Tietze A, et al. Modification and de novo design of non-ribosomal peptide synthetases using specific assembly points within condensation domains. Nat Chem, 2019, 11(7): 653-661. DOI:10.1038/s41557-019-0276-z
|
| [33] |
Hahn M, Stachelhaus T. Selective interaction between nonribosomal peptide synthetases is facilitated by short communication-mediating domains. Proc Natl Acad Sci USA, 2004, 101(44): 15585-15590. DOI:10.1073/pnas.0404932101
|
| [34] |
Hahn M, Stachelhaus T. Harnessing the potential of communication-mediating domains for the biocombinatorial synthesis of nonribosomal peptides. Proc Natl Acad Sci USA, 2006, 103(2): 275-280. DOI:10.1073/pnas.0508409103
|
| [35] |
Chiocchini C, Linne U, Stachelhaus T. In vivo biocombinatorial synthesis of lipopeptides by COM domain-mediated reprogramming of the surfactin biosynthetic complex. Chem Biol, 2006, 13(8): 899-908. DOI:10.1016/j.chembiol.2006.06.015
|
| [36] |
Liu HX, Gao L, Han JZ, et al. Biocombinatorial synthesis of novel lipopeptides by COM domain-mediated reprogramming of the plipastatin NRPS complex. Front Microbiol, 2016, 7: 1801.
|
| [37] |
Cai XF, Nowak S, Wesche F, et al. Entomopathogenic bacteria use multiple mechanisms for bioactive peptide library design. Nat Chem, 2017, 9(4): 379-386. DOI:10.1038/nchem.2671
|
| [38] |
Hacker C, Cai XF, Kegler C, et al. Structure-based redesign of docking domain interactions modulates the product spectrum of a rhabdopeptide- synthesizing NRPS. Nat Commun, 2018, 9: 4366. DOI:10.1038/s41467-018-06712-1
|
| [39] |
Watzel J, Hacker C, Duchardt-Ferner E, et al. A new docking domain type in the peptide- antimicrobial-Xenorhabdus peptide producing nonribosomal peptide synthetase from Xenorhabdus bovienii. ACS Chem Biol, 2020, 15(4): 982-989. DOI:10.1021/acschembio.9b01022
|
| [40] |
Cai XF, Zhao L, Bode HB. Reprogramming promiscuous nonribosomal peptide synthetases for production of specific peptides. Org Lett, 2019, 21(7): 2116-2120. DOI:10.1021/acs.orglett.9b00395
|
| [41] |
Kegler C, Bode HB. Artificial splitting of a non-ribosomal peptide synthetase by inserting natural docking domains. Angew Chem Int Ed, 2020, 59(32): 13463-13467. DOI:10.1002/anie.201915989
|
| [42] |
Gidijala L, Kiel JAKW, Douma RD, et al. An engineered yeast efficiently secreting penicillin. PLoS ONE, 2009, 4(12): e8317. DOI:10.1371/journal.pone.0008317
|
| [43] |
Penn J, Li X, Whiting A, et al. Heterologous production of daptomycin in Streptomyces lividans. J Ind Microbiol Biotechnol, 2006, 33(2): 121-128. DOI:10.1007/s10295-005-0033-8
|
| [44] |
Barkei JJ, Kevany BM, Felnagle EA, et al. Investigations into viomycin biosynthesis by using heterologous production in Streptomyces lividans. ChemBioChem, 2009, 10(2): 366-376. DOI:10.1002/cbic.200800646
|
| [45] |
Watanabe K, Hotta K, Praseuth AP, et al. Total biosynthesis of antitumor nonribosomal peptides in Escherichia coli. Nat Chem Biol, 2006, 2(8): 423-428. DOI:10.1038/nchembio803
|
| [46] |
Butz D, Schmiederer T, Hadatsch B, et al. Module extension of a non-ribosomal peptide synthetase of the glycopeptide antibiotic balhimycin produced by Amycolatopsis balhimycina. ChemBioChem, 2008, 9(8): 1195-1200. DOI:10.1002/cbic.200800068
|
| [47] |
Mootz HD, Kessler N, Linne U, et al. Decreasing the ring size of a cyclic nonribosomal peptide antibiotic by in-frame module deletion in the biosynthetic genes. J Am Chem Soc, 2002, 124(37): 10980-10981. DOI:10.1021/ja027276m
|
| [48] |
Beer R, Herbst K, Ignatiadis N, et al. Creating functional engineered variants of the single-module non-ribosomal peptide synthetase IndC by T domain exchange. Mol BioSyst, 2014, 10(7): 1709-1718. DOI:10.1039/C3MB70594C
|
| [49] |
Drake EJ, Miller BR, Shi C, et al. Structures of two distinct conformations of holo-non-ribosomal peptide synthetases. Nature, 2016, 529(7585): 235-238. DOI:10.1038/nature16163
|
| [50] |
Reimer JM, Aloise MN, Harrison PM, et al. Synthetic cycle of the initiation module of a formylating nonribosomal peptide synthetase. Nature, 2016, 529(7585): 239-242. DOI:10.1038/nature16503
|
| [51] |
Tarry MJ, Haque AS, Bui KH, et al. X-ray crystallography and electron microscopy of cross- and multi-module nonribosomal peptide synthetase proteins reveal a flexible architecture. Structure, 2017, 25(5): 783-793. DOI:10.1016/j.str.2017.03.014
|
| [52] |
Reimer JM, Eivaskhani M, Harb I, et al. Structures of a dimodular nonribosomal peptide synthetase reveal conformational flexibility. Science, 2019, 366(6466): eaaw4388. DOI:10.1126/science.aaw4388
|
| [53] |
Bloudoff K, Alonzo DA, Schmeing TM. Chemical probes allow structural insight into the condensation reaction of nonribosomal peptide synthetases. Cell Chem Biol, 2016, 23(3): 331-339. DOI:10.1016/j.chembiol.2016.02.012
|
| [54] |
Huguenin-Dezot N, Alonzo DA, Heberlig GW, et al. Trapping biosynthetic acyl-enzyme intermediates with encoded 2, 3-diaminopropionic acid. Nature, 2019, 565(7737): 112-117. DOI:10.1038/s41586-018-0781-z
|
 2021, Vol. 37
2021, Vol. 37