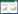中国科学院微生物研究所、中国微生物学会主办
文章信息
- 郝天怡, 赫卫清
- Hao Tianyi, He Weiqing
- 大环内酯类抗生素代谢工程的研究进展
- Advances in metabolic engineering of macrolide antibiotics
- 生物工程学报, 2021, 37(5): 1737-1747
- Chinese Journal of Biotechnology, 2021, 37(5): 1737-1747
- 10.13345/j.cjb.200686
-
文章历史
- Received: October 26, 2020
- Accepted: January 28, 2021
大环内酯类抗生素(Macrolide antibiotics,MA)是临床常用的一类抗菌药物,它是由一个多元碳的内酯环附着一个或多个脱氧糖所构成的聚酮类抗生素,常见的MA是指14–16元环的MA (如红霉素类衍生物、乙酰螺旋霉素等)。红霉素是第一个用于临床的MA,其后问世的地红霉素、麦迪霉素等第一代MA对胃酸不稳定,生物利用度低。第二代如罗红霉素、阿奇霉素等提高了对胃酸的稳定性和生物利用度,抗菌活性增强。随着很多细菌对MA出现了较普遍的耐药性,因此开发出了第三代酮内酯类药物,包括泰利霉素、喹红霉素等,它们对第一、二代MA的耐药菌有良好作用,且抗菌谱更广。但是第三代大环内酯类药物毒性偏大,通过研究证实,泰利霉素碱性侧链的吡啶环会阻断烟碱型胆碱受体,从而导致肝毒性。目前去掉吡啶环的新药物索利霉素正在进行Ⅲ期临床试验,有望成为治疗细菌性肺炎的第四代大环内酯类药物[1]。这些药物大部分是通过化学修饰法、半合成法获得,但以红霉素为基础再改造出新的衍生物已经非常困难。随着生物技术的快速发展,通过代谢工程的方法改造MA的生物合成基因可以获得大量MA新衍生物。本文将对利用代谢工程解析并改造MA的生物合成基因来获得新的MA衍生物以及利用代谢工程的方法提高MA产量进行总结,并详细介绍了我国自主研发的大环内酯类新药可利霉素的特点及利用代谢工程构建和改造其产生菌的过程。
1 代谢工程与MA的改造代谢工程是在1991年Bailey[2]首先提出的概念,主要指利用重组DNA技术,有目的地操纵细胞的酶、转运和调控功能,从而改善细胞的活性。经过近30年的发展,其外延已得到了极大拓展,在改造MA的生物合成获得新衍生物以及在提高其产量方面显示出巨大优势。早期代谢工程的方法改造MA产生菌时,主要通过对MA生物合成途径中的关键基因进行遗传改造来提高其产量。如通过阻断泰乐菌素生物合成基因簇中的负调控基因可以提高泰乐菌素的产量[3]。随着DNA测序和生物信息学的快速发展,代谢工程融入了各种组学技术,能够从基因、RNA、蛋白、代谢物等多个层次系统地分析微生物代谢,极大地推动了代谢工程和微生物发酵工业的发展[4]。近些年来,合成生物学技术[5]发展迅速,并与代谢工程既交叉融合又相互促进。合成生物学是以工程学理论为依据,设计和合成新的生物元件,或是设计改造已经存在的生物系统。一些合成生物学方法也广泛应用在MA产生菌的代谢工程改造中。例如利用成簇规律间隔短回文重复序列及其相关蛋白9 (Clustered regularly interspaced short palindromic repeats/CRISPR-associated protein 9,CRISPR/Cas9) 技术在红霉素的生物合成基因簇中插入双向启动子元件来代替天然启动子,并结合CRISPR干扰技术对基因簇进行调控,显著提高了红色糖多孢菌Saccharopolyspora erythraea中红霉素的产量[6]。
目前,代谢工程在MA生物合成研究方面主要包括阐明MA的生物合成机制、提高产量以及结构的定向改造等。随着生物信息学的发展,一些次级代谢产物生物合成基因簇预测软件的出现,为解析次级代谢产物的生物合成机制带来了极大便利。如antiSMASH (Antibiotics and secondary metabolite analysis shell),是目前使用率最高的基因簇预测和分析的数据库[7],通过antiSMASH分析,可以快速鉴定出基因组序列中负责次级代谢产物的生物合成基因簇。对解析后的生物合成基因簇,可以通过改造其生物合成基因或利用组合生物合成的方法获得新的衍生物,也可以改造其调控基因来提高产量。
2 MA的作用靶点与结构优化绝大多数MA的结合靶点在细菌核糖体50S大亚基上,位于与肽酰转移酶活性中心相邻的新生肽链的通道附近,它们可逆地干扰新生肽链的延伸[8-9]。以核糖体为靶点的MA可以分为抑菌型和杀菌型,这与药物从核糖体上的解离动力学有关,解离慢的MA通常具有杀菌作用,而相对快速解离的MA只有抑菌效果[10]。为深入了解大环内酯类药物的作用方式,用高浓度MA处理大肠杆菌,对其进行全基因组水平的核糖体图谱分析,发现MA依赖的翻译阻滞位点是由肽基转移酶中心的氨基酸残基性质决定,而不是由出口通道中新生肽链的氨基酸序列决定[11]。MA上的糖基侧链与核糖体的相互作用对其生物学活性至关重要(图 1)[12],但红霉素的L-红霉糖侧链在酸性环境中常常会被缓慢水解而失去活性。为了获得活性更强的红霉素衍生物,将3位的易水解糖基进行改造,或在红霉素4″位加上碱性侧链来增加活性,因为这个碱性侧链会伸到肽酰转移酶的活性中心区域[13]。
在16元MA中,碳霉糖(Mycarose) 的“延伸”能够与核糖体发生额外的相互作用,这是与14元环MA的主要区别[14]。因此,这两种大环内酯类化合物呈现出不同的作用方式:16元MA是肽键形成的抑制剂,而14元MA是结合在新生肽链的出口,导致肽链过早终止[15]。16元环MA的结构与抗菌活性研究表明,在螺旋霉素(Mycarose)的4″位上增加酰基侧链可增加与核糖体靶点的相互作用,而且酰基链的长度越长抗菌活性越好[16-17]。16元环MA与核糖体大亚基的三维结构研究中也证实碳霉素(Carbomycin) 的4″的异戊酰基侧链可以干扰到肽酰转移酶中心的A位点[12] (图 1)。王以光团队从生米卡链霉菌Streptomyces mycarofaciens 1748中克隆了4″-O-丙酰转移酶基因,通过生物转化螺旋霉素的方法,首次获得4″位为丙酰基修饰的丙酰螺旋霉素[18];之后又从耐热链霉菌Streptomyces thermotolerans中克隆到4″-O-异戊酰基转移酶基因(4″-O-isovaleryltransferase gene,ist)[19-20],将其整合至从我国土壤中分离到的螺旋链霉菌Streptomyces spiramyceticus的染色体上,发酵产生4″异戊酰化修饰的螺旋霉素为主组分的可利霉素(原名为生技霉素或必特螺旋霉素)[21]。泰乐菌素通过微生物转化的方法在其4″端加上异戊酰基后,其活性也明显提高[22]。
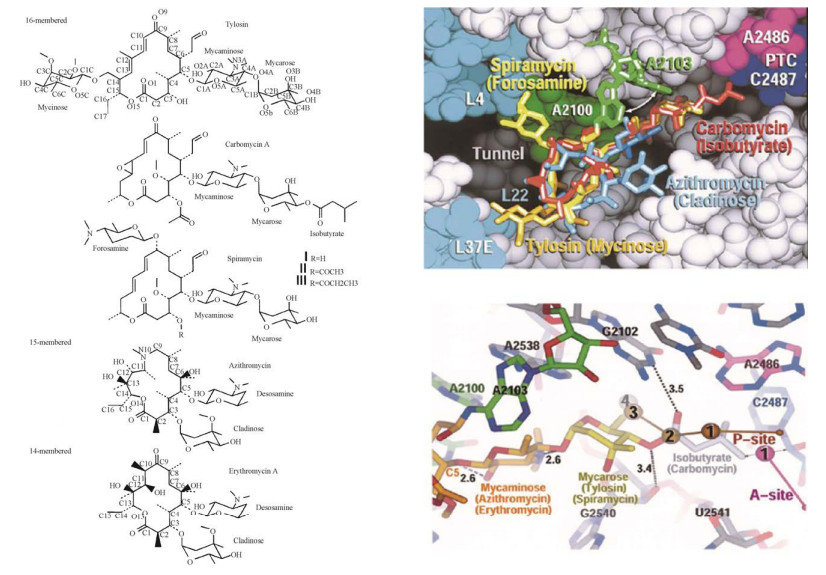
3 MA的生物合成与改造 3.1 MA的生物合成MA生物合成过程可以分为两个步骤,首先是利用PKS合成大环内酯骨架,之后是对大环内酯骨架进行PKS后修饰,包括糖基化、羟基化、氧化和甲基化等。以红霉素为代表的MA生物合成是利用模块化的Ⅰ型聚酮合酶(Polyketide synthase,PKS) 由α-羧酰辅酶A (CoA) 构建单元衍生而来,通过连续地脱羧缩合来催化合成6-脱氧红霉内酯B (6-deoxyerythronolide B synthase,DEBS)[23-26](图 2)。红霉素生物合成的Ⅰ型PKS由3个蛋白亚基组成,分别是DEBS1、DEBS2和DEBS3,各亚基单元分别催化一轮碳链的延伸,其中每个亚基单元包含多个执行特定催化功能的结构域,负责对碳链进行装配和修饰,各单元内对每个碳链的缩合和还原周期包含有完整的活性位点。PKS催化的终产物碳链的长度及立体化学构型是由单元的组成和数目决定的,每个单元均含有酮基合成酶(Ketosynthase,KS)、酰基转移酶(Acyltransferase,AT)、酰基载体蛋白(Acyl carrier protein,ACP),有的PKS还含有酮基还原酶(Ketoreduetase,KR)、烯醇还原酶(Enoyl reductase,ER)、羟基脱氢酶(Dehydrogenase,DH) 中的一个或几个,硫酯酶(Thioesterase,TE) 位于最末端,行使碳链延长终止和内酯环化的功能。
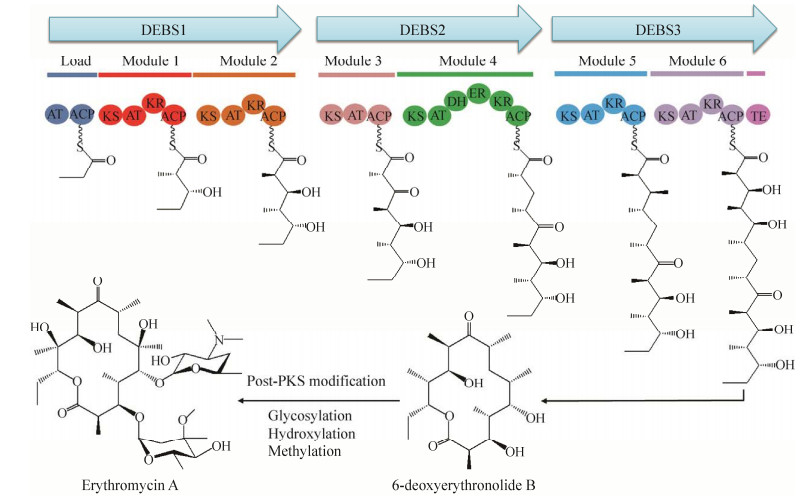
|
| 图 2 红霉素A的生物合成 Fig. 2 The biosynthesis of erythromycin A. |
| |
一般来讲,荷载域的AT具有较大的底物宽容性,可以将荷载域进行替换,从而识别不同的前体物来定向实现组合生物合成。PKS模块中KS的专一性与AT相似,具有较严格的底物专一性,但是不同模块的KS对底物的专一性有所不同。而KR、DH和ER可以接受许多不同的天然产物,因此在PKS模块之间增减这些结构域,就可以改变链延伸单位化学键的饱和度。在PKS模块中TE结构域是负责结束PKS合成的,将变构菌素(Tautomycetin) 的TE结构域替换为苦霉素(Pikromycin) 的TE结构域不仅能产生线性的变构菌素,还产生了环形的变构菌素[27]。迄今为止,改造PKS模块的方法主要为构建异源AT结构域[28-31]、异源KR结构域[32-34]或其他异源结构域[28, 32, 35-37]的嵌合PKS。这些结果表明,通过识别、连接和催化结构域的组合,可以成功构建活性PKS嵌合体,但不同AT结构域的替换常常导致杂合酶的低效或失活。2017年Yuzawa等[31]系统鉴定出了高度保守的AT结构域的边界,这就使AT结构域的替换变得更加简单高效。
随着体外克隆、生物信息学等技术的快速发展,近年来建立PKS定向、理性设计的研究受到广泛关注。AT结构域的定向改造是通过替换氨基酸残基来获得具有底物宽泛性的PKS,而不是通过改变AT结构域来替换对底物的选择性。Koryakina等[38]通过直接从红霉素DEBS中AT结构域的活性位点饱和文库中筛选突变体来改变延伸单位选择性,发现了一系列可显著影响PKS选择性而仅适度降低产品收率的单氨基酸取代位点。其中,DEBS3的AT6结构域中Tyr189处的特定突变使其选择性地向非天然扩展单元转移,从而导致产物更加多样化。Eng等[39]开发了一个集群CAD计算平台,它利用Ⅰ型PKS的共线结构和确定性逻辑,应用于组合生物合成。用户可以通过搜索已知的一个模块预测得到一个与目的化合物结构最为相似的聚酮肽中间体,根据与PKS亲本的序列相似性,选择供体模块进行催化结构域交换。
近期,Kudo等[40]利用CRISPR/Cas9与Gibson组装相结合的新技术应用于PKS的模块化编辑,再通过异源表达,最终建立了一套适用于各种天然产物合成的模块编辑系统。首先利用体外CRISPR/Cas9技术在包含有雷帕霉素生物合成基因簇的载体上精确地剪切模块9的AT结构域,然后将消化后的基因簇片段与另一个AT结构域的DNA片段进行Gibson组装,由此将雷帕霉素PKS模块9的丙二酰-CoA的延伸单元AT结构域替换为甲基丙二酰-CoA单元延伸的AT结构域。研究者利用此方法也尝试了模块的敲除和插入,得到了多种雷帕霉素的衍生物。
3.3 PKS后修饰过程的改造MA的PKS后修饰主要由单个或少数几个基因来负责,增加或者替换其他适宜的后修饰基因就会对内酯环进行新的修饰,从而获得具有新活性的衍生物。MA的化学结构中都至少包含一个糖基侧链,糖基化可以有效改善化合物的疏水性、溶解度和立体构型,且常直接参与化合物与靶点的相互作用,进而影响其生物活性。因此,对MA的糖基化改造已经开展了大量研究。Melançon等[41]将灰色链霉菌Streptomyces griseus中的TDP-双羟基链霉糖(TDP-D-dihydrostreptose) 的生物合成基因或弗氏链霉菌Streptomyces fradiae中的TDP-mycaminose生物合成基因导入到委内瑞拉链酶菌Streptomyces venezuelae的desI基因突变株中,可以产生新的酒霉素(Methymycin) 或苦霉素(Pikromycin) 衍生物。美加米星(Megolomicin) 与红霉素结构相似,利用其产生菌中美加糖胺基转移酶改造红霉素和阿奇霉素,获得了6-O-美加糖胺红霉素/阿奇霉素,这些新的糖基化修饰的衍生物还产生了抗疟原虫活性[42]。该团队还在大肠杆菌中建立异源脱氧糖基的合成系统,对红霉素上的糖基进行替换,获得了一系列新的糖基化产物[43]。另外,利用泰乐菌素、蔷薇霉素等几种不同大环内酯类化合物的P450单加氧酶对泰乐菌素大环内酯进行催化,可以实现不同位点的氧化,从而获得一系列新修饰的抗生素[44]。
4 利用代谢工程提高MA产量MA的发酵产量直接影响药物的后续研发,通过代谢工程的方法可增强目标MA的生物合成,主要从以下几个方面进行改造:1) 改善或提高限速步骤相关酶的活性,或增加单位细胞内控制限速步骤相关酶的基因拷贝数,以解除或一定程度上解除该酶的限速性[45];2) 通过引入高表达量的抗性基因、调控因子、激活蛋白来提高抗生素的产量,也可通过抑制负调控蛋白的表达或敲除编码负调控蛋白的基因[46-48];3) 链霉菌是好氧菌,其发酵过程中溶氧水平对抗生素的产量极为重要,通过添加外源透明颤菌血红蛋白基因可以提高供氧量,从而提高抗生素的产量[49-50];4) 通过敲除或破坏产生次要组分的生物合成基因来消除或减少次要组分;5) 将PKS的底物甲基丙二酰辅酶A等的降解途径进行阻断,如在红色糖多孢菌Streptomyces erythraea HL3168 E3中敲除编码甲基丙二酰辅酶A歧化酶的基因mutB,可以将红霉素产量提高1.6倍[51];6) 用强启动子替换相关生物合成基因簇中的弱启动子等。
另外,调控生长期与稳定期的细菌代谢途径,也是实现高效生产抗生素的有效手段。聚酮类抗生素是链霉菌的一类重要的次级代谢产物,其生物合成过程受到严格的调控,其产量与发酵培养时期以及初级代谢产物的产量具有紧密的联系。近期,Wang等[52]发现,链霉菌在进入生长稳定期后,内源三酰甘油(Triacylglycerols,TAGs) 由积累转变为降解,从而为聚酮类次级代谢产物的合成提供前体和还原力。研究人员通过人工控制内源TAGs的降解,显著提高了阿维菌素等聚酮类药物的产量。Tan等[53]通过采用转录组学和蛋白质组学技术,分析了多杀菌素的生物合成基因在链霉菌寄主以及异源宿主中的表达,并通过代谢组学分析研究了多杀菌素的前体物质和辅因子,以及生物合成中间产物,进而揭示了多杀菌素异源合成的几个限速步骤,通过替换限速步骤中关键基因的启动子,使多杀菌素在异源宿主中的产量增加了近千倍。
5 16元环的MA 5.1 16元环MA的特点由于14或15元环容易产生诱导耐药,而16元环MA分子柔性好、无诱导耐药性、交叉耐药性小,特别是对由靶点修饰、外排作用介导的耐药菌,有较好的抗菌活性[54]。14元环MA已被广泛研究,相比之下16元MA的研究较少。16元MA主要用于兽医学(如泰乐菌素A),只有少数已成功应用于临床治疗,如乙酰螺旋霉素等。目前临床应用的16元大环内酯类药物是交沙霉素、螺旋霉素和麦迪霉素,以及半合成的米奥卡霉素、罗基塔霉素、替米考星和替利匹罗辛等[55-56]。16元环的MA与14和15元环的红霉素及衍生物相比,具有相似的抗菌特性,主要针对革兰氏阳性菌和部分革兰氏阴性菌。但16元环的MA还有另外一些优势,包括更好的胃肠道耐受性、降低药物与药物的相互作用、与核糖体有更多的结合位点而具有更多的相互作用来对抗某些耐药菌株。16元环的MA除具有抗菌活性外,泰乐菌素A以及一些地霉素衍生物也显示出抗疟活性,这种抗疟活性为这些药物作为廉价和有效的抗疟药物进行研究提供了机会[56]。
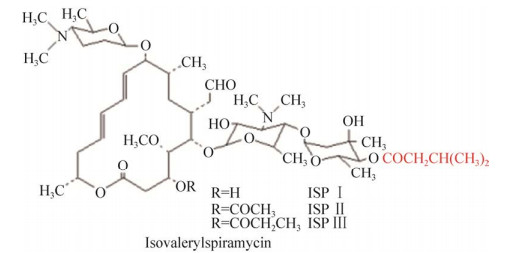
5.2 16元环大环内酯类新药—可利霉素可利霉素是由中国医学科学院医药生物技术研究所与沈阳同联集团有限公司共同开发的国家一类新药,于2019年6月获批上市。它是我国首个利用组合生物合成研发的大环内酯类创新药物。可利霉素的成功首先在于定向选用安全有效的螺旋霉素母核作为研发基础,根据药物特性和作用靶点进行针对性改造。可利霉素是利用组合生物合成方法将异源ist基因导入到螺旋链霉菌种,通过发酵产生的酰化螺旋霉素,其中4ʺ-O-异戊酰螺旋霉素(Isovalerylspiramycin,ISP) Ⅰ、Ⅱ和Ⅲ (图 3) 为主组分,还包括一些在4ʺ位进行其他酰化修饰的小组分。临床试验证实可利霉素在体内外抗菌活性均明显优于乙酰螺旋霉素和阿奇霉素。药物代谢动力学研究也证实它比螺旋霉素体内半衰期更长,部分代谢产物仍然保持抗菌活性;组织渗透性增强,还具有明显的抗生素后效应[57-58]。
|
|
图 3 异戊酰螺旋霉素的化学结构
Fig. 3 The chemical structure of isovalerylspiramycin.
|
| |
药效学研究表明,可利霉素对某些耐药菌(例如对产β-内酰胺酶的金黄色葡萄球菌、耐红霉素金黄色葡萄球菌等) 有效,主要用于治疗呼吸系统的感染,与红霉素和阿奇霉素等大环内酯类抗生素不完全交叉耐药,诱导耐药率低。可利霉素除对耐药的革兰氏阳性菌有效外,对部分阴性耐药菌,如产超广谱β-内酰胺酶的大肠杆菌感染也有一定疗效。可利霉素还对部分异烟肼和利福平耐药结核杆菌也有活性[59]。在今年的新冠肺炎疫情期间的药物筛选过程中,发现可利霉素对新型冠状病毒(SARS-CoV2) 有很好的抑制活性,并进行了临床试验[60]。
可利霉素最初构建的质粒型工程菌的发酵效价只有(450±70.8) μg/mL左右[61],经过同源双交换将ist基因整合到宿主染色体上后获得稳定的工程菌株S. spiramyceticus WSJ-1,效价为(838± 65.9) μg/mL,提高近2倍[21]。许多经过基因操作的工程菌发酵单位均会明显降低,这也是许多工程菌发酵产物无法推广应用的重要原因。在筛选可利霉素高产工程菌方面,采用了多种方法进行遗传育种。在常规诱变育种基础上,还开展了核糖体工程、全基因组Shuffling、太空育种[62]、微波诱变[63]等方法提高工程菌生产可利霉素的能力,最终获得基因工程高产菌株WSJ-195。
对可利霉素工程菌也进行了全基因组测序,其基因组为线型,序列全长7 183 802 bp,包含6 515个基因。通过anti-SMASH分析和基因功能鉴定得到可利霉素的生物合成基因簇[64],还发现有31个与次级代谢产物生物合成相关基因簇,说明该菌株具备合成多种次级代谢产物的潜能。将可利霉素低产菌株与高产菌株的基因组进行对比分析,发现高产菌株染色体中有30多个基因发生了点突变,这为今后构建新的高产菌株奠定了基础。
5.3 可利霉素产生菌的升级改造由于螺旋霉素3位酰基化酶底物的特异性不强,可以同时识别乙酰基和丙酰基底物,导致可利霉素出现了3种主组分ISP Ⅰ、Ⅱ和Ⅲ。可利霉素作为一种多种结构类似成分的混合物,研究其作用机制难度较大,对提取纯化工艺的控制要求比较高,难以制作为注射剂型等其他剂型。经研究证实ISP Ⅰ的药物活性与可利霉素相当,因此可以作为一种单组分药物进行开发,这也降低了药物在发酵产物提纯和质控方面的难度。通过阻断WSJ-1中螺旋霉素3位酰基化酶基因bsm4,获得只产ISP Ⅰ的基因工程菌株WSJ-2[65]。但此菌株ISP Ⅰ的产量很低,发酵液的效价只有(280±20) μg/mL。为了提高ISP Ⅰ的发酵产量,又将ist基因和其正调控基因acyB2整合到上述ISP Ⅰ菌株的染色体上,通过育种筛选,获得效价为(1 160±108)μg/mL的高产菌株WSJ-IA[66]。但在WSJ-IA高产菌株中已经包含硫链丝菌素和安普霉素两种抗性基因,利用现有的链霉菌质粒很难再对其进行定向遗传操作。因此采用新型等离子诱变和传统诱变相结合的方法对WSJ-IA菌株进行诱变育种,获得效价为(2 000±200) μg/mL的高产菌株IA-425[67],基本满足工业发酵的要求。但此菌株共进行3次遗传操作,包含了2个抗性基因,在菌株染色体的两个不同位置中还包含3个ist基因,在传代过程中有发生同源重组的可能性,不利于菌株的稳定遗传。因此利用CRISPR/Cas9编辑系统构建出一种没有抗性的ISP Ⅰ产生菌[68],将来可以重复利用CRISPR/Cas9系统对此菌株进行升级改造。
6 总结与展望MA是重要的一线临床用药,但获得新的大环内酯类化合物的难度越来越大,通过代谢工程解析其生物合成机制,重构PKS合成大环内酯的步骤以及后修饰过程,可以获得大量的衍生物。另外重新组装的PKS可以通过优化底盘菌株(或生物系统) 和表达系统来实现。因此,未来应将新兴的合成生物学技术与传统的代谢工程相结合,将使我们有能力创造出具有多种骨架的大环内酯类化合物来抵御后抗生素时代的威胁。
| [1] |
Fernandes P, Martens E, Pereira D. Nature nurtures the design of new semi-synthetic macrolide antibiotics. J Antibiot (Tokyo), 2017, 70(5): 527-533. DOI:10.1038/ja.2016.137
|
| [2] |
Bailey JE. Toward a science of metabolic engineering. Science, 1991, 252(5013): 1668-1675. DOI:10.1126/science.2047876
|
| [3] |
Stratigopoulos G, Cundliffe E. Inactivation of a transcriptional repressor during empirical improvement of the tylosin producer, Streptomyces fradiae. J Ind Microbiol Biotechnol, 2002, 28(4): 219-224. DOI:10.1038/sj.jim.7000234
|
| [4] |
张学礼. 代谢工程发展20年. 生物工程学报, 2009, 25(9): 1285-1295. Zhang XL. Twenty years development of metabolic engineering—a review. Chin J Biotech, 2009, 25(9): 1285-1295 (in Chinese). DOI:10.3321/j.issn:1000-3061.2009.09.002 |
| [5] |
Endy D. Foundations for engineering biology. Nature, 2005, 438(7067): 449-453. DOI:10.1038/nature04342
|
| [6] |
Liu Y, Ren CY, Wei WP, et al. A CRISPR-Cas9 strategy for activating the Saccharopolyspora erythraea erythromycin biosynthetic gene cluster with knock-in bidirectional promoters. ACS Synth Biol, 2019, 8(5): 1134-1143. DOI:10.1021/acssynbio.9b00024
|
| [7] |
Blin K, Shaw S, Steinke K, et al. antiSMASH 5.0: updates to the secondary metabolite genome mining pipeline. Nucleic Acids Res, 2019, 47(W1): W81-W87. DOI:10.1093/nar/gkz310
|
| [8] |
Dinos GP. The macrolide antibiotic renaissance. Br J Pharmacol, 2017, 174(18): 2967-2983. DOI:10.1111/bph.13936
|
| [9] |
Jelić D, Antolović R. From erythromycin to azithromycin and new potential ribosome-binding antimicrobials. Antibiotics (Basel), 2016, 5(3): 29. DOI:10.3390/antibiotics5030029
|
| [10] |
Svetlov MS, Vázquez-Laslop N, Mankin AS. Kinetics of drug-ribosome interactions defines the cidality of macrolide antibiotics. Proc Natl Acad Sci USA, 2017, 114(52): 13673-13678. DOI:10.1073/pnas.1717168115
|
| [11] |
Kannan K, Kanabar P, Schryer D, et al. The general mode of translation inhibition by macrolide antibiotics. Proc Natl Acad Sci USA, 2014, 111(45): 15958-15963. DOI:10.1073/pnas.1417334111
|
| [12] |
Hansen JL, Ippolito JA, Ban N, et al. The structures of four macrolide antibiotics bound to the large ribosomal subunit. Mol Cell, 2002, 10(1): 117-128. DOI:10.1016/S1097-2765(02)00570-1
|
| [13] |
Park JW, Yoon YJ. Recent advances in the discovery and combinatorial biosynthesis of microbial 14-membered macrolides and macrolactones. J Ind Microbiol Biotechnol, 2019, 46(3/4): 445-458.
|
| [14] |
Gharbi-Benarous J, Evrard-Todeschi N, Ladam P, et al. Conformational analysis of josamycin, a 16-membered macrolide free in solution and bound to bacterial ribosomes. J Chem Soc Perkin Trans 2, 1999, 2(3): 529-543.
|
| [15] |
Selmer M, Liljas A. Exit biology: battle for the nascent chain. Structure, 2008, 16(4): 498-500. DOI:10.1016/j.str.2008.03.002
|
| [16] |
Sano H, Sunazuka T, Tanaka H, et al. Chemical modification of spiramycins. Ⅳ. Synthesis and in vitro and in vivo activities of 3", 4"-diacylates and 3, 3", 4"-triacylates of spriamycin Ⅰ. J Antibiot (Tokyo), 1984, 37(7): 760-772. DOI:10.7164/antibiotics.37.760
|
| [17] |
Ōmura S, Sano H, Sunazuka T. Structure activity relationships of spiramycins. J Antimicrob Chemother, 1985, 16(Suppl.A)): 1-11.
|
| [18] |
王以光, 金莲舫, 徐小敏, 等. 基因工程丙酰螺旋霉素. 中国抗生素杂志, 1994, 19(2): 109-116. Wang YG, Jin LF, Xu XM, et al. A genetically engineered hybrid antibiotic propionylspiramycin. Chin J Antibiot, 1994, 19(2): 109-116 (in Chinese). |
| [19] |
Epp JK, Huber MLB, Turner JR, et al. Production of a hybrid macrolide antibiotic in Streptomyces ambofaciens and Streptomyces lividans by introduction of a cloned carbomycin biosynthetic gene from Streptomyces thermotolerans. Gene, 1989, 85(2): 293-301. DOI:10.1016/0378-1119(89)90421-6
|
| [20] |
Arisawa A, Kawamura N, Tsunekawa H, et al. Cloning and nucleotide sequences of two genes involved in the 4″-O-acylation of macrolide antibiotics from Streptomyces thermotolerans. Biosci Biotechnol Biochem, 1993, 57(12): 2020-2025. DOI:10.1271/bbb.57.2020
|
| [21] |
Shang GD, Dai JL, Wang YG. Construction and physiological studies on a stable bioengineered strain of shengjimycin. J Antibiot (Tokyo), 2001, 54(1): 66-73. DOI:10.7164/antibiotics.54.66
|
| [22] |
Arisawa A, Kawamura N, Narita T, et al. Direct fermentative production of acyltylosins by genetically-engineered strains of Streptomyces fradiae. J Antibiot (Tokyo), 1996, 49(4): 349-354. DOI:10.7164/antibiotics.49.349
|
| [23] |
Hopwood DA. Genetic contributions to understanding polyketide synthases. Chem Rev, 1997, 97(7): 2465-2498. DOI:10.1021/cr960034i
|
| [24] |
Cane DE. Programming of erythromycin biosynthesis by a modular polyketide synthase. J Biol Chem, 2010, 285(36): 27517-27523. DOI:10.1074/jbc.R110.144618
|
| [25] |
Valenzano CR, You YO, Garg A, et al. Stereospecificity of the dehydratase domain of the erythromycin polyketide synthase. J Am Chem Soc, 2010, 132(42): 14697-14699. DOI:10.1021/ja107344h
|
| [26] |
Keatinge-Clay AT. Polyketide synthase modules redefined. Angew Chem Int Ed Engl, 2017, 56(17): 4658-4660. DOI:10.1002/anie.201701281
|
| [27] |
Tripathi A, Choi SS, Sherman DH, et al. Thioesterase domain swapping of a linear polyketide tautomycetin with a macrocyclic polyketide pikromycin in Streptomyces sp. CK4412. J Ind Microbiol Biotechnol, 2016, 43(8): 1189-1193. DOI:10.1007/s10295-016-1790-2
|
| [28] |
McDaniel R, Thamchaipenet A, Gustafsson C, et al. Multiple genetic modifications of the erythromycin polyketide synthase to produce a library of novel "unnatural" natural products. Proc Natl Acad Sci USA, 1999, 96(5): 1846-1851. DOI:10.1073/pnas.96.5.1846
|
| [29] |
Hans M, Hornung A, Dziarnowski A, et al. Mechanistic analysis of acyl transferase domain exchange in polyketide synthase modules. J Am Chem Soc, 2003, 125(18): 5366-5374. DOI:10.1021/ja029539i
|
| [30] |
Dunn BJ, Khosla C. Engineering the acyltransferase substrate specificity of assembly line polyketide synthases. J Roy Soc Interface, 2013, 10(85): 20130297. DOI:10.1098/rsif.2013.0297
|
| [31] |
Yuzawa S, Deng K, Wang G, et al. Comprehensive in vitro analysis of acyltransferase domain exchanges in modular polyketide synthases and its application for short-chain ketone production. ACS Synth Biol, 2017, 6(1): 139-147. DOI:10.1021/acssynbio.6b00176
|
| [32] |
Kao CM, McPherson M, McDaniel RN, et al. Alcohol stereochemistry in polyketide backbones is controlled by the β-ketoreductase domains of modular polyketide synthases. J Am Chem Soc, 1998, 120(10): 2478-2479. DOI:10.1021/ja973913a
|
| [33] |
Kellenberger L, Galloway IS, Sauter G, et al. A polylinker approach to reductive loop swaps in modular polyketide synthases. ChemBioChem, 2008, 9(16): 2740-2749. DOI:10.1002/cbic.200800332
|
| [34] |
Eng CH, Yuzawa S, Wang G, et al. Alteration of polyketide stereochemistry from anti to syn by a ketoreductase domain exchange in a Type Ⅰ modular polyketide synthase subunit. Biochemistry, 2016, 55(12): 1677-1680. DOI:10.1021/acs.biochem.6b00129
|
| [35] |
McDaniel R, Kao CM, Hwang SJ, et al. Engineered intermodular and intramodular polyketide synthase fusions. Chem Biol, 1997, 4(9): 667-674. DOI:10.1016/S1074-5521(97)90222-2
|
| [36] |
Gaisser S, Kellenberger L, Kaja AL, et al. Direct production of ivermectin-like drugs after domain exchange in the avermectin polyketide synthase of Streptomyces avermitilis ATCC31272. Org Biomol Chem, 2003, 1(16): 2840-2847. DOI:10.1039/b304022d
|
| [37] |
Hagen A, Poust S, De Rond T, et al. Engineering a polyketide synthase for in vitro production of adipic acid. ACS Synth Biol, 2016, 5(1): 21-27. DOI:10.1021/acssynbio.5b00153
|
| [38] |
Koryakina I, Kasey C, McArthur JB, et al. Inversion of extender unit selectivity in the erythromycin polyketide synthase by acyltransferase domain engineering. ACS Chem Biol, 2017, 12(1): 114-123. DOI:10.1021/acschembio.6b00732
|
| [39] |
Eng CH, Backman TWH, Bailey CB, et al. ClusterCAD: a computational platform for type Ⅰ modular polyketide synthase design. Nucleic Acids Res, 2018, 46(D1): D509-D515. DOI:10.1093/nar/gkx893
|
| [40] |
Kudo K, Hashimoto T, Hashimoto J, et al. In vitro Cas9-assisted editing of modular polyketide synthase genes to produce desired natural product derivatives. Nat Commun, 2020, 11: 4022. DOI:10.1038/s41467-020-17769-2
|
| [41] |
Melançon CE 3rd, Yu WL, Liu HW. TDP-mycaminose biosynthetic pathway revised and conversion of desosamine pathway to mycaminose pathway with one gene. J Am Chem Soc, 2005, 127(35): 12240-12241. DOI:10.1021/ja053835o
|
| [42] |
Goodman CD, Useglio M, Peirú S, et al. Chemobiosynthesis of new antimalarial macrolides. Antimicrob Agent Chemother, 2013, 57(2): 907-913. DOI:10.1128/AAC.01825-12
|
| [43] |
Fang L, Zhang GJ, El-Halfawy O, et al. Broadened glycosylation patterning of heterologously produced erythromycin. Biotechnol Bioeng, 2018, 115(11): 2771-2777. DOI:10.1002/bit.26735
|
| [44] |
Lowell AN, DeMars Ⅱ MD, Slocum ST, et al. Chemoenzymatic total synthesis and structural diversification of tylactone-based macrolide antibiotics through late-stage polyketide assembly, tailoring, and C-H functionalization. J Am Chem Soc, 2017, 139(23): 7913-7920. DOI:10.1021/jacs.7b02875
|
| [45] |
Jiang H, Wang YY, Ran XX, et al. Improvement of natamycin production by engineering of phosphopantetheinyl transferases in Streptomyces chattanoogensis L10. Appl Environ Microbiol, 2013, 79(11): 3346-3354. DOI:10.1128/AEM.00099-13
|
| [46] |
Bate N, Stratigopoulos G, Cundliffe E. Differential roles of two SARP-encoding regulatory genes during tylosin biosynthesis. Mol Microbiol, 2002, 43(2): 449-458. DOI:10.1046/j.1365-2958.2002.02756.x
|
| [47] |
Aigle B, Pang XH, Decaris B, et al. Involvement of AlpV, a new member of the streptomyces antibiotic regulatory protein family, in regulation of the duplicated type Ⅱ polyketide synthase alp gene cluster in Streptomyces ambofaciens. J Bacteriol, 2005, 187(7): 2491-2500. DOI:10.1128/JB.187.7.2491-2500.2005
|
| [48] |
Horinouchi S, Beppu T. Hormonal control by a-factor of morphological development and secondary metabolism in Streptomyces. Proc Jpn Acad Ser B, 2007, 83(9/10): 277-295. DOI:10.2183/pjab.83.277
|
| [49] |
Tabakov VI, Emel'ianova LK, Antonova SV, et al. Effect of the gene for bacterial hemoglobin vhb on the effectiveness of the process of Escherichia coli-Streptomyces interspecies conjugation and production of antibiotics in Streptomycetes. Genetika, 2001, 37(3): 422-425.
|
| [50] |
崔风文, 杨胜利. 利用ORF438启动子在链霉菌中表达透明颤菌血红蛋白. 生物工程学报, 1997, 13(4): 443-445. Cui FW, Yang SL. Expression of vitreoscilla hemoglobin in Streptomyces by ORF438 promoter. Chin J Biotech, 1997, 13(4): 443-445 (in Chinese). |
| [51] |
Chen CC, Hong M, Chu J, et al. Blocking the flow of propionate into TCA cycle through a mutB knockout leads to a significant increase of erythromycin production by an industrial strain of Saccharopolyspora erythraea. Bioprocess Biosyst Eng, 2017, 40(2): 201-209. DOI:10.1007/s00449-016-1687-5
|
| [52] |
Wang WS, Li SS, Li ZL, et al. Harnessing the intracellular triacylglycerols for titer improvement of polyketides in Streptomyces. Nat Biotechnol, 2020, 38(1): 76-83. DOI:10.1038/s41587-019-0335-4
|
| [53] |
Tan GY, Deng KH, Liu XH, et al. Heterologous biosynthesis of spinosad: an omics-guided large polyketide synthase gene cluster reconstitution in Streptomyces. ACS Synth Biol, 2017, 6(6): 995-1005. DOI:10.1021/acssynbio.6b00330
|
| [54] |
王建强, 马淑涛. 16元大环内酯类抗生素的研究进展. 中国药学杂志, 2006, 41(24): 1841-1844. Wang JQ, Ma ST. Progress of 16-membered macrolide antibiotics. Chin Pharm J, 2006, 41(24): 1841-1844 (in Chinese). DOI:10.3321/j.issn:1001-2494.2006.24.001 |
| [55] |
Kirst HA. Semi-synthetic derivatives of 16-membered macrolide antibiotics. Prog Med Chem, 1994, 31: 265-295.
|
| [56] |
Arsic B, Barber J, Čikoš A, et al. 16-membered macrolide antibiotics: a review. Int J Antimicrob Agents, 2018, 51(3): 283-298. DOI:10.1016/j.ijantimicag.2017.05.020
|
| [57] |
Shi XG, Fawcett JP, Chen XY, et al. Structural identification of bitespiramycin metabolites in rat: a single oral dose study. Xenobiotica, 2005, 35(4): 343-358. DOI:10.1080/00498250500087580
|
| [58] |
Shi XG, Sun YM, Zhang YF, et al. Tissue distribution of bitespiramycin and spiramycin in rats. Acta Pharmacol Sin, 2004, 25(11): 1396-1401.
|
| [59] |
王以光, 姜洋, 赵小峰, 等. 可利霉素在抗结核分枝杆菌感染中的应用: 中国, 201511030787.7. 2018-02-13. Wang YG, Jiang Y, Zhao XF, et al. Application of kelimycin in Mycobacterium tuberculosis infection resistance: CN, 201511030787.7. 2018-02-13 (in Chinese). |
| [60] |
Tay MZ, Poh CM, Rénia L, et al. The trinity of COVID-19: immunity, inflammation and intervention. Nat Rev Immunol, 2020, 20(6): 363-374. DOI:10.1038/s41577-020-0311-8
|
| [61] |
Sun CH, Jiang W, Huang J, et al. Shengjimycins: a group of hybrid antibiotics, 4"-acylspiramycins. Actinomycetologica, 1999, 13(2): 120-125. DOI:10.3209/saj.13_120
|
| [62] |
王红远, 周红霞, 戴剑漉, 等. 必特霉素基因工程菌航天育种的研究. 药物生物技术, 2007, 14(1): 10-13. Wang HY, Zhou HX, Dai JL, et al. Studies on space-flight breeding of genetically engineered strain of biotechmycin. Chin J Pharm Biotechnol, 2007, 14(1): 10-13 (in Chinese). DOI:10.3969/j.issn.1005-8915.2007.01.003 |
| [63] |
戴剑漉, 李瑞芬, 武临专, 等. 新一代必特螺旋霉素基因工程菌的微波诱变. 中国抗生素杂志, 2009, 34(7): 406-410, 428. Dai JL, Li RF, Wu LZ, et al. Improvement of new generation of bitespiramycin producing strain by microwave radiation. Chin J Antibiot, 2009, 34(7): 406-410, 428 (in Chinese). |
| [64] |
Dai JL, Wang YG, Liu JJ, et al. The regulatory genes involved in spiramycin and bitespiramycin biosynthesis. Microbiol Res, 2020, 240: 126532. DOI:10.1016/j.micres.2020.126532
|
| [65] |
Ma CY, Zhou HX, Li JY, et al. Construction of 4″-isovalerylspiramycin-Ⅰ-producing strain by in-frame partial deletion of 3-O-acyltransferase gene in Streptomyces spiramyceticus WSJ-1, the bitespiramycin producer. Curr Microbiol, 2011, 62(1): 16-20. DOI:10.1007/s00284-010-9664-8
|
| [66] |
戴剑漉, 卢智黎, 林灵, 等. 利用异源正调控基因acyB2构建埃莎霉素Ⅰ高产菌株. 中国生物工程杂志, 2017, 37(3): 65-72. Dai JL, Lu ZL, Lin L, et al. Construction of the isomycin Ⅰ high-yield strain by introducing a heterologous positive regulatory gene acyB2. China Biotechnol, 2017, 37(3): 65-72 (in Chinese). |
| [67] |
戴剑漉, 张晓婷, 卢智黎, 等. 新型常压室温等离子体—紫外复合诱变选育埃莎霉素Ⅰ高产菌株. 中国抗生素杂志, 2018, 43(2): 182-188. Dai JL, Zhang XT, Lu ZL, et al. Breeding of high isomycin-Ⅰ-producing strain by MPMS composite mutagenesis with plasma and UV. Chin J Antibiot, 2018, 43(2): 182-188 (in Chinese). DOI:10.3969/j.issn.1001-8689.2018.02.011 |
| [68] |
张晓婷, 张妍, 戴剑漉, 等. 利用CRISPR-Cas9系统构建新型异戊酰螺旋霉素Ⅰ产生菌. 生物工程学报, 2019, 35(3): 472-481. Zhang XT, Zhang Y, Dai JL, et al. Construction of a new isovalerylspiramycin-Ⅰ-producing strain by CRISPR-Cas9 system. Chin J Biotech, 2019, 35(3): 472-481 (in Chinese). |
 2021, Vol. 37
2021, Vol. 37