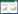中国科学院微生物研究所、中国微生物学会主办
文章信息
- 李金根, 刘倩, 刘德飞, 武敏, 田朝光
- Li Jingen, Liu Qian, Liu Defei, Wu Min, Chaoguang Tian
- 丝状真菌代谢工程研究进展
- Advances in metabolic engineering of filamentous fungi
- 生物工程学报, 2021, 37(5): 1637-1658
- Chinese Journal of Biotechnology, 2021, 37(5): 1637-1658
- 10.13345/j.cjb.200723
-
文章历史
- Received: November 11, 2020
- Accepted: January 29, 2021
2. 国家合成生物技术创新中心,天津 300308
2. National Technology Innovation Center of Synthetic Biology, Tianjin 300308, China
丝状真菌(Filamentous fungi) 作为重要的工业发酵微生物,具有能够利用廉价原料发酵、蛋白质分泌能力强等优势。目前,丝状真菌已经被用于生产多种大宗发酵产品,如工业蛋白质(纤维素酶、糖化酶等),大宗有机酸(柠檬酸、衣康酸等) 以及抗生素等次级代谢产物。由于丝状真菌遗传背景复杂,天然菌种的诱变筛选是发酵菌种选育的重要手段,在抗生素等多个产品的菌种选育中取得了巨大进步。1991年Bailey等率先提出了代谢工程的概念,旨在通过遗传工程系统修饰特定的生化反应或引进新的生化反应,以改善细胞性能,实现更高效的生物化学转化,提升菌种生产能力[1]。随后,这一概念逐渐发展为一个全新领域——代谢工程学科。
与前期基因工程不同的是代谢工程更加关注细胞代谢途径改造过程中的整体性、系统性和迭代性。近年来,代谢工程研究被大量用于丝状真菌改造,极大地提高了研究者认识、设计和改造细胞代谢的能力,在此过程中发展的大量使能技术,促进了高性能工业丝状真菌细胞工厂的构建,拓宽了丝状真菌的研究和应用领域(图 1)。2001年,模式丝状真菌粗糙脉孢菌Neurospora crassa基因组序列的公布[2-3],标志着丝状真菌“后基因组时代”的开启,其后大量丝状真菌基因组完成测序,基因组序列信息及注释信息促进了研究者对微生物生理和代谢特性在全局水平上的认识,加快了丝状真菌代谢途径改造,进一步推动了丝状真菌在工业发酵中的应用。
|
| 图 1 丝状真菌代谢工程及使能技术发展概图 Fig. 1 Overview of the development of metabolic engineering and enabling technologies for filamentous fungi. |
| |
鉴于丝状真菌在工业发酵中的独特优势,丝状真菌代谢工程研究已成为现代工业生物技术领域重要研究方向之一,本文将首先对丝状真菌代谢工程改造中的使能技术进行回顾,随后简述丝状真菌代谢工程在微生物细胞工厂创建中的应用,并讨论丝状真菌代谢工程中的关键问题并展望其未来发展,以期为丝状真菌代谢工程研究提供参考。
1 丝状真菌代谢工程中的使能技术 1.1 丝状真菌遗传转化技术相比于大肠杆菌、酿酒酵母等单细胞微生物,丝状真菌遗传背景较为复杂,形态特异,加上细胞壁较厚,导入外源基因片段较为困难。因此发展高效的遗传转化技术是实现丝状真菌代谢工程改造的前提。近年来,已开发了多种遗传转化方法用于丝状真菌的遗传改造,包括:CaCl2/聚乙二醇(PEG) 介导原生质体法(Protoplast-mediated transformation,PMT)、根癌农杆菌介导的转化方法(Agrobacterium tumefaciens-mediated transformation,ATMT)、电穿孔转化方法(Electroporation transformation,EP)、以及基因枪法(Biolistic transformation,BT)。由于基因枪转化方法操作比较复杂,且对设备的要求较高,现在前3种转化方法在实验室中最为常用,经过不断优化后其转化效率均得到了显著提高(表 1)。
| Methoda | Principle | Advantage | Disadvantage | Fungal species |
| PMT | Transformation of DNA fragment into protoplast with the aid of CaCl2 and PEG | Efficient; suitable for most fungi; high copy number of inserted DNA | Requires special lytic enzyme; each step of protoplast preparation is optimized; regeneration of protoplast was needed | A. niger T. reesei A. oryzae N. crassa M. thermophila[4-8] |
| ATMT | DNA fragment is integrated into T-vector and transformed into blastospore by A. tumefaciens | Efficient; copy number of DNA is inserted low; high homologous recombination efficiency | Time-consuming and tedious; it requires to prepare binary vectors; some fungi was sensive to acetosyringone | A. awamori A. fumigatus M. thermophila M. anisopliae[9-12] |
| EP | Uptake of DNA fragment is mediated by reversible permeabilization of biomembranes induced by s electric pulses | High copy number of inserted DNA; more convenient; different cell types (spores, blastospore and protoplast) can be used | Low efficient; special equipment was needed; buffer conditions need to be optimized | A. niger A. oryzae N. crassa[13-15] |
| aPMT: protoplast-mediated transformation; ATMT: Agrobacterium tumefaciens-mediated transformation; EP: electroporation transformation. | ||||
CaCl2/PEG介导的原生质体转化方法(PMT)一般以原生质体为感受态,在CaCl2和PEG的作用下将外源DNA片段转入细胞中。PMT方法于1978年开发,最先用于酿酒酵母Saccharomyces cerevisiae转化[16],而后逐渐用于丝状真菌,如黑曲霉Aspergillus niger,里氏木霉Tricherdoma reesei,嗜热毁丝霉Myceliophthora thermophila等[4-6]。该方法转化效率较高,且能够同时转化多个DNA片段,导入的基因片段通过宿主自身修复方式随机插入或定点插入至宿主基因组。通过PMT的方法可在宿主基因组中插入多拷贝目标基因片段。但PMT方法存在一定限制,由于丝状真菌细胞壁成分和结构的不同,很多菌株原生质体制备较为困难,并且原生质体不易储存,一般需要现用现制备。另外,原生质体再生效率较低且其条件具有菌株特异性。
根瘤农杆菌介导法(ATMT) 最先用于植物的遗传转化,经过改良后用于丝状真菌,如泡盛曲霉Aspergillus awamori、烟曲霉Aspergillus fumigatus、嗜热毁丝霉M. thermophila、绿僵菌Metarhizium anisopliae[9-12, 17]。但较为遗憾的是,ATMT方法不适合用于重要工业丝状真菌黑曲霉。ATMT方法主要利用根癌农杆菌对丝状真菌芽生孢子的侵染,将外源T-DNA转入宿主细胞中。ATMT方法相比于PMT法具有诸多优势,如ATMT方法不需要制备原生质体,且该技术以丝状真菌孢子为转化对象,且假阳性转化子较少;导入的外源基因拷贝较低,容易获得单拷贝外源基因插入的转化子,且可介导大片段DNA的导入。据报道在泡盛曲霉遗传转化中,ATMT方法的转化效率为PMT方法的600倍[9]。但ATMT方法耗时较长,需要制备含有目标T-DNA的根瘤农杆菌,每次仅能导入单个目标DNA片段,另外,一些丝状真菌对ATMT方法所用到的乙酰丁香酮较为敏感,导致转化效率较低。
电穿孔转化(EP) 方法利用瞬时高效电脉冲在生物膜上形成短暂通道,促进目标DNA的导入,该方法最早应用于酿酒酵母转化[18],而后逐渐应用于丝状真菌,如粗糙脉孢菌N. crassa等[13]。EP方法会对细胞造成高度损伤,以原生质体为转化对象时,死亡率可达50%以上。经过不断优化改良后,成功实现了以丝状真菌分生孢子感受态作为转化对象,简化了操作步骤并提高了转化效率。
丝状真菌代谢工程改造中一般需要对宿主细胞进行迭代改造,以改善其细胞性能,满足高效生产目标产物的目的。但多数丝状真菌缺乏自主复制质粒或人工染色体,而且导入的筛选标记基因随外源基因整合至宿主基因组中,因此充足的筛选标记基因对丝状真菌的代谢途径改造至关重要。丝状真菌遗传转化筛选标记主要包括营养缺陷型标记基因,如乳清酸核苷-5-磷酸脱羧酶pyr-4、色氨酸生物合成酶基因trp-1,乙酰胺酶基因amds 等,以及药物抗性标记基因,如潮霉素B抗性基因hph、博来霉素抗性基因ble、寡霉素抗性基因oliC、benomyl 抗性基因bml、博来霉素或遗传霉素(G418) 抗性基因neo、草甘膦抗性基因bar、诺尔斯菌素抗性基因nat等。但值得注意的是,由于不同菌株遗传背景的特异性,并非每种筛选标记都可以适合所有丝状真菌的遗传转化。潮霉素B抗性基因hph常用于工业真菌黑曲霉和里氏木霉的转化子筛选,但是在嗜热毁丝霉遗传转化,以hph为筛选基因,所获得转化子一般均为异核体,且假阳性率较高[12]。相反的是,草甘膦抗性基因bar 可作为嗜热毁丝霉遗传转化的筛选标记[12, 19],但不适合于黑曲霉。目前每种丝状真菌一般有2–3个筛选标记适合其转化子筛选,对同一菌株进行多轮遗传改造时,出现了筛选标记无法满足使用的情况,严重限制了丝状真菌代谢工程改造。基于此,发展抗性标记重复使用技术成为实现对丝状真菌代谢途径进行迭代改造的关键之一。Aigner团队利用Cre-loxP系统在里氏木霉实现了多轮基因操作的抗性筛选基因重复使用[20]。随后山东大学方诩团队对其进行了优化改良,在草酸青霉中实现了多基因操作中吡啶硫胺素抗性标签的多轮重复利用[21]。天津科技大学刘浩团队在黑曲霉中实现了潮霉素B抗性基因hph 的循环使用[22]。笔者团队开发了Camr技术(CRISPR-Cas-assisted marker recycling technology),交替使用筛选标记基因neo和bar,对嗜热毁丝霉基因组进行了多轮改造,最终实现了超过10个靶标基因的改造[23]。
1.2 丝状真菌基因敲除技术同源重组(Homologous recombination,HR)是生物体修复DNA突变的内在机制之一,构成了早期丝状真菌基因敲除技术的分子基础,即在外源DNA片段(如筛选标记基因) 两侧添加同源臂,通过遗传转化技术导入菌株后,与目标靶点发生同源重组,使得目标基因片段被外源DNA片段替代,以达到敲除目标基因的目的。基因敲除技术在改造丝状真菌基因组、研究菌丝发育、鉴定基因功能以及创制新型发酵菌株中发挥着重要作用。在丝状真菌中,Ku/Mus蛋白介导的非同源末端连接(Non-homologous ending-joining,NHEJ) 修复机制在DNA损伤修复中占据主导地位,外源DNA片段容易随机整合至其他基因组位点,导致同源重组发生频率非常低,限制了精准改造基因组的效率[24-25]。研究者通常采用失活Ku/Mus蛋白和加大同源臂的方式,来提升目标基因的敲除效率[12]。
研究表明,在基因组目标靶点上人为引入双链断裂,能够显著提升细胞的同源重组效率[26]。近年来,人工核酸内切酶技术相继用于基因组编辑,其中包括人工核酸酶介导的锌指核酸酶(Zinc finger nucleases,ZFNs) 技术、类转录激活因子效应物核酸酶(Transcription activator-like effector nucleases,TALENs) 技术和RNA介导的CRISPR/Cas (Clustered regulatory interspaced short palindromic repeats/CRISPR-associated protein,CRISPR/Cas) 技术[27-28]。利用核酸内切酶对DNA损伤后产生的双链断裂激活细胞内固有的非同源末端连接或同源重组两种不同的修复机制对损伤的DNA进行修复,从而实现对基因组的定点改造。核酸内切酶的使用极大提高了丝状真菌同源重组的效率,DNA片段两侧同源臂可由1–2 kb缩短至30 bp,为敲除载体的构建提供了便利[29]。
1.3 丝状真菌基因组编辑技术近几年,迅速发展的基于CRISPR/Cas的基因编辑以及基因表达调控技术为丝状真菌代谢工程提供了技术支撑。自2015年,基因组编辑技术已经相继应用于一些重要模式丝状真菌以及工业丝状真菌,包括里氏木霉、稻瘟病菌、黑曲霉、烟曲霉、米曲霉、粗糙脉孢菌、产黄青霉等[29-33]。笔者团队在嗜热毁丝霉挖掘了高效RNA聚合酶U6启动子,用于调控单向导RNA (Single guide RNA,sgRNA) 的转录,在嗜热毁丝霉中成功构建了基于CRISPR/Cas9和CRISPR/Cas12a (Cpf1) 的基因组编辑技术体系[23, 34],利用该体系可快速、高效地对毁丝霉基因组中的多基因同时进行编辑,其中单基因敲除的同源重组效率高达100%,双基因缺失的同源重组效率为61%−69%,极大推进了嗜热毁丝霉的代谢工程改造,目前已构建了蛋白质(纤维素酶、糖化酶)[35-36]和有机酸(苹果酸、富马酸) 高效合成的工程菌株[19, 37]。
1.4 组学技术2001年,第一株丝状真菌粗糙脉孢菌的完整基因组序列被公布,随后黑曲霉、里氏木霉等重要工业丝状真菌相继完成全基因组测序,标志着丝状真菌“后基因组时代”的来临。目前,完成全基因组测序的丝状真菌超过1 000种,且具有相对较好的注释信息。大量丝状真菌全基因组数据催生了比较基因组学、转录组学、蛋白质组学、代谢组学等多组学技术的发展。在此基础上,研究者能够从基因、RNA、蛋白质、代谢网络等多个层次分析丝状真菌的生理代谢,为丝状真菌代谢工程的发展创造了前所未有的机遇。
比较基因组学:通过对微生物基因组序列进行深入的比较研究,可以推测基因功能、表达调控机理和物种进化规律。进入后基因组时代后,越来越多的丝状真菌基因组序列及注释信息在各类数据库中公开,如NCBI (https://www.ncbi.nlm.nih.gov/)、AspDB (http://www.aspergillusgenome.org/)、FungiDB (https://fungidb.org/fungidb/app) 以及JGI (https://genome.jgi.doe.gov/portal/)等,为比较基因组分析提供了大量数据。Pel等利用比较基因组学分析,结合代谢网络的重构,预测了黑曲霉基因组中大量与DNA复制、物质转运以及蛋白质表达分泌等生理过程相关的基因[38]。Andersen研究组对23个曲霉基因组序列进行分析,预测了598个碳水化合物水解基因及其大量的次级代谢产物基因簇(估计每个菌种约含有73个)[39]。
转录组学:基因组水平分析细胞在特定条件下基因表达水平的技术。通过分析不同菌株或同一菌株在不同培养条件、不同时空的基因表达差异,有助于发现影响菌种发酵性能的关键基因,为代谢工程改造提供候选靶点。高密度基因芯片技术和二代测序技术(RNA-seq) 先后成为丝状真菌转录组学分析的主要工具。2009年,加州大学伯克利分校Glass实验室利用高密度DNA芯片分析了粗糙脉孢菌在不同条件下的转录组学数据,对纤维素降解丝状真菌进行了全基因组学水平的系统研究,预测了粗糙脉孢菌中主要纤维素酶和半纤维素酶基因及其调控因子[40]。近期,笔者团队分析了嗜热毁丝霉苹果酸高产菌和野生型菌株的转录数据,揭示糖转运蛋白是提高有机酸合成的关键靶点,过表达葡萄糖转运蛋白显著促进了葡萄糖和纤维素条件下的苹果酸合成[19]。
蛋白质组学:通过2D-聚丙烯酰胺凝胶电泳和质谱(GC/LS-MS) 等技术,系统研究细胞蛋白质图谱,能够获得蛋白质翻译加工及修饰、蛋白质间相互作用等信息。蛋白质是细胞中各类生化反应的直接参与者,从蛋白质水平系统研究微生物代谢,能够为丝状真菌改造提供更直接的参考。在丝状真菌遗传改造中,蛋白组学技术多用于分析丝状真菌分泌的蛋白质,如纤维素酶和糖化酶等。目前,已有多个工业丝状真菌的分泌蛋白质组分析报道,包括黑曲霉、里氏木霉、草酸青霉、嗜热毁丝霉等[41-46],鉴定了诸多生物质降解相关水解酶。Christopher等利用定量蛋白质组学技术分析了粗糙脉孢菌纤维素酶系的主要成分及比例,在总蛋白中纤维二糖水解酶占53%,纤维素内切酶占13%,外切酶占5.9%,揭示在纤维素降解过程中纤维二糖水解酶占据主导地位,为改善粗糙脉孢菌酶系进而提升纤维素降解效率的遗传改造提供了重要参考[47]。
代谢组学:利用核磁共振(NMR) 以及质谱(MS、GC-MS和GC-MS) 等对胞内代谢物质进行高通量定量分析的技术。胞内代谢物浓度直接和生物反应相关,代谢组学更能反映细胞内代谢的动态变化,为微生物代谢工程改造及其发酵过程优化提供依据。Askenazi等利用代谢组学及转录组学技术分析了土曲霉在合成洛伐他汀过程中的细胞生理变化,并根据代谢组数据提出改造策略,使其产量提高50%[48]。Akihiko Kondo研究组利用代谢组学分析了米曲霉中线粒体丙酮酸转运蛋白失活对细胞代谢的扰动,而后通过强化乳酸合成途径,显著提高了宿主的乳酸合成水平[49]。
代谢通量组学:一般包含以同位素标记的碳源或氮源为底物的菌株培养,胞内同位素标记的代谢物分析,整合代谢网络模型计算等步骤。代谢通量组学(又称代谢流分析) 相比于代谢组学更能反映细胞内代谢物动态流向。Knuf等通过代谢流分析,发现米曲霉细胞质中的苹果酸含量与副产物丁二酸的合成呈正相关,并计算出了细胞质中以l-苹果酸为底物合成丁二酸的通量,为加强l-苹果酸合成,降低副产物丁二酸提供了思路[50]。Noorman团队分析了黑曲霉糖化酶高产菌株与野生型菌株的代谢通量,发现了黑曲霉胞内能量水平与糖化酶的高效合成相关[51]。Nielsen研究组利用代谢流技术分析了黑曲霉草酸合成途径敲除对菌株代谢的影响,提出草酰乙酸水解酶是黑曲霉柠檬酸高产菌株中消除草酸合成的关键靶点[52]。
多组学整合分析:结合基因组学、转录组学、蛋白质组学等多组学技术,从不同维度分析微生物代谢,更真实地反映细胞的时空生理与调控机制。吕雪峰研究组结合转录组学和蛋白质组学技术,鉴定了土曲霉中衣康酸降解途径关键酶基因(IctA、IchA和cclA),为提高衣康酸合成的代谢工程改造提供了新靶点[53]。笔者团队综合利用转录组学和蛋白质组学分析了嗜热毁丝霉野生型菌株与调控因子clr-4突变体在纤维素条件下的响应,解析了转录因子Clr-4在丝状真菌纤维素酶分泌的调控机制[36]。
1.5 丝状真菌代谢网络模型构建随着大量组学数据的产出,基因组尺度代谢网络模型(Genome-scale metabolic network models,GEMs) 已成为系统生物学研究强有力的工具[54]。GEMs是通过整合基因组学、蛋白质组学等数据,基于反应计量关系和质量平衡方程进行建模,是基因-蛋白质-反应关系的集合[55]。GEMs的构建可以从细胞全局代谢网络角度对微生物代谢问题进行探究。
丝状真菌作为发酵工业重要微生物,是工业酶制剂、有机酸和抗生素生产的重要体系,其GEMs的构建工作也得到了重视。2007年Sun等根据两株黑曲霉基因组注释信息,首次构建了黑曲霉的GEM[56]。随后Andersen等[57]基于文献信息、生理生化数据和基因组注释信息,构建了更加完善的黑曲霉GEM iMA871。利用此模型成功预测了黑曲霉生物量得率和草酸、柠檬酸的产量,并模拟了黑曲霉产柠檬酸过程中的胞内代谢物通量和合成电子传递链的变化。时至今日,研究者已对黑曲霉GEM进行了多次完善[58-60]。目前,其他丝状真菌,如粗糙脉孢菌、米曲霉、青霉菌、里氏木霉等,都已经相继建立了GEM的初级版本。
基于GEMs指导的菌株设计和优化,从全细胞水平指导代谢工程改造,通过计算机设计模拟,提出改造策略,并预测改造基因靶点,从而寻找最佳的优化策略,为新型菌株的构建提供思路。目前GEM在丝状真菌代谢工程研究改造方面总体处在初期阶段,但已经开启了有益尝试。Lu等[61]使用黑曲霉GEM iHL1210,并结合多组学数据对黑曲霉在氧限制情况下产酶能力提高的分子机理进行了分析。结果表明在氧限制条件下,糖酵解途径代谢通量增加,为菌体在限氧环境下提供必要的前体物质和能量。同时,乙醛酸支路通量增加,可以减少三羧酸循环(Tricarboxylic acid cycle,TCA cycle) 中NADH的形成,利于限氧条件下细胞还原力的平衡。Sui等[62]结合黑曲霉GEM对代谢物通量进行模拟,通过辅因子工程,增加细胞质中NADPH,从而提高黑曲霉糖化酶产量。Pakula等利用里氏木霉GEM预测了该菌的纤维素酶的产率,并提出硫同化和半胱氨酸代谢是提高纤维素酶产量的潜在靶点[63]。为该菌蛋白质生产从代谢网络角度提供了新的视角。
与传统代谢工程策略相比,GEM指导的代谢途径设计提供包括基因敲除、基因表达调控、新途径构建、外源途径添加等工程策略[64],实现产品从无到有,从有到优的代谢改造过程。GEM的出现使得传统代谢工程向理性设计方向转变。丝状真菌GEM的发展和应用仍方兴未艾,随着GEM的构建方法的完善,多组学数据的整合和其准确性、有效性的提升,GEM在丝状真菌代谢工程改造中的应用会更加广泛。
2 丝状真菌代谢工程在构建发酵菌种中的应用近年来,微生物代谢工程使能技术的发展极大地推动了丝状真菌细胞工厂的构建,接下来简述丝状真菌代谢工程在生产有机酸(柠檬酸、衣康酸及苹果酸),蛋白质及次级代谢物等生物基产品方面的进展。
2.1 有机酸柠檬酸(Citric acid) 是三羧酸循环的中间产物,是具有重要商业价值的有机酸,被广泛应用于食品、医药、洗涤剂和化妆品等领域。1784年瑞典化学家Carl Scheele首次从柠檬汁分离出柠檬酸。直到1891年Wehmer等发现霉菌能够生产有机酸,但由于菌种易退化、易染菌等原因未能实现生产。随后,柠檬酸首次被认定为霉菌代谢的产物。1913年,Zahorsky获得了第一个用黑曲霉生产柠檬酸的发明专利。1917年,Currie发现黑曲霉可以在高浓度糖培养基上(初始pH 2.5–3.5) 大量合成柠檬酸,进而实现了柠檬酸的工业化发酵[65],自此掀起了以黑曲霉为底盘细胞生产柠檬酸的研究热潮。
在细胞中糖酵解途径形成的丙酮酸进入线粒体后,通过三羧酸循环合成柠檬酸。柠檬酸的理论转化率为1 mol/mol葡萄糖。每产生一分子柠檬酸,释放一分子ATP和3分子NADH,过剩的NADH在呼吸链中被氧化[66]。诱变筛选在黑曲霉柠檬酸工业菌种选育过程中举足轻重,通过诱变筛选,菌种发酵性能取得了巨大进步,为发酵法生产柠檬酸达到每年近200万t规模奠定了核心基础。进入后基因组时代后,丝状真菌遗传转化技术、基因组编辑技术及其代谢工程改造策略的开发进一步推进了黑曲霉柠檬酸合成菌株的改造及优化。近年来,黑曲霉柠檬酸合成菌株的代谢工程改造策略主要包含以下4种:(1) 提升碳源利用,强化发酵原料(玉米淀粉) 的降解。天津科技大学王德培团队在黑曲霉中过表达葡糖淀粉酶基因glaA,柠檬酸产量提高16.9%[67]。(2) 提高胞内前体物质:线粒体乙酰辅酶A和草酰乙酸是柠檬酸合成前体,Nielsen团队证实强化黑曲霉草酰乙酸的合成能够显著促进柠檬酸的合成[68]。(3) 降低反馈抑制:糖酵解途径中的关键酶受到反馈调控作用,如己糖激酶(HXK) 活性受到海藻糖-6-磷酸的抑制,果糖-6-磷酸激酶(FPK) 活性受ATP以及柠檬酸的抑制,因此解除糖酵解途径中的反馈抑制作用能够显著提高柠檬酸的合成。Capuder等利用蛋白质工程技术对FPK进行了改造,获得的突变体mtPFKA10 (T89D) 解除了柠檬酸和ATP的抑制作用,将突变体蛋白导入黑曲霉后其柠檬酸产量相比于出发菌株提高了70%[69]。(4) 调控细胞呼吸作用:黑曲霉合成柠檬酸过程中伴随了净ATP和NADH产生,胞内过量的ATP会抑制呼吸作用,导致细胞中NAD+含量降低,进而影响细胞代谢及产物合成。Hou等通过过表达交替氧化酶基因aox1降低呼吸作用合成ATP的效率,提升了柠檬酸的产量[70]。
经过多年来持续的菌株改造及发酵条件优化,在工业发酵中黑曲霉柠檬酸转化率已经接近理论水平,产量可以达到170 g/L,工业生产发酵周期缩短为72 h,甚至更短。现在常规代谢工程改造对柠檬酸工业菌株的提升空间有限,要继续提升柠檬酸发酵水平,需要设计更高效的新途径。
衣康酸(Itaconic acid) 学名为甲叉琥珀酸、亚甲基丁二酸,是一种不饱和二元有机酸,具有活泼的化学性质,是化学合成和化工生产的重要工业原料。目前衣康酸全球年产量达到8–10万t,我国是世界上衣康酸的最大生产国和出口国。衣康酸生产菌株主要包括土曲霉A. terreus、衣康酸曲霉Aspergillus itaconicus、假丝酵母Candida mycoderma、玉米黑粉菌Ustilago maydis、查尔斯青霉Penicillum charlesii和黑曲霉A. niger。但是,只有土曲霉的产酸能力可以达到工业化生产的标准。1931年,日本学者Kinoshita报道了利用A. itaconicus生产衣康酸的研究[71],随后,Raistrick成功利用A. terreus进行衣康酸的合成[72]。Bentley等通过14C标记示踪研究了衣康酸合成途径,他们发现在微生物体内衣康酸主要通过异柠檬酸脱羧形成[73],这为后期通过代谢工程优化衣康酸合成途径奠定了基础。Park等在2008年从土曲霉中第一次克隆了衣康酸合成关键酶基因——顺式乌头酸脱羧酶基因cds。之后,荷兰学者Li等通过基因芯片技术鉴定了土曲霉中衣康酸合成相关基因。在土曲霉中,柠檬酸在乌头酸酶的作用下形成乌头酸,而后在细胞质中脱羧形成衣康酸,但是柠檬酸和乌头酸作为TCA途径的中间物质一般存在于线粒体,因此前体物质的供应是丝状真菌合成衣康酸的关键因素。2011年,该团队首次在黑曲霉中构建了衣康酸代谢途径,将线粒体乌头酸转运蛋白、乌头酸脱羧酶以及衣康酸转运蛋白基因导入黑曲霉后实现了衣康酸的合成,但是其产量(0.05 g/L) 与土曲霉相差甚远[74],随后该团队通过加强细胞糖酵解途径,同时提高血红蛋白含量促进了土曲霉衣康酸的合成,但其产量仅仅达到1.96 g/L[75]。2014年,中国科学院青岛生物能源与过程所刘建军和吕雪峰团队将土曲霉中3个关键基因依次进行了过表达,发现衣康酸合成均有所提高,但是每个基因起到的作用有所不同,其中过表达乌头酸脱羧酶基因后衣康酸产量提高最为显著[76],同年该团队在土曲霉中过表达淀粉酶基因,以玉米淀粉水解为发酵原料时,衣康酸产量达到80 g/L[77]。近期,荷兰科学家Hossain通过转录组学分析发现在黑曲霉细胞质中存在第二个柠檬酸合酶,并通过过表达细胞质柠檬酸合酶提升了细胞质柠檬酸浓度,衣康酸产量提高至26.2 g/L,为黑曲霉合成衣康酸的最高水平[78]。除此之外,玉米黑粉菌也是天然的衣康酸生产菌株,其合成途径与土曲霉不同。在黑粉菌中顺式乌头酸首先经过异构酶形成反式乌头酸,而后经过脱羧形成衣康酸,目前玉米黑粉菌衣康酸的最高水平为53 g/L。
此外,发酵条件对土曲霉衣康酸生产具有重要影响[79-80],如pH [81]、无机盐离子浓度[82]、溶氧[83]等。德国Kuenz团队通过优化发酵培养基的无机盐离子浓度,在微好氧条件下衣康酸产量达到87.7 g/L,生产强度为1.88 g/(L·h)[83]。2017年,该团队致力于开发具有产业化水平的衣康酸发酵技术,研究发现初始培养基的pH对菌丝生长及其衣康酸的合成具有重要影响,在最适条件下衣康酸产量达到160 g/L,这是迄今为止已报道的衣康酸的最高水平。
苹果酸(Malic acid) 又名2-羟基丁二酸,为白色晶体或粉末,因1785年由Scheele从未成熟的苹果中分离而得名。苹果酸具有口感柔和等特点,在食品、医药等领域具有广泛的用途,已成为继柠檬酸、乳酸之后用量排第3位的食品酸味剂。丝状真菌在l-苹果酸发酵方面具有显著的优势,目前已在嗜热毁丝霉、米曲霉、黑曲霉、青霉、黑粉菌等丝状真菌体系进行合成[7, 19, 22, 84-87],多体系同时研发,有望推进发酵法合成苹果酸的产业化进程(表 2)。
| Fungal species | Strategy for metabolic engineering | Feedstock | Fermentation | Titer (g/L) | Productivity (g/L/h) | Yield (g/g) | References |
| Aspergillus oryzae NRRL 3488 | Overexpressing native pyruvate carboxylase PYC, malate dehydrogenase MDH and a C4-dicarboxylate transporter Mae | Glucose | 2 l-fermentor | 154 | 0.94 | 1.03 | [84] |
| Aspergillus oryzae NRRL 3488 | Enhanceing rTCA pathway (overexpressing Aopyc Aomdh, Ecppc, and Ecpck), improving C4-dicarboxylate export (overexpressing Aomae and Spmae1) and then overexpressing pfk | Glucose | 3 l-fermentor | 165 | 1.38 | 0.68 | [7] |
| Aspergillus oryzae GAAF41 | Improving pyruvate carboxylation (overexpressing pyc in the cytosol and mitochondria) and glyoxylate bypass, downregulating TCA cycle, and then overexpressing sfc1 and Linox | Corn starch | Shake-flask | 117.2 | 1.17 | 0.9 | [85] |
| Aspergillus carbonarius ITEM5010 | Overexpressing a native C4-dicarboxylate transporter Dct | Glucose | Shake-flask | 32 | – | 0.404 | [94] |
| wheat straw hydrolysate | Shake-flask | 20 | – | 0.179 | |||
| Aspergillis niger ATCC 1015 | Deleting oxaloacetate acetylhydrolase gene oahA and overexpressing pyc, mdh3 and C4t318 | Glucose | 2 l-fermentor | 201.24 | 0.93 | 0.945 | [22] |
| Aspergillis niger S575 | Eliminating the accumulation of citric acid by deleting cexA and enhancing glycolytic flux by overexpressing mstC, hxkA, pfkA and pkiA | Glucose | 2 l-fermentor | 201.13 | 1.05 | 1.22 | [95] |
| Ustilago trichophora TZ1 | Overexpression of PYC, MDH (cytoplasmic Mdh1 and mitochondrial Mdh2) and two malic acid transporters Ssu1 and Ssu2 | glycerol | 2.5 l-fermentor | 134 | 0.56 | 0.42 | [87] |
| Myceliophthora thermophila ATCC 42464 | Overexpressing pyruvate carboxylase PYC and a C4-dicarboxylate transporter Mae | Avicel | 5 l-fermentor | 181 | 0.87 | 0.99 | [19] |
| Corncob | 5 l-fermentor | 105 | 0.66 | 0.38 | |||
| Myceliophthora thermophila JG207 | Enhancing cellobiose uptake by overexpressing cdt-1 and phosphorolytic pathway and deleting bgl2 and bgl3 | Cellobiose | Shake-flask | 101.2 | – | 1.35 | [96] |
| Avicel | Shake-flask | 77.4 | – | 1.03 |
1962年,美国Abe团队首先报道了利用黄曲霉合成苹果酸的研究,并申请了第一个利用丝状真菌生产苹果酸的专利。1988年,Goldberg等通过13C标记对黄曲霉中苹果酸合成途径进行了分析,发现草酰乙酸是苹果酸合成的关键前体物质[88]。随后(1990年和1991年),该团队探讨了曲霉属菌株中丙酮酸羧化酶的亚细胞定位与苹果酸合成之间的关系[89]。在一些真核生物细胞质中,在丙酮酸羧化酶的作用下,通过固定一分子CO2将丙酮酸转化生成草酰乙酸,之后由苹果酸脱氢酶催化形成苹果酸。ATP与辅因子NADH在整个合成过程中是平衡的,加上二氧化碳的固定,使得苹果酸的理论转化率达到2 mol/mol葡萄糖。在微生物代谢工程过程中,一般以此途径为基础对目标菌株进行改造,以期获得高产工业菌株。Brown团队利用代谢工程手段对A. oryzae NRRL 3488进行了改造,其苹果酸产量相比于出发菌得到了显著提高。在2 L发酵罐中,转化子(2103a-68) 利用葡萄糖为碳源在164 h内苹果酸产量达到了154 g/L,产率为0.94 g/(L·h),转化率达到理论值的69% (1.38 mol/mol葡萄糖)。
在苹果酸合成过程中,碳酸钙与苹果酸反应将培养基的pH保持在6.5左右,释放的二氧化碳可以作为丙酮酸羧化酶的底物。培养基中碳酸钙的浓度对苹果酸合成具有重要影响,形成的苹果酸钙更有利于发酵液中苹果酸的积累。在此过程中,Ca2+作为重要的信号分子,也可能有着重要调控作用,但具体调控机理仍需要进一步的研究。除此之外,瑞典科学家Nielsen等研究了不同氮源对米曲霉苹果酸合成的影响,通过转录组学分析发现,当处于氮饥饿状态时,米曲霉细胞质中苹果酸合成相关基因均上调,其中丙酮酸羧化酶受到底物及转录水平的调节,被鉴定为细胞质苹果酸合成途径的限速酶[90]。
为了提高苹果酸发酵水平,降低发酵成本,研究者对苹果酸发酵培养基进行了多项优化。瑞典科学家Nielsen等利用A. oryzae DSM1863以木糖或甘油为原料进行发酵,其苹果酸产量分别达到了45.4 g/L和39.4 g/L。当木糖浓度为4.5 g/L,副产物富马酸产量降到了最低,而在甘油生长条件下,其富马酸产量显著上升[50]。2015年,美国科学家West分析了不同黑曲霉菌株以甘油为底物苹果酸产量,其中黑曲霉ATCC 12486的苹果酸产量达到24 g/L[91]。自此之后,以工业粗甘油为底物合成苹果酸得到了越来越多的关注。2016年,德国Wierckx团队对74株Ustilaginaceae菌株进行筛选获得了苹果酸高产菌株Ustilago trichophora TZ1,通过适应进化后,该菌株在甘油条件下的生长速率和苹果酸合成效率分别提高2.5倍和6.6倍,苹果酸的最终产量达到196 g/L,转化率为0.82 g/L,但是其较低的生产效率(0.39 g/(L·h)) 限制了其规模化生产[92]。随后该团队优化了苹果酸发酵条件,合理调整初始碳氮比例并改善中和剂,U. trichophora TZ1利用甘油为底物合成苹果酸的生产效率增加到(0.74±0.06) g/(L·h),但以粗甘油为碳源时,其苹果酸产量急剧降低[93]。
在国内,多家单位利用丝状真菌体系,通过代谢工程研发了苹果酸发酵菌种。江南大学刘龙团队以米曲霉体系为出发菌株,通过过表达丙酮酸羧化酶和苹果酸脱氢酶强化了还原型TCA途径,苹果酸产量达到42.3 g/L,而后该团队进一步加强了糖酵解途径及苹果酸转运能力,使得菌株的苹果酸产量进一步提升到165 g/L[7]。天津科技大学刘浩团队利用黑曲霉体系,通过强化rTCA途径及苹果酸转运系统,结合代谢支路草酸合成途径的敲除,获得了苹果酸高产菌株,在发酵罐中苹果酸产量达到201.24 g/L[22]。笔者团队以纤维素降解高温真菌嗜热毁丝霉为体系,通过强化rTCA途径及l-苹果酸转运模块,同时构建CO2浓缩转运模块,获得发酵法生产苹果酸的嗜热真菌细胞工厂。该体系发酵温度为45–50 ℃,相比于中温的曲霉系统(34 ℃发酵) 可以显著节省冷却用能,降低能耗成本。而且,该菌株不仅可以利用葡萄糖为底物高效合成苹果酸(发酵浓度超过200 g/L),而且能够直接以纤维素为原料进行发酵,苹果酸产量达到181 g/L,糖酸转化率为0.99 g/g[4],是目前以木质纤维素为原料直接发酵生产大宗有机酸的最高水平,一旦成功产业化,将是生物炼制领域的重要突破。
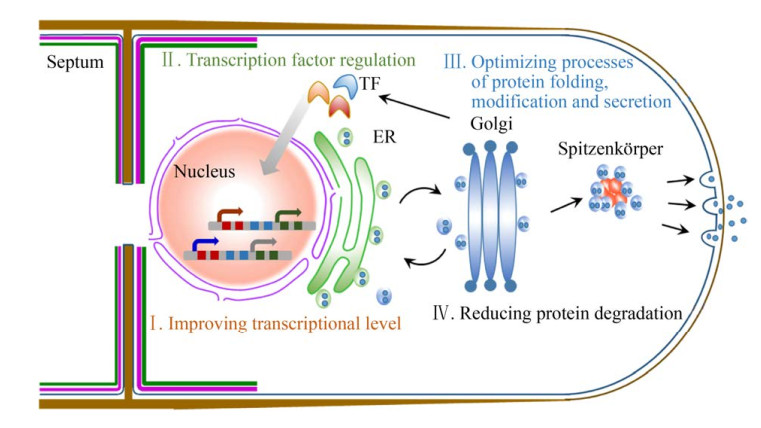
2.2 蛋白质丝状真菌具有成熟的蛋白分泌修饰系统,是工业酶制剂等蛋白质产品的重要生产者。近年来,随着代谢工程和分子生物学的快速发展,越来越多的丝状真菌如粗糙脉孢菌、黑曲霉、嗜热毁丝霉和里氏木霉开展了纤维素酶、糖化酶、抗体等蛋白质产品合成研究[2, 6, 97-99]。据报道丝状真菌工业菌种蛋白分泌量相当惊人,分泌量已超过100 g/L[6, 100-101]。在快速发展的后基因组时代,利用代谢组学、转录组学等技术,结合系统生物学方法深入解析丝状真菌细胞的蛋白分泌过程(图 2),极大地促进了丝状真菌蛋白质工业菌株的理性设计与改造[102]。
|
| 图 2 丝状真菌遗传改造提升蛋白质合成 Fig. 2 Genetic engineering of filamentous fungi for improved protein production. TF: transcription factor; ER: endoplasmic reticulum; Golgi: Golgi apparatus. |
| |
高效表达元件策略:在构建蛋白质细胞工厂过程中,选用丝状真菌本身强启动子增强目标蛋白基因的转录水平,是提高目标蛋白质合成的有效策略之一。目前所使用的启动子主要包括组成型启动子和诱导性启动子。进入后基因组时代后,不同条件下的转录组数据为研究者选取高效启动子提供了有力的参考。在曲霉中,淀粉糖化酶基因启动子PglaA、转录延伸因子启动子Ptef1、3-磷酸甘油脱氢酶基因启动子PgpdA、酸性磷酸酶基因启动子PpacA等常被认为是具有很强启动效率的组成型启动子,而木聚糖内切酶基因启动子PexlA是很好的诱导型启动子,在诱导条件下PexlA的启动效率为PglaA的3倍[103]。在一些纤维素酶分泌菌株中,如里氏木霉、粗糙脉孢菌、草酸青霉、嗜热毁丝霉等,纤维素酶基因启动子常用于目标蛋白质表达,如纤维二糖水解酶基因启动子PcbhⅠ和PcbhⅡ,内切葡聚糖酶基因启动子Pegl2等。另外,为了满足高水平合成目标蛋白的需要,研究者还开发了多拷贝启动子、启动子组合模块、人工合成启动子等策略,用于提升目标蛋白的合成水平。
转录调控机器改造:在丝状真菌中以转录因子为核心的调控机器在蛋白质表达调控网络中处于主要地位,如丝状真菌纤维素酶的产量主要是在转录层面上受到严谨的调控,目前改造转录调控网络成为增强蛋白质合成的重要策略。里氏木霉、曲霉、粗糙脉孢菌以及嗜热毁丝霉等重要工业丝状真菌中的关键纤维素酶转录因子陆续被报道,包括激活因子(Clr1、Clr2、XInR、Ace2等) 和抑制因子(Cre1、Ace1等),为增强丝状真菌蛋白质表达提供了诸多靶点。在里氏木霉中,山东大学刘巍峰团队通过过表达转录因子Xyr1能够使里氏木霉摆脱对诱导碳源的依赖,在葡萄糖条件下也能高水平地表达纤维素酶[104]。奥地利维也纳技术大学Kubicek实验室报道了转录激活因子(Xry1、Ace3和Vel1) 以及甲基化转移酶Lae1的过表达均能够增加菌株纤维素酶的产量[105]。上海交通大学白凤武、赵心清团队通过构建人工锌指蛋白库并转化至纤维素酶高产突变株里氏木霉RUT-C30,获得了U3突变株,其纤维素酶活力提高55%,β-葡萄糖苷酶活力提升了8.1倍[106],随后2017年该团队将Cre1的DNA结合域和Xyr1进行融合,使里氏木霉在葡萄糖培养基中的纤维素酶产量提高了12.75倍[107]。在粗糙脉孢菌中,美国加州大学伯克利分校Glass实验室发现2个全新纤维素酶调控元件Clr-1和Clr-2,该团队在粗糙脉孢菌过表达clr-2能够显著提高纤维素酶的活力;过表达Xlr-1突变型(V828F) 能显著提高木聚糖酶的表达水平[108]。在嗜热毁丝霉中,深圳大学刘刚团队利用RNA干扰技术沉默了Mtcre1基因后,菌株的纤维素酶产量得到了显著提高[109]。研究发现,转录因子可以通过相互作用,形成调控机器,协同调控蛋白(如纤维素酶) 的表达。山东大学曲音波团队利用转录因子单突变菌株库,从中筛选到包括ClrB、CreA、XlnR、Ace1、AmyR等20个对草酸青霉纤维素酶的表达起到调控作用的转录因子,并利用分子生物学手段对纤维素酶表达调控网络涉及的多个转录因子进行了协同改造[110]。该研究表明,clrB和xlnR的同时过表达、xlnR 的单独过表达、amyR和creA的双基因缺失均能实现菌株纤维素酶表达量的增加,将草酸青霉的clrB进行过表达,同时敲除creA和bgl2,纤维素酶活力和蛋白质分泌量分别提高了20倍和10倍[111]。笔者团队分析了嗜热毁丝霉在微晶纤维素、葡萄糖和无碳源条件下的转录组,鉴定了143个结晶纤维素基因,并对一个全新的纤维素酶转录激活因子MtClr-4开展功能研究[36],结果表明该元件直接调控2个关键的转录因子MtClr-2和MtXyr-1,说明在嗜热毁丝霉纤维素酶的表达分泌受到多种调控因子的协同控制。该团队利用基因组编辑技术对9个纤维素酶表达分泌相关基因进行了调控,包括cre-1、Mtxyr-1、hcr-1等转录因子,使菌株的蛋白质分泌水平与纤维素酶活分别提高9倍和18.5倍[23]。
增强蛋白质折叠修饰策略:在翻译水平上,加强蛋白质的正确折叠及其在内质网、高尔基体中正确修饰和分泌是提高目标蛋白生产的又一有效策略。其中内质网应激过程中未折叠蛋白质反应(Unfolded protein response,UPR) 以及ER偶联的蛋白质降解途径(ER-associated protein degradation,ERAD) 研究较多,途径中多个关键基因被鉴定,为提高宿主蛋白质分泌的工程改造提供诸多靶点。在黑曲霉中过表达UPR相关基因,如hacA、sstC、pdiA、gptA、ostA、eroA或cnxA等,能够显著提高突变体异源蛋白的产量[102]。其中,将UPR中心激活因子HacA组成型表达后,异源牛凝乳酶产量提高近3倍,变色栓菌来源的漆酶产量提高7倍[112]。当同时过表达与UPR相关的寡糖基转移酶SttC和敲除ERAD调控因子DoaA后,与野生型菌株相比,内源葡糖淀粉酶和内切蛋白酶的活性显著提高[113]。
降低宿主蛋白酶策略:丝状真菌体内含有大量的酸性/碱性蛋白酶,能快速降解目标蛋白,造成异源蛋白表达产量较低,因此,筛选或构建蛋白酶缺失突变株作为宿主是提高蛋白表达水平的重要手段之一。Johannes等针对黑曲霉中3个酸性蛋白酶基因(PepA、PepB和PepE) 进行了敲除,所获得的突变体(ΔpepA、ΔpepB和ΔpepE),与野生型相比,胞外蛋白酶活性分别降低到16%、94%和32%[114]。同时,王勇超等利用比较基因组技术鉴定了黑曲霉的4个天冬氨酸蛋白酶基因(pepAa、pepAb、pepAc和pepAd),而后进行了敲除实验,获得了其中3个基因的单突变体(ΔpepAa、ΔpepAb和ΔpepAd),与出发菌株相比,突变株异源漆酶的发酵水平分别增加了21%、42%和30%,糖水解酶产量分别增加了约18.7%、37.0%和5.20%[115]。
另外,研究者还通过蛋白融合表达、筛选高转录基因整合位点、调控5′-UTR区域以及终止子序列、开发低背景底盘表达系统等不同的方式提升目标蛋白合成。随着使能技术的不断发展以及人们对蛋白质表达修饰和分泌过程的深入认识,丝状真菌作为在工业蛋白表达系统将会发挥越来越重要的作用。
2.3 次级代谢产物自青霉素发现以来,丝状真菌次级代谢产物成为药物分子的重要来源,例如产黄青霉素Penicillium chrysogenum和顶头孢霉菌Acremonium chrysogenum产的青霉素和头孢菌素具有广谱抗菌活性,是重要抗生素品种,土曲霉A. terreus和红曲霉Monascus spp.发酵生产的洛伐他汀及其衍生物是常用的降血脂药物,而来源于木霉属Trichoderma和弯颈霉属Tolypocladium的环孢菌素是一种被广泛用于预防器官移植排斥的免疫抑制剂[116]。丝状真菌的次级代谢产物按照其合成途径主要分为聚酮类(例如他汀类、黄曲霉毒素)、非核糖体肽类(例如青霉素、头孢菌素和环孢菌素)、萜类化合物(例如胡萝卜素) 等。真菌聚酮化合物由聚酮合酶(PKS) 催化乙酰辅酶A和丙二酰辅酶A (或其衍生物) 合成的一类化合物。非核糖体肽(Nnribosomal peptides,NRPs) 是在非核糖体肽合成酶(Nonribosomal peptide synthases,NRPSs) 催化作用下经非核糖体途径合成的多肽类小分子化合物[117]。丝状真菌中萜类化合物由萜烯环化酶催化异戊二烯单元产生[118],首先通过甲羟戊酸(MVA) 途径合成二甲基丙烯焦磷酸(DMAPP) 和异戊烯焦磷酸(IPP),之后IPP在异构酶作用下可以转化为DMAPP。以DMAPP起始,在异戊烯基转移酶作用下与IPP首尾连接聚合形成线性聚异戊烯基焦磷酸前体,然后在萜类环化酶的作用下进行环化或重排形成萜类骨架,进一步后修饰(如氧化、脱水等) 生成各种萜类化合物[119]。
抗生素的发现和大量工业生产及临床应用拯救了无数人的生命,青霉素的发现被誉为“20世纪医学界最伟大的收获”。1928年,英国科学家Fleming在实验研究中最早发现了青霉素[120],之后Norman Heatley、Howard Florey、Ernst Chain等科学家对青霉素进行了分离和纯化,并证明了其有效的抗菌临床效用[121]。在这些研究成果的推动下,青霉素很快被美国制药公司投入工业生产,但是一开始产量很低,不足以应用于临床。为了提高青霉素的产量,研究者们除了优化发酵条件,还通过不断的诱变筛选(如紫外辐射、X-射线辐射和氮芥气),以及增加青霉素合成基因簇拷贝数(已知的现代工业菌株有超过12–14个拷贝数) 的方法,获得高产青霉素的工业菌株P. chrysogenum (高于50 g/L)。Rodriguez-Saiz等通过比较不同的产黄青霉菌工业菌株发现,在诱变过程中编码乙酸苯酯羟化酶基因pahA的失活,推测此点突变降低了青霉素-G合成前体物苯乙酸的降解,从而提升了青霉素的产量[122]。武敏等发现在菌种诱变筛选过程中多个氨基酸代谢途径相关基因发生了突变,其中编码丝氨酸/苏氨酸脱氨酸的两个基因和一个编码色氨酸合成酶的基因均被单个点突变失活,消除了丝氨酸/苏氨酸的降解途径,从而让更多的代谢流流向半胱氨酸合成途径[123]。半胱氨酸作为合成青霉素的三大前体物质之一,是影响青霉素产量最重要的因素之一。另外,Salo等发现在菌种诱变筛选过程中,许多其他的次级代谢途径由于引入的点突变被“沉默”。例如,一个编码聚酮合酶的基因在菌种诱变阶段突变失活,消除了一种黄色色素(Sorbicillinoid) 的形成,这种色素被认为是青霉素生产过程中的杂质[124]。除了青霉素以外,顶头孢霉菌A. chrysogenum产生的头孢菌素(Cephalosporin) 及其衍生物是另一类被广泛应用的抗生素,对维护人类健康有着重要意义[125]。然而,微生物对于抗生素的耐药性已经成为亟待解决的世界性难题。Jens Christian Nielsen等研究者发现,丝状真菌含有上千个次级代谢产物基因簇,具有发现新型抗生素及其他药物(如抗肿瘤药物) 的潜力,鉴定其功能和激活“沉默”基因簇是丝状真菌次级代谢产物领域的新研究热点[126]。
他汀类药物是丝状真菌另一类重要的次生代谢产物,如洛伐他汀、辛伐他汀等,已成为治疗心血管疾病、降低胆固醇的主要药物,目前其市场价值已超过200亿美元。洛伐他汀为第一个上市的他汀类药物,于1987年获得美国食品药品监督管理局(FDA) 批准,其市场药品名称定为“Mevacor”。相关洛伐他汀的研究最早起始于红曲霉[127]。默克(Merck) 公司通过筛选不同来源的土曲霉菌株,获得了洛伐他汀高产菌株,最终成功应用于工业生产。目前,土曲霉已成为了洛伐他汀的主要生产菌株。1999年,Kennedy等报道了土曲霉合成洛伐他汀的关键途径[128]。在土曲霉中,洛伐他汀的生物合成涉及18个基因,构成了长度为64 kb的基因簇,受特异性转录调控因子LovE的调控。其中,聚酮合酶(Polyketide synthases,PKS) LovB和LovF是洛伐他汀合成的关键酶。尽管土曲霉洛伐他汀合成途径已被鉴定,但目前其工业菌株的优化主要通过诱变育种的方式获得,通过代谢工程改造,提升菌株生产性能的案例较少,并且主要集中在产量较低的实验室菌种中。Hasan等通过提升产物合成前体物质的策略,在土曲霉中运用强启动子过表达了乙酰-CoA羧化酶ACCase,将洛伐他汀产量由62.7 mg/L提高至88 mg/L[129]。Zhgun等在土曲霉ATCC 20542中将转录因子LovE组成型过表达,其洛伐他汀的产量提高了10倍,达到了1 g/L[130]。1992年默克公司推出了第二代他汀类药物辛伐他汀,该药列名于世界卫生组织基本药物清单,成为基础医疗体系的必备药物之一,每年的市场销售额达到30亿美元。辛伐他汀为洛伐他汀的半衍生物,通过在辛伐他汀前体物质monacolin J上添加侧链(2, 2-二甲基丁酸酯) 合成。在工业生产中,通过碱水解方式除去洛伐他汀侧链(2-甲基丁醇),从而获得monacolin J,随后利用化学或酶催化方法添加新侧链来合成辛伐他汀。洛伐他汀碱处理工艺较为复杂,且在此过程中使用了大量的有机试剂,对环境造成严重污染。为此,利用生物合成monacolin J成为高效绿色生产辛伐他汀的关键。中国科学院青岛生物能源与过程研究所吕雪峰团队通过关键酶筛选,鉴定了洛伐他汀水解酶PcEST,在土曲霉洛伐他汀工业菌株过表达后,其转化子能够将95%的洛伐他汀水解为成monacolin J[131]。随后,该团队利用比较基因组学法方法,鉴定了洛伐他汀侧链(2-甲基丁醇) 合成关键基因lovF,敲除该基因后,实现了土曲霉monacolin J的高效积累。土曲霉中洛伐他汀合成基因簇受特异调控因子LovE的调控,利用强启动子PgpdAt过表达基因lovE后,monacolin J产量提高了52.5%,达到16.36 mmol/L[132]。
次级代谢产物的合成是一个复杂的过程,需要多个基因参与调控。与散布在整个真菌基因组中的初级代谢产物合成所需的基因相反,次级代谢产物合成途径中的基因大多以基因簇(BGC)的方式连续排列[133]。一般情况下,丝状真菌基因组中的次级代谢产物基因簇均为沉默状态,通过调控因子激活基因簇或直接过表达基因簇中全部/部分基因,成为提高丝状真菌次级代谢物产量的常用策略。Juguet等将合成杀刚果锥虫素(Congocidine) 基因簇克隆到载体pBelo-BAC11中,分别转入天蓝色链霉菌Streptomyces coelicolor M145、杀真菌链霉菌Streptomyces fungicidicus B-5477和白色链霉菌Streptomyces albus中,发酵产物中均得到了杀刚果锥虫素[134]。研究发现,在已挖掘的真菌次级代谢物合成基因簇中,60%以上基因簇受特异性调控基因控制[135]。Westpha等通过过表达镰刀菌素(Aurofusarin) 基因簇转录因子AurR1,使得镰刀菌素合成途径中的5种关键酶的表达水平显著上调,转化子色素产量提高了3倍[136]。白灰制菌素(Leucinostatin) 生物合成基因簇中存在一个特异性调控基因lcsF,高表达lcsF 使白灰制菌素产量增加了1.5倍[137]。黄体素(Verticillins) 是由罗杰斯无性穗霉Clonostachys rogersoniana产生的多硫代二酮哌嗪(ETP) 类化合物[138],黄体素生物合成基因簇受特异性转录因子VerZ的调控[139]。
丝状真菌次级代谢产物虽然不是菌株正常生长所必需的化合物,却是菌株发育、环境应答等过程的重要参与者,尽管近几年对真菌次级代谢产物的合成及其调控研究已经取得了很大的成就,然而其合成与调控机制非常复杂,除转录水平调控外,丝状真菌次级代谢还与组蛋白乙酰化修饰水平以及诸多环境因素(氮源、碳源、pH值、温度、光照以及不同微生物间的刺激) 相关。将来有必要利用转录组学、蛋白质组学及代谢组学等组学技术进一步研究次级代谢物合成及调控机制,为其代谢工程改提供目标靶点及理论依据。
3 丝状真菌代谢工程中的关键问题及展望 3.1 丝状真菌代谢工程中的关键问题丝状真菌特有的遗传特点、生长方式以及发酵形态,使得该体系的代谢工程改造所针对的问题及采取策略有别于酿酒酵母等单细胞微生物,未来在以下方面值得发力(图 3)。

|
| 图 3 丝状真菌系统代谢工程的未来发展 Fig. 3 Future perspectives for systems metabolic engineering of filamentous fungi. |
| |
(1) 进一步增强基因组水平编辑能力:丝状真菌一般存在多核现象,在遗传改造中易产生异核转化子现象,从而导致后续菌种发酵性状不稳定。因此,在丝状真菌代谢工程中需要采用高效的遗传操作技术。日益成熟的基因组编辑技术及真菌高通量筛选技术为丝状真菌分子改造奠定了技术基础。
(2) 丰富和深化代谢改造的层级:不仅集中在代谢途径,可以尝试从其他层次考虑,比如丝状真菌为真菌微生物,存在大量的细胞器,其代谢物和生物反应分布在不同亚细胞结构中。某一产品合成途径的关键酶及中间产物可能存在于单一或多个细胞器中,产物的快速合成需要代谢物在亚细胞间进行高效运输,给代谢途径改造及优化带来了一定的难度。另外,同一生化反应可能存在于多个细胞器中,不利于消除发酵副产物合成反应。丝状真菌全基因组分析,代谢网络模型构建及其代谢组学分析技术在解决这一问题中起到重要作用。尤其是随着现在检测技术的发展,人们能够获得微生物在不同时期的生理变化信息,为代谢网络模型的优化提供数据支持,从而指导如何优化目标产物合成途径。
(3) 提升计算生物学的介入深度:丝状真菌的代谢调控较为复杂,需要大力发展计算生物学,构建可靠的代谢网络模型、适合真菌代谢工程研究的算法以及代谢流分析工具等,结合多种高通量组学数据,积极探讨机器学习和人工智能在丝状真菌代谢途径设计重构中的介入,从整体水平制定其代谢工程改造策略,减少盲目性,提升代谢改造效率和菌种代谢能力上限。
(4) 拓宽丝状真菌发酵产品:目前丝状真菌的目标产物有限,有必要开发全新产品来扩宽丝状真菌在工业发酵中应用。其关键问题在于如何设计生物基产品合成途径并获取相关关键元件,如何实现目标产品合成途径与丝状真菌底盘细胞原有代谢适配。不出意外,丝状真菌代谢工程将和发展迅猛的合成生物学深度融合,在新一代工业丝状真菌底盘构建和一批生物基化学品细胞工厂构建等方面获得更大进展。
3.2 展望日益严峻的环境及能源问题,使得生物制造对人类经济社会的可持续发展尤为重要。代谢工程旨在通过优化代谢网络和调控网络,提高细胞代谢能力,推进工业菌种发酵水平进步,进一步提升生物制造水平和能力,迎接生物经济时代的到来。丝状真菌作为重要工业微生物,在发酵生产生物基化学品方面具有诸多优势,具有强大的蛋白质合成分泌能力,可以利用廉价的生物质进行发酵等。合成生物学的迅猛发展,使研究者能够从DNA、RNA、蛋白质和代谢物等不同层面和基因组水平系统分析、改造和设计细胞代谢,提升对丝状真菌整体代谢调控的理解能力和改造水平,相信丝状真菌代谢工程研究在未来生物经济时代必将发挥越来越重要的作用。
| [1] |
Bailey JE. Toward a science of metabolic engineering. Science, 1991, 252(5013): 1668-1675. DOI:10.1126/science.2047876
|
| [2] |
Galagan JE, Calvo SE, Borkovich KA, et al. The genome sequence of the filamentous fungus Neurospora crassa. Nature, 2003, 422(6934): 859-868. DOI:10.1038/nature01554
|
| [3] |
Kelkar HS, Griffith J, Case ME, et al. The Neurospora crassa genome: cosmid libraries sorted by chromosome. Genetics, 2001, 157(3): 979-990. DOI:10.1093/genetics/157.3.979
|
| [4] |
Gruber F, Visser J, Kubicek CP, et al. The development of a heterologous transformation system for the cellulolytic fungus Trichoderma reesei based on a pyrG-negative mutant strain. Curr Genet, 1990, 18(1): 71-76. DOI:10.1007/BF00321118
|
| [5] |
Ruiz-Díez B. Strategies for the transformation of filamentous fungi. J Appl Microbiol, 2002, 92(2): 189-195. DOI:10.1046/j.1365-2672.2002.01516.x
|
| [6] |
Visser H, Joosten V, Punt PJ, et al. Development of a mature fungal technology and production platform for industrial enzymes based on a Myceliophthora thermophila isolate, previously known as Chrysosporium lucknowense C1. Ind Biotechnol, 2011, 7(3): 214-223. DOI:10.1089/ind.2011.7.214
|
| [7] |
Liu JJ, Xie ZP, Shin HD, et al. Rewiring the reductive tricarboxylic acid pathway and L-malate transport pathway of Aspergillus oryzae for overproduction of L-malate. J Biotechnol, 2017, 253: 1-9. DOI:10.1016/j.jbiotec.2017.05.011
|
| [8] |
Case ME, Schweizer M, Kushner SR, et al. Efficient transformation of Neurospora crassa by utilizing hybrid plasmid DNA. Proc Natl Acad Sci USA, 1979, 76(10): 5259-5263. DOI:10.1073/pnas.76.10.5259
|
| [9] |
Gouka RJ, Gerk C, Hooykaas PJJ, et al. Transformation of Aspergillus awamori by Agrobacterium tumefaciens-mediated homologous recombination. Nat Biotechnol, 1999, 17(6): 598-601. DOI:10.1038/9915
|
| [10] |
Fang WG, Pei Y, Bidochka MJ. Transformation of Metarhizium anisopliae mediated by Agrobacterium tumefaciens. Can J Microbiol, 2006, 52(7): 623-626. DOI:10.1139/w06-014
|
| [11] |
Sugui JA, Chang YC, Kwon-Chung KJ. Agrobacterium tumefaciens-mediated transformation of Aspergillus fumigatus: an efficient tool for insertional mutagenesis and targeted gene disruption. Appl Environ Microbiol, 2005, 71(4): 1798-1802. DOI:10.1128/AEM.71.4.1798-1802.2005
|
| [12] |
Xu J, Li JG, Lin LC, et al. Development of genetic tools for Myceliophthora thermophila. BMC Biotechnol, 2015, 15: 35.
|
| [13] |
Colot HV, Park G, Turner GE, et al. A high-throughput gene knockout procedure for Neurospora reveals functions for multiple transcription factors. Proc Natl Acad Sci USA, 2006, 103(27): 10352-10357. DOI:10.1073/pnas.0601456103
|
| [14] |
Chakraborty BN, Patterson NA, Kapoor M. An electroporation-based system for high-efficiency transformation of germinated conidia of filamentous fungi. Can J Microbiol, 1991, 37(11): 858-863. DOI:10.1139/m91-147
|
| [15] |
Ozeki K, Kyoya F, Hizume K, et al. Transformation of intact Aspergillus niger by electroporation. Biosci Biotechnol Biochem, 1994, 58(12): 2224-2227. DOI:10.1271/bbb.58.2224
|
| [16] |
Hinnen A, Hicks JB, Fink GR. Transformation of yeast. Proc Natl Acad Sci USA, 1978, 75(4): 1929-1933. DOI:10.1073/pnas.75.4.1929
|
| [17] |
De Groot MJA, Bundock P, Hooykaas PJJ, et al. Agrobacterium tumefaciens-mediated transformation of filamentous fungi. Nat Biotechnol, 1998, 16(9): 839-842. DOI:10.1038/nbt0998-839
|
| [18] |
Karube I, Tamiya E, Matsuoka H. Transformation of Saccharomyces cerevisiae spheroplasts by high electric pulse. FEBS Lett, 1985, 182(1): 90-94. DOI:10.1016/0014-5793(85)81160-1
|
| [19] |
Li JG, Lin LC, Sun T, et al. Direct production of commodity chemicals from lignocellulose using Myceliophthora thermophila. Metab Eng, 2019, 61: 416-426.
|
| [20] |
Steiger MG, Vitikainen M, Uskonen P, et al. Transformation system for Hypocrea jecorina (Trichoderma reesei) that favors homologous integration and employs reusable bidirectionally selectable markers. Appl Environ Microbiol, 2011, 77(1): 114-121. DOI:10.1128/AEM.02100-10
|
| [21] |
Jiang BJ, Zhang RQ, Feng D, et al. A Tet-on and Cre-loxP based genetic engineering system for convenient recycling of selection markers in Penicillium oxalicum. Front Microbiol, 2016, 7: 485.
|
| [22] |
Xu YX, Shan L, Zhou YT, et al. Development of a Cre-loxP-based genetic system in Aspergillus niger ATCC1015 and its application to construction of efficient organic acid-producing cell factories. Appl Microbiol Biotechnol, 2019, 103(19): 8105-8114. DOI:10.1007/s00253-019-10054-3
|
| [23] |
Liu Q, Zhang YL, Li FY, et al. Upgrading of efficient and scalable CRISPR-Cas-mediated technology for genetic engineering in thermophilic fungus Myceliophthora thermophila. Biotechnol Biofuels, 2019, 12: 293. DOI:10.1186/s13068-019-1637-y
|
| [24] |
Shrivastav M, De Haro LP, Nickoloff JA. Regulation of DNA double-strand break repair pathway choice. Cell Res, 2008, 18(1): 134-147. DOI:10.1038/cr.2007.111
|
| [25] |
Meyer V. Genetic engineering of filamentous fungi —Progress, obstacles and future trends. Biotechnol Adv, 2008, 26(2): 177-185. DOI:10.1016/j.biotechadv.2007.12.001
|
| [26] |
Porteus MH, Baltimore D. Chimeric nucleases stimulate gene targeting in human cells. Science, 2003, 300(5620): 763. DOI:10.1126/science.1078395
|
| [27] |
Hsu PD, Lander ES, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering. Cell, 2014, 157(6): 1262-1278. DOI:10.1016/j.cell.2014.05.010
|
| [28] |
Komor AC, Badran AH, Liu DR. CRISPR-based technologies for the manipulation of eukaryotic genomes. Cell, 2017, 168(1/2): 20-36.
|
| [29] |
Zhang C, Meng XH, Wei XL, et al. Highly efficient CRISPR mutagenesis by microhomology-mediated end joining in Aspergillus fumigatus. Fungal Genet Biol, 2016, 86: 47-57. DOI:10.1016/j.fgb.2015.12.007
|
| [30] |
Liu R, Chen L, Jiang YP, et al. Efficient genome editing in filamentous fungus Trichoderma reesei using the CRISPR/Cas9 system. Cell Discov, 2015, 1: 15007.
|
| [31] |
Nødvig CS, Nielsen JB, Kogle ME, et al. A CRISPR-Cas9 system for genetic engineering of filamentous fungi. PLoS ONE, 2015, 10(7): e0133085. DOI:10.1371/journal.pone.0133085
|
| [32] |
Arazoe T, Miyoshi K, Yamato T, et al. Tailor-made CRISPR/Cas system for highly efficient targeted gene replacement in the rice blast fungus. Biotechnol Bioeng, 2015, 112(12): 2543-2549. DOI:10.1002/bit.25662
|
| [33] |
Wang Q, Zhao QQ, Liu Q, et al. CRISPR/Cas9- mediated genome editing in Penicillium oxalicum and Trichoderma reesei using 5S rRNA promoter- driven guide RNAs. Biotechnol Lett, 2020. DOI:10.1007/s10529-020-03024-7
|
| [34] |
Liu Q, Gao RR, Li JG, et al. Development of a genome-editing CRISPR/Cas9 system in thermophilic fungal Myceliophthora species and its application to hyper-cellulase production strain engineering. Biotechnol Biofuels, 2017, 10: 1. DOI:10.1186/s13068-016-0693-9
|
| [35] |
Xu GB, Li JG, Liu Q, et al. Transcriptional analysis of Myceliophthora thermophila on soluble starch and role of regulator AmyR on polysaccharide degradation. Bioresour Technol, 2018, 265: 558-562. DOI:10.1016/j.biortech.2018.05.086
|
| [36] |
Liu Q, Li JG, Gao RR, et al. CLR-4, a novel conserved transcription factor for cellulase gene expression in ascomycete fungi. Mol Microbiol, 2019, 111(2): 373-394. DOI:10.1111/mmi.14160
|
| [37] |
Gu SY, Li JG, Chen BC, et al. Metabolic engineering of the thermophilic filamentous fungus Myceliophthora thermophila to produce fumaric acid. Biotechnol Biofuels, 2018, 11: 323. DOI:10.1186/s13068-018-1319-1
|
| [38] |
Pel HJ, De Winde JH, Archer DB, et al. Genome sequencing and analysis of the versatile cell factory Aspergillus niger CBS 513.88. Nat Biotechnol, 2007, 25(2): 221-231. DOI:10.1038/nbt1282
|
| [39] |
Kjærbølling I, Vesth T, Frisvad JC, et al. A comparative genomics study of 23 Aspergillus species from section Flavi. Nat Commun, 2020, 11: 1106. DOI:10.1038/s41467-019-14051-y
|
| [40] |
Tian CG, Beeson WT, Iavarone AT, et al. Systems analysis of plant cell wall degradation by the model filamentous fungus Neurospora crassa. Proc Natl Acad Sci USA, 2009, 106(52): 22157-22162. DOI:10.1073/pnas.0906810106
|
| [41] |
Adav SS, Chao LT, Sze SK. Quantitative secretomic analysis of Trichoderma reesei strains reveals enzymatic composition for lignocellulosic biomass degradation. Mol Cell Proteomics, 2012, 11(7): M111.012419.
|
| [42] |
Adav SS, Li AA, Manavalan A, et al. Quantitative iTRAQ secretome analysis of Aspergillus niger reveals novel hydrolytic enzymes. J Proteome Res, 2010, 9(8): 3932-3940. DOI:10.1021/pr100148j
|
| [43] |
Jun H, Kieselbach T, Jönsson LJ. Enzyme production by filamentous fungi: analysis of the secretome of Trichoderma reesei grown on unconventional carbon source. Microb Cell Fact, 2011, 10: 68. DOI:10.1186/1475-2859-10-68
|
| [44] |
Berka RM, Grigoriev IV, Otillar R, et al. Comparative genomic analysis of the thermophilic biomass-degrading fungi Myceliophthora thermophila and Thielavia terrestris. Nat Biotechnol, 2011, 29(10): 922-927. DOI:10.1038/nbt.1976
|
| [45] |
Liu GD, Zhang L, Wei XM, et al. Genomic and secretomic analyses reveal unique features of the lignocellulolytic enzyme system of Penicillium decumbens. PLoS ONE, 2013, 8(2): e55185. DOI:10.1371/journal.pone.0055185
|
| [46] |
Marx IJ, Van Wyk N, Smit S, et al. Comparative secretome analysis of Trichoderma asperellum S4F8 and Trichoderma reesei Rut C30 during solid-state fermentation on sugarcane bagasse. Biotechnol Biofuels, 2013, 6(1): 172. DOI:10.1186/1754-6834-6-172
|
| [47] |
Phillips CM, Iavarone AT, Marletta MA. Quantitative proteomic approach for cellulose degradation by Neurospora crassa. J Proteome Res, 2011, 10(9): 4177-4185. DOI:10.1021/pr200329b
|
| [48] |
Askenazi M, Driggers EM, Holtzman DA, et al. Integrating transcriptional and metabolite profiles to direct the engineering of lovastatin-producing fungal strains. Nat Biotechnol, 2003, 21(2): 150-156. DOI:10.1038/nbt781
|
| [49] |
Zhang SL, Wakai S, Sasakura N, et al. Pyruvate metabolism redirection for biological production of commodity chemicals in aerobic fungus Aspergillus oryzae. Metab Eng, 2020, 61: 225-237. DOI:10.1016/j.ymben.2020.06.010
|
| [50] |
Knuf C, Nookaew I, Remmers I, et al. Physiological characterization of the high malic acid-producing Aspergillus oryzae strain 2103a-68. Appl Microbiol Biotechnol, 2014, 98(8): 3517-3527. DOI:10.1007/s00253-013-5465-x
|
| [51] |
Lu HZ, Liu XY, Huang MZ, et al. Integrated isotope-assisted metabolomics and 13C metabolic flux analysis reveals metabolic flux redistribution for high glucoamylase production by Aspergillus niger. Microb Cell Fact, 2015, 14: 147. DOI:10.1186/s12934-015-0329-y
|
| [52] |
Pedersen H, Christensen B, Hjort C, et al. Construction and characterization of an oxalic acid nonproducing strain of Aspergillus niger. Metab Eng, 2000, 2(1): 34-41. DOI:10.1006/mben.1999.0136
|
| [53] |
Chen M, Huang XN, Zhong CW, et al. Identification of an itaconic acid degrading pathway in itaconic acid producing Aspergillus terreus. Appl Microbiol Biotechnol, 2016, 100(17): 7541-7548. DOI:10.1007/s00253-016-7554-0
|
| [54] |
Kitano H. Computational systems biology. Nature, 2002, 420(6912): 206-210. DOI:10.1038/nature01254
|
| [55] |
Thiele I, Palsson BØ. A protocol for generating a high-quality genome-scale metabolic reconstruction. Nat Protoc, 2010, 5(1): 93-121. DOI:10.1038/nprot.2009.203
|
| [56] |
Sun JB, Lu X, Rinas U, et al. Metabolic peculiarities of Aspergillus niger disclosed by comparative metabolic genomics. Genome Biol, 2007, 8(9): R182. DOI:10.1186/gb-2007-8-9-r182
|
| [57] |
Andersen MR, Nielsen ML, Nielsen J. Metabolic model integration of the bibliome, genome, metabolome and reactome of Aspergillus niger. Mol Syst Biol, 2008, 4(1): 178. DOI:10.1038/msb.2008.12
|
| [58] |
Brandl J, Aguilar-Pontes MV, Schöpe P, et al. A community-driven reconstruction of the Aspergillus niger metabolic network. Fungal Biol Biotechnol, 2018, 5: 16. DOI:10.1186/s40694-018-0060-7
|
| [59] |
Lu HZ, Cao WQ, Ouyang LM, et al. Comprehensive reconstruction and in silico analysis of Aspergillus niger genome-scale metabolic network model that accounts for 1210 ORFs. Biotechnol Bioeng, 2017, 114(3): 685-695. DOI:10.1002/bit.26195
|
| [60] |
Upton DJ, McQueen-Mason SJ, Wood AJ. In silico evolution of Aspergillus niger organic acid production suggests strategies for switching acid output. Biotechnol Biofuels, 2020, 13: 27. DOI:10.1186/s13068-020-01678-z
|
| [61] |
Lu HZ, Cao WQ, Liu XY, et al. Multi-omics integrative analysis with genome-scale metabolic model simulation reveals global cellular adaptation of Aspergillus niger under industrial enzyme production condition. Sci Rep, 2018, 8: 14404. DOI:10.1038/s41598-018-32341-1
|
| [62] |
Sui YF, Schütze T, Ouyang LM, et al. Engineering cofactor metabolism for improved protein and glucoamylase production in Aspergillus niger. Microb Cell Fact, 2020, 19: 198. DOI:10.1186/s12934-020-01450-w
|
| [63] |
Pakula TM, Nygren H, Barth D, et al. Genome wide analysis of protein production load in Trichoderma reesei. Biotechnol Biofuels, 2016, 9: 132. DOI:10.1186/s13068-016-0547-5
|
| [64] |
Kim B, Kim WJ, Kim DI, et al. Applications of genome-scale metabolic network model in metabolic engineering. J Ind Microbiol Biotechnol, 2015, 42(3): 339-348. DOI:10.1007/s10295-014-1554-9
|
| [65] |
Anastassiadis S, Morgunov IG, Kamzolova SV, et al. Citric acid production patent review. Recent Pat Biotechnol, 2008, 2(2): 107-123. DOI:10.2174/187220808784619757
|
| [66] |
Yin X, Li JH, Shin HD, et al. Metabolic engineering in the biotechnological production of organic acids in the tricarboxylic acid cycle of microorganisms: Advances and prospects. Biotechnol Adv, 2015, 33(6): 830-841. DOI:10.1016/j.biotechadv.2015.04.006
|
| [67] |
Wang L, Cao ZL, Hou L, et al. The opposite roles of agdA and glaA on citric acid production in Aspergillus niger. Appl Microbiol Biotechnol, 2016, 100(13): 5791-5803. DOI:10.1007/s00253-016-7324-z
|
| [68] |
De Jongh WA, Nielsen J. Enhanced citrate production through gene insertion in Aspergillus niger. Metab Eng, 2008, 10(2): 87-96. DOI:10.1016/j.ymben.2007.11.002
|
| [69] |
Capuder M, Šolar T, Benčina M, et al. Highly active, citrate inhibition resistant form of Aspergillus niger 6-phosphofructo-1-kinase encoded by a modified pfkA gene. J Biotechnol, 2009, 144(1): 51-57. DOI:10.1016/j.jbiotec.2009.04.004
|
| [70] |
Hou L, Liu L, Zhang HF, et al. Functional analysis of the mitochondrial alternative oxidase gene (aox1) from Aspergillus niger CGMCC 10142 and its effects on citric acid production. Appl Microbiol Biotechnol, 2018, 102(18): 7981-7995. DOI:10.1007/s00253-018-9197-9
|
| [71] |
Kinoshita K. Production of itaconic acid and mannitol by Aspergillus itaconicus. J Chem Soc Japan, 1929, 50: 583-593.
|
| [72] |
Calam CT, Oxford AE, Raistrick H. Studies in the biochemistry of micro-organisms: itaconic acid, a metabolic product of a strain of Aspergillus terreus Thom. Biochem J, 1939, 33(9): 1488-1495. DOI:10.1042/bj0331488
|
| [73] |
Bentley R, Thiessen CP. Biosynthesis of itaconic acid in Aspergillus terreus. Ⅰ. Tracer studies with C14-labeled substrates. J Biol Chem, 1957, 226(2): 673-687. DOI:10.1016/S0021-9258(18)70850-8
|
| [74] |
Li A, Van Luijk N, Ter Beek M, et al. A clone-based transcriptomics approach for the identification of genes relevant for itaconic acid production in Aspergillus. Fungal Genet Biol, 2011, 48(6): 602-611. DOI:10.1016/j.fgb.2011.01.013
|
| [75] |
Li A, Pfelzer N, Zuijderwijk R, et al. Enhanced itaconic acid production in Aspergillus niger using genetic modification and medium optimization. BMC Biotechnol, 2012, 12: 57. DOI:10.1186/1472-6750-12-57
|
| [76] |
Huang XN, Lu XF, Li YM, et al. Improving itaconic acid production through genetic engineering of an industrial Aspergillus terreus strain. Microb Cell Fact, 2014, 13: 119. DOI:10.1186/s12934-014-0119-y
|
| [77] |
Huang XN, Chen M, Lu XF, et al. Direct production of itaconic acid from liquefied corn starch by genetically engineered Aspergillus terreus. Microb Cell Fact, 2014, 13: 108. DOI:10.1186/s12934-014-0108-1
|
| [78] |
Hossain AH, Li A, Brickwedde A, et al. Rewiring a secondary metabolite pathway towards itaconic acid production in Aspergillus niger. Microb Cell Fact, 2016, 15: 130. DOI:10.1186/s12934-016-0527-2
|
| [79] |
Krull S, Hevekerl A, Kuenz A, et al. Process development of itaconic acid production by a natural wild type strain of Aspergillus terreus to reach industrially relevant final titers. Appl Microbiol Biotechnol, 2017, 101(10): 4063-4072. DOI:10.1007/s00253-017-8192-x
|
| [80] |
Kuenz A, Gallenmüller Y, Willke T, et al. Microbial production of itaconic acid: developing a stable platform for high product concentrations. Appl Microbiol Biotechnol, 2012, 96(5): 1209-1216. DOI:10.1007/s00253-012-4221-y
|
| [81] |
Hevekerl A, Kuenz A, Vorlop KD. Influence of the pH on the itaconic acid production with Aspergillus terreus. Appl Microbiol Biotechnol, 2014, 98(24): 10005-10012. DOI:10.1007/s00253-014-6047-2
|
| [82] |
Karaffa L, Díaz R, Papp B, et al. A deficiency of manganese ions in the presence of high sugar concentrations is the critical parameter for achieving high yields of itaconic acid by Aspergillus terreus. Appl Microbiol Biotechnol, 2015, 99(19): 7937-7944. DOI:10.1007/s00253-015-6735-6
|
| [83] |
Hevekerl A, Kuenz A, Vorlop KD. Filamentous fungi in microtiter plates—an easy way to optimize itaconic acid production with Aspergillus terreus. Appl Microbiol Biotechnol, 2014, 98(16): 6983-6989. DOI:10.1007/s00253-014-5743-2
|
| [84] |
Brown SH, Bashkirova L, Berka R, et al. Metabolic engineering of Aspergillus oryzae NRRL 3488 for increased production of L-malic acid. Appl Microbiol Biotechnol, 2013, 97(20): 8903-8912. DOI:10.1007/s00253-013-5132-2
|
| [85] |
Liu JJ, Li JH, Liu YF, et al. Synergistic rewiring of carbon metabolism and redox metabolism in cytoplasm and mitochondria of Aspergillus oryzae for increased L-malate production. ACS Synth Biol, 2018, 7(9): 2139-2147. DOI:10.1021/acssynbio.8b00130
|
| [86] |
Khan I, Nazir K, Wang ZP, et al. Calcium malate overproduction by Penicillium viticola 152 using the medium containing corn steep liquor. Appl Microbiol Biotechnol, 2014, 98(4): 1539-1546. DOI:10.1007/s00253-013-5326-7
|
| [87] |
Zambanini T, Tehrani H, Geiser E, et al. Metabolic engineering of Ustilago trichophora TZ1 for improved malic acid production. Metab Eng Commun, 2017, 4: 12-21. DOI:10.1016/j.meteno.2017.01.002
|
| [88] |
Peleg Y, Barak A, Scrutton MC, et al. Malic acid accumulation by Aspergillus flavus. Ⅲ. 13C nmr and isoenzyme analyses. Appl Microbiol Biotechnol, 1989, 30(2): 176-183.
|
| [89] |
Bercovitz A, Peleg Y, Battat E, et al. Localization of pyruvate carboxylase in organic acid-producing Aspergillus strains. Appl Environ Microbiol, 1990, 56(6): 1594-1597. DOI:10.1128/AEM.56.6.1594-1597.1990
|
| [90] |
Knuf C, Nookaew I, Brown SH, et al. Investigation of malic acid production in Aspergillus oryzae under nitrogen starvation conditions. Appl Environ Microbiol, 2013, 79(19): 6050-6058. DOI:10.1128/AEM.01445-13
|
| [91] |
West TP. Fungal biotransformation of crude glycerol into malic acid. Z Naturforsch C J Biosci, 2015, 70(5/6): 165-167.
|
| [92] |
Zambanini T, Sarikaya E, Kleineberg W, et al. Efficient malic acid production from glycerol with Ustilago trichophora TZ1. Biotechnol Biofuels, 2016, 9: 67. DOI:10.1186/s13068-016-0483-4
|
| [93] |
Zambanini T, Kleineberg W, Sarikaya E, et al. Enhanced malic acid production from glycerol with high-cell density Ustilago trichophora TZ1 cultivations. Biotechnol Biofuels, 2016, 9: 135. DOI:10.1186/s13068-016-0553-7
|
| [94] |
Yang L, Christakou E, Vang J, et al. Overexpression of a C4-dicarboxylate transporter is the key for rerouting citric acid to C4-dicarboxylic acid production in Aspergillus carbonarius. Microb Cell Fact, 2017, 16: 43. DOI:10.1186/s12934-017-0660-6
|
| [95] |
Xu YX, Zhou YT, Cao W, et al. Improved production of malic acid in Aspergillus niger by abolishing citric acid accumulation and enhancing glycolytic flux. ACS Synth Biol, 2020, 9(6): 1418-1425. DOI:10.1021/acssynbio.0c00096
|
| [96] |
Li JG, Gu SY, Zhao Z, et al. Dissecting cellobiose metabolic pathway and its application in biorefinery through consolidated bioprocessing in Myceliophthora thermophila. Fungal Biol Biotechnol, 2019, 6: 21. DOI:10.1186/s40694-019-0083-8
|
| [97] |
Guillemette T, Ram AFJ, Carvalho NDSP, et al. Methods for investigating the UPR in filamentous fungi. Methods Enzymol, 2011, 490: 1-29.
|
| [98] |
Galagan JE, Calvo SE, Cuomo C, et al. Sequencing of Aspergillus nidulans and comparative analysis with A. oryzae. Nature, 2005, 438(7071): 1105-1115. DOI:10.1038/nature04341
|
| [99] |
Bischof RH, Ramoni J, Seiboth B. Cellulases and beyond: the first 70 years of the enzyme producer Trichoderma reesei. Microb Cell Fact, 2016, 15: 106. DOI:10.1186/s12934-016-0507-6
|
| [100] |
Gutiérrez-Rojas I, Moreno-Sarmiento N, Montoya D. Mechanisms and regulation of enzymatic hydrolysis of cellulose in filamentous fungi: classical cases and new models. Rev Iberoam Micol, 2015, 32(1): 1-12. DOI:10.1016/j.riam.2013.10.009
|
| [101] |
Fonseca LM, Parreiras LS, Murakami MT. Rational engineering of the Trichoderma reesei RUT-C30 strain into an industrially relevant platform for cellulase production. Biotechnol Biofuels, 2020, 13: 93. DOI:10.1186/s13068-020-01732-w
|
| [102] |
Heimel K. Unfolded protein response in filamentous fungi-implications in biotechnology. Appl Microbiol Biotechnol, 2015, 99(1): 121-132. DOI:10.1007/s00253-014-6192-7
|
| [103] |
Gouka RJ, Hessing JGM, Punt PJ, et al. An expression system based on the promoter region of the Aspergillus awamori 1, 4-β-endoxylanase A gene. Appl Microbiol Biotechnol, 1996, 46(1): 28-35. DOI:10.1007/s002530050779
|
| [104] |
Lv XX, Zheng FL, Li CY, et al. Characterization of a copper responsive promoter and its mediated overexpression of the xylanase regulator 1 results in an induction-independent production of cellulases in Trichoderma reesei. Biotechnol Biofuels, 2015, 8: 67. DOI:10.1186/s13068-015-0249-4
|
| [105] |
Druzhinina IS, Kubicek CP. Genetic engineering of Trichoderma reesei cellulases and their production. Microb Biotechnol, 2017, 10(6): 1485-1499. DOI:10.1111/1751-7915.12726
|
| [106] |
Zhang F, Bai FW, Zhao XQ. Enhanced cellulase production from Trichoderma reesei Rut-C30 by engineering with an artificial zinc finger protein library. Biotechnol J, 2016, 11(10): 1282-1290. DOI:10.1002/biot.201600227
|
| [107] |
Zhang XY, Li YH, Zhao XQ, et al. Constitutive cellulase production from glucose using the recombinant Trichoderma reesei strain overexpressing an artificial transcription activator. Bioresour Technol, 2017, 223: 317-322. DOI:10.1016/j.biortech.2016.10.083
|
| [108] |
Craig JP, Coradetti ST, Starr TL, et al. Direct target network of the Neurospora crassa plant cell wall deconstruction regulators CLR-1, CLR-2, and XLR-1. mBio, 2015, 6(5): e01452-15. DOI:10.1128/mBio.01452-15
|
| [109] |
Yang F, Gong YF, Liu G, et al. Enhancing cellulase production in thermophilic fungus Myceliophthora thermophila ATCC42464 by RNA interference of cre1 gene expression. J Microbiol Biotechnol, 2015, 25(7): 1101-1107. DOI:10.4014/jmb.1501.01049
|
| [110] |
Li ZH, Yao GS, Wu RM, et al. Synergistic and dose-controlled regulation of cellulase gene expression in Penicillium oxalicum. PLoS Genet, 2015, 11(9): e1005509. DOI:10.1371/journal.pgen.1005509
|
| [111] |
Yao GS, Li ZH, Gao LW, et al. Redesigning the regulatory pathway to enhance cellulase production in Penicillium oxalicum. Biotechnol Biofuels, 2015, 8: 71. DOI:10.1186/s13068-015-0253-8
|
| [112] |
Valkonen M, Ward M, Wang HM, et al. Improvement of foreign-protein production in Aspergillus niger var. awamori by constitutive induction of the unfolded-protein response. Appl Environ Microbiol, 2003, 69(12): 6979-6986. DOI:10.1128/AEM.69.12.6979-6986.2003
|
| [113] |
Jacobs DI, Olsthoorn MMA, Maillet I, et al. Effective lead selection for improved protein production in Aspergillus niger based on integrated genomics. Fungal Genet Biol, 2009, 46(1): S141-S152. DOI:10.1016/j.fgb.2008.08.012
|
| [114] |
Van Den Hombergh JPTW, Gelpke MDS, Van De Vondervoort PJI, et al. Disruption of three acid proteases in Aspergillus niger-effects on protease spectrum, intracellular proteolysis, and degradation of target proteins. Eur J Biochem, 1997, 247(2): 605-613. DOI:10.1111/j.1432-1033.1997.00605.x
|
| [115] |
Wang YC, Xue W, Sims AH, et al. Isolation of four pepsin-like protease genes from Aspergillus niger and analysis of the effect of disruptions on heterologous laccase expression. Fungal Genet Biol, 2008, 45(1): 17-27. DOI:10.1016/j.fgb.2007.09.012
|
| [116] |
Raja HA, Miller AN, Pearce CJ, et al. Fungal identification using molecular tools: A primer for the natural products research community. J Nat Prod, 2017, 80(3): 756-770. DOI:10.1021/acs.jnatprod.6b01085
|
| [117] |
Lebeau J, Venkatachalam M, Fouillaud M, et al. Production and new extraction method of polyketide red pigments produced by ascomycetous fungi from Terrestrial and Marine habitats. J Fungi (Basel), 2017, 3(3): 34. DOI:10.3390/jof3030034
|
| [118] |
Keller NP. Fungal secondary metabolism: regulation, function and drug discovery. Nat Rev Microbiol, 2019, 17(3): 167-180. DOI:10.1038/s41579-018-0121-1
|
| [119] |
Chang MC, Keasling JD. Production of isoprenoid pharmaceuticals by engineered microbes. Nat Chem Biol, 2006, 2(12): 674-681. DOI:10.1038/nchembio836
|
| [120] |
Fleming A. On the antibacterial action of cultures of a penicillium, with special reference to their use in the isolation of B. influenzae. 1929. Bull World Health Organ, 2001, 79(8): 780-790.
|
| [121] |
Abraham EP, Chain E, Fletcher CM, et al. Further observations on penicillin. Lancet, 1941, 238(6155): 177-189. DOI:10.1016/S0140-6736(00)72122-2
|
| [122] |
Rodríguez-Sáiz M, Barredo JL, Moreno MA, et al. Reduced function of a phenylacetate-oxidizing cytochrome P450 caused strong genetic improvement in early phylogeny of penicillin-producing strains. J Bacteriol, 2001, 183(19): 5465-5471. DOI:10.1128/JB.183.19.5465-5471.2001
|
| [123] |
Wu M, Crismaru CG, Salo O, et al. Impact of classical strain improvement of Penicillium rubens on amino acid metabolism during β-lactam production. Appl Environ Microbiol, 2020, 86(3): e01561-19.
|
| [124] |
Salo O, Guzmán-Chávez F, Ries MI, et al. Identification of a polyketide synthase involved in sorbicillin biosynthesis by Penicillium chrysogenum. Appl Environ Microbiol, 2016, 82(13): 3971-3978. DOI:10.1128/AEM.00350-16
|
| [125] |
Brakhage AA. Molecular regulation of beta-lactam biosynthesis in filamentous fungi. Microbiol Mol Biol Rev, 1998, 62(3): 547-585. DOI:10.1128/MMBR.62.3.547-585.1998
|
| [126] |
Nielsen JC, Grijseels S, Prigent S, et al. Global analysis of biosynthetic gene clusters reveals vast potential of secondary metabolite production in Penicillium species. Nat Microbiol, 2017, 2: 17044. DOI:10.1038/nmicrobiol.2017.44
|
| [127] |
Endo A, Negishi Y, Iwashita T, et al. Biosynthesis of ML-236B (compactin) and monacolin K. J Antibiot (Tokyo), 1985, 38(3): 444-448. DOI:10.7164/antibiotics.38.444
|
| [128] |
Kennedy J, Auclair K, Kendrew SG, et al. Modulation of polyketide synthase activity by accessory proteins during lovastatin biosynthesis. Science, 1999, 284(5418): 1368-1372. DOI:10.1126/science.284.5418.1368
|
| [129] |
Hasan H, Abd Rahim MH, Campbell L, et al. Overexpression of acetyl-CoA carboxylase in Aspergillus terreus to increase lovastatin production. N Biotechnol, 2018, 44: 64-71. DOI:10.1016/j.nbt.2018.04.008
|
| [130] |
Zhgun AA, Nuraeva GK, Dumina MV, et al. 1, 3-diaminopropane and spermidine upregulate lovastatin production and expression of lovastatin biosynthetic genes in Aspergillus terreus via LaeA regulation. Appl Biochem Microbiol, 2019, 55(3): 243-254. DOI:10.1134/S0003683819020170
|
| [131] |
Huang XN, Liang YJ, Yang Y, et al. Single-step production of the simvastatin precursor monacolin J by engineering of an industrial strain of Aspergillus terreus. Metab Eng, 2017, 42: 109-114. DOI:10.1016/j.ymben.2017.06.005
|
| [132] |
Huang XN, Tang S, Zheng LH, et al. Construction of an efficient and robust Aspergillus terreus cell factory for monacolin J production. ACS Synth Biol, 2019, 8(4): 818-825. DOI:10.1021/acssynbio.8b00489
|
| [133] |
Osbourn A. Secondary metabolic gene clusters: evolutionary toolkits for chemical innovation. Trends Genet, 2010, 26(10): 449-457. DOI:10.1016/j.tig.2010.07.001
|
| [134] |
Awaad AS, Al-Zaylaee HM, Alqasoumi SI, et al. Anti-leishmanial activities of extracts and isolated compounds from Drechslera rostrata and Eurotium tonpholium. Phytother Res, 2014, 28(5): 774-780. DOI:10.1002/ptr.5096
|
| [135] |
Fox EM, Howlett BJ. Secondary metabolism: regulation and role in fungal biology. Curr Opin Microbiol, 2008, 11(6): 481-487. DOI:10.1016/j.mib.2008.10.007
|
| [136] |
Westphal KR, Wollenberg RD, Herbst FA, et al. Enhancing the production of the fungal pigment aurofusarin in Fusarium graminearum. Toxins (Basel), 2018, 10(11): 485. DOI:10.3390/toxins10110485
|
| [137] |
Wang G, Liu ZG, Lin RM, et al. Biosynthesis of antibiotic leucinostatins in bio-control fungus Purpureocillium lilacinum and their inhibition on phytophthora revealed by genome mining. PLoS Path, 2016, 12(7): e1005685. DOI:10.1371/journal.ppat.1005685
|
| [138] |
Wang Y, Hu PJ, Pan YY, et al. Identification and characterization of the verticillin biosynthetic gene cluster in Clonostachys rogersoniana. Fungal Genet Biol, 2017, 103: 25-33. DOI:10.1016/j.fgb.2017.03.007
|
| [139] |
Guo Z, Hao TC, Wang Y, et al. VerZ, a Zn(Ⅱ)2Cys6 DNA-binding protein, regulates the biosynthesis of verticillin in Clonostachys rogersoniana. Microbiology, 2017, 163(11): 1654-1663. DOI:10.1099/mic.0.000557
|
 2021, Vol. 37
2021, Vol. 37