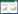中国科学院微生物研究所、中国微生物学会主办
文章信息
- 姚瑞莲
- Yao Ruilian
- 13C代谢流量分析发展30年
- Thirty years development of 13C metabolic flux analysis:
- 生物工程学报, 2021, 37(5): 1510-1525
- Chinese Journal of Biotechnology, 2021, 37(5): 1510-1525
- 10.13345/j.cjb.200741
-
文章历史
- Received: November 23, 2020
- Accepted: February 19, 2021
- Published: February 24, 2021
基于DNA重组技术基础上的代谢工程,是一种通过理性设计和改造细胞代谢网络,从而改善细胞性能、增强产品合成的技术[1-2]。近些年,随着系统生物学的快速发展,科研工作者能够利用基因、RNA、蛋白、代谢物和代谢流量等多个层次的数据,从全局规模上分析微生物代谢,并以此为基础进一步设计、修饰和重构细胞,使代谢工程逐渐步入系统代谢工程时代[3-5]。
作为系统生物学最下游的组学——代谢流量组学,是细胞代谢反应速率的集合,反映基因-蛋白-代谢物之间的相互作用对代谢网络的综合效应[6]。代谢流量分析在严格评价细胞表型、深入解析代谢调控规律、提供预测目标产物高产的改造方案、使代谢流量最大限度地流入目标产物的合成途径、推动代谢工程从穷举试错法到理性设计转变等方面发挥至关重要的作用[5]。通过该技术成功改造相关宿主生产的目标产物包括异戊二烯[7]、维生素B2[8]、可可浆[9]、丙酮醇[10]、乙醛[11]、异丙醇[12]、脂质[13]、琥珀酸[14]、赖氨酸[15]和脂肪醇[16]等。此外,由于这项技术能够用于识别代谢通路和发现新的代谢途径,揭示疾病(非小细胞肺癌[17]、恶性胶质瘤[18]、大肠腺癌[19]、乳腺癌[20]、胰腺导管肺癌[21]、糖尿病[22]、代谢综合征[23]和视网膜退行性疾病[24]等) 发生发展过程中的主要通路变化,追踪药物(达卡他韦[25]和扑热息痛[26]等) 在组织或器官中参与的代谢途径,也已成功应用于生物医药领域。
本文首先简述建立13C代谢流量分析平台的流程、13C代谢流量分析的发展历程和最新技术的研究,然后系统综述代谢流量组学技术在工业生物技术和生物医药与大健康领域中的应用,最后对13C代谢流量分析的未来发展方向进行展望。
1 建立13C代谢流量分析平台的流程细胞内的代谢流量不能被直接检测,需要利用同位素示踪技术、结合代谢组学分析(仅非稳态13C代谢流量分析拟合需要)、进而进行数学建模计算得到代谢流量组[27]。建立13C代谢流量分析平台,即在培养基中加入13C标记底物,经过一段时间培养,收集细胞,提取胞内代谢物,利用质谱或核磁共振波谱检测细胞内中间代谢物或蛋白氨基酸的13C同位素标记异构体的分布,基于代谢反应中碳原子转移特征建立13C分布的数学模型,结合实验测量值和数学模型系统地定量细胞内代谢流量的分布(图 1)。
|
|
图 1 13C代谢流量分析平台
Fig. 1 13C metabolic flux analysis platform.
|
| |
整个流程可归纳为4个模块:外部速率的确定、代谢网络的构建、13C标记实验(也称为稳定同位素示踪实验)、流量计算和统计分析[28-29]。外部代谢速率包括比生长速率、底物比消耗速率、和产物比生成速率。基于同位素标记异构体分布模型的代谢网络包含生物化学反应和相关的原子位移矩阵,可从KEGG、BioCyc数据库和已有文献获得相应信息。网络构建的最低目标是阐明检测到代谢物碳原子的来源[29]。代谢途径包括底物摄取途径、中心碳代谢途径、产物生成途径和氨基酸合成途径。生物量合成的反应经常被合并到一起,比仅检测细胞组分精确[30]。对于未被研究透彻的生物体,在网络构建最低目标的基础上进行流量计算后,引入卡方检验对模型进行评估,检查是否出现代谢物失拟,为查找缺失反应提供有效线索。如果中间代谢物同位素标记异构体模拟计算值和实际测量值之间的残差平方和过大,需要将最初设置为失活的途径改为激活,或依据文献,把其他生物相关途径移植至该生物,修正模型后重新进行流量计算和统计分析。
13C标记实验包含4个步骤[28-29]:(1) 标记底物的设计;(2) 取样时间点的选择;(3) 中间代谢物同位素标记的质量分布向量的测定;(4) 同位素标记异构体天然同位素丰度的矫正。标记底物必须根据研究目的和代谢途径仔细选择。在代谢网络和代谢流量测量值/假定值的基础上,基于D-优化准则,使标记实验能够最大程度地提供代谢途径分析的信息[31-32]。当含有标记底物的培养基开始补入以后,细胞内所有代谢物的同位素标记异构体的分布逐步增加至一定程度,方可进行取样分析。当分批培养时,标记底物在一开始就加入,在对数生长期的中后期可以同时达到代谢稳态和同位素稳态。当连续培养时,达到代谢稳态后,非标记底物被替换成标记底物,取样的时间点越晚越接近同位素稳态[28]。中间代谢物同位素标记的质量分布向量(Mass distribution vector,MDV) 的测量,是较为重要的步骤,在下文3.2中详细介绍。由于天然稳定同位素也会造成较高的质量数。例如,测量得到的M+1,有可能是1个碳原子被13C标记,也有可能是1个氢原子被2H标记,或者1个氧原子被17O标记的所有可能情况的丰度总和。因此需要对原始的质谱数据,矫正元素的天然同位素丰度,以获得真实的质量分布向量[33-34]。
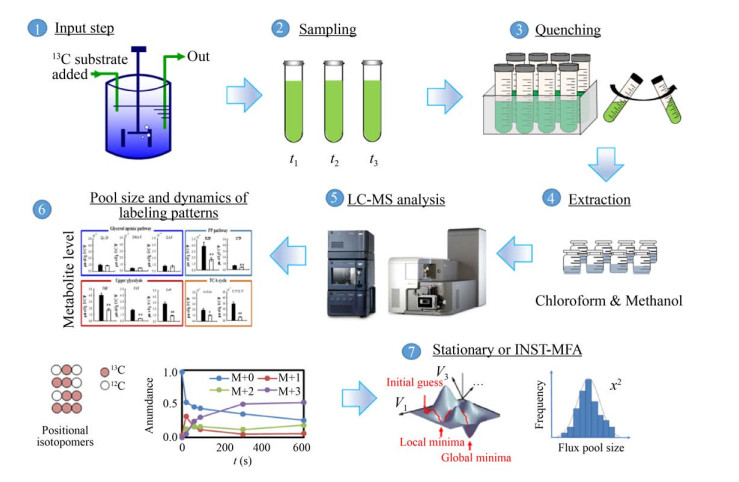
在确定外部速率、代谢网络和13C标记实验的基础上,进行流量计算与统计分析,使用参数迭代拟合的方法,建立代谢流量和同位素标记异构体测量值之间的数学关系,比较蛋白氨基酸或中间代谢物同位素标记异构体模拟计算值和实际测量值,利用非线性最小二乘法进行迭代计算,寻找全局最优解,使两者之间的差别最小。利用蒙特卡罗、线性搜索和非线性搜索的方法进行统计分析,计算置信区间[35] (图 2)。
|
|
图 2 同位素标记异构体和代谢物基元的算法
Fig. 2 A schematic view of the algorithm for the isotopomer and elementary metabolite units.
|
| |
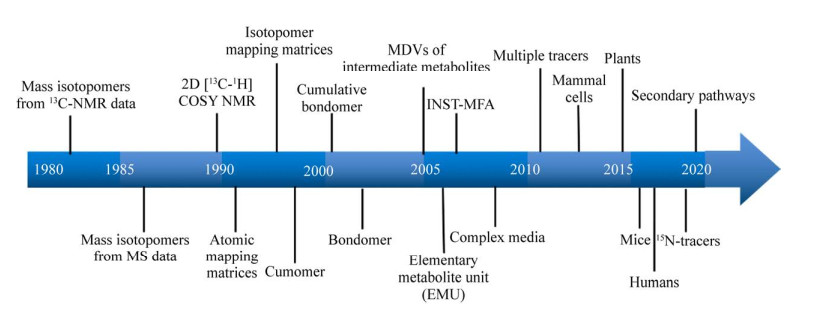
自1980年起,伴随着稳定同位素示踪和检测技术的快速发展,13C代谢流量分析在算法上也形成了颠覆式创新,突破了仅能解析中心碳代谢、适用于基本培养基和应用于微生物系统的原有限制(图 3)。
|
|
图 3 13C代谢流量分析的发展历程
Fig. 3 Development of 13C-MFA.
|
| |
稳定同位素示踪技术的快速发展,促进了13C代谢流量分析的发展。从1980年起,稳定同位素示踪技术由于其具有安全性、易操作性、从单一实验可获取较多同位素丰度的数据等优势,逐渐替代放射性示踪的标记技术[36],为流量计算提供了大量信息。
2.2 同位素标记异构体分布检测的技术平台核磁共振波谱和质谱是中间代谢物同位素标记异构体的检测平台。精确定量同位素标记异构体至关重要[37]。对于n个碳原子的化合物,可以产生2n种同位素标记异构体[38]。早期,由波谱技术所获取的同位体分布信息非常有限,2D [13C, 1H] COSY NMR技术的出现有效地改善了这一状况[39],然而得到全部信息仍是一个巨大挑战。近年来,质谱技术已成为主流分析平台。发展最为成熟的气相色谱-质谱联用仪(Gas chromatography-mass spectrometry,GC-MS),借助于衍生化技术,能够广泛适用于生物样本中氨基酸和有机酸等小分子代谢物的分析检测[40],是基于蛋白氨基酸数据拟合的13C-MFA的重要测定技术,已成为13C-MFA最常用的平台。国际上已建立了通用的化合物库(美国国家标准技术研究院质谱数据库,http://www.nist.gov/srd/nist1a.cfm),极大地方便了代谢产物的结构鉴定。然而,此技术不适用于难挥发和高极性代谢物的分析,尤其是基于中间代谢物同位体分布信息的13C-MFA所必需的磷酸糖类。毛细管电泳-质谱联用仪(Capillary electrophoresis-mass spectrometry,CE-MS) 结合了毛细管电泳的分离效率高、分离速度快、样品消耗量少以及质谱检测的高灵敏度和强结构分析能力等优点,已成为少量代谢流量组学研究者关注的新型微量分析技术[41]。与其他色谱-质谱联用技术相比,液相色谱-质谱联用(Liquid chromatography- mass spectrometry,LC-MS) 技术具有高灵敏度、高选择性且测试时不受样品稳定性和挥发性影响,所覆盖的代谢物种类多等优点,已成为研究代谢流量组学的功能最全面的分析平台[42],未来将有可能替代GC-MS,推进基于中间代谢物同位体分布信息的13C代谢流量分析方法的建立。串联质谱可获得重要代谢物片段的同位体质量分布信息,更为有效地提升了测量精度[43-44]。
2.3 13C代谢流量分析的算法和软件包利用同位体分布信息进行代谢流量的定量分析包括局部流比率[45]和整体迭代拟合法[27],前者已由花强等[46]系统综述了其算法和在代谢工程中的应用。本文将重点论述后者的发展历程。20世纪90年代中期,最早出现的Isotopomer算法是基于原子映射矩阵(Atom mapping matrix,AMM) 和同位素标记异构体映射矩阵(Isotopomer mapping matrix,IMM) 对代谢网络中同位素标记异构体分布进行建模[38, 47],已由花强等[46]作了详尽的分析(图 2)。然而,两分子或三分子反应引起的非线性和大量的同位素标记异构体是求解同位素标记异构体的平衡方程的两大难点[48]。特定同位素标记构体组[49] (Cumomer)、键标记物[50] (Bondomer)、累积键标记物[51] (Cumulative bondomer) 和代谢物基元[52] (Elementary metabolite units,EMU) 的出现,将耦合非线性方程转移至仅包含线性方程的子集,有效求解非线性方程。其中,EMU基于高效的分解方法(图 2),在保证信息完整的情况下,利用代谢网络反应中原子转移信息来确定反应网络中模拟同位素标记所需要的最少信息,产生确定流量和同位素测量之间关系的系统方程,极大地减少了计算时间,100 s EMUs vs. 1 000 s isotopomers。Cumomer和Cumulative bondomer算法于2D [13C, 1H] COSY NMR获得的数据,而Isotopomer和EMU算法同时适用于NMR和MS数据,应用范围较为广泛。基于以上算法,研究者开发了相应的软件包[53-60],提供较为全面的分析平台,可实现稳态和非稳态13C代谢流量分析,帮助初学者进行模式生物的13C代谢流量分析(表 1)。然而,当研究对象、研究问题与模式生物的中心碳代谢网络相差较大时,例如复合培养基、补料-分批培养、高等生物的区室化、次级代谢途径、途径存在代谢物通道和基因组尺度代谢网络,需要研究者对13C代谢流量分析有全面深入的理解,不能简单套用已有模型。
| Software | Isotopomer method | Statistical analysis | Programming language | References |
| 13CFLUX | Isotopomer | Linear | C++ | [53] |
| 13CFLUX2 | Cumomer/EMU | Linear/Monte Carlo | C++ | [54] |
| OpenFlux | EMU | Non-linear search/Monte Carlo | Matlab | [56] |
| OpenFlux2 | EMU | Linear/Monte Carlo | Matlab | [57] |
| influx_s | Cumomer/EMU | Linear/Monte Carlo | R & Python | [58] |
| INCA | EMU | Non-linear search/Monte Carlo | Matlab | [59] |
| OpenMebius | EMU | Non-linear search | Matlab | [60] |
| Metran | EMU | Non-linear/Monte Carlo | Matlab | [29] |
随着稳定同位素示踪和检测技术平台以及算法的发展,13C代谢流量分析的新技术相继出现:基于中间代谢物同位体分布信息的13C代谢流量分析方法、非稳态13C代谢流量分析、应用于复合培养基的13C代谢流量分析和多底物标记的13C代谢流量分析。
2.4.1 基于中间代谢物同位体分布信息的13C代谢流量分析方法用蛋白氨基酸数据拟合中间代谢物同位体分布可能会导致计算偏差,对除中心碳代谢途径外的其他代谢产物合成的特殊途径,无法发挥其作用。然而,中间代谢物的低浓度、样品前处理过程中胞内代谢物的降解和基质效应等因素导致同位素标记异构体难以检测[61]。随着样品预处理方法的优化,高灵敏度和稳定性好的高分辨质谱的应用,色谱分离及质谱条件的优化,提高了氨基酸以外的中间代谢物同位素标记异构体检测的灵敏度、准确性、特异性和重现性[42, 62]。基于中间代谢物同位体分布信息的13C代谢流量分析已有成功的应用[10, 63-64],有效地提高了计算精度。
2.4.2 非稳态13C代谢流量分析传统的稳态13C代谢流量分析需要同时满足代谢稳态和同位素稳态的苛刻条件,需要大量的同位素标记物,实验时间漫长,也难以应用到动物细胞、自养生物、工业生产以及高通量实验中。非稳态13C代谢流量分析仅需要在代谢稳态的条件下,利用同位素动态示踪技术快速取样,淬灭和提取胞内代谢物,构建代谢组学和胞内代谢物同位素标记异构体动态分布的常微分方程组模型,结合实测值与模型预估值,获得最优代谢流量组[65-66]。非稳态13C代谢流量分析极大地缩短同位素标记所需要的时间,节约实验成本;贴切反映真实发酵条件下细胞的生理特性[66],为后续应用到复杂培养条件,建立工业菌株代谢网络的代谢流集成平台奠定基础。
Nöh等[65]最先提出了非稳态13C代谢流量分析的概念,通过在野生型大肠杆菌培养过程中突然增加13C标记底物的比例,通过自动取样装置在16 s内的11个时间点快速取样,同时得到中间代谢物的浓度和动态分布的大量信息。在这些数据的基础上,作者开发了新的基于综合碳标记网络模型的流量分析工具13C FLUX/INST来计算代谢流量。Young等[67]将EMU方法应用于同位素非稳态流量分析,建立了仅依靠外部流量和动态标记数据估计流量值的模型,在棕色脂肪细胞上得到成功验证。在此基础上,开发了非稳态13C代谢流量的分析工具INCA[59],并发表了一系列的相关研究[11, 68-69]。
2.4.3 多底物标记的13C代谢流量分析为增强发酵技术的经济竞争力,需要微生物能够代谢具有价格优势的原料(甘油、葡萄糖、木质纤维素和蔗糖等),转化为高附加值的化学品。两种底物高效共利用,可以充分发挥每种底物的优势,同时满足菌体生长和目标产物生产的需要。由于单一底物标记,所获得标记的同位体分布信息少,最优解精度低。传统13C-MFA方法不适用于培养基中存在两种或多种底物混合培养的情况。因此,发展多种及两种底物标记的代谢流量分析方法非常必要,而且随着代谢流量分析技术的快速发展,建立上述方法也成为可能。
Cordova等[70]通过单一标记葡萄糖([1, 6-13C]葡萄糖)、单一标记木糖([5-13C]木糖)、和混合标记葡萄糖([1, 6-13C]葡萄糖) 和木糖([5-13C]木糖),对能同时消耗葡萄糖-木糖的嗜热栖热菌的适应性进化菌株进行流量分析,发现该菌的上游途径有糖供给的柔性,而下游途径具有刚性的调控结构。该研究首次报道了木质纤维素生物质获得的水解主要产物葡萄糖和木糖的流量分布,为其他微生物利用上述糖类的深入研究提供了基础。
Yao等[10]以一株高效共利用甘油和葡萄糖的大肠杆菌为研究对象,设计了[1-13C]葡萄糖和[1, 3-13C]甘油的底物标记模式:[1-13C]葡萄糖被用于区分糖酵解途径与磷酸戊糖途径;[1, 3-13C]甘油用于区分[1-13C]葡萄糖产生的中间代谢物同位体分布信息。在大肠杆菌普适性代谢网络模型(糖酵解途径、磷酸戊糖途径、三羧酸(TCA) 循环、乙醛酸支路、总生物量合成和转氢反应) 的基础上,添加了甘油代谢途径。笔者基于Isotopomer算法,使用内部软件计算流量:在稀释率为0.1 h–1和0.35 h–1的实验中,分别使用116个和120个蛋白氨基酸碎片的质量分布向量进行拟合,自由净流量为18和17;将稀释率、葡萄糖摄取速率、甘油摄取速率和乙酸生成速率作为固定值添加到模型中。各种蛋白氨基酸的代谢物同位素标记异构体测量值与拟合值近似,拟合效果很好,计算精确度高。进一步,笔者用蒙特卡罗方法计算置信区间,用以分析统计学特点。该研究阐明了混合利用葡萄糖和甘油的机制:碳源和能量代谢在高稀释率下进行再分布;快速的甘油摄取影响了NADPH/NADH的比值,降低了ATP浓度,激活了ArcA调节系统,抑制了TCA循环,引起乙酸溢流;葡萄糖从糖酵解途径流向磷酸戊糖途径,说明以甘油为主要碳源时需添加其他碳源以增加NADPH的供应。该研究思路为扩展13C-MFA的应用范围从单一底物到多种底物提供了完整的研究路线,对研究微生物代谢以生物柴油副产物甘油和其他碳源,以生产高价值化学品提供借鉴。
Ahn等[71]采用同时标记[1, 2-13C]葡萄糖和[U-13C]谷氨酰胺的策略:葡萄糖流入糖酵解途径的代谢物达到同位素稳态需要1.5 h,谷氨酰胺流入TCA循环的代谢物达到同位素稳态需要3 h。中国仓鼠细胞从生长状态转变到非生长状态,磷酸戊糖途径、回补途径、氨基酸代谢和脂肪酸合成途径的流量都发生较大变化。
2.4.4 应用于复合培养基的13C代谢流量分析微生物的工业发酵生产大宗化学品培养条件复杂。传统的13C-MFA的局限于最小培养基,限制了其应用范围。应用于复合培养基的13C-MFA能够理解细胞调控网络和工业环境信号之间的关系,展现出广阔的应用前景。针对复合培养基中存在未知的碳源,设计不同平行标记实验和选择最优的分析平台成为解决策略[72]。
真菌棉阿舒囊霉是产维生素B2的重要工业生产菌株,其培养基包括植物油、酵母提取物、谷氨酸、甘氨酸和甲酸。Schwechheimer等[8]结合平行标记实验([U-13C] 酵母提取物和[1, 3-13C]谷氨酸) 和基于GC-MS、13C NMR和1H NMR分析培养基中的组分对产物维生素B2的相对贡献,发现酵母提取物是合成代谢的主要碳源。流量计算结果显示三羧酸循环在生长和产物生成阶段具有高度的代谢活性,糖异生途径和磷酸戊糖途径的流量较低。该研究是将代谢流量分析应用到工业微生物的复杂培养条件的重要起始点。
Adler[9]借助平行标记[1, 2-13C]葡萄糖,[U-13C]葡萄糖和[U-13C]果糖,分别定量发酵乳杆菌NCC 575和528和植物乳杆菌NCC 2829和1295的流量分布。研究发现,发酵乳杆菌利用大量果糖合成木糖醇,仅余4%–6%进入糖酵解途径,而植物乳杆菌的大部分糖酵解流量由果糖摄取提供。进一步对微生物群落在可可浆工业发酵条件下进行了宏代谢流量组分析,发现发酵乳杆菌NCC 575贡献了群落总体流量的96%。
2.5 13C代谢流量分析的研究对象的扩展随着复杂生物体的稳定同位素注入技术的发展,13C代谢流量分析的应用不再局限于微生物系统,而是扩展至动植物细胞、植物,甚至小鼠和人等复杂生物体。Okahashi等[73]采用GC-NCI-MS检测磷酸糖类,通过优化GC温度梯度,获得基本骨架全部碳原子的片段,使戊糖磷酸和己糖磷酸的最低检测限达到10 mmol/L。作者以此检测方法为基础,定量人乳腺癌细胞系MCF-7的代谢流量分布,有效地提高了糖酵解途径和磷酸戊糖途径净流量和可逆流量的计算精度。Ma等[68]首次将非稳态13C-MFA应用到陆地植物系统拟南芥,从37个代谢物获得1 400个碎片,拟合136个流量(54个自由净流量)。全局光合作用碳流量的变化揭示了叶片对强光干扰时的代谢响应:羧化速率倍增,而光呼吸速率降低。Liu等[74]使用多种实验条件下的小鼠体内的同位素标记数据,构建13C-MFA数学模型,发现葡萄糖对TCA循环的相对贡献超过乳酸。进一步地,该结论在不同动物品系和组织中得到了验证。Faubert等[75]通过对非小细胞肺癌病人在手术前静脉注入13C标记乳酸,发现病人肿瘤样本中的TCA循环中间代谢物被广泛标记,进一步对移植人肺癌的小鼠静脉同时注入13C标记乳酸和葡萄糖,发现乳酸对于TCA循环的贡献甚至超过葡萄糖对其的贡献,从而揭示了乳酸是促进肿瘤体内生长、增殖甚至转移的主要能量原料。
3 13C代谢流量分析的应用随着代谢工程技术的不断推进,生物体内固有的各种代谢通路将会被不断重构设计,对细胞工厂的优化也将会进一步升级(图 3)。因此,基于13C代谢流量分析的认识和设计,是目前工业生物技术的关键研究策略[3]。在大健康领域,健康研究的原因和复杂的疾病的出现,并在此基础上为早期发现、预防、诊断和治疗开发新策略,需要临床前模型。13C代谢流量分析,能够准确定量细胞内各个代谢反应的活性,有助于阐明这些重大疾病的发生、发展和转归的机制[76] (图 4)。本节将依据具体案例分析13C代谢流量分析在工业生物技术和生物医药领域的应用。
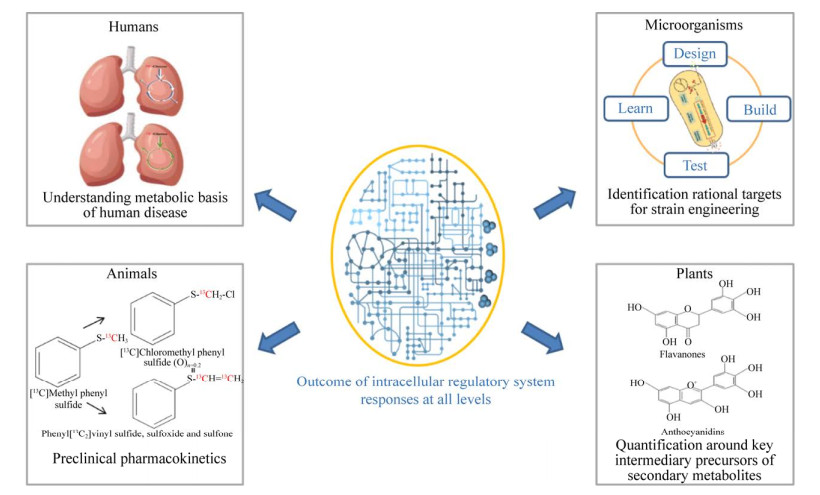
|
|
图 4 13C代谢流量分析的应用
Fig. 4 Applications of 13C-MFA.
|
| |
稳定同位素示踪技术能够通过示踪底物,将原子标记通过代谢反应传递到中间代谢物和产物,获得代谢途径的信息。基于此特性,代谢流量数据已经在辨别新型代谢反应和途径、完善代谢网络模型、验证代谢预测正确与否,得到了广泛的应用,尤其对生产菌株和非模式菌株的生理代谢特性有了系统的研究和认识[77]。
Roell等[78]借助13C途径分析、13C脉冲示踪和13C-MFA分析红球菌PD630代谢苯酚的机制:苯酚通过邻位剪切途径进行代谢;苯酚利用需要高活性的TCA循环;NADPH主要通过异柠檬酸脱氢酶产生;瀑布反应增加了TCA循环的柔性;2-酮-3-脱氢-6-磷酸葡糖酸途径反向,与糖异生协同作用。该研究首次报道了13C-MFA在微生物以苯酚为唯一碳源的全链条应用,为深入研究木质素的利用提供新思路。
Zhang等[79]利用动态15N及13C代谢流量与代谢组分析技术,建立数学模型,研究了蓝藻对外界氮源扰动的代谢响应,通过鸟氨酸标记动力学数据失拟,发现细胞内鸟氨酸和精氨酸之间存在活跃的代谢循环。进而发现该循环包含一步新的生化反应,即精氨酸双水解酶催化精氨酸水解生成鸟氨酸和氨。当模型加入此反应,鸟氨酸的拟合优度高。鸟氨酸-氨循环具有氮存储和活化的功能,对于蓝藻适应环境氮源缺乏和变化极其重要。鸟氨酸-氨循环在蓝藻中广泛存在,包括许多海洋固氮蓝藻,因此这一代谢途径对于海洋氮固定乃至地球的氮循环具有重要贡献。
Cui等[13]对比了产脂质隐甲藻在添加和不添加乙醇胺条件下的流量分布,发现添加乙醇胺增强了糖酵解途径和柠檬酸-丙酮酸循环的流量,降低了磷酸戊糖途径和TCA循环的流量。理解化学调节剂对隐甲藻的调控作用,为提高脂质的积累和廿二碳六烯酸的产量提供有效指导。
3.1.2 改造靶点鉴定和途径优化13C-MFA除了能够用于辨别新的代谢途径和阐明代谢网络结构外,还能用于分析控制代谢流量分布的调控节点,成为设计-创建-测试-学习(Design-Build-Test-Learn,DBTL) 的关键步骤,对于细胞工厂的系统性代谢网络改造至关重要。在完成代谢途径的遗传改造后,还要对细胞的生理变化、代谢通量进行详细分析,以此来决定下一步遗传改造的靶点。通过多个循环,不断提高细胞的生产性能[72]。
Cheah等[11]利用动态代谢流量分析定量了产乙醛蓝细菌的代谢流量,发现4个与丙酮酸节点相邻的反应变化明显,即与产物合成正相关的丙酮酸激酶和乙酰乳酸合成酶、与产物合成负相关的丙酮酸脱氢酶和磷酸烯醇式丙酮酸羧化酶。进一步验证4个靶点,过表达正相关的酶未增加乙醛产量,而通过反义表达RNA技术下调负相关的酶使产量提升了40%–60%。
Okahashi等[12]借助13C-MFA成功解析了产异丙醇大肠杆菌细胞内的代谢调控机制,发现NADPH的再生限制了目标产物的合成。代谢网络包括糖酵解途径、磷酸戊糖途径、三羧酸循环、2-酮-3-脱氧-6-磷酸葡糖酸途径、回补途径、有机酸分泌途径、异丙醇合成途径、氨基酸代谢和生物量合成反应。研究使用EMU算法、OpenMebius软件包,同时拟合蛋白氨基酸(丙氨酸、天冬氨酸、谷氨酸、苯丙氨酸、甘氨酸、酪氨酸和色氨酸) 和中间代谢物(延胡索酸、苹果酸、柠檬酸、α-酮戊二酸、甘油酸-3-磷酸、葡萄糖-6-磷酸,磷酸烯醇式丙酮酸、果糖-6-磷酸、景天庚酮糖-7-磷酸、果糖-1, 6-二磷酸) 碎片的同位体分布信息。出发菌株生长期和非生长期的自由净流量为25和26,冗余测量为43和38,模拟计算值和实际测量值之间的加权的残差平方和为40和48.9;优良工程菌株在生长期和非生长期的自由净流量都为27,冗余测量为41和45,模拟计算值和实际测量值之间的加权的残差平方和为21.3和53.3。残差平方和都在系统建模不确定度的上界和下界之间,通过卡方检验验证了系统模型的准确性。研究者循序渐进地提高胞内NADPH的水平:(1) 利用氮源限制的培养条件降低用于生物合成的NADPH的消耗;(2) 激活2-酮-3-脱氢-6-磷酸葡糖酸途径;(3) 组合使用上述两种方法。最终,异丙醇的产率达到0.67 mmol/(g·h),对葡萄糖的得率达到0.55 mol/mol。
Yang等[7]发掘在大肠杆菌同时表达甲基磷酸赤藓糖醇途径(Methylerythritol phosphate pathway,MEP) 和甲羟戊酸途径(Mevalonate pathway,MVA) 时,异戊二烯的产率大幅增加。13C代谢流量分析的结果揭示MEP和MVA的流量增加,进一步通过膦胺霉素处理和甲羟戊酸添加,验证了两个途径的协同作用。最终实现异戊二烯的产量达到24 g/L,产物对葡萄糖的得率达到0.267 g/g。
3.1.3 发酵工艺的优化13C代谢流量分析能够阐明代谢网络结构及调控特性,深入认识复杂系统。通过细致刻画不同阶段代谢流分布,阐明合成产物的菌株代谢生理与发酵产量的关系,进一步进行精准调控,该技术逐步应用到工业发酵优化,尤其是抗体的生产中[80-81]。受限于标记底物的价格,13C代谢流量分析在其他化学品的宏观尺度代谢调控和发酵优化未完全铺开应用。
Liu等[63]通过平行标记[1-13C]乙酸和[U-13C]乙酸,定量耶氏解脂酵母在生长阶段和产甘油三酯阶段的代谢流量分布。研究发现,从生长阶段转移到脂质生产阶段,糖异生途径作用从提供生物合成原料转变为支撑磷酸戊糖途径供给脂质合成需要的NADPH,流量需求增加。其动态和快速的变化通过苹果酸转运酶和丙酮酸激酶的活性严格调控,多余的乙醛酸支路流量返排TCA循环用以供能。为提高甘油三酯的产量,细胞需要有效分配碳源,优化生产阶段流量分布,实现能量和产物合成的竞争均衡。
Templeton等[80]对中国仓鼠细胞接近工业过程的批次流加培养的4个阶段(对数生长期早期、对数生长期晚期、稳定期和衰亡期) 进行了代谢流量分析。细胞生长峰值与糖酵解流量相关,而抗体的生产峰值与高度氧化状态相关。当培养从生长阶段跨入抗体生产阶段,乳酸代谢从净生产转变为净消耗,能量源自氧化磷酸化,与增加的氧化磷酸戊糖途径的活性关联。在稳定期,比生长速率持续降低,TCA循环流量和抗体生产达到峰值。基于上述结果,作者提出了中国仓鼠细胞的工业培养策略,增加TCA循环和磷酸戊糖途径氧化分支的流量,以提高抗体生产能力和降低乳酸的积累。
3.1.4 代谢流量组学与其他组学的整合分析影响目标产物合成的调控机制是复杂的多层次的非线性网络变化,使得任何一种建立在单一学科上的研究方法都难以完整地揭示规律。代谢流量组学与其他组学的整合分析,实现从基因组变异-表达变化-代谢流调节-网络适应性的全流程分析,在定量的水平上来理解生物体是如何作为一个构造严密的系统,从整体上对代谢进行调控,从而指导代谢工程与合成生物学的设计和改造。
Yao等[10]通过13C-MFA与代谢组学结合使用,成功解析了产丙酮醇大肠杆菌的代谢调控规律。通过比较分析初代菌株和对照菌株,发现在初代菌株中,转氢反应流向为NADH→NADPH,与对照菌株相反,NADPH浓度和NADPH/NADP+降低,由此确定转氢途径是限速步骤。利用表达转氢途径中的nadK、pntAB以及串联表达nadK和pntAB基因,丙酮醇的产量分别提高了65%、141%和208%。进一步比较了5株菌的流量分布,发现糖酵解上游途径、磷酸戊糖途径和TCA循环的流量保守,说明了维持生物合成的必需流量,彰显代谢网络的刚性。二羟丙酮磷酸为柔性节点,通过此节点,碳流量从糖酵解下游途径转移至产物丙酮醇生成途径。乙酰辅酶A也是一个柔性节点,通过它的再分配使TCA流量保守。
Wang等[82]利用转录组、代谢组和代谢流量组,深度解析了链霉菌甘油三酯(Triacylglycerols,TAGs) 在衔接初级代谢和聚酮合成过程的关键作用:TAGs在初级代谢阶段大量积累,当菌体生长进入稳定期开始合成聚酮时,TAGs则开始降解;TAGs的降解不但能为聚酮合成提供必要的前体和还原力,还能调节更多的碳流转向聚酮合成。基于上述基础,作者提出了一个精准动态控制内源TAGs水平提高聚酮产量的工程策略,实现了若干Ⅰ型聚酮类药物(阿维菌素和米尔倍霉素) 和Ⅱ型聚酮类药物(土霉素和杰多霉素) 的高产菌株构建。
D’Espaux等[16]借助代谢流量组和蛋白组学分析,揭示产脂肪醇酿酒酵母出发菌株的代谢瓶颈。通过pull-push-block策略,引入活性最高的外源脂肪酸还原酶,敲除消耗脂酰辅酶A或脂肪醇的竞争途径,过表达乙酰辅酶A羧化酶,优化辅因子利用,最终实现脂肪醇的产量在摇瓶中达到1.2 g/L,在发酵罐中达到6.0 g/L。
3.2 13C代谢流量分析在生物医药领域的应用13C代谢流量分析能够用于研究细胞在营养条件改变、胁迫条件施加及药物投放等环境扰动时的代谢动态响应及其调控机制。通过13C代谢流量分析揭示代谢活动的基本规律,为各类代谢性疾病和肿瘤发生提供进行干预的理论依据,已在生物医药领域取得了令人瞩目的进展[83-84]。
Spinelli等[85]利用15N动态示踪和代谢组分析技术,发现乳腺癌细胞能够将代谢废弃物-氨通过谷氨酸脱氢酶进行再利用,从而满足其快速生长的需要;通过对接受人乳腺癌移植的小鼠进行15N同位素腹部注入和动态示踪,发现肿瘤将其微环境中大量积累的氨直接用于合成氨基酸,从而揭示了氨对于乳腺癌细胞生长具有重要的促进作用。Christen等[20]通过小鼠注入13C标记葡萄糖,检测TCA循环中间代谢物的标记模式,建立数学模型,发现肺转移的丙酮酸羧化酶的流量显著高于原发性乳腺癌。这个突破性的进展说明乳腺癌细胞在体内肺部微环境能够依赖丙酮酸羧化酶进行回补。进一步验证机制,发现该响应由丙酮酸羧化酶表达的变化和线粒体中丙酮酸的浓度介导。
4 总结与展望随着代谢流量组学技术的出现,催生代谢科学理论研究和生物产业发展的更深层次的需求和进步,成功改造和优化重要微生物的生理和代谢功能,显著提升特定代谢产物的生产水平,实现从天然产物到高值化合物的低成本生物制造。随着相关技术的不断发展,13C代谢流量分析主要有以下发展趋势:
1) 13C代谢流量分析的标准化问题
尽管花强等[46],Zamboni[28]和Long[29]等已系统描述了13C代谢流量分析平台的建立,然而所基于的是模式生物。针对不同的科学问题和高等生物,从标记指导、样本收集(动态代谢流量分析)与检测、流量计算和统计分析的流程,都需要纳入标准化。代谢组学和动态代谢流量分析需要捕捉到代谢物的高度瞬间态,因此样本收集的时间点和前处理的准确性决定是否具有后续分析的价值。代谢组学和代谢流量组学检测使用的技术平台是一致的。对于LC-MS,Aligent、Thermo Fisher Scientific、SCIEX、Waters和Bruker都有自己的分析软件和数据处理方法,但无法进行交叉验证。只有通过解决这些问题,才能促使13C代谢流量分析技术本身和相关领域的研究水平得到提升,全面进入精准医学。
2) 高通量分析
培养系统的逐渐小型甚至到微型化节省了培养基的利用、培养空间和标记底物的花费。Fischer等[45]首先使用微量滴定板技术,与摇瓶发酵的计算获得的流量具有很好的一致性。Fischer等[86]应用这项技术,对137株枯草芽孢杆菌基因缺失菌株进行了大规模的代谢流量分析,极大地提高了对于细菌代谢网络恒定性和灵活性的认识。整合13C代谢流量分析与高通量技术是将代谢流量组学能否作为快速筛选工具的关键步骤。
3) 工业生产
与高通量分析相反,在工业生产情况下应用13C代谢流量分析需要将培养扩大化,以反映真实发酵条件下的流量分布。昂贵的标记底物价格成为了限制性因素。针对这一问题,El Massaoudi等[87]通过操作“传感器反应器”,在大规模反应器进行生产,成功将13C-MFA应用于氨基酸的工业生产[88-89],确定磷酸烯醇式丙酮酸节点的流量变化与氨基酸的产率相关。Yuan等[90]使用GC-C-IRMS的方法,提高同位素检测的灵敏度到3个量级,有效降低标记底物用量。研究高效的微生物菌株在工业发酵条件最佳生理性能,有效扩大了13C代谢流量分析的应用范围。
4) 基因组尺度的13C-MFA
基因组尺度的代谢网络模型(Genome-scale metabolic models,GEMs) 已成为代谢工程研究的重要工具之一,然而大部分模型并未经过严格的实验验证[91]。同位素标记异构体分布模型被简化为核心模型[92]。Martín等[92]系统定义了反应的核心子集,设定其单向偶联周边的基因组尺度的反应,满足同位素标记信息检测的质量和数量,对解空间设置合理约束。GEMs的综合范围和13C-MFA的数据整合能力的有效结合,从整体上对大规模生物学数据进行全面、系统地分析和整理,在系统生物学中发挥了举足轻重的作用。
13C代谢流量分析经过30年的发展,突破了最初设置的许多限制,研究范围从简单的微生物系统扩展到了高等生物,应用从学术研究到形成产品线。未来,随着其在代谢科学的引领作用和大健康领域引起的关注,必将迎来更加蓬勃的发展。
| [1] |
Bailey JE. Toward a science of metabolic engineering. Science, 1991, 252(5013): 1668-1675. DOI:10.1126/science.2047876
|
| [2] |
Stephanopoulos G, Stafford DE. Metabolic engineering: a new frontier of chemical reaction engineering. Chem Eng Sci, 2002, 57(14): 2595-2602. DOI:10.1016/S0009-2509(02)00088-X
|
| [3] |
Choi KR, Jang WD, Yang D, et al. Systems metabolic engineering strategies: integrating systems and synthetic biology with metabolic engineering. Trends Biotechnol, 2019, 37(8): 817-837. DOI:10.1016/j.tibtech.2019.01.003
|
| [4] |
李寅. 代谢工程: 一项不断发展的菌株改造技术. 生物工程学报, 2009, 25(9): 1281-1284. Li Y. Metabolic engineering: an evolving technology for strain improvement. Chin J Biotech, 2009, 25(9): 1281-1284 (in Chinese). DOI:10.3321/j.issn:1000-3061.2009.09.001 |
| [5] |
Chae TU, Choi SY, Kim JW, et al. Recent advances in systems metabolic engineering tools and strategies. Curr Opin Biotechnol, 2017, 47: 67-82. DOI:10.1016/j.copbio.2017.06.007
|
| [6] |
Sauer U. Metabolic networks in motion: 13C-based flux analysis. Mol Syst Biol, 2006, 2: 62. DOI:10.1038/msb4100109
|
| [7] |
Yang C, Gao X, Jiang Y, et al. Synergy between methylerythritol phosphate pathway and mevalonate pathway for isoprene production in Escherichia coli. Metab Eng, 2016, 37: 79-91. DOI:10.1016/j.ymben.2016.05.003
|
| [8] |
Schwechheimer SK, Becker J, Peyriga L, et al. Metabolic flux analysis in Ashbya gossypii using 13C-labeled yeast extract: industrial riboflavin production under complex nutrient conditions. Microb Cell Fact, 2018, 17(1): 162. DOI:10.1186/s12934-018-1003-y
|
| [9] |
Adler P, Bolten CJ, Dohnt K, et al. Core fluxome and metafluxome of lactic acid bacteria under simulated cocoa pulp fermentation conditions. Appl Environ Microbiol, 2013, 79(18): 5670-5681. DOI:10.1128/AEM.01483-13
|
| [10] |
Yao R, Li J, Feng L, et al. 13C metabolic flux analysis-guided metabolic engineering of Escherichia coli for improved acetol production from glycerol. Biotechnol Biofuels, 2019, 12: 29. DOI:10.1186/s13068-019-1372-4
|
| [11] |
Cheah YE, Xu Y, Sacco SA, et al. Systematic identification and elimination of flux bottlenecks in the aldehyde production pathway of Synechococcus elongatus PCC 7942. Metab Eng, 2020, 60: 56-65. DOI:10.1016/j.ymben.2020.03.007
|
| [12] |
Okahashi N, Matsuda F, Yoshikawa K, et al. Metabolic engineering of isopropyl alcohol-producing Escherichia coli strains with 13C-metabolic flux analysis. Biotechnol Bioeng, 2017, 114(12): 2782-2793. DOI:10.1002/bit.26390
|
| [13] |
Cui J, Diao J, Sun T, et al. 13C metabolic flux analysis of enhanced lipid accumulation modulated by ethanolamine in Crypthecodinium cohnii. Front Microbiol, 2018, 9: 956. DOI:10.3389/fmicb.2018.00956
|
| [14] |
Lange A, Becker J, Schulze D, et al. Bio-based succinate from sucrose: High-resolution 13C metabolic flux analysis and metabolic engineering of the rumen bacterium Basfia succiniciproducens. Metab Eng, 2017, 44: 198-212. DOI:10.1016/j.ymben.2017.10.003
|
| [15] |
Hoffmann SL, Jungmann L, Schiefelbein S, et al. Lysine production from the sugar alcohol mannitol: design of the cell factory Corynebacterium glutamicum SEA-3 through integrated analysis and engineering of metabolic pathway fluxes. Metab Eng, 2018, 47: 475-487. DOI:10.1016/j.ymben.2018.04.019
|
| [16] |
D'Espaux L, Ghosh A, Runguphan W, et al. Engineering high-level production of fatty alcohols by Saccharomyces cerevisiae from lignocellulosic feedstocks. Metab Eng, 2017, 42: 115-125. DOI:10.1016/j.ymben.2017.06.004
|
| [17] |
Sellers K, Fox MP, Bousamra M, et al. Pyruvate carboxylase is critical for non-small-cell lung cancer proliferation. J Clin Invest, 2015, 125(2): 687-698. DOI:10.1172/JCI72873
|
| [18] |
Tardito S, Oudin A, Ahmed SU, et al. Glutamine synthetase activity fuels nucleotide biosynthesis and supports growth of glutamine-restricted glioblastoma. Nat Cell Biol, 2015, 17(12): 1556-1568. DOI:10.1038/ncb3272
|
| [19] |
Wang Y, Nasiri AR, Damsky WE, et al. Uncoupling hepatic oxidative phosphorylation reduces tumor growth in two murine models of colon cancer. Cell Rep, 2018, 24(1): 47-55. DOI:10.1016/j.celrep.2018.06.008
|
| [20] |
Christen S, Lorendeau D, Schmieder R, et al. Breast cancer-derived lung metastases show increased pyruvate carboxylase-dependent anaplerosis. Cell Rep, 2016, 17(3): 837-848. DOI:10.1016/j.celrep.2016.09.042
|
| [21] |
Davidson SM, Jonas O, Keibler MA, et al. Direct evidence for cancer-cell-autonomous extracellular protein catabolism in pancreatic tumors. Nat Med, 2017, 23(2): 235-241. DOI:10.1038/nm.4256
|
| [22] |
Neinast MD, Jang C, Hui S, et al. Quantitative analysis of the whole-body metabolic fate of branched-chain amino acids. Cell Metab, 2019, 29(2): 417-429. DOI:10.1016/j.cmet.2018.10.013
|
| [23] |
Jang C, Hui S, Lu W, et al. The small intestine converts dietary fructose into glucose and organic acids. Cell Metab, 2018, 27(2): 351-361. DOI:10.1016/j.cmet.2017.12.016
|
| [24] |
Yam M, Engel AL, Wang Y, et al. Proline mediates metabolic communication between retinal pigment epithelial cells and the retina. J Biol Chem, 2019, 294(26): 10278-10289. DOI:10.1074/jbc.RA119.007983
|
| [25] |
Jiang H, Zeng J, Li W, et al. Practical and efficient strategy for evaluating oral absolute bioavailability with an intravenous microdose of a stable isotopically-labeled drug using a selected reaction monitoring mass spectrometry assay. Anal Chem, 2012, 84(22): 10031-10037. DOI:10.1021/ac3024558
|
| [26] |
Unkefer CJ, Martinez RA. The use of stable isotope labelling for the analytical chemistry of drugs. Drug Test Anal, 2012, 4(3/4): 303-307.
|
| [27] |
Dalman T, Wiechert W, Nöh K. A scientific workflow framework for 13C metabolic flux analysis. J Biotechnol, 2016, 232: 12-24. DOI:10.1016/j.jbiotec.2015.12.032
|
| [28] |
Zamboni N, Fendt SM, Rühl M, et al. 13C-based metabolic flux analysis. Nat Protoc, 2009, 4(6): 878-892. DOI:10.1038/nprot.2009.58
|
| [29] |
Long CP, Antoniewicz MR. High-resolution 13C metabolic flux analysis. Nat Protoc, 2019, 14(10): 2856-2877. DOI:10.1038/s41596-019-0204-0
|
| [30] |
Long CP, Antoniewicz MR. Quantifying biomass composition by gas chromatography/mass spectrometry. Anal Chem, 2014, 86(19): 9423-9427. DOI:10.1021/ac502734e
|
| [31] |
Crown SB, Long CP, Antoniewicz MR. Optimal tracers for parallel labeling experiments and 13C metabolic flux analysis: a new precision and synergy scoring system. Metab Eng, 2016, 38: 10-18. DOI:10.1016/j.ymben.2016.06.001
|
| [32] |
Toya Y, Ohashi S, Shimizu H. Optimal 13C-labeling of glycerol carbon source for precise flux estimation in Escherichia coli. J Biosci Bioeng, 2018, 125(3): 301-305. DOI:10.1016/j.jbiosc.2017.09.009
|
| [33] |
van Winden WA, Wittmann C, Heinzle E, et al. Correcting mass isotopomer distributions for naturally occurring isotopes. Biotechnol Bioeng, 2002, 80(4): 477-479. DOI:10.1002/bit.10393
|
| [34] |
Millard P, Letisse F, Sokol S, et al. IsoCor: correcting MS data in isotope labeling experiments. Bioinformatics, 2012, 28(9): 1294-1296. DOI:10.1093/bioinformatics/bts127
|
| [35] |
Heux S, Bergès C, Millard P, et al. Recent advances in high-throughput 13C-fluxomics. Curr Opin Biotechnol, 2017, 43: 104-109. DOI:10.1016/j.copbio.2016.10.010
|
| [36] |
Matthews DE, Bier DM. Stable isotope methods for nutritional investigation. Annu Rev Nutr, 1983, 3: 309-339. DOI:10.1146/annurev.nu.03.070183.001521
|
| [37] |
Christensen B, Nielsen J. Isotopomer analysis using GC-MS. Metab Eng, 1999, 1(4): 282-290. DOI:10.1006/mben.1999.0117
|
| [38] |
Schmidt K, Carlsen M, Nielsen J, et al. Modeling isotopomer distributions in biochemical networks using isotopomer mapping matrices. Biotechnol Bioeng, 1997, 55(6): 831-840. DOI:10.1002/(SICI)1097-0290(19970920)55:6<831::AID-BIT2>3.0.CO;2-H
|
| [39] |
Szyperski T. Biosynthetically directed fractional 13C-labeling of proteinogenic amino acids. An efficient analytical tool to investigate intermediary metabolism. Eur J Biochem, 1995, 232(2): 433-448. DOI:10.1111/j.1432-1033.1995.tb20829.x
|
| [40] |
Becker J, Wittmann C. GC-MS-based 13C metabolic flux analysis. New York, NY: Springer New York, 2014: 165-174.
|
| [41] |
Toya Y, Nakahigashi K, Tomita M, et al. Metabolic regulation analysis of wild-type and arcA mutant Escherichia coli under nitrate conditions using different levels of omics data. Mol Biosyst, 2012, 8(10): 2593-2604. DOI:10.1039/c2mb25069a
|
| [42] |
Jaiswal D, Mittal A, Nagrath D, et al. Liquid chromatography methods for separation of polar and charged intracellular metabolites for 13C metabolic flux analysis. Methods Mol Biol, 2020, 2088: 33-50.
|
| [43] |
Rühl M, Rupp B, Nöh K, et al. Collisional fragmentation of central carbon metabolites in LC-MS/MS increases precision of C metabolic flux analysis. Biotechnol Bioeng, 2012, 109(3): 763-771. DOI:10.1002/bit.24344
|
| [44] |
McCloskey D, Young JD, Xu S, et al. MID max: LC-MS/MS method for measuring the precursor and product mass isotopomer distributions of metabolic intermediates and cofactors for metabolic flux analysis applications. Anal Chem, 2016, 88(2): 1362-1370. DOI:10.1021/acs.analchem.5b03887
|
| [45] |
Fischer E, Zamboni N, Sauer U. High-throughput metabolic flux analysis based on gas chromatography-mass spectrometry derived 13C constraints. Anal Biochem, 2004, 325(2): 308-316. DOI:10.1016/j.ab.2003.10.036
|
| [46] |
花强, 杨琛. 代谢流量比率分析及其在代谢工程中的应用. 生物工程学报, 2009, 25(9): 1303-1311. Hua Q, Yang C. Application of metabolic flux ratio analysis in metabolic engineering—a review. Chin J Biotechnol, 2009, 25(9): 1303-1311 (in Chinese). DOI:10.3321/j.issn:1000-3061.2009.09.004 |
| [47] |
Zupke C, Stephanopoulos G. Modeling of isotope distributions and intracellular fluxes in metabolic networks using atom mapping matrices. Biotechnol Prog, 1994, 10(5): 489-498. DOI:10.1021/bp00029a006
|
| [48] |
Dai Z, Locasale JW. Understanding metabolism with flux analysis: from theory to application. Metab Eng, 2017, 43(Pt B): 94-102.
|
| [49] |
Wiechert W, Möllney M, Isermann N, et al. Bidirectional reaction steps in metabolic networks: Ⅲ. explicit solution and analysis of isotopomer labeling systems. Biotechnol Bioeng, 1999, 66(2): 69-85. DOI:10.1002/(SICI)1097-0290(1999)66:2<69::AID-BIT1>3.0.CO;2-6
|
| [50] |
Sriram G, Shanks JV. Improvements in metabolic flux analysis using carbon bond labeling experiments: bondomer balancing and Boolean function mapping. Metab Eng, 2004, 6(2): 116-132. DOI:10.1016/j.ymben.2004.02.003
|
| [51] |
van Winden WA, Heijnen JJ, Verheijen PJ. Cumulative bondomers: a new concept in flux analysis from 2D[13C, 1H] COSY NMR data. Biotechnol Bioeng, 2002, 80(7): 731-745. DOI:10.1002/bit.10429
|
| [52] |
Antoniewicz MR, Kelleher JK, Stephanopoulos G. Elementary metabolite units (EMU): a novel framework for modeling isotopic distributions. Metab Eng, 2007, 9: 68-86. DOI:10.1016/j.ymben.2006.09.001
|
| [53] |
Wiechert W, Möllney M, Petersen S, et al. A universal framework for 13C metabolic flux analysis. Metab Eng, 2001, 3(3): 265-283. DOI:10.1006/mben.2001.0188
|
| [54] |
Weitzel M, Nöh K, Dalman T, et al. 13CFLUX2-High performance software suite for 13C-metabolic flux analysis. Bioinformatics, 2013, 29: 143-145. DOI:10.1093/bioinformatics/bts646
|
| [55] |
Zamboni N, Fischer E, Sauer U. FiatFlux—a software for metabolic flux analysis from 13C-glucose experiments. BMC Bioinformatics, 2005, 6: 209. DOI:10.1186/1471-2105-6-209
|
| [56] |
Quek LE, Wittmann C, Nielsen LK, et al. OpenFLUX: efficient modelling software for 13C-based metabolic flux analysis. Microb Cell Fact, 2009, 8: 25. DOI:10.1186/1475-2859-8-25
|
| [57] |
Shupletsov MS, Golubeva LI, Rubina SS, et al. OpenFLUX2: 13C-MFA modeling software package adjusted for the comprehensive analysis of single and parallel labeling experiments. Microb Cell Fact, 2014, 13: 152.
|
| [58] |
Sokol S, Millard P, Portais JC. Influx_s: increasing numerical stability and precision for metabolic flux analysis in isotope labelling experiments. Bioinformatics, 2012, 28(5): 687-693. DOI:10.1093/bioinformatics/btr716
|
| [59] |
Young JD. INCA: a computational platform for isotopically non-stationary metabolic flux analysis. Bioinformatics, 2014, 30(9): 1333-1335. DOI:10.1093/bioinformatics/btu015
|
| [60] |
Kajihata S, Furusawa C, Matsuda F, et al. OpenMebius: an open source software for isotopically nonstationary 13C-based metabolic flux analysis. Biomed Res Int, 2014, 2014: 627014.
|
| [61] |
Millard P, Massou S, Wittmann C, et al. Sampling of intracellular metabolites for stationary and non-stationary 13C metabolic flux analysis in Escherichia coli. Anal Biochem, 2014, 465: 38-49. DOI:10.1016/j.ab.2014.07.026
|
| [62] |
Teleki A, Takors R. Quantitative profiling of endogenous metabolites using hydrophilic interaction liquid chromatography-tandem mass spectrometry (HILIC-MS/MS). Methods Mol Biol, 2019, 1859: 185-207.
|
| [63] |
Liu N, Qiao K, Stephanopoulos G. 13C metabolic flux analysis of acetate conversion to lipids by Yarrowia lipolytica. Metab Eng, 2016, 38: 86-97. DOI:10.1016/j.ymben.2016.06.006
|
| [64] |
He L, Xiu Y, Jones JA, et al. Deciphering flux adjustments of engineered E. coli cells during fermentation with changing growth conditions. Metab Eng, 2017, 39: 247-256. DOI:10.1016/j.ymben.2016.12.008
|
| [65] |
Nöh K, Grönke K, Luo B, et al. Metabolic flux analysis at ultra short time scale: isotopically non-stationary 13C labeling experiments. J Biotechnol, 2007, 129(2): 249-267. DOI:10.1016/j.jbiotec.2006.11.015
|
| [66] |
Cheah YE, Young JD. Isotopically nonstationary metabolic flux analysis (INST-MFA): putting theory into practice. Curr Opin Biotechnol, 2018, 54: 80-87.
|
| [67] |
Young JD, Walther JL, Antoniewicz MR, et al. An elementary metabolite unit (EMU) based method of isotopically nonstationary flux analysis. Biotechnol Bioeng, 2008, 99(3): 686-699. DOI:10.1002/bit.21632
|
| [68] |
Ma F, Jazmin LJ, Young JD, et al. Isotopically nonstationary 13C flux analysis of changes in Arabidopsis thaliana leaf metabolism due to high light acclimation. Proc Natl Acad Sci USA, 2014, 111(47): 16967-16972. DOI:10.1073/pnas.1319485111
|
| [69] |
Jazmin LJ, Xu Y, Cheah YE, et al. Isotopically nonstationary 13C flux analysis of cyanobacterial isobutyraldehyde production. Metab Eng, 2017, 42: 9-18. DOI:10.1016/j.ymben.2017.05.001
|
| [70] |
Cordova LT, Lu J, Cipolla RM, et al. Co-utilization of glucose and xylose by evolved Thermus thermophilus LC113 strain elucidated by 13C metabolic flux analysis and whole genome sequencing. Metab Eng, 2016, 37: 63-71. DOI:10.1016/j.ymben.2016.05.001
|
| [71] |
Ahn WS, Antoniewicz MR. Parallel labeling experiments with[1, 2-13C]glucose and[U-13C]glutamine provide new insights into CHO cell metabolism. Metab Eng, 2013, 15: 34-47. DOI:10.1016/j.ymben.2012.10.001
|
| [72] |
Schwechheimer SK, Becker J, Wittmann C. Towards better understanding of industrial cell factories: novel approaches for 13C metabolic flux analysis in complex nutrient environments. Curr Opin Biotechnol, 2018, 54: 128-137. DOI:10.1016/j.copbio.2018.07.001
|
| [73] |
Okahashi N, Maeda K, Kawana S, et al. Sugar phosphate analysis with baseline separation and soft ionization by gas chromatography-negative chemical ionization-mass spectrometry improves flux estimation of bidirectional reactions in cancer cells. Metab Eng, 2019, 51: 43-49. DOI:10.1016/j.ymben.2018.08.011
|
| [74] |
Liu S, Dai Z, Cooper DE, et al. Quantitative analysis of the physiological contributions of glucose to the TCA cycle. Cell Metab, 2020, 32(4): 619-628.e21. DOI:10.1016/j.cmet.2020.09.005
|
| [75] |
Faubert B, Li KY, Cai L, et al. Lactate metabolism in human lung tumors. Cell, 2017, 171(2): 358-371.e9. DOI:10.1016/j.cell.2017.09.019
|
| [76] |
Badur MG, Metallo CM. Reverse engineering the cancer metabolic network using flux analysis to understand drivers of human disease. Metab Eng, 2018, 45: 95-108. DOI:10.1016/j.ymben.2017.11.013
|
| [77] |
Nie X, Hua Q, Xu P, et al. Biological insights into non-model microbial hosts through stable-isotope metabolic flux analysis. Curr Opin Biotechnol, 2020, 64: 32-38. DOI:10.1016/j.copbio.2019.09.010
|
| [78] |
Roell GW, Carr RR, Campbell T, et al. A concerted systems biology analysis of phenol metabolism in Rhodococcus opacus PD630. Metab Eng, 2019, 55: 120-130. DOI:10.1016/j.ymben.2019.06.013
|
| [79] |
Zhang H, Liu Y, Nie X, et al. The cyanobacterial ornithine-ammonia cycle involves an arginine dihydrolase. Nat Chem Biol, 2018, 14(6): 575-581. DOI:10.1038/s41589-018-0038-z
|
| [80] |
Templeton N, Dean J, Reddy P, et al. Peak antibody production is associated with increased oxidative metabolism in an industrially relevant fed-batch CHO cell culture. Biotechnol Bioeng, 2013, 110(7): 2013-2024. DOI:10.1002/bit.24858
|
| [81] |
Templeton N, Xu S, Roush DJ, et al. 13C metabolic flux analysis identifies limitations to increasing specific productivity in fed-batch and perfusion. Metab Eng, 2017, 44: 126-133. DOI:10.1016/j.ymben.2017.09.010
|
| [82] |
Wang W, Li S, Li Z, et al. Harnessing the intracellular triacylglycerols for titer improvement of polyketides in Streptomyces. Nat Biotechnol, 2020, 38(1): 76-83. DOI:10.1038/s41587-019-0335-4
|
| [83] |
Charidemou E, Ashmore T, Griffin JL. The use of stable isotopes in the study of human pathophysiology. Int J Biochem Cell Biol, 2017, 93: 102-109. DOI:10.1016/j.biocel.2017.07.012
|
| [84] |
Lai M, Gruetter R, Lanz B. Progress towards in vivo brain 13C-MRS in mice: metabolic flux analysis in small tissue volumes. Anal Biochem, 2017, 529: 229-244. DOI:10.1016/j.ab.2017.01.019
|
| [85] |
Spinelli JB, Yoon H, Ringel AE, et al. Metabolic recycling of ammonia via glutamate dehydrogenase supports breast cancer biomass. Science, 2017, 358(6365): 941-946. DOI:10.1126/science.aam9305
|
| [86] |
Fischer E, Sauer U. Large-scale in vivo flux analysis shows rigidity and suboptimal performance of Bacillus subtilis metabolism. Nat Genet, 2005, 37(6): 636-640. DOI:10.1038/ng1555
|
| [87] |
El Massaoudi M, Spelthahn J, Drysch A, et al. Production process monitoring by serial mapping of microbial carbon flux distributions using a novel sensor reactor approach: Ⅰ—Sensor reactor system. Metab Eng, 2003, 5(2): 86-95. DOI:10.1016/S1096-7176(03)00004-1
|
| [88] |
Drysch A, El Massaoudi M, Mack C, et al. Production process monitoring by serial mapping of microbial carbon flux distributions using a novel Sensor Reactor approach: Ⅱ—13C-labeling-based metabolic flux analysis and L-lysine production. Metab Eng, 2003, 5(2): 96-107. DOI:10.1016/S1096-7176(03)00005-3
|
| [89] |
Wahl A, El Massaoudi M, Schipper D, et al. Serial 13C-based flux analysis of an L-phenylalanine- producing E. coli strain using the sensor reactor. Biotechnol Prog, 2004, 20(3): 706-714. DOI:10.1021/bp0342755
|
| [90] |
Yuan Y, Yang TH, Heinzle E. 13C metabolic flux analysis for larger scale cultivation using gas chromatography-combustion-isotope ratio mass spectrometry. Metab Eng, 2010, 12(4): 392-400. DOI:10.1016/j.ymben.2010.02.001
|
| [91] |
Buescher JM, Antoniewicz MR, Boros LG, et al. A roadmap for interpreting 13C metabolite labeling patterns from cells. Curr Opin Biotechnol, 2015, 34: 189-201. DOI:10.1016/j.copbio.2015.02.003
|
| [92] |
Martín HG, Kumar VS, Weaver D, et al. A method to constrain genome-scale models with 13C labeling data. PLoS Comput Biol, 2015, 11(9): e1004363. DOI:10.1371/journal.pcbi.1004363
|
 2021, Vol. 37
2021, Vol. 37