中国科学院微生物研究所、中国微生物学会主办
文章信息
- 郑小梅, 郑平, 孙际宾
- Zheng Xiaomei, Zheng Ping, Sun Jibin
- 基于CRISPR/Cas系统的黑曲霉基因组编辑技术
- CRISPR/Cas-based genome editing in Aspergillus niger
- 生物工程学报, 2021, 37(3): 980-990
- Chinese Journal of Biotechnology, 2021, 37(3): 980-990
- 10.13345/j.cjb.200613
-
文章历史
- Received: September 25, 2020
- Accepted: December 25, 2020
2. 中国科学院系统微生物工程重点实验室,天津 300308;
3. 中国科学院大学,北京 100049;
4. 国家合成生物技术创新中心,天津 300308
2. Key Laboratory of Systems Microbial Biotechnology, Chinese Academy of Sciences, Tianjin 300308, China;
3. University of Chinese Academy of Sciences, Beijing 100049, China;
4. National Technology Innovation Center of Synthetic Biology, Tianjin 300308, China
黑曲霉Aspergillus niger是重要的工业生产菌株,广泛用于有机酸与酶制剂的生产[1]。在有机酸生产方面,黑曲霉是柠檬酸[2]的主力生产菌株,其生产的柠檬酸全球年产量达200万t,产值超过20亿美元,并以每年5%的速度递增[2]。在酶制剂方面,2015年全球丝状真菌酶制剂的产值约35亿欧元,其中,黑曲霉所生产的葡糖淀粉酶、葡萄糖氧化酶与植酸酶等大宗酶制剂占有较大的市场份额[3]。黑曲霉具有极端环境耐受性、高生产经济性、强发酵鲁棒性和高食品安全性(拥有美国FDA的GRAS标识) 等,这些优势使得黑曲霉成为不可多得的细胞工厂[4]。黑曲霉基因组信息的公布与多组学数据的大量涌现,为揭示黑曲霉细胞工厂高效运转机制提供了知识储备[4-5]。但这些基于组学数据的高产机制假设还需在黑曲霉中进行分子验证,进而解析出分子机制。另外,在黑曲霉细胞工厂创建与改造的过程中,往往涉及碳源利用的优化、关键酶的表达强化、副产物途径的敲除与弱化以及菌丝体形态优化等多个方面[2],因此,也往往涉及基因组中多个不同基因的改造。由此可见,黑曲霉高效遗传操作的建立成为基因功能研究与细胞工厂改造优化的限制瓶颈。
近年来,基于CRISPR/Cas系统(Clustered Regularly Interspaced Short Palindromic Repeats (CRISPR)/CRISPR associated (Cas) genome editing system) 的基因组编辑技术,以其操作简单、靶向特异、编辑高效、通用性广等优势广泛应用于不同物种中[6]。在该技术中,Cas蛋白在引导RNA (Guide RNA,gRNA) 的招募下对基因组特定位点切割,产生DNA双链断裂(Double strand break,DSB)[6]。在真核生物中,DSB可通过体内DNA修复系统如非同源末端连接(Non-homologous end-joining,NHEJ) 或同源定向修复(Homology directed repair,HDR) 得以修复,实现基因组原位编辑[6]。在NHEJ修复系统中,异源二聚体Ku70-Ku80结合于DSB的DNA末端,然后招募DNA依赖的蛋白激酶催化亚基(DNA-dependent protein kinase catalytic subunit DNA-PKC) 与DNA连接酶LigD等,进行易错修复,引入不确定数量的碱基插入或缺失,导致基因失活。当细胞中存在供体DNA (Donor DNA) 时,可由HDR系统以供体DNA为修复模板进行同源重组修复,可对特定位点进行精确的基因编辑。利用Cas蛋白、引导RNA与供体DNA的合理设计,来实现基因组编辑。
随着CRISPR/Cas系统的快速发展,黑曲霉中也陆续建立了不同的基因组编辑技术体系。本文聚焦于该技术在黑曲霉中的开发与应用,重点综述不同技术体系中的关键设计策略,以及其在黑曲霉细胞工厂基因编辑与表达调控上的应用。最后,展望了该技术在黑曲霉细胞工厂改造优化中的未来发展方向。
1 基于CRISPR/Cas系统的黑曲霉基因组编辑技术基于CRISPR/Cas系统的基因组编辑技术中,核心组分主要包括Cas蛋白、引导RNA与供体DNA等。本节重点就该技术在黑曲霉中的开发优化及其核心技术设计策略进行综述。
1.1 Cas蛋白天然CRISPR/Cas系统是细菌与古菌中的获得性免疫系统,在CRISPR序列的引导下,Cas蛋白或Cas蛋白复合体识别并切割降解外源入侵的DNA。根据Cas蛋白与识别作用机制的不同,目前,CRISPR/Cas系统可分为两大类(Class 1与Class 2),6个不同类型(TypeⅠ、Ⅱ、Ⅲ、Ⅳ、Ⅴ、Ⅵ)[7]。第一大类CRISPR/Cas系统包括Ⅰ型、Ⅲ型与Ⅳ型,由多个Cas蛋白共同形成Cas复合体来参与外源DNA的识别与降解。而第二类CRISPR/Cas系统(Ⅱ型、Ⅴ型与Ⅵ型) 则仅需一个Cas蛋白在RNA的介导下即可发挥干扰功能[7]。因此,Ⅱ型系统中的Cas9[8-10]、Ⅴ型系统中的Cas12a (Cpf1)[11]已被用于黑曲霉基因编辑系统的建立。Cas9蛋白或Cas12a对基因组特定位点的切割依赖于引导RNA中靶向DNA序列上游或下游的PAM (Protospacer adjacent motif) 序列。目前,黑曲霉中常用的来源于酿脓链球菌Streptococcus pyogenes的SpCas9的PAM序列为5′-NGG-3′[12],而来源于毛螺菌Lachnospiraceae bacterium的LbCas12a (LbCpf1) 的PAM序列为5′-TTTN-3′[13]。因此,基于不同Cas蛋白的基因编辑技术为扩展黑曲霉基因组的可编辑范围提供了技术支撑。
Cas蛋白的突变体及其与不同功能蛋白的融合,可进一步扩展基因组编辑的能力。比如Cas9的核酸酶结构域RuvC与HNH[12],可分别对gRNA的DNA互补链与非互补链进行切割,位于RuvC结构域中的D10A突变体与位于HNH结构域的H840A突变体可使相应的结构域失活,因此使Cas9成为切口酶nCas9,仅可对DNA进行单链切割[12]。Huang等[14]在nCas9 (D10A) 的N端融合了大鼠来源的胞嘧啶脱氨酶APOBEC1,实现了不依赖于供体DNA的黑曲霉基因组原位碱基突变。
在黑曲霉中,建立CRISPR/Cas基因组编辑技术时,Cas蛋白的表达需要考虑以下3点:1)由于Cas蛋白来源细菌或古菌,需要根据黑曲霉密码子偏好性进行密码子优化以保证Cas蛋白的正常翻译。2) 基因组编辑发生于细胞核,需在Cas蛋白的N端和/或C端添加核定位信号(Nuclear localization signal,NLS),如来源于SV40病毒的NLS (PKKKRKV),以保证Cas蛋白的细胞核定位[9]。3) 用于Cas蛋白表达的启动子,多为组成型强启动子如Ptef1[8]、PcoxA[15]与PpkiA[16]等,或诱导型启动子如PglaA[17]等(表 1)。
| gRNA expression | Cas protein expression(expression cassette, SM) | Donor DNA(SM, homology arm size (bp/nt)) | DNA repair systema | Gene editing typeb | Efficiency | References |
| sgRNA expression derived by II type promoters | ||||||
| PgpdA-HH-sgRNA-HDV-TtrpC | Ptefl-cas9-Ttefl, hph | \ | NHEJ | GD | Success | [8] |
| PmbfA-HDV-sgRNA-HH-TtrpC | PcoxA-cas9-TtrpC, hph, | pyrG\690(U), 834(D) | HDR | KI | 100% | [15] |
| PgpdA-HH-sgRNA-HDV-TtrpC | Ptefl-cas9-Ttefl, hph | Marker-free\90 SSD | HDR | GD, BE | 60% | [29] |
| sgRNA expression derived by III type promoters | ||||||
| PafU3-tRNA-sgRNA-tRNA-TafU3 | Ptefl-Lbcpfl-Ttefl, pyrG | Marker-free\90 SSD | HDR | GD, BE | 80% | [11] |
| Marker-free\60 | HDR | KI | Success | |||
| PhU6-sgRNA-TU6 | PglaA-cas9-TglaA, amdS | \ | NHEJ | GD | 15% | [9] |
| PyU6-sgRNA-TU6 | PglaA-cas9-TglaA, amdS | \ | NHEJ | GD | 20% | |
| PanU6-sgRNA-TU6 | PglaA-cas9-TglaA, amdS | \ | NHEJ | GD | 23% | |
| PanU6-sgRNA-TU6 | PglaA-cas9-TglaA, amdS | hph\ 40 | HDR | GD | 36% | |
| PaoU6-sgRNA-TU6 | Ptefl -rAPOBECl-nCas9-UGI-Ttefl, hph | \ | \ | BE | 47.4%-100% | [14] |
| P5S rRNA-sgRNA -TU6 | PglaA-cas9-TglaA, amdS | \ | NHEJ | GD | 95.28%-100% | [10] |
| hph\ 40 | HDR | GD, KO | 100% | |||
| hph\ 40 | HDR | GD | 45.83% (Double) | |||
| P5S rRNA-sgRNA-TU6 | PglaA-cas9-TglaA, hph | hph\ 40 | HDR | GD | 100% | [17] |
| hph\ 40 | HDR | GD | 25% (WT) | |||
| PtRNA-sgRNA -TtRNA | PpkiA-cas9-TglaA, pyrG | \ | NHEJ | GD | 13%—96% | [16] |
| Marker-free\600 | HDR | GR | 42% (WT) 93% 11)kns. 11 | |||
| PtRNA-sgRNA-TtRNA | PpkiA-cas9-TglaA, pyrG | Marker-free\60 SSD | HDR | BE | Success | [39] |
| PtRNA-sgRNA-TtRNA | Ptefl-cas9-Ttefl, hph | Marker-free\ 1 000 | HDR | KO | 66.7%-100% | [36] |
| sgRNA in vitro transcription | ||||||
| In vitro transcription | Ptefl-cas9-Ttefl, hph | pyrG\1500 | HDR | KO | 70%-80% | [37] |
| In vitro transcription | Ptefl-cas9-Ttefl, hph | pyrG\1500 | HDR | KO | 27.5%-100% | [23] |
| In vitro transcription | Ptefl-cas9-Ttefl, hph | 731 (UP)/419(DW) | HDR | BE | Success | [28] |
| In vitro transcription | Cas9 protein expressed in Escherichia coli | pyrG\500 | HDR | KI | 25% | [31] |
| pyrG\1000 | HDR | KI | 100% | |||
| pyrG\1500 | HDR | KI | 100% | |||
| pyrG\1500 | HDR | KI | 13%—25% (Double) | |||
| pyrG\1500 | HDR | KI | 17% (Triple) | |||
| Note: (a) DNA repair system. NHEJ: non-homologous end-joining system; HDR: homology directed repair system. (b) Gene editing type. GD: gene disruption; KO: gene knock-out; KI: gene knock-in; GR: gene replacement; BE: base editing. | ||||||
在细菌或古菌的天然CRISPR/Cas系统中,引导RNA是以CRISPR阵列(CRISPR array) 的方式存在。比如,在CRISPR/Cas9系统中,CRISPR阵列转录形成crRNA的初级转录本,然后在RNase Ⅲ的参与下加工为成熟的crRNA[18]。crRNA与反式激活RNA (tracrRNA) 结合形成crRNA: tracrRNA二聚体后,被Cas9识别并结合[18]。在CRISPR/Cas9基因组编辑技术中,常常将crRNA: tracrRNA设计为一条包含靶向信息的引导RNA (Single guide RNA,sgRNA),利用sgRNA中靶向DNA序列与基因组特定位置的互补配对来定位Cas9[12]。sgRNA中靶向DNA序列可利用Cas-Designer (http://www.rgenome.net/cas-designer/)[19]、Cas- OFFinder (http://www.rgenome.net/cas-offinder/)[20]、sgRNACas9 (https://sourceforge.net/projects/sgrna cas9/)[21]等软件分析来设计,以提高靶向编辑效率。
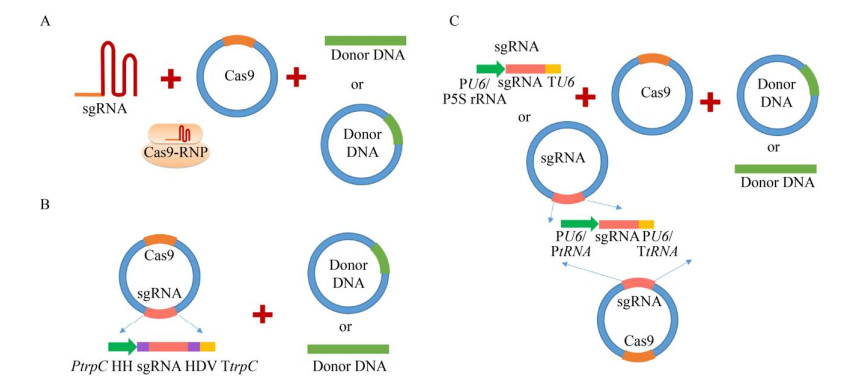
黑曲霉非编码RNA转录调控机制的研究较少,因此,sgRNA的表达成为建立基于CRISPR/Cas系统的基因组编辑技术的关键瓶颈问题。针对这一问题,目前多个不同sgRNA表达策略陆续被报道,主要分为体外转录[22-23]与RNA聚合酶Ⅱ或RNA聚合酶Ⅲ所识别的启动子介导的体内表达[8-10, 15-16] (图 1,表 1)。在sgRNA的体外转录方面,Kuivanen等[22-23]采用T7启动子在体外对sgRNA进行合成,然后与Cas9表达质粒或Cas9重组蛋白共转化至黑曲霉中来进行基因编辑(图 1A,表 1)。这一策略仅能用于瞬时编辑,且sgRNA稳定性与转化效率也会影响基因组编辑效率[24]。
|
| 图 1 基于CRISPR/Cas9系统的黑曲霉基因组编辑技术 Fig. 1 CRISPR/Cas9 genome editing system in A. niger. Three sgRNA expression strategies have been established in A. niger. (A) sgRNA was expressed by in vitro transcription and co-transformed into A. niger protoplasts with the Cas9 recombinant protein or Cas9 expression plasmids. (B) sgRNA was expressed under the control of promoters recognized by RNA polymerase Ⅱ, and then processed by the fused ribozymes. (C) sgRNA was expressed by the promoters recognized by RNA polymerase Ⅲ. sgRNA: single guide RNA; Cas9 RNP: Cas9/sgRNA ribonucleoprotein; HH: hammerhead ribozymes; HDV: hepatitis delta virus ribozymes. |
| |
在RNA聚合酶Ⅱ所识别的启动子介导的体内表达方面,最早Nodvig等[8]利用来自于构巢曲霉Aspergillus nidulans的RNA聚合酶Ⅱ所识别的强启动子PgpdA来进行sgRNA的表达(图 1B,表 1)。但由于RNA聚合酶Ⅱ用于小RNA转录时往往引起通读,而严重影响sgRNA的构象,需在sgRNA的5′-端与3′-端添加可用于自我切割的核酶。后续,Sarkari等[15]也采用类似策略,以PmbfA用于sgRNA的表达。
在sgRNA体内表达中,除RNA聚合酶Ⅱ所识别的启动子外,目前RNA聚合酶Ⅲ识别的启动子也不断被鉴定,并用于黑曲霉sgRNA的表达(图 1C,表 1)。Zheng等[9]测试发现来源于人类、酵母及黑曲霉内源的PU6均可实现黑曲霉albA基因的失活,但基因失活效率相对较低,仅为15%-36%。Song等[16]也测试了37个tRNA启动子的sgRNA表达效果,发现除tRNAGly5以外,其他tRNA启动子可起始sgRNA的转录,但基因失活效率在2%-13%之间。针对sgRNA表达水平低的问题,Zheng等[10]首次提出5S rRNA启动子来起始sgRNA的转录,发现内源5S rRNA启动子可显著提高sgRNA的表达水平,使基因靶向失活效率达100%。
1.3 供体DNACas蛋白在gRNA的引导下产生DSB后,若存在带有同源臂的供体DNA,可在HDR系统下,实现靶基因的定向精准编辑[25]。在丝状真菌中,DNA的DSB修复以非同源末端连接为主,同源重组效率低于5%[26-27]。在传统基因编辑中,通过增加同源臂的长度可提高同源重组效率,比如在黑曲霉AB4.1中,当同源臂的长度由100 bp提高至1 500 bp时,基因敲除效率从4%提高至29%[27]。为简化供体DNA的构建,Zheng等[10]直接利用带有同源臂的引物一步PCR扩增即可获得供体DNA,发现利用基于5S rRNA的CRISPR/Cas9系统,即使短至4 bp的同源臂在野生型与NHEJ失活菌株中的精准编辑效率也可分别达到75%与100% (表 1)。Vanegas等[11]也发现借助CRISPR/LbCpf1系统,仅含有60 bp的同源臂的无痕供体DNA,也可实现荧光蛋白的定点插入(表 1)。除双链供体DNA外,超短的单链寡核苷酸DNA (Single stranded DNA,ssDNA) 也作为供体DNA用于黑曲霉的碱基编辑[11, 28-29] (表 1)。
1.4 Cas蛋白与引导RNA的转化方式外源DNA转化至黑曲霉细胞中是遗传改造的关键环节。黑曲霉DNA转化方式主要包括原生质体介导的转化、农杆菌介导的转化与电击转化等。目前,原生质体转化是黑曲霉CRISPR/Cas基因编辑技术中的主要转化方法[30]。基于CRISPR/ Cas系统的黑曲霉基因组编辑技术中,DNA转化方式也多采用原生质体转化的方法,根据转化组分元件的不同,主要包括以下3种:1) Cas蛋白表达质粒与sgRNA表达质粒或表达盒以DNA的方式共转化至原生质体中,Cas蛋白/sgRNA的表达盒可整合至基因组[9-10],或存在于含有自主复制元件AMA1的质粒上[8] (表 1)。2) sgRNA体外转录后,以RNA的方式与Cas蛋白表达质粒共转化至原生质体中,sgRNA在体内瞬时存在[23] (表 1)。3) sgRNA通过体外转录获得,Cas蛋白通过在大肠杆菌中进行重组表达与纯化制备获得,sgRNA与Cas蛋白首先在体外形成RNP复合体(Cas/sgRNA ribonucleoprotein complex),然后以蛋白-RNA复合体的方式转化至原生质体中,sgRNA与Cas蛋白在体内均为瞬时存在[31] (表 1)。
Kwon等[32]在嗜热毁丝霉Myceliophthora thermophila中比对了Cas蛋白/sgRNA表达质粒的转化方式与RNP复合体的转化方式的优缺点,为黑曲霉中DNA转化方式的选择提供了借鉴。Kwon等[32]发现通过质粒转化的方式在体内表达Cas蛋白与sgRNA,需要构建针对不同靶序列的sgRNA的表达质粒,具有操作简单、质粒可长期保存的优点;而使用转化RNP复合体的方式,需要体外转录sgRNA,对操作技术的要求相对较高。另外,关于Cas蛋白和sgRNA的遗传稳定性,由表达质粒携带的Cas蛋白与sgRNA可以在子代细胞中持续表达;而RNP复合体在细胞内为瞬时存在,仅能满足瞬时编辑,但其优点是可降低Cas蛋白的脱靶风险。关于转化效率与基因编辑效率,编辑多基因时,质粒转化效率较高,而不同sgRNA的RNP复合体的共转化效率则相对较低,因而导致其多基因编辑效率较低。
1.5 脱靶效应基于CRISPR/Cas系统的基因组编辑技术,虽然操作简便,基因编辑效率相对较高,但其脱靶风险(Off-effect) 也值得引起关注。以CRISPR/Cas9系统为例,脱靶效应可主要与Cas9/sgRNA复合体与基因组上目标基因的识别特异性相关。比如,当sgRNA与非靶向序列存在错配碱基时,若Cas9仍可对这一非靶向序列进行识别,则会产生脱靶效应。Fu等[33]发现在人类细胞中Cas9可对存在5个错配碱基的非靶向序列进行切割,引起严重的脱靶效应。因此,如何降低脱靶效应是基因组编辑中的重要问题。
目前,降低脱靶效应的策略主要包括筛选使用高识别特异性Cas蛋白突变体、合理设计选择sgRNA、调控Cas蛋白与sgRNA表达量、合理选择Cas蛋白与sgRNA的转化方式等。首先,在Cas蛋白的识别特异性方面,Kleinstiver等[34]筛选获得了识别特异性高的Cas9突变体SpCas9-HF1,显著降低了Cas9与存在错配碱基的非靶向序列的结合,从而降低了脱靶风险。其次,在sgRNA的靶序列设计方面,可借助sgRNA设计软件或脱靶风险预测软件来分析靶向序列在基因组中的特异性,通过选择高特异性与低脱靶风险的sgRNA来提高基因靶向编辑效率。比如,sgRNAcas9软件[21]可预测sgRNA中靶向DNA序列在基因组中的潜在脱靶位点(Potential off-target cleavage sites,POT)。再次,在调控sgRNA与Cas蛋白的表达量方面,降低细胞中sgRNA与Cas蛋白的浓度可能会降低脱靶风险[12]。Shen等[35]通过设计严谨人工开关自我调控Cas9蛋白转录与翻译水平,可显著降低脱靶效率。最后,在Cas蛋白与sgRNA的转化方式方面,如上文所述,当Cas蛋白与sgRNA在体外组装后以RNP复合体的形式转化到胞内时,由于其为瞬时存在,也可以降低脱靶效率[32]。这些策略为降低黑曲霉基因组编辑脱靶效应提供了有效的方案。
2 黑曲霉基因组编辑技术的应用基于CRISPR/Cas系统的基因编辑技术可通过Cas蛋白在基因组的特定位置引入DSB,然后利用体内DNA修复系统,可实现基因失活、基因敲除、基因插入、碱基编辑以及基因表达调控等(表 1)。本节将就该技术在黑曲霉基因组编辑与基因表达调控中的应用进行综述。
2.1 基因失活与基因敲除基因失活与基因敲除是功能基因组研究与代谢途径改造的重要方法。首先,基因失活可通过NHEJ系统介导的易错修复[8]、HDR系统介导基因内部插入一段供体DNA[10]、单链寡核苷酸供体中引入一个终止密码子或移码突变[11]等不同策略来实现(表 1)。在建立基因组编辑技术时,常通过NHEJ系统介导的易错修复来失活具有明显表型变化的基因来测试基因组编辑的效果,如参与孢子色素合成的albA基因(黑色孢子/白色孢子)[8]、brnA基因(黑色孢子/棕色孢子)[36],或者营养缺陷筛选标记pyrG基因[17]。
基因敲除通常是指基因组中目标基因的去除。比如,Kuivanen等[23]利用1 500 bp同源臂介导的同源重组与体外转录sgRNA的方式对转录组预测出的半乳糖二酸(Galactaric acid) 分解途径的基因进行敲除,鉴定出半乳糖二酸下游降解的关键基因(表 1)。另外,基因敲除还可通过“双切割(Two-CUT)”策略来实现,即在靶基因(簇) 的上下游分别设计相应的靶序列来构建相应sgRNA,引导Cas蛋白在靶基因的上下游两个位点同时进行切割,然后利用同源重组来敲除靶基因。Zheng等[10]利用这一“双切割(Two-CUT)”策略成功实现长至48 kb基因簇的敲除。为实现基因的无痕敲除,van Leeuwe等[36]以上下游同源臂融合的无筛选标记供体DNA为修复模板,在NHEJ失活菌株中,成功实现靶基因的无痕敲除;Leynaud-Kieffer等[37]通过在筛选标记pyrG基因的上下游引入一段300 bp的同向重复序列,利用5′-氟乳清酸的反向筛选,实现了靶基因的无痕敲除(表 1)。
2.2 基因插入基因插入是细胞工厂构建中的重要基因编辑方式,可实现关键基因或代谢途径的定点插入、荧光蛋白标签的原位基因融合等。在基因的定点插入方面,Sarkari等[15]利用CRISPR/Cas9系统结合pyrG筛选标记断裂技术,建立可定向在pyrG基因位点插入外源基因表达盒的基因插入策略,将来源于土曲霉Aspergillus terreus的线粒体顺乌头酸转运蛋白插入到pyrG位点后,改变黑曲霉的中心代谢途径,造成顺乌头酸的胞外积累(表 1)。在基因的多位点插入方面,Kuivanen等[31]通过将细菌来源的糖醛酸脱氢酶(Uronate dehydrogenase,UDH) 的表达盒直接插入至半乳糖酸竞争途径的3个关键酶中,成功实现了竞争途径的失活与前体供给的强化(表 1)。另外,在基因的无痕插入方面,Vanegas等[11]测试发现CRISPR/LbCpf1系统可在albA基因的内部无痕插入红色荧光蛋白(表 1)。荧光蛋白的原位融合可实现对靶基因的亚细胞定位跟踪分析或不同条件下的靶基因表达变化的直观表征[38]。
2.3 碱基编辑基因组的定向碱基编辑可通过供体DNA的设计与Cas融合蛋白的设计这两种不同的方式来实现。在通过供体DNA引入碱基编辑方面,首先,Nodvig等[29]发现含有突变的90 nt的单链寡核苷酸供体DNA可在曲霉基因组中引入碱基突变(表 1)。后续,Alazi等[28]黑曲霉中利用带有突变的供体DNA可成功在转录因子GaaR中引入W361R,导致果胶酶的持续表达(表 1)。最近,Kun等[39]进一步发现60 nt的单链寡核苷酸供体DNA也可成功在转录因子XlnR与GaaR中引入点突变(表 1)。在通过Cas融合蛋白来突变基因组中的碱基方面,如前所述,Huang等[14]通过nCas9 (D10A) 与胞嘧啶脱氨酶APOBEC1的融合,可将sgRNA靶序列的胞嘧啶C突变为胸腺嘧啶T,编辑窗口为第4-9位的胞嘧啶C,根据基因的不同,碱基突变的效率可达到47.36%-100% (表 1)。
2.4 基因表达调控除基因序列的定向编辑外,基因表达调控也是细胞工厂构建与优化的重要方式。由于启动子是调控基因表达的重要元件,基因表达调控首先可通过利用同源重组将目标基因的天然启动子替换为目标强度的启动子来实现。其中,诱导启动子的替换是基因表达优化与基因功能定量分析的有力方法。Zhang等[17]将黑曲霉pyrG基因的启动子原位替换为Tet-on诱导启动子,利用不同诱导剂的添加获得pyrG表达水平与柠檬酸发酵的定量关系(表 1)。类似地,Cairns等[40]定量分析了γ-衔接蛋白AplD的表达与黑曲霉菌球形态与菌丝发育的关系。其次,基于CRISPR/Cas系统的转录调控还可利用dCas蛋白与转录激活域或转录抑制域的融合蛋白。目前,Roux等[41]已在构巢曲霉中通过将dLbCas12a (D156R) 与转录激活域VPR (VP64-P65ad-Rta) 融合,建立了可激活靶基因转录的CRISPRa系统。这些基于CRISPR/Cas系统的转录调控策略为黑曲霉细胞工厂中代谢途径的精细调控与动态调控提供了技术支撑。
3 总结与展望自2015年,Nodvig等[8]首次在黑曲霉中建立基于CRISPR/Cas9系统的基因组编辑技术。在短短5年间,黑曲霉基因编辑调控技术不断快速地开发优化,在Cas蛋白、sgRNA、供体DNA与转化方式等方面都涌现出了不同的设计策略,打破了黑曲霉高效遗传操作的屏障,使黑曲霉基因组中的基因失活、基因插入、碱基编辑与启动子替换等基因编辑与表达调控得以实现,进而为黑曲霉基因研究与细胞工厂的构建优化提供了丰富的技术工具。
针对黑曲霉细胞工厂的机制解析与构建优化的需求,无痕基因编辑、多基因编辑、基因表达的精细调控是黑曲霉基因组编辑技术的重要发展方向。在无痕基因编辑方面,随着黑曲霉基因组编辑技术的发展,无痕供体DNA的设计将成为实现基因无痕编辑的重要方法[11, 28-29, 36]。另外,针对黑曲霉基因组编辑过程中组分元件的残留问题,虽然Cas蛋白与sgRNA在体外形成RNP复合体后,再转化至黑曲霉中介导的瞬时编辑可解决这一问题,但其编辑效率相对较低且操作相对繁琐[31]。因此,采用带有AMA1自主复制元件的Cas/sgRNA表达质粒[8],在基因组编辑后,再进行质粒丢失,成为解决组分元件残留的有效策略。在多基因编辑方面,多个sgRNA表达盒的设计与共转化效率是黑曲霉多基因同时编辑效率的重要影响因素[42]。最后,在基因表达的精细调控方面,启动子替换是基因表达调控的有效策略,尤其是利用Tet-on诱导启动子[43]等可通过诱导剂的添加可获得靶基因的最优表达水平。此外,CRISPRa与CRISPRi系统也可成为多基因协同表达调控的重要策略[42]。综上所述,基于CRISPR/ Cas系统的基因组编辑技术的不断发展将大大推动黑曲霉高效运转分子机制的研究,并进一步加速黑曲霉细胞工厂的系统改造优化。
| [1] |
Meyer V, Fiedler M, Nitsche B, et al. The cell factory Aspergillus enters the big data era: opportunities and challenges for optimising product formation//Krull R, Bley T, Eds. Filaments in Bioprocesses. Cham: Springer, 2015, 149: 91-132.
|
| [2] |
Tong ZY, Zheng XM, Tong Y, et al. Systems metabolic engineering for citric acid production by Aspergillus niger in the post-genomic era. Microb Cell Fact, 2019, 18: 28. DOI:10.1186/s12934-019-1064-6
|
| [3] |
Meyer V, Andersen MR, Brakhage AA, et al. Current challenges of research on filamentous fungi in relation to human welfare and a sustainable bio-economy: a white paper. Fungal Biol Biotechnol, 2016, 3: 6. DOI:10.1186/s40694-016-0024-8
|
| [4] |
郭艳梅, 郑平, 孙际宾. 黑曲霉作为细胞工厂: 知识准备与技术基础. 生物工程学报, 2010, 26(10): 1410-1418. Guo YM, Zheng P, Sun JB. Aspergillus niger as a potential cellular factory: prior knowledge and key technology. Chin J Biotech, 2010, 26(10): 1410-1418 (in Chinese). |
| [5] |
郑小梅, 郑平, 孙际宾. 面向工业生物技术的系统生物学. 生物工程学报, 2019, 35(10): 1955-1973. Zheng XM, Zheng P, Sun JB. Systems biology for industrial biotechnology. Chin J Biotech, 2019, 35(10): 1955-1973 (in Chinese). |
| [6] |
郑小梅, 张晓立, 于建东, 等. CRISPR-Cas9介导的基因组编辑技术的研究进展. 生物技术进展, 2015, 5(1): 1-9. Zheng XM, Zhang XL, Yu JD, et al. CR ISPR-Cas9-based genome engineering. Curr Biotech, 2015, 5(1): 1-9 (in Chinese). |
| [7] |
Hille F, Richter H, Wong SP, et al. The biology of CRISPR-cas: backward and forward. Cell, 2018, 172(6): 1239-1259. DOI:10.1016/j.cell.2017.11.032
|
| [8] |
Nodvig CS, Nielsen JB, Kogle ME, et al. A CRISPR-Cas9 system for genetic engineering of filamentous fungi. PLoS ONE, 2015, 10(7): e0133085. DOI:10.1371/journal.pone.0133085
|
| [9] |
Zheng XM, Zheng P, Sun JB, et al. Heterologous and endogenous U6 snRNA promoters enable CRISPR/Cas9 mediated genome editing in Aspergillus niger. Fungal Biol Biotechnol, 2018, 5: 2. DOI:10.1186/s40694-018-0047-4
|
| [10] |
Zheng XM, Zheng P, Zhang K, et al. 5S rRNA promoter for guide RNA expression enabled highly efficient CRISPR/Cas9 genome editing in Aspergillus niger. ACS Synth Biol, 2019, 8(7): 1568-1574. DOI:10.1021/acssynbio.7b00456
|
| [11] |
Vanegas KG, Jarczynska ZD, Strucko T, et al. Cpf1 enables fast and efficient genome editing in Aspergilli. Fungal Biol Biotechnol, 2019, 6: 6. DOI:10.1186/s40694-019-0069-6
|
| [12] |
Hsu PD, Scott DA, Weinstein JA, et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol, 2013, 31(9): 827-832. DOI:10.1038/nbt.2647
|
| [13] |
Zetsche B, Gootenberg JS, Abudayyeh OO, et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-cas system. Cell, 2015, 163(3): 759-771. DOI:10.1016/j.cell.2015.09.038
|
| [14] |
Huang LG, Dong HZ, Zheng JW, et al. Highly efficient single base editing in Aspergillus niger with CRISPR/Cas9 cytidine deaminase fusion. Microbiol Res, 2019, 223-225: 44-50. DOI:10.1016/j.micres.2019.03.007
|
| [15] |
Sarkari P, Marx H, Blumhoff ML, et al. An efficient tool for metabolic pathway construction and gene integration for Aspergillus niger. Bioresour Technol, 2017, 245: 1327-1333. DOI:10.1016/j.biortech.2017.05.004
|
| [16] |
Song LT, Ouedraogo JP, Kolbusz M, et al. Efficient genome editing using tRNA promoter-driven CRISPR/Cas9 gRNA in Aspergillus niger. PLoS ONE, 2018, 13(8): e0202868. DOI:10.1371/journal.pone.0202868
|
| [17] |
Zhang LH, Zheng XM, Cairns TC, et al. Disruption or reduced expression of the orotidine-5'-decarboxylase gene pyrG increases citric acid production: a new discovery during recyclable genome editing in Aspergillus niger. Microb Cell Fact, 2020, 19: 76. DOI:10.1186/s12934-020-01334-z
|
| [18] |
Deltcheva E, Chylinski K, Sharma CM, et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase Ⅲ. Nature, 2011, 471(7340): 602-607. DOI:10.1038/nature09886
|
| [19] |
Park J, Bae S, Kim JS. Cas-Designer: a web-based tool for choice of CRISPR-Cas9 target sites. Bioinformatics, 2015, 31(24): 4014-4016.
|
| [20] |
Bae S, Park J, Kim JS. Cas-OFFinder: a fast and versatile algorithm that searches for potential off-target sites of Cas9 RNA-guided endonucleases. Bioinformatics, 2014, 30(10): 1473-1475. DOI:10.1093/bioinformatics/btu048
|
| [21] |
Xie SS, Shen B, Zhang CB, et al. sgRNAcas9: a software package for designing CRISPR sgRNA and evaluating potential off-target cleavage sites. PLoS ONE, 2014, 9(6): e100448. DOI:10.1371/journal.pone.0100448
|
| [22] |
Kuivanen J, Arvas M, Richard P. Clustered genes encoding 2-keto-l-gulonate reductase and l-idonate 5-dehydrogenase in the novel fungal D-glucuronic acid pathway. Front Microbiol, 2017, 8: 225.
|
| [23] |
Kuivanen J, Wang YMJ, Richard P. Engineering Aspergillus niger for galactaric acid production: elimination of galactaric acid catabolism by using RNA sequencing and CRISPR/Cas9. Microb Cell Fact, 2016, 15: 210. DOI:10.1186/s12934-016-0613-5
|
| [24] |
Pohl C, Kiel JAKW, Driessen AJM, et al. CRISPR/Cas9 based genome editing of Penicillium chrysogenum. ACS Synth Biol, 2016, 5(7): 754-764. DOI:10.1021/acssynbio.6b00082
|
| [25] |
Hsu PD, Lander ES, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering. Cell, 2014, 157(6): 1262-1278. DOI:10.1016/j.cell.2014.05.010
|
| [26] |
Fuller KK, Chen S, Loros JJ, et al. Development of the CRISPR/Cas9 system for targeted gene disruption in Aspergillus fumigatus. Eukaryot Cell, 2015, 14(11): 1073-1080. DOI:10.1128/EC.00107-15
|
| [27] |
Meyer V, Arentshorst M, El-Ghezal A, et al. Highly efficient gene targeting in the Aspergillus niger kusA mutant. J Biotechnol, 2007, 128(4): 770-775. DOI:10.1016/j.jbiotec.2006.12.021
|
| [28] |
Alazi E, Niu J, Otto SB, et al. W361R mutation in GaaR, the regulator of D-galacturonic acid-responsive genes, leads to costitutive production of pectinases in Aspergillus niger. Microbiology Open, 2019, 8(5): e00732. DOI:10.1002/mbo3.732
|
| [29] |
Nødvig CS, Hoof JB, Kogle ME, et al. Efficient oligo nucleotide mediated CRISPR-Cas9 gene editing in Aspergilli. Fungal Genet Biol, 2018, 115: 78-89. DOI:10.1016/j.fgb.2018.01.004
|
| [30] |
Song RJ, Zhai Q, Sun L, et al. CRISPR/Cas9 genome editing technology in filamentous fungi: progress and perspective. Appl Microbiol Biotechnol, 2019, 103(17): 6919-6932. DOI:10.1007/s00253-019-10007-w
|
| [31] |
Kuivanen J, Korja V, Holmström S, et al. Development of microtiter plate scale CRISPR/Cas9 transformation method for Aspergillus niger based on in vitro assembled ribonucleoprotein complexes. Fungal Biol Biotechnol, 2019, 6: 3. DOI:10.1186/s40694-019-0066-9
|
| [32] |
Kwon MJ, Schütze T, Spohner S, et al. Practical guidance for the implementation of the CRISPR genome editing tool in filamentous fungi. Fungal Biol Biotechnol, 2019, 6: 15. DOI:10.1186/s40694-019-0079-4
|
| [33] |
Fu YF, Foden JA, Khayter C, et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat Biotechnol, 2013, 31: 822-826. DOI:10.1038/nbt.2623
|
| [34] |
Kleinstiver BP, Pattanayak V, Prew MS, et al. High-fidelity CRISPR-Cas9 nucleases with no detectable genome-wide off-target effects. Nature, 2016, 529(7587): 490-495. DOI:10.1038/nature16526
|
| [35] |
Shen CC, Hsu MN, Chang CW, et al. Synthetic switch to minimize CRISPR off-target effects by self-restricting Cas9 transcription and translation. Nucleic Acids Res, 2019, 47(3): e13. DOI:10.1093/nar/gky1165
|
| [36] |
van Leeuwe TM, Arentshorst M, Ernst T, et al. Efficient marker free CRISPR/Cas9 genome editing for functional analysis of gene families in filamentous fungi. Fungal Biol Biotechnol, 2019, 6: 13. DOI:10.1186/s40694-019-0076-7
|
| [37] |
Leynaud-Kieffer LMC, Curran SC, Kim I, et al. A new approach to Cas9-based genome editing in Aspergillus niger that is precise, efficient and selectable. PLoS ONE, 2019, 14(1): e0210243. Wang Q, Coleman JJ. Progress and challenges: development and implementation of CRISPR/Cas9 technology in filamentous fungi. Computat Struct Biotechnol J, 2019, 17: 761-769.
|
| [38] |
Kun RS, Meng JL, Salazar-Cerezo S, et al. CRISPR/Cas9 facilitates rapid generation of constitutive forms of transcription factors in Aspergillus niger through specific on-site genomic mutations resulting in increased saccharification of plant biomass. Enzyme Microb Technol, 2020, 136: 109508. DOI:10.1016/j.enzmictec.2020.109508
|
| [39] |
Cairns TC, Feurstein C, Zheng XM, et al. A quantitative image analysis pipeline for the characterization of filamentous fungal morphologies as a tool to uncover targets for morphology engineering: a case study using aplD in Aspergillus niger. Biotechnol Biofuels, 2019, 12: 149. DOI:10.1186/s13068-019-1473-0
|
| [40] |
Roux I, Woodcraft C, Hu JY, et al. CRISPR-mediated activation of biosynthetic gene clusters for bioactive molecule discovery in filamentous fungi. ACS Synth Biol, 2020, 9(7): 1843-1854. DOI:10.1021/acssynbio.0c00197
|
| [41] |
Lian JZ, HamediRad M, Zhao HM. Advancing metabolic engineering of Saccharomyces cerevisiae using the CRISPR/Cas system. Biotechnol J, 2018, 13(9): 1700601. DOI:10.1002/biot.201700601
|
| [42] |
Meyer V, Wanka F, van Gent J, et al. Fungal gene expression on demand: an inducible, tunable, and metabolism-independent expression system for Aspergillus niger. Appl Environ Microbiol, 2011, 77(9): 2975-2983. DOI:10.1128/AEM.02740-10
|
 2021, Vol. 37
2021, Vol. 37