中国科学院微生物研究所、中国微生物学会主办
文章信息
- 郭树奇, 费强
- Guo Shuqi, Fei Qiang
- 甲烷生物利用及嗜甲烷菌的工程改造
- Bioconversion of methane by metabolically engineered methanotrophs
- 生物工程学报, 2021, 37(3): 816-830
- Chinese Journal of Biotechnology, 2021, 37(3): 816-830
- 10.13345/j.cjb.200639
-
文章历史
- Received: October 8, 2020
- Accepted: January 5, 2021
2. 陕西省能源化工过程强化重点实验室,陕西 西安 710049
2. Shaanxi Key Laboratory of Energy Chemical Process Intensification, Xi'an Jiaotong University, Xi'an 710049, Shaanxi, China
随着石化资源过度消耗、环境问题日趋严重以及全球气候变暖,节能减排已成为全球发展共识[1]。研究表明,在导致全球变暖的温室气体中,甲烷(CH4)占比高达20%[2],而以20年期计算,甲烷的全球变暖潜力值是二氧化碳(CO2) 的86倍[3]。目前,在甲烷的总排放量中,人为排放占63% (化石燃料和生物质燃烧、畜牧业、垃圾填埋场),自然来源占37% (湿地、海洋、河流、湖泊和永久冻土)。由于气体不易运输,甲烷利用面临的最主要技术问题是气体-液体(Gas to liquid,GTL) 的转化[4]。虽然化学方法能够实现甲烷的气液转换,但该类技术所需的能耗和反应条件及其转化成本较高,严重制约了甲烷的广泛应用[4-5]。现阶段我国甲烷(沼气、天然气) 仍以燃烧利用为主,方式较单一,利用率有待提高,同时也增加了温室气体排放(每燃烧1 mol甲烷同时释放1 mol二氧化碳)。随着我国沼气产量和天然气(页岩气、可燃冰) 探明储量的不断提高,甲烷的绿色利用途径和技术急需补充和拓展。嗜甲烷菌(Methanotroph) 是天然的甲烷利用菌株,能够以甲烷为唯一的碳源和能源进行生长和代谢。嗜甲烷菌利用其天然甲烷转化途径,可以在温和条件下实现甲烷的生物利用,因而具有较好的发展潜力[6]。此外,随着我国沼气生产技术和页岩气开采技术的不断提升、糖类底物成本上涨以及全球粮食问题日益加重,生物制造过程迫切需要开发廉价的、非食品的新一代碳原料,上述背景为甲烷的生物固定和转化技术的发展创造了新的契机[7]。同时,以生物工程为基础的甲烷生物转化技术的兴起,也为甲烷利用提供了变革性的解决方案[7-8]。近年来,研究人员应用先进的生物技术,已对不同模式嗜甲烷菌的代谢网络进行了理性设计和重构,实现了多种大宗化学品、平台化合物和液体燃料(前体) 的高效合成和积累[7, 9-10],为实现甲烷气体的生物开发利用提供了新的思路[11-12]。本文通过讨论嗜甲烷菌代谢途径的工程化改造应用,深入地介绍了嗜甲烷菌催化氧化甲烷合成生物基化学品的相关研究进展,同时探讨了高效嗜甲烷菌人工细胞的构建策略、存在的挑战和发展方向。
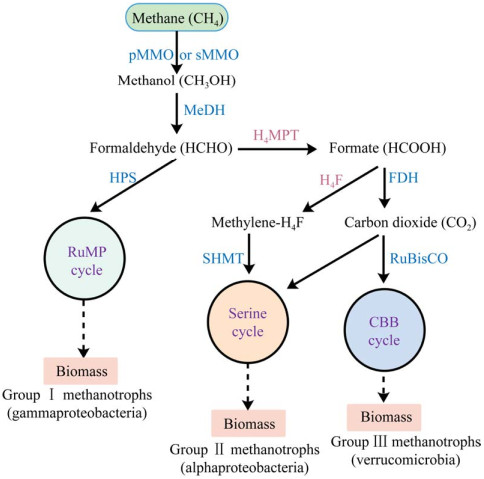
1 嗜甲烷菌嗜甲烷菌属于革兰氏阴性菌,该类细菌大多数是从污水、沼泽等富含甲烷的环境中被分离出来的,在厌氧和好氧条件下均能实现对甲烷的利用[13]。目前,已报道的好氧嗜甲烷菌主要分为3组(图 1):Group Ⅰ、Group Ⅱ和Group Ⅲ。其中,Group Ⅰ主要包括γ-变形菌(Gammaproteobacteria),该类细菌通过单磷酸核酮糖(Ribulose monophosphate,RuMP) 循环对甲烷进行单碳同化;Group Ⅱ主要包括α-变形菌(Alphaproteobacteria),该类细菌通过丝氨酸循环(Serine cycle) 同化甲烷单一碳源;Group Ⅲ主要包括疣微菌门(Verrucomicrobia),该类细菌能够利用氧化甲烷产生的代谢能量和二氧化碳,通过卡尔文循环(Calvin-Benson-Bassham cycle,CBB cycle) 以吸收二氧化碳实现碳源的同化[13-14]。
|
| 图 1 嗜甲烷菌中甲烷的代谢途径(参考文献[14]修改) Fig. 1 Methane metabolism in methanotrophs (modified from [14]). pMMO: particulate methane monooxygenase; sMMO: soluble methane monooxygenase; MeDH: methanol dehydrogenase; HPS: 3-hexulose-6-phosphate synthase; H4MPT: tetrahydromethanopterin; H4F: tetrahydrofolate; FDH: formate dehydrogenase; RuBisCO: ribulose-1, 5- bisphosphate carboxylase; SHMT: serine hydroxymethyltransferase; RuMP: ribulose monophosphate; CBB: calvin- benson-bassham. |
| |
嗜甲烷菌能够利用甲烷,主要是由于该类菌株中含有特定的甲烷单加氧化酶(Methane monooxygenase,MMO),该酶的主要功能是以氧气作为电子受体氧化剂,将甲烷氧化为甲醇,从而形成甲烷代谢途径的第一个中间体。目前,在嗜甲烷菌中已发现的MMO主要包括两种类型,存在于细胞质中的可溶性甲烷单加氧化酶sMMO以及与膜结合或者部分结合的颗粒状甲烷单加氧化酶pMMO[15]。研究发现,虽然sMMO和pMMO催化相同的甲烷氧化反应过程,但是酶结构及催化机制并不相同[13, 16]。例如,大多数嗜甲烷菌都能表达pMMO,只有少数菌株可以同时产生sMMO和pMMO,而且sMMO也只有在低铜含量(< 0.005 mol/L) 条件下才能表达;相反铜离子是pMMO的转录激活因子,pMMO表达水平随铜离子浓度的增加而增加[17]。
sMMO是一种可溶性复合蛋白,由mmoXYBZDC基因编码,包含有3个不同蛋白,分别为羟化酶(sMMO hydroxylase,MMOH)、调节蛋白(sMMO regulatory protein,MMOB) 和还原酶(sMMO reductase,MMOR)[18]。作为一种NAD(P)H依赖的双铁酶,sMMO需要通过这3个不同的蛋白相互作用来协调电子、氧气、质子和蛋白质传递到催化部位以完成对不同底物特异性的氧化,因而sMMO具有复杂的生化机制[19]。除了氧化甲烷外,sMMO还能氧化多种不同种类的有机底物,包括烯烃和苯类化合物等[20]。
尽管pMMO属于膜结合蛋白[14],但相对于sMMO,pMMO酶系统结构较为简单。其包含PmoB、PmoA和PmoC三个亚基,分别由pmoCAB基因编码。与sMMO相比,pMMO作为一种铜离子依赖酶,对甲烷具有更强的亲和性[20-21],pMMO也是自然界中发现的最有效的甲烷催化剂。除了甲烷,pMMO酶对一系列其他底物也表现出催化活性,例如,其可以将C1-C5链碳氢化合物转化为相应的醇或将少数线性烯烃(乙烯、丙烯、1-丁烯、2-丁烯) 环氧化[22]。虽然pMMO底物范围相较于sMMO较窄,但是除了对底物的独特化学专一性外,pMMO在烷烃羟基化反应中表现出显著的区域专一性和立体选择性[20]。目前,pMMO已经作为催化剂被开发利用,其在外界环境条件下能够有效且有选择性的催化甲烷氧化。到目前为止,基于甲烷单加氧酶开发的平台技术,不仅能够将甲烷液化为甲醇,而且可以将天然气转化为醇和酮[23-24]。通过甲烷单加氧化酶的催化机制和调控网络的研究,也能够促进高效微生物甲烷催化剂的设计和开发,从而满足工业规模的利用天然气生产甲醇的需求。
在嗜甲烷菌中,MMO将甲烷转化为甲醇后,由周质中存在的以吡咯喹啉醌为辅因子的甲醇脱氢酶(MeDH) 进一步氧化为甲醛[14],其中钙离子依赖的甲醇脱氢酶(MxaF-MDH) 发现于Group Ⅰ和Group Ⅱ菌,而镧离子依赖的甲醇脱氢酶(XoxF-MDH) 则发现于Group Ⅲ菌。由于甲醛是一种有毒的中间体,细胞内必须保持在无毒水平,因此甲醛在嗜甲烷菌体内会被进一步转化。在Group Ⅰ菌中,甲醛直接进入RuMP循环被利用。在Group Ⅱ菌中,甲醛通过四氢甲烷蝶呤(Tetrahydromethanopterin,H4MPT) 转化为甲酸后,在以四氢叶酸(Tetrahydrofolate,H4F) 为辅因子的甲酸四氢叶酸连接酶(Formate-tetrahydrofolate ligase) 等系列酶的催化作用下转化为亚甲基四氢叶酸(Methylene-H4F),进而在丝氨酸羟甲基转移酶(Serine hydroxymethyltransferas,SHMT) 等催化酶作用下进入丝氨酸循环。此外,甲醛也可以由甲酸脱氢酶(Formate dehydrogenase,FDH) 进一步氧化为二氧化碳进入CBB循环[14, 25-26]。
1.2 甲烷碳同化途径在甲烷氧化途径中,嗜甲烷菌通过其体内的氧化过程逐步将甲烷转化为甲醛或者甲酸,之后根据菌种差异,分别以3种不同代谢途径实现甲烷同化。目前,嗜甲烷菌中已发现的甲烷同化途径主要包括RuMP循环、丝氨酸循环[13]以及CBB循环[27]。
1.2.1 RuMP循环Group Ⅰ嗜甲烷菌主要通过RuMP循环直接对甲醛进行同化(图 1)[14]。RuMP循环主要包括3个步骤:碳固定、裂解和重排。在碳固定途径中,甲醛在己酮糖-6-磷酸合酶(3-hexulose-6-phosphate synthase,HPS) 的催化作用下与5-磷酸核酮糖(Ribulose-5-phosate,Ru5P) 缩合产生6-磷酸己酮糖(Hexulose-6-phosphate,H6P)。随后H6P被磷酸己糖异构酶(Phosphohexose isomerase,PHI) 转化为6-磷酸果糖(Fructose-6-phosphate,F6P)。裂解阶段,F6P通过糖酵解途径(Embden- Meyerhof-Parnas,EMP) 转化为1, 6-二磷酸果糖(Fructose-1, 6-bisphosphate,F-1, 6-BP),随后在果糖二磷酸醛缩酶(Fructose-bisphosphate aldolase,FBA) 的催化作用下裂解为3-磷酸甘油醛(Glyceraldehyde-3-phosphate,GAP) 和磷酸二羟丙酮(Dihydroxyacetone phosphate,DHAP);F6P也能够通过ED (Entner-Doudoroff) 途径转化为2-酮-3-脱氧-6-磷酸葡糖酸(2-keto-3-deoxy-6- phosphogluconate,KDPG),接着在KDPG醛缩酶的作用下裂解为GAP和丙酮酸(Pyruvate)。在碳源重排过程中,RuMP循环通过转酮醇酶(Transketolase,TKT) 催化F6P和GAP生成木酮糖-5-磷酸(Xylulose-5-phosphate,Xu5P) 和赤藓糖-4-磷酸(Erythrose-4-phosphate,E4P),E4P在转醛醇酶(Transaldolase,TAL) 催化下和另一分子F6P反应生成景天庚酮-7-磷酸(Sedoheptulose- 7-phosphate,S7P)。随后,S7P和GAP通过转酮醇酶的催化作用生成Xu5P和核糖-5-磷酸(Ribose-5-phosphate,R5P),Xu5P和R5P分别在相应催化酶的作用下再次转化生成Ru5P[14, 25, 28]。
1.2.2 丝氨酸循环Group Ⅱ嗜甲烷菌主要通过丝氨酸循环对甲烷碳源进行同化(图 1)[14]。首先,甲酸通过耗能过程在甲酸四氢叶酸连接酶(Formate-tetrahydrofolate ligase,FTFL)、NADPH依赖的亚甲基四氢叶酸脱氢酶(Methylenetetrahydrofolate dehydrogenase,MTDA) 等一系列催化酶的作用下转化为亚甲基四氢叶酸。随后丝氨酸循环的第一步是在丝氨酸羟甲基转移酶(Serine-hydroxymethyltransferase,SHMT) 的催化作用下,由亚甲基四氢叶酸及甘氨酸合成丝氨酸,接着丝氨酸通过丝氨酸-乙醛酸盐氨基转移酶(Serine-glyoxylate aminotransferase,SGAA) 的转氨作用转化为羟基丙酮酸,随后在NADH依赖的羟丙酮酸还原酶(Hydroxypyruvate reductas,HPRA) 及甘油酸激酶(Glycerate 2-kinase,GCKA) 的催化作用下转化为2分子2-磷酸甘油酸,该过程需要消耗ATP。之后,1分子2-磷酸甘油酸被同化进入下游途径,另1分子2-磷酸甘油酸在烯醇酶(Enolase,ENO) 的催化作用下转化为磷酸烯醇丙酮酸(Phosphoenolpyruvate,PEP)。PEP通过进一步的羧化作用产生草酰乙酸(Oxaloacetate),继而转化为苹果酸(Malate) 和苹果酰辅酶A (Malyl-CoA)。Malyl-CoA在苹果酰辅酶A裂解酶(Malyl-CoA lyase,MCL) 的催化作用下生成乙醛酸盐(Glyoxylate) 和乙酰辅酶A (Acetyl-CoA)。丝氨酸循环的最后一步是甘氨酸作为主要受体与丝氨酸-乙醛酸氨基转移过程中产生的乙醛酸完成缩合[13, 25, 28]。在丝氨酸循环中,嗜甲烷菌细胞以1︰1的比例利用二氧化碳和甲醛,并用于产生乙酰辅酶A和生物质合成。
1.2.3 卡尔文循环Group Ⅲ菌中,甲酸通过NAD依赖的甲酸脱氢酶转化为二氧化碳后通过卡尔文循环以二氧化碳形式实现甲烷单一碳源的同化(图 1)[14]。研究发现在富马酸甲酯菌Methylacidiphilum fumariolicum SolV细胞内含有CBB循环特有的催化酶核酮糖-1, 5-二磷酸羧化酶/加氧酶(Ribulose-1, 5-bisphosphate carboxylase/oxygenase,RuBisCO),该酶能够催化二氧化碳进入CBB循环,通过二氧化碳与1, 5-二磷酸核酮糖(Ribulose-1, 5-bisphosphate,RuBP) 的整合完成甲烷单一碳源的同化[27, 29-30]。值得注意的是,菌株M.fumariolicum SolV以二氧化碳为唯一碳源生长,以甲烷作为能源。在甲烷存在的情况下,当二氧化碳浓度低于1%时,M.fumariolicum SolV细胞生长受限;而当二氧化碳浓度低于0.3%时,未观察到该菌株生长,CBB循环中甲烷碳源通过转化为二氧化碳后被同化[27]。
对比不同类型的甲烷同化途径,其中Ⅰ型嗜甲烷菌细胞内所有碳源均以甲醛氧化的形式被吸收。Ⅰ型菌通过直接耦合机制,将电子从甲醇氧化过程直接转移到pMMO;而Ⅱ型嗜甲烷菌中pMMO通过氧化还原臂机制从泛醌池中提取电子,这也导致菌体的生物量低于Ⅰ型菌。同时,由于RuMP循环中的所有反应均为放能过程,所以Ⅰ型嗜甲烷菌比Ⅱ型嗜甲烷菌更具有能量效率优势。因此,目前大多数代谢工程的应用研究都只集中于Ⅰ型嗜甲烷菌。而Ⅱ型嗜甲烷菌在乙酰辅酶A池和甲烷-二氧化碳共利用方面具有明显的生化优势,多用于乙酰辅酶A衍生化合物的合成[31-32]。如典型的Ⅱ型嗜甲烷菌Methylosinus trichosporium OB3b使用丝氨酸循环作为甲烷同化的主要途径,其培养基中乙酰辅酶A浓度在指数中期可达到519.3 μmol/L[33]。目前关于M.fumariolicum SolV菌株遗传操作的报道相对较少,该菌株多被用于将甲烷转化为甲醇的研究中[27, 34]。
2 嗜甲烷菌代谢工程遗传改造工具代谢工程遗传改造工具在嗜甲烷菌工业菌株及生物催化剂研究中发挥着关键作用[35]。尽管与大肠杆菌Escherichia coli等已深入研究的宿主相比,嗜甲烷菌中发现的高效遗传操作工具还相对有限,但是基于好氧嗜甲烷菌仍有大量可用的遗传改造工具不断被开发和使用。目前,研究人员在好氧嗜甲烷菌体系中已经开发了多种用途广泛的质粒载体、基因转移技术以及无痕基因敲除和整合技术。例如:用于结合转移及电转化技术的广宿主质粒pBHR和pAWP89、用于基因转移的辅助质粒pRK2013、用于基因复制的质粒pBBR和IncQ以及用于基因无痕敲除的蔗糖致死基因sacB筛选体系[36]。这些基因操作工具和研究进展在前期的综述中进行了详细的概括[12, 14, 25, 37]。
近期,基于嗜甲烷菌体系中优化的基因操作工具也不断被开发。例如,Liu等在嗜甲烷菌Methylotuvimicrobium buryatense 5GB1C中利用pheS基因开发了一种反向筛选标记,该研究不仅在嗜甲烷菌实现了基因的无痕敲除,而且大幅度提升了基因敲除操作效率(阳性率高达92%,操作周期缩短至8 d)[38]。Nguyen等通过研究不同细胞生长阶段、电穿孔溶液、感受态细胞、DNA浓度、电穿孔条件和外源DNA类型等多种因素对Methylomonas sp. DH-1电穿孔效率的影响,显著提高了嗜甲烷菌遗传改造工具的效率。该研究发现在2.5 kV、25 μF、400 Ω条件下,指数期中期细胞的转化效率高于平台期细胞,无菌水和10%甘油相较于9.3%蔗糖、10% PEG 1 000、10% PEG 6 000转化效率更高;当转化体系为200 μL,DNA浓度为500 ng且DNA体积不超过2 μL时,Methylomonas sp. DH-1电穿孔率最佳[39]。
基于基因表达元件优化,Garg等通过筛选不同启动子(诱导性和组成型)、核糖体结合位点(RBS1、RBS2、RBS3和RBS4) 及质粒载体(pCAH01和pAWP87),在M.buryatense 5GB1中构建了3种不同组合的表达载体pTETR、pCMR和pAMR。通过不同元件的组合优化,该研究使M.buryatense 5GB1菌株中从甲烷合成乳酸的产量提高了14倍[8]。在通过系统代谢工程将甲烷转化为产物2, 3-丁二醇(2, 3-butanediol) 过程中,Nguyen等基于嗜甲烷菌Methylomicrobium alcaliphilum 20Z菌株首次利用计算机模拟指导的菌株基因尺度模型i20ZR-BDO对2, 3-丁二醇的合成途径进行了优化,使其产量提高了20%[40]。另外,高通量组学分析技术的应用也有效促进了好氧嗜甲烷菌的研究。Hu等利用转录组技术分析了CH4/O2的供气比对好氧嗜甲烷细胞生长和代谢途径的影响,结果发现,在分批培养过程中,当CH4/O2供应摩尔比为0.93时,M.buryatense 5GB1菌株生长速率最高;保持碳氧供应平衡是促进菌株生长的关键。同时,该研究分析显示最佳的CH4/O2比例可以增加菌株碳、氮、磷代谢途径相关基因表达,从而促进菌株的生长[41]。这些基因操作方法(插入和敲除基因、电转化)[38-39]、基因表达系统(启动子开发和其他调节元件)[8]以及诱变技术的开发,以及高通量测序技术的发展和应用,进一步推动了理性改造嗜甲烷菌工业菌株的研究,促进了代谢工程改造甲烷碳代谢通量高效率流向目标产品(化学品、生物液体燃料等)[35]。
3 嗜甲烷菌的代谢工程改造目前,通过生物法将甲烷转化为高附加值产品的研究,有望成为节能减排最有前景的策略之一。嗜甲烷菌利用天然途径可以将甲烷转化为多种具有较高市场价值的化学品,主要包括可溶性的代谢产物如四氢嘧啶(Ectoine)、单细胞蛋白(Single-cell protein,SCP)、生物塑料和甲醇等[35]。其中,四氢嘧啶作为生物分子和细胞的保护剂,具有广泛的科学和商业价值。嗜甲烷菌为了应对高盐度的生长培养基,能够在细胞质内积累大量的渗透压保护物质四氢嘧啶[42]。单细胞蛋白由多种氨基酸组成,主要来源于嗜甲烷菌的生物质。由于甲烷可由储量充足且价格低廉的天然气及沼气供应,研究发现从天然气中生产单细胞蛋白在工业规模上是可行的[43]。除了生产单细胞蛋白,Liang等通过经济技术分析(Techno-economic analysis) 发现,利用高碳通量的嗜甲烷菌将甲烷转化为乳酸的高效生产工艺,也具有商业化的应用潜力。该研究分析表明,甲烷的转换效率、乳酸发酵浓度、产率以及气体流量对乳酸生产成本的影响最为显著,根据这些影响因素以甲烷生产乳酸的最低销售价格在5.83美元/kg–2.17美元/kg之间,其中2.17美元/kg的最低价格优于目前乳酸的市场价格(3.00美元/kg)[44]。聚羟基丁酸是(Poly-3-hydroxybutyrate,PHB) 是一种可降解塑料,Group Ⅰ和Group Ⅱ嗜甲烷菌均能产生PHB,其中Group Ⅱ中的丝氨酸循环是PHB生物合成最有效的代谢途径[45]。另外,甲醇作为嗜甲烷菌中甲烷代谢的初始产物,仍需要更高的产率以满足经济生产过程的需要[35]。
除了嗜甲烷菌中的甲烷天然途径合成的生物基产品,随着嗜甲烷菌系统生物学及生物技术的发展,通过理性代谢工程改造,好氧嗜甲烷菌以甲烷为唯一碳源也已实现了多种大宗化学品和生物燃料的合成(表 1)。
| Pathway | Product | Performance | Methanotrophs | References |
| Pyruvate-derived products | Lactic acid | 370 mg/L | Methylococcus capsulatus | [46] |
| Lactic acid | 800 mg/L | M. buryatense | [47] | |
| Lactic acid | 600 mg/L | M.buryatense | [8] | |
| Isobutanola | 220 mg/L | M.capsulatus Bath | [48] | |
| Isobutanolb | 1 mg/L | M.capsulatusBath | [48] | |
| 2, 3-butanediol | 86.2 mg/L | M.alcaliphilum 20Z | [40] | |
| Acetyl-CoA-derived products | Fatty acids | 111 mg/g DCW | M.buryatense 5GB1 | [49] |
| Crotonic acid | 70 mg/L | M.buryatense 5GB1S | [50] | |
| 3-HPc | 60.59 mg/L | M.trichosporium OB3b | [33] | |
| TCA cycle-derived products | Succinic acid | 105 mg/L | Methylococcus capsulatus | [51] |
| Succinic acid | 195 mg/L | Methylomonas sp. DH-1 | [39] | |
| Putrescine | 98.08 mg/L | M.alcaliphilum 20Z | [52] | |
| 1, 4-butanediol | - | M.capsulatus | [53] | |
| Mep-derived products | Isoprene | 10 mg/L | M.capsulatus Bath | [54] |
| Isoprene | 0.056 mg/L | Methylomonas sp. | [55] | |
| Limonene | 0.5 mg/L | Methylomonas sp. 16a | [56] | |
| Farnesene | - | Methylomonas sp. 16a | [12] | |
| Castaxanthin | 2.4 mg/g DCW | Methylomonas sp. 16a | [57] | |
| Castaxanthin | 2.0 mg/g DCW | Methylomonas sp. 16a | [58] | |
| Shikimic acid-derived products | Muconic acid | 12.4 mg/L | M.buryatense | [59] |
| Glycerophosphoric acid derived products | Lipid | 45.4 mg/L/h | M.buryatense | [60] |
| a: Two genes pathway; b: Five genes pathway; c: 3HP: 3-hydroxypropionic acid. | ||||
根据前体的来源不同,这些转化甲烷合成的目标产物主要分为6大类:丙酮酸衍生产物[12],包括乳酸(Lactic acid)、异丁醇(Isobutanol)[61]以及2, 3-丁二醇(2, 3-butanediol)[40]等;乙酰辅酶A衍生产物,包括脂肪酸(Fatty acids)、丁烯酸(Crotonic acid)[62]以及3-羟基丙酸(3HP)[63]等;TCA循环衍生产物,主要包括琥珀酸(Succinic acid)[64]、1, 4-丁二醇(1, 4-butanediol)[65]以及四氢嘧啶(Ectoine)[66-67]等;甲基赤藓糖醇-4-磷酸途径衍生产物,主要包括异戊二烯(Isoprene)、柠檬烯(Limonene)[68]、法尼烯(Farnesene)[69]、角黄素(Canthaxanthin)、虾青素(Astaxanthin)[57]以及番茄红素(Lycopene)[70]等;莽草酸途径衍生产物[71],例如黏糠酸(Muconic acid)[72];以及甘油磷酸途径衍生产物,主要包括脂质(Lipids)、磷脂酰乙醇胺(Phosphatidyl ethanolamine,PE)、磷脂酰甘油(Phosphatidyl glycerol,PG) 等[13, 73]。
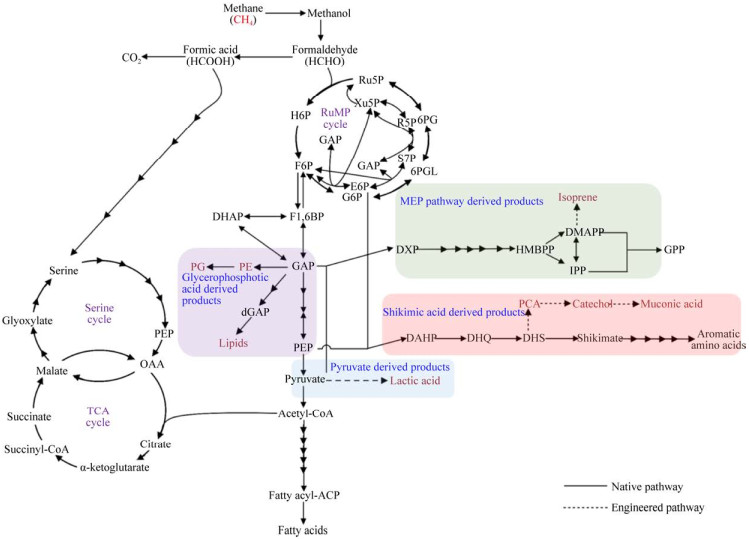
本文以嗜甲烷菌利用甲烷合成大宗化学品乳酸、黏糠酸、生物燃料异戊二烯以及脂质的研究为例(表 1,图 2)。
|
| 图 2 已报道的嗜甲烷菌以甲烷为碳源合成化学品和燃料的代谢途径(参考文献[12]修改) Fig. 2 The reported metabolic pathway for production of chemicals and fuels by methanotrophs from methane (modified from [12]). Ru5P: Ribulose-5-phosphate; H6P: Hexulose-6-phosphate; F6P: Fructose-6-phosphate; F1, 6BP: Fructose bisphosphate; G6P: Glucose-6-phosphate; 6PGL: 6-phospho-gluconolactone; 6PG: 6-phosphogluconate; DHAP: Dihydroxyacetone phosphate; GAP: Glyceraldehyde-3-phosphate; Xu5P: Xylulose-5-phosphate; E4P: Erythrose- 4-phosphate; S7P: Sedoheptulose-7-phosphate; DAHP: Dihydroxyacetone phosphate; DHQ: 3-dehydroquianate; DHS: 3-dehydroshikimate; PCA: Protocatechuic acid; DXP: 1-deoxy-d-xylulose 5-phosphate; HMBPP: 4-hydroxy-3-methyl-2- butenyl pyrophosphate; DMAPP: Dimethylallyl pyrophosphate; IPP: Isopentenyl pyrophosphate; GPP: Geranyl pyrophosphate; OAA: Oxaloacetic acid; PEP: Phosphoenolpyruvic acid; dGAP: Diacylglycerol 3-phosphate; FPP: Farnesyl pyrophosphate; GGPP: Geranylgeranyl pyrophosphate; PE: Phosphatidyl ethanolamine; PG: Phosphatidyl glycerol. |
| |
乳酸作为最重要的有机酸之一,具有羧基和羟基两个不同的官能团,是一种多功能合成中间体,在工业和生物技术领域有广泛的应用,如用于药物传递系统,可生物降解塑料、食品和健康领域等[74]。因此,通过嗜甲烷菌利用甲烷生产乳酸也成为代谢工程及工业应用的研究热点[12]。在嗜甲烷菌中,利用乳酸脱氢酶(Lactate dehydrogenase) 能够将丙酮酸转化为乳酸。Henard等分别将来源于E. coli、长双歧杆菌Bifidobacterium longum以及瑞士乳杆菌Lactobacillus helveticus菌株中的乳酸脱氢酶经密码子优化后在嗜甲烷菌M.buryatense中进行异源表达,成功将丙酮酸转化为乳酸。该研究同时发现来源于L. helveticus中的乳酸脱氢酶催化丙酮酸产生乳酸效率最高。在5 L发酵罐中,以甲烷为碳源,改造后M.buryatense菌株中乳酸得率为0.05 g乳酸/g甲烷,乳酸浓度为0.8 g/L,连续通气发酵产率为0.008 g/(L·h)[47]。之后,Garg等[8]研究发现使用Hungate试管对M. buryatense菌株进行中高通量培养时,其生长速率较传统血清瓶培养提高了2倍。该研究组通过代谢工程策略对乳酸合成模块的启动子及核糖体结合位点进行筛选,使乳酸的产量较前期报道提高了14倍。最后,通过利用廉价的铵代替硝酸盐作为氮源,在工业培养基中L-乳酸的合成通量比之前报道的生物反应器系统高出约50倍,发酵产量达到0.6 g/L[8]。在沼气的利用过程中,Henard等通过敲除丙酮酸脱氢酶使M.alcaliphilum 20Z菌株突变株中乳酸的产量达到0.027 g/(g DCW·h)[75] (图 2)。由此可见,丙酮酸作为嗜甲烷菌RuMP途径中重要的中间化合物,具有较高的碳通量。因此,在嗜甲烷菌中合成丙酮酸相关衍生物具有显著优势。
3.2 黏糠酸黏糠酸是一种重要的平台化合物以及药物中间体,可转化为多种高价值的化学品,如己二酸、对苯二甲酸、尼龙等[72]。基于微生物莽草酸途径,利用代谢工程改造策略已经在多种异养微生物中实现了黏糠酸的高效合成[72, 76-77]。Henard等通过在M.buryatense 5GB1中引入来源于苏云金芽孢杆菌Bacillus thuringiensis中的二羟基莽草酸脱水酶AsbF、来源于阴沟肠杆菌Enterobacter cloacae中的原儿茶酸脱羧酶AroY以及来源于不动杆菌属Acinetobacter sp.中的儿茶酚双加氧酶CatAP76A,首次成功将嗜甲烷菌莽草酸途径中间产物二羟基莽草酸转化为黏糠酸,最终通过途径改造,以甲烷为唯一碳源,黏糠酸的最高得率为2.8 mg/g[59]。同时,He等发现M.alcaliphilum 20Z菌株中黏糠酸的产量相较于理论最大值明显较低(比理论值低166.7倍)[59],该菌株中可能包含有莽草酸合成途径中的同工酶或替代途径需要进一步挖掘。从整体来看,目前关于嗜甲烷菌中莽草酸途径代谢产物的研究相对较少,该合成途径的代谢机制、合成潜力以及转化甲烷合成高值化学品对甲烷碳代谢通量的调控作用均需要深入研究以获得更加准确的嗜甲烷菌代谢模型,并进一步指导菌株改造。
3.3 异戊二烯异戊二烯是一种广泛研究的挥发性五碳烃类,是生产聚异戊二烯、弹性材料、合成橡胶、粘合剂及药品的重要平台化学品[78]。基于MEP (Methylerythritol 4-phosphate-derived products) 途径,Leonard等将来源于山葛Pueraria montana中的基因ispS通过密码子优化后,利用MDH启动子在嗜甲烷菌M.capsulatus Bath进行了重组表达,结果发现重组菌株中能够有效积累异戊二烯(Isoprene)[54]。之后,Donaldson等利用氯霉素抗性的pBHR1载体,将密码子优化的来源于白杨Populus alba中的基因ispS以及来源于酿酒酵母中的idi基因在菌株Methylomonas进行表达,重组菌株以甲烷为碳源,Isoprene的产量为0.056 mg/L[55]。另外,基于Ⅰ型嗜甲烷菌M.alcaliphilum 20Z中的DXP (1-deoxy-d-xylulose 5-phosphate) 途径,Song等通过引入来源于甘薯Ipomoea batatas中的异戊二烯合成酶(Isoprene synthase,ISPS),也成功构建了以甲烷为唯一碳源生产异戊二烯的合成途径[79]。目前,在嗜甲烷菌体内发现有MEP途径或MVA (mevalonate) 途径,这两种途径均能够用于生物燃料和药物的合成。其中,已报道的嗜甲烷菌中以MEP途径为主,且MEP途径合成IPP的理论最大值高于MVA途径[6, 12]。因此,从分析结果来看,嗜甲烷菌中的MEP更适合于生产异戊二烯相关产物。
3.4 脂质脂质来源于细胞内膜,是生产绿色柴油的关键前体。甘油糖脂类物质磷脂酰乙醇胺和磷脂酰甘油均为嗜甲烷菌生长过程中产生的细胞膜成分,这些细胞膜成分可以通过加氢除氧的催化过程转化为生物柴油。目前,通过实验室生物炼制工艺已经成功将M.buryatense菌株生产的细胞膜脂转化为绿色柴油[13, 73]。Fei等在研究脂质合成过程中发现,菌株M.buryatense 5GB1和AP18 (5GB1CΔglgA1 ∆glgA2Δsps) 中细胞膜脂质主要由肉豆蔻酸(Myristic acid,C14:0)、棕榈酸(Palmitic acid,C16:0) 和棕榈烯酸(Palmitoleic acid,C16:1) 等脂肪酸组成,通过高密度发酵以及连续通气,M. buryatense 5GB1的最高细胞干重达到21.4 g/L,脂质的最高合成速率为45.4 mg/(L·h),较前期文献报道分别提高了2倍和3倍[60]。
从以上研究可以看出,基于嗜甲烷菌中的甲烷代谢途径,通过代谢工程改造更有利于产生以丙酮酸、乙酰辅酶A以及TCA循环代谢物为前体合成的化合物。下一步关于嗜甲烷菌的理性代谢工程改造策略应该集中于对嗜甲烷菌中单加氧酶进行有针对性的改进,提高代谢途径中甲烷同化和能量转换效率。此外,由于大多数生物燃料和高价值化学中间体来自丙酮酸或乙酰辅酶A代谢节点,嗜甲烷菌常以糖原的形式积累大量生物量,这会显著降低目标产物的潜在碳通量,因此通过敲除碳源竞争途径以及绕过脱羧过程,能最大限度地将甲烷碳源转化为中间代谢物,有效促进甲烷生物基产品的合成[7, 25, 80-82]。
4 展望开发和改善工业微生物的鲁棒性是发展生物产业的基础[83]。嗜甲烷菌具有抗污染能力强、培养条件耐受范围广、生长迅速的特点(倍增时间为3 h)[50, 82, 84],是甲烷生物转化极具应用前景的宿主。现阶段,通过代谢工程改造的嗜甲烷菌,虽然有潜力成为一种多功能且高效的甲烷转化平台,但这些生物技术在生产力方面仍无法与传统发酵和物理-化学方法竞争[85]。为此,基于嗜甲烷菌生物转化平台,提高生产竞争力应该注重培养过程优化、菌种改良以及先进嗜甲烷菌生物技术的开发和应用[86]。
首先,在嗜甲烷菌培养过程中,除了培养基组分复杂之外,培养条件对嗜甲烷菌的细胞生长、碳代谢流、还原力以及能量再生等过程影响显著[35]。Hu等研究发现[41],当CH4/O2供应摩尔比为0.93时,嗜甲烷菌M.buryatense 5GB1的比生长速率提高了24%,达到0.287/h。而在此条件下,M.buryatense 5GB1胞内的甲烷代谢、磷酸盐吸收以及固氮相关基因的表达量显著上调,该研究为进一步优化培养条件,提高甲烷转化效率提供了研究基础。此外,培养组分价格昂贵也是制约嗜甲烷菌规模化应用的因素之一。以氮源为例,嗜甲烷菌能够以硝酸盐、氨等作为氮源。然而,氨是甲烷的竞争性抑制剂,会抑制MMO的活性,导致嗜甲烷菌在以氨为氮源条件下生长速率明显降低(倍增时间为12 h),因此,在常规培养条件下嗜甲烷菌以硝酸盐为氮源。目前,硝酸钠的售价为200–400美元/t,而氯化铵的价格仅为80–150美元/t[8, 82]。已有研究表明,嗜甲烷菌可以氯化铵替换硝酸盐为氮源,高效转化甲烷合成生物乳酸[8],这为有效降低工业菌株的氮源成本并提高甲烷转化的经济可行性提供了实践基础。值得注意的是,目前已有相关报道验证嗜甲烷菌可以转化氮气作为辅助氮源进行代谢和生长[41, 87],如能在此基础上对嗜甲烷菌生物固氮功能进行深度挖掘,有望进一步降低其生产应用的成本。当前,关于嗜甲烷菌的固氮作用及其固氮机理的研究还鲜有报道,下一步需要推动嗜甲烷菌的固氮作用机制解析以及工程改造利用的相关研究。
其次,尽管研究人员在嗜甲烷菌的代谢工程改造研究中已取得了一些进步,但是基于嗜甲烷菌广泛可用的工业生物技术仍十分有限,仍缺乏对嗜甲烷菌生理生化特性、代谢网络及调控机制的深入认识,例如甲烷单加氧酶的电子受体及活性调控机制仍不清楚等[8]。研究人员曾尝试将来源于嗜甲烷菌的sMMO和pMMO在大肠杆菌或红球菌Rhodococcus erythropolis等不同宿主中表达,但均未检测到酶活性[88-89]。这主要是由于嗜甲烷菌MMO的结构特异,使其难以在异源宿主中有效折叠。加之甲烷氧化过程复杂且受到严格控制,需要保持细胞内甲烷氧化和同化的平衡,Cu、Fe等金属离子依赖、电子供体及催化机制不清楚等因素均导致了MMO在非原生宿主中的功能性表达至今尚未成功,阻碍了非天然嗜甲烷菌人工细胞的构建[90-91]。下一阶段,通过功能改造获得在异源宿主体内具有活性的MMO,将为人工合成甲烷营养菌提供新的机遇。
最后,嗜甲烷菌的低代谢活性和低碳源转化效率是制约甲烷生物催化的最主要因素。现有研究表明,单一的菌株代谢工程改造并不是实现甲烷生物转化产业化的捷径。甲烷和氧气的传质限制也是导致目标产物发酵浓度及产率较低的关键因素[12]。因此,在嗜甲烷菌研究中,不仅要注重对菌株培养、生理代谢以及生物技术的探索和创新,而且需要在嗜甲烷菌发酵工艺设计中有所突破,开发出适合嗜甲烷工程菌工业应用的甲烷气体发酵系统,以克服传质限制。针对嗜甲烷菌研究中面临的问题,通过优化培养体系探索嗜甲烷菌合成目标产物的最佳培养条件、利用多组学分析挖掘嗜甲烷菌中高效的基因表达元件和明确代谢网络[41]以及采用经济技术分析评估和优化甲烷合成目标产物的生产工艺等[44]研究尤为必要。未来,开发高效的分子生物学技术、解析嗜甲烷菌的生理代谢机制、应用理性的代谢工程改造策略以及通过生物反应器设计,强化甲烷及氧气从气相到培养基液相的传质,优化传质、动力学、细胞生长和目标产品的生产之间的联系,从而建立先进的气-液发酵工艺等研究对提高甲烷碳源转化生产率,推动甲烷生物利用产业化至关重要。
| [1] |
史硕博, 孟琼宇, 乔玮博, 等. 塑造低碳经济的第三代固碳生物炼制. 合成生物学, 2020, 1(1): 44-59. Shi SB, Meng QY, Qiao WB, et al. Establishing carbon dioxide-based third-generation biorefinery for a sustainable low-carbon economy. Syn Bio J, 2020, 1(1): 44-59 (in Chinese). |
| [2] |
Yvon-Durocher G, Allen AP, Bastviken D, et al. Methane fluxes show consistent temperature dependence across microbial to ecosystem scales. Nature, 2014, 507(7493): 488-491. DOI:10.1038/nature13164
|
| [3] |
E PA. Understanding global warming potentials. Environmental Protection Agency, 2017.
|
| [4] |
Conrado RJ, Gonzalez R. Envisioning the bioconversionof methane to liquid fuels. Science, 2014, 343(6171): 621-623. DOI:10.1126/science.1246929
|
| [5] |
Haynes CA, Gonzalez R. Rethinking biological activation of methane and conversion to liquid fuels. Nat Chem Biol, 2014, 10(5): 331-339. DOI:10.1038/nchembio.1509
|
| [6] |
Jeon YC, Nguyen AD, Lee EY. Bioproduction of Isoprenoids and other secondary metabolites using methanotrophic bacteria as an alternative microbial cell factory option: current stage and future aspects. Catalysts, 2019, 9(11): 883. DOI:10.3390/catal9110883
|
| [7] |
Strong PJ, Xie S, Clarke WP. Methane as a resource: can the methanotrophs add value?. Environ Sci Technol, 2015, 49(7): 4001-4018.
|
| [8] |
Garg S, Clomburg JM, Gonzalez R. A modular approach for high-flux lactic acid production from methane in an industrial medium using engineered Methylomicrobium buryatense 5GB1. J Ind MIcrobiol Biot, 2018, 45(6): 379-391. DOI:10.1007/s10295-018-2035-3
|
| [9] |
Lee JW, Na D, Park JM, et al. Systems metabolic engineering of microorganisms for natural and non-natural chemicals. Nat Chem Biol, 2012, 8(6): 536-546. DOI:10.1038/nchembio.970
|
| [10] |
Choi KR, Jang WD, Yang D, et al. Systems metabolic engineering strategies: integrating systems and synthetic biology with metabolic engineering. Trends Biotechnol, 2019, 37(8): 817-837. DOI:10.1016/j.tibtech.2019.01.003
|
| [11] |
Ko YS, Kim JW, Lee JA, et al. Tools and strategies of systems metabolic engineering for the development of microbial cell factories for chemical production. Chem Soc Rev, 2020, 49(14): 4615-4636. DOI:10.1039/D0CS00155D
|
| [12] |
Lee OK, Nguyen DTN, Lee EY. Metabolic engineering of methanotrophs for the production of chemicals and fuels//Lee EY, Ed. Methanotrophs. Cham: Springer, 2019: 163-203.
|
| [13] |
Fei Q, Guarnieri MT, Tao L, et al. Bioconversion of natural gas to liquid fuel: Opportunities and challenges. Biotechnol Adv, 2014, 32(3): 596-614. DOI:10.1016/j.biotechadv.2014.03.011
|
| [14] |
Kalyuzhnaya MG, Puri AW, Lidstrom ME. Metabolic engineering in methanotrophic bacteria. Metab Eng, 2015, 29: 142-152. DOI:10.1016/j.ymben.2015.03.010
|
| [15] |
Sirajuddin S, Rosenzweig AC. Enzymatic oxidation of methane. Biochemistry, 2015, 54(14): 2283-2294. DOI:10.1021/acs.biochem.5b00198
|
| [16] |
Culpepper MA, Rosenzweig AC. Architecture and active site of particulate methane monooxygenase. Crit Rev Biochem Mol Biol, 2012, 47(6): 483-492. DOI:10.3109/10409238.2012.697865
|
| [17] |
Kao WC, Chen YR, Yi EC, et al. Quantitative proteomic analysis of metabolic regulation by copper ions in Methylococcus capsulatus (Bath). J Biol Chem, 2004, 279(49): 51554-51560. DOI:10.1074/jbc.M408013200
|
| [18] |
Gassner GT, Lippard SJ. Component interactions in the soluble methane monooxygenase system from Methylococcus capsulatus (Bath). Biochemistry, 1999, 38(39): 12768-12785. DOI:10.1021/bi990841m
|
| [19] |
Lee SJ, McCormick MS, Lippard SJ, et al. Control of substrate access to the active site in methane monooxygenase. Nature, 2013, 494(7437): 380-384. DOI:10.1038/nature11880
|
| [20] |
Chan SI, Lee SJ. The biochemistry of methane monooxygenases//Lee E, Ed. Methanotrophs. Cham: Springer, 2019: 71-120.
|
| [21] |
Trotsenko YA, Murrell JC. Metabolic aspects of aerobic obligate methanotrophy. Adv Appl Microb, 2008, 63: 183-229.
|
| [22] |
Elliott SJ, Zhu M, Tso L, et al. Regio- and stereoselectivity of particulate methane monooxygenase from Methylococcus capsulatus (Bath). J Am Chem Soc, 1997, 119(42): 9949-9955. DOI:10.1021/ja971049g
|
| [23] |
Liu CC, Mou CY, Yu SSF, et al. Heterogeneous formulation of the tricopper complex for efficient catalytic conversion of methane into methanol at ambient temperature and pressure. Energ Environ Sci, 2016, 9(4): 1361-1374. DOI:10.1039/C5EE03372A
|
| [24] |
Liu CC, Janmanchi D, Wen DR, et al. Catalytic oxidation of light alkanes mediated at room temperature by a tricopper cluster complex immobilized in mesoporous silica nanoparticles. Acs Sustainable Chem Eng, 2018, 6(4): 5431-5440. DOI:10.1021/acssuschemeng.8b00270
|
| [25] |
Lee OK, Hur DH, Nguyen DTN, et al. Metabolic engineering of methanotrophs and its application to production of chemicals and biofuels from methane. Biofuel Bioprod Bior, 2016, 10(6): 848-863. DOI:10.1002/bbb.1678
|
| [26] |
De La Torre A, Metivier A, Chu F, et al. Genome-scale metabolic reconstructions and theoretical investigation of methane conversion in Methylomicrobium buryatense strain 5G (B1). Microb Cell Fact, 2015, 14: 188. DOI:10.1186/s12934-015-0377-3
|
| [27] |
Khadem AF, Pol A, Wieczorek A, et al. Autotrophic methanotrophy in Verrucomicrobia: Methylacidiphilum fumariolicum SolV uses the calvin-benson-bassham cycle for carbon dioxide fixation. J Bacteriol, 2011, 193(17): 4438-4446. DOI:10.1128/JB.00407-11
|
| [28] |
胡礼珍, 王佳, 袁波, 等. 碳一气体生物利用进展. 生物加工过程, 2017, 15(6): 17-25. Hu LZ, Wang J, Yuan B, et al. Production of biofuels and chemicals from C1 gases by microorganisms: status and prospects. Chin J Bio Eng, 2017, 15(6): 17-25 (in Chinese). DOI:10.3969/j.issn.1672-3678.2017.06.003 |
| [29] |
Anvar SY, Frank J, Pol A, et al. The genomic landscape of the verrucomicrobial methanotroph Methylacidiphilum fumariolicum SolV. BMC Genomics, 2014, 15: 914. DOI:10.1186/1471-2164-15-914
|
| [30] |
Rasigraf O, Kool DM, Jetten MSM, et al. Autotrophic carbon dioxide fixation via the calvin-benson-bassham cycle by the denitrifying methanotroph "candidatus Methylomirabilis oxyfera". Appl Environ Microbiol, 2014, 80(8): 2451-2460. DOI:10.1128/AEM.04199-13
|
| [31] |
Akberdin IR, Thompson M, Hamilton R, et al. Methane utilization in Methylomicrobium alcaliphilum 20ZR: a systems approach. Sci Rep, 2018, 8: 4753. DOI:10.1038/s41598-018-23088-w
|
| [32] |
Bordel S, Rodríguez Y, Hakobyan A, et al. Genome scale metabolic modeling reveals the metabolic potential of three Type Ⅱ methanotrophs of the genus Methylocystis. Metab Eng, 2019, 54: 191-199. DOI:10.1016/j.ymben.2019.04.001
|
| [33] |
Nguyen DTN, Lee OK, Lim C, et al. Metabolic engineering of type Ⅱ methanotroph, Methylosinus trichosporium OB3b, for production of 3-hydroxypropionic acid from methane via a malonyl-CoA reductase-dependent pathway. Metab Eng, 2020, 59: 142-150. DOI:10.1016/j.ymben.2020.02.002
|
| [34] |
Hogendoorn C, Pol A, Nuijten GHL, et al. Methanol production by "Methylacidiphilum fumariolicum" SolV under different growth conditions. Appl Environ Microbiol, 2020, 86(18): e01188-20.
|
| [35] |
Fei Q, Pienkos PT. Bioconversion of methane for value-added products//Sani RK, Rathinam NK, Eds. Extremophilic microbial processing of lignocellulosic feedstocks to biofuels, value-added products, and usable power. Cham: Springer, 2018: 145-162.
|
| [36] |
Puri AW, Owen S, Chu F, et al. Genetic tools for the industrially promising methanotroph Methylomicrobium buryatense. Appl Environ Microbiol, 2015, 81(5): 1775-1781. DOI:10.1128/AEM.03795-14
|
| [37] |
Hwang IY, Nguyen AD, Nguyen TT, et al. Biological conversion of methane to chemicals and fuels: technical challenges and issues. Appl Microbiol Biotechnol, 2018, 102(7): 3071-3080. DOI:10.1007/s00253-018-8842-7
|
| [38] |
Liu YC, He XR, Zhu PP, et al. pheSAG based rapid and efficient markerless mutagenesis in Methylotuvimicrobium. Front Microbiol, 2020, 11: 441. DOI:10.3389/fmicb.2020.00441
|
| [39] |
Nguyen DTN, Lee OK, Hadiyati S, et al. Metabolic engineering of the type Ⅰ methanotroph Methylomonas sp. DH-1 for production of succinate from methane. Metab Eng, 2019, 54: 170-179. DOI:10.1016/j.ymben.2019.03.013
|
| [40] |
Nguyen AD, Hwang IY, Lee OK, et al. Systematic metabolic engineering of Methylomicrobium alcaliphilum 20Z for 2, 3-butanediol production from methane. Metab Eng, 2018, 47: 323-333. DOI:10.1016/j.ymben.2018.04.010
|
| [41] |
Hu L, Yang Y, Yan X, et al. Molecular mechanism associated with the impact of methane/oxygen gas supply ratios on cell growth of Methylomicrobium buryatense 5GB1 through RNA-Seq. Front Bioeng Biotech, 2020, 8: 263. DOI:10.3389/fbioe.2020.00263
|
| [42] |
Pastor JM, Salvador M, Argandoña M, et al. Ectoines in cell stress protection: uses and biotechnological production. Biotechnol Adv, 2010, 28(6): 782-801. DOI:10.1016/j.biotechadv.2010.06.005
|
| [43] |
Vallenet D, Belda E, Calteau A, et al. MicroScope—an integrated microbial resource for the curation and comparative analysis of genomic and metabolic data. Nucleic Acids Res, 2013, 41(D1): D636–-D647. DOI:10.1093/nar/gks1194
|
| [44] |
Fei Q, Liang BB, Tao L, et al. Biological valorization of natural gas for the production of lactic acid: Techno-economic analysis and life cycle assessment. Biochem Eng J, 2020, 158: 107500. DOI:10.1016/j.bej.2020.107500
|
| [45] |
Wendlandt KD, Jechorek M, Helm J, et al. Producing poly-3-hydroxybutyrate with a high molecular mass from methane. J Biotechnol, 2001, 86(2): 127-133. DOI:10.1016/S0168-1656(00)00408-9
|
| [46] |
Subbian E. Production of lactic acid from organic waste or biogas or methane using recombinant methanotrophic bacteria: US, WO/2015/155790. 2019-01-29.
|
| [47] |
Henard CA, Smith H, Dowe N, et al. Bioconversion of methane to lactate by an obligate methanotrophic bacterium. Sci Rep, 2016, 6: 21585. DOI:10.1038/srep21585
|
| [48] |
Coleman WJ, Vidanes GM, Cottarel G, et al. Biological conversion of multi-carbon compounds from methane: US, 20140273128. 2014-09-18.
|
| [49] |
Demídenko A, Akberdin IR, Allemann M, et al. Fatty acid biosynthesis pathways in Methylomicrobium buryatense 5G(B1). Front Microbiol, 2017, 7: 2167.
|
| [50] |
Garg S, Wu H, Clomburg JM, et al. Bioconversion of methane to C-4 carboxylic acids using carbon flux through acetyl-CoA in engineered Methylomicrobium buryatense 5GB1C. Metab Eng, 2018, 48: 175-183. DOI:10.1016/j.ymben.2018.06.001
|
| [51] |
Subbian E. Production of succinic acid from organic waste or biogas or methane using recombinant methanotrophic bacterium: US, 20170121740. 2017-05-04.
|
| [52] |
Nguyen LT, Lee EY. Biological conversion of methane to putrescine using genome-scale model-guided metabolic engineering of a methanotrophic bacterium Methylomicrobium alcaliphilum 20Z. Biotechnol Biofuels, 2019, 12: 147. DOI:10.1186/s13068-019-1490-z
|
| [53] |
Held MA, Zhao X, Chao LY, et al. New genetically modified microorganism comprising a heterologous gene under the control of a molecular switch, useful for making a multicarbon product, e. g. 2, 3-butanediol, 1, 4-butanediol, and isobutyraldehyde: US, 0040344. 2020-02-06.
|
| [54] |
Leonard E, Minshull J, Ness J, et al. Compositions and methods for biological production of isoprene. US, WO/2014/138419. 2014-09-12.
|
| [55] |
Donaldson GK, Hollands K, Picataggio SK. Biocatalyst for conversion of methane and methanol to isoprene: US, 20150225743. 2015-08-13.
|
| [56] |
Dicosimo DJ, Koffas M, Odom JM, et al. Production of cyclic terpenoids: US, 6818424. 2004-11-16.
|
| [57] |
Ye RW, Yao H, Stead K, et al. Construction of the astaxanthin biosynthetic pathway in a methanotrophic bacterium Methylomonas sp strain 16a. J Ind Microbiol Biot, 2007, 34(4): 289-299. DOI:10.1007/s10295-006-0197-x
|
| [58] |
Sharpe PL, DiCosimo D, Bosak MD, et al. Use of transposon promoter-probe vectors in the metabolic engineering of the obligate methanotroph Methylomonas sp. strain 16a for enhanced C-40 carotenoid synthesis. Appl Environ Microbiol, 2007, 73(6): 1721-1728. DOI:10.1128/AEM.01332-06
|
| [59] |
Henard CA, Akberdin IR, Kalyuzhnaya MG, et al. Muconic acid production from methane using rationally-engineered methanotrophic biocatalysts. Green Chem, 2019, 21(24): 6731-6737. DOI:10.1039/C9GC03722E
|
| [60] |
Fei Q, Puri AW, Smith H, et al. Enhanced biological fixation of methane for microbial lipid production by recombinant Methylomicrobium buryatense. Biotechnol Biofuels, 2018, 11: 129. DOI:10.1186/s13068-018-1128-6
|
| [61] |
Sherkhanov S, Korman TP, Chan S, et al. Isobutanol production freed from biological limits using synthetic biochemistry. Nat Commun, 2020, 11(1): 4292. DOI:10.1038/s41467-020-18124-1
|
| [62] |
Wang L, Zong Z, Liu Y, et al. Metabolic engineering of Yarrowia lipolytica for the biosynthesis of crotonic acid. Bioresource Technol, 2019, 287: 121484. DOI:10.1016/j.biortech.2019.121484
|
| [63] |
Kim JW, Ko YS, Chae TU, et al. High-level production of 3-hydroxypropionic acid from glycerol as a sole carbon source using metabolically engineered Escherichia coli. Biotechnol Bioeng, 2020, 117(7): 2139-2152. DOI:10.1002/bit.27344
|
| [64] |
Ahn JH, Seo H, Park W, et al. Enhanced succinic acid production by Mannheimia employing optimal malate dehydrogenase. Nat Commun, 2020, 11: 19701.
|
| [65] |
Li W, Narancic T, Kenny ST, et al. Unraveling 1, 4-butanediol metabolism in Pseudomonas putida KT2440. Front Microbiol, 2020, 11: 382. DOI:10.3389/fmicb.2020.00382
|
| [66] |
Ma H, Zhao YQ, Huang WZ, et al. Rational flux-tuning of Halomonas bluephagenesis for co-production of bioplastic PHB and ectoine. Nat Commun, 2020, 11(1): 3313. DOI:10.1038/s41467-020-17223-3
|
| [67] |
Khmelenina VN, Kalyuzhnaya MG, Sakharovsky VG, et al. Osmoadaptation in halophilic and alkaliphilic methanotrophs. Arch Microbiol, 1999, 172(5): 321-329. DOI:10.1007/s002030050786
|
| [68] |
Ren YY, Liu SS, Jin GJ, et al. Microbial production of limonene and its derivatives: Achievements and perspectives. Biotechnol Adv, 2020, 44(15): 107628.
|
| [69] |
Liu SC, Liu ZJ, Wei LJ, et al. Pathway engineering and medium optimization for alpha-farnesene biosynthesis in oleaginous yeast Yarrowia lipolytica. J Biotechnol, 2020, 319: 74-81. DOI:10.1016/j.jbiotec.2020.06.005
|
| [70] |
Guo W, Li D, He R, et al. Synthesizing value-added products from methane by a new Methylomonas. J Appl Microbiol, 2017, 123(5): 1214-1227. DOI:10.1111/jam.13581
|
| [71] |
Huccetogullari D, Luo ZW, Lee SY. Metabolic engineering of microorganisms for production of aromatic compounds. Microb Cell Fact, 2019, 18: 41. DOI:10.1186/s12934-019-1090-4
|
| [72] |
Bentley GJ, Narayanan N, Jha RK, et al. Engineering glucose metabolism for enhanced muconic acid production in Pseudomonas putida KT2440. Metab Eng, 2020, 59: 64-75. DOI:10.1016/j.ymben.2020.01.001
|
| [73] |
Dong T, Fei Q, Genelot M, et al. A novel integrated biorefinery process for diesel fuel blendstock production using lipids from the methanotroph, Methylomicrobium buryatense. Energ Convers Manage, 2017, 140: 62-70. DOI:10.1016/j.enconman.2017.02.075
|
| [74] |
Jang YS, Kim B, Shin JH, et al. Bio-based production of C2-C6 platform chemicals. Biotechnol Bioeng, 2012, 109(10): 2437-2459. DOI:10.1002/bit.24599
|
| [75] |
Henard CA, Franklin TG, Youhenna B, et al. Biogas biocatalysis: methanotrophic bacterial cultivation, metabolite profiling, and bioconversion to lactic acid. Front Microbiol, 2018, 9: 2610. DOI:10.3389/fmicb.2018.02610
|
| [76] |
Wang GK, Øzmerih S, Guerreiro R, et al. Improvement of cis, cis-muconic acid production in Saccharomyces cerevisiae through biosensor-aided genome engineering. Acs Synth Biol, 2020, 9(3): 634-646. DOI:10.1021/acssynbio.9b00477
|
| [77] |
Fujiwara R, Noda S, Tanaka T, et al. Metabolic engineering of Escherichia coli for shikimate pathway derivative production from glucose-xylose co-substrate. Nat Commun, 2020, 11(1): 1037. DOI:10.1038/s41467-020-14710-5
|
| [78] |
Gao X, Gao F, Liu D, et al. Engineering the methylerythritol phosphate pathway in cyanobacteria for photosynthetic isoprene production from CO2. Energ Environ Sci, 2016, 9(4): 1400-1411. DOI:10.1039/C5EE03102H
|
| [79] |
Song JY, Cho KK, Lee KS, et al. Method for producing isoprene using recombinant halophilic methanotroph: US, 9994869. 2018-06-12.
|
| [80] |
Kalyuzhnaya MG, Yang S, Rozova ON, et al. Highly efficient methane biocatalysis revealed in methanotrophic bacterium. Nat Commun, 2013, 4: 2758. DOI:10.1038/ncomms3758
|
| [81] |
Henard CA, Freed EF, Guarnieri MT, et al. Phosphoketolase pathway engineering for carbon-efficient biocatalysis. Curr Opin Biotech, 2015, 36: 183-188. DOI:10.1016/j.copbio.2015.08.018
|
| [82] |
Gilman A, Laurens LM, Puri AW, et al. Bioreactor performance parameters for an industrially-promising methanotroph Methylomicrobium buryatense 5GB1. Microb Cell Fact, 2015, 14: 182. DOI:10.1186/s12934-015-0372-8
|
| [83] |
蔺玉萍, 王钦宏. 工业微生物遗传和环境扰动的调控和适应进化. 生物工程学报, 2019, 35(10): 1925-1941. Lin YP, Wang QH. Regulation and adaptive evolution of industrial microorganisms towards genetic and environmental disturbances. Chin J Biotech, 2019, 35(10): 1925-1941 (in Chinese). |
| [84] |
Kaluzhnaya M, Khmelenina V, Eshinimaev B, et al. Taxonomic characterization of new alkaliphilic and alkalitolerant methanotrophs from soda lakes of the Southeastern Transbaikal region and description of Methylomicrobium buryatense sp. nov. Syst Appl Microbiol, 2001, 24(2): 166-176. DOI:10.1078/0723-2020-00028
|
| [85] |
Cantera S, Bordel S, Lebrero R, et al. Bio-conversion of methane into high profit margin compounds: an innovative, environmentally friendly and cost-effective platform for methane abatement. World J Microb Biot, 2019, 35(1): 16. DOI:10.1007/s11274-018-2587-4
|
| [86] |
Meruvu H, Wu H, Jiao ZY, et al. From nature to nurture: Essence and methods to isolate robust methanotrophic bacteria. Syn Sys Biot, 2020, 5(3): 173-178.
|
| [87] |
Hoefman S, van der Ha D, Boon N, et al. Niche differentiation in nitrogen metabolism among methanotrophs within an operational taxonomic unit. BMC Microbiol, 2014, 14: 83. DOI:10.1186/1471-2180-14-83
|
| [88] |
Balasubramanian R, Smith SM, Rawat S, et al. Oxidation of methane by a biological dicopper centre. Nature, 2010, 465(7294): 115-131. DOI:10.1038/nature08992
|
| [89] |
Gou Z, Xing X, Luo M, et al. Functional expression of the particulate methane mono-oxygenase gene in recombinant Rhodococcus erythropolis. FEMS Microbiol Lett, 2006, 263(2): 136-141. DOI:10.1111/j.1574-6968.2006.00363.x
|
| [90] |
Nguyen AD, Lee EY. Engineered Methanotrophy: A sustainable solution for methane-based industrial biomanufacturing. Trends Biotechnol, 2020.
|
| [91] |
Nguyen AD, Hwang IY, Chan JY, et al. Reconstruction of methanol and formate metabolic pathway in non-native host for biosynthesis of chemicals and biofuels. Biotechnol Bioproce, 2016, 21(4): 477-482. DOI:10.1007/s12257-016-0301-7
|
 2021, Vol. 37
2021, Vol. 37