中国科学院微生物研究所、中国微生物学会主办
文章信息
- 王丽君, 闫思翰, 杨套伟, 徐美娟, 张显, 邵明龙, 李华钟, 饶志明
- Wang Lijun, Yan Sihan, Yang Taowei, Xu Meijuan, Zhang Xian, Shao Minglong, Li Huazhong, Rao Zhiming
- 代谢改造重组谷氨酸棒杆菌C4途径高效合成5-氨基乙酰丙酸
- Engineering the C4 pathway of Corynebacterium glutamicum for efficient production of 5-aminolevulinic acid
- 生物工程学报, 2021, 37(12): 4314-4328
- Chinese Journal of Biotechnology, 2021, 37(12): 4314-4328
- 10.13345/j.cjb.210057
-
文章历史
- Received: January 18, 2021
- Accepted: March 8, 2021
- Published: March 8, 2021
5-氨基乙酰丙酸(5-aminolevulinic acid,5-ALA) 是四氢吡咯化合物如血红素、叶绿素和维生素B12等的共同前体[1],在医药和农业等方面有着非常重要的作用。在医药行业,5-ALA主要被应用于肿瘤定位和多种癌症的光动力诊断治疗等[2]。在农业领域,它被用作一种可生物降解的除草剂、杀虫剂或对作物、动物和人类无毒的生长调节剂[3]。针对合成方式而言,化学合成法存在试剂贵、毒性高、产率低和反应条件苛刻的弊端,所以环保高效的微生物发酵法逐渐成为主流的生产方式[4]。
据报道5-ALA有C4途径和C5途径两种生物合成方式(图 1),其中C4途径存在于酿酒酵母Saccharomyces cerevisiae[5]等菌株中,主要是以5-磷酸吡哆醛为辅因子,由甘氨酸和琥珀酰CoA通过5-氨基乙酰丙酸合酶(ALAS) 缩合形成5-ALA。5-ALA生物合成途径目前已在大肠杆菌Escherichia coli[6]、谷氨酸棒杆菌Corynebacterium glutamicum[7]和天蓝色链霉菌Streptomyces coelicolor[8]等模式菌株中成功构建。另一条C5途径则存在于E. coli等菌株中,主要通过谷氨酰tRNA合成酶GluRS (由gltX编码),NADPH依赖的谷氨酰-tRNA-还原酶HemA (由HemA编码) 和谷氨酸-1-半醛氨基转移酶HemL (由HemL编码) 3种酶,以谷氨酸为底物经过3步反应生成5-ALA[9],但此途径会受终产物血红素的反馈抑制作用[10]。Kang等通过同时表达亚利桑那沙门菌Salmonella arizonae来源的HemA、E. coli来源的HemL和ALA胞外运输蛋白基因rhtA构建重组E. coli工程菌,可积累4.13 g/L的5-ALA[11]。但是,由于C5途径包含以ATP和NADPH作为辅因子的复杂酶反应,且依赖于tRNA-Glu合成酶,与能量代谢、氧化还原状态和蛋白质合成的复杂关系使菌株改良变得困难。而C4途径只需在E. coli中过表达ALAS即可合成5-ALA,易于进一步优化改造,得到了更多的关注和研究[12-13]。C. glutamicum作为GRAS菌株是生产5-ALA的理想宿主,Yang等[7]构建了利用C4途径合成5-ALA的重组C. glutamicum,运用分批补料发酵和两阶段发酵策略优化5-ALA的合成,产量达14.7 g/L,生产率达0.92 g/(L·h),为迄今为止报道的C. glutamicum以葡萄糖为碳源合成5-ALA的最高产率。Chen等[14]则进一步使用酶水解的木薯蔗渣代替葡萄糖作为碳源使碳源成本降低了90.1%,5-ALA产量增加到了18.5 g/L。另外,弱化5-ALA下游代谢路径,减少胆色素原(Porphobilinogen,PBG) 产生的策略可解决5-ALA在发酵后期易被消耗合成血红素[15-16]的问题,可进一步促进5-ALA的积累。
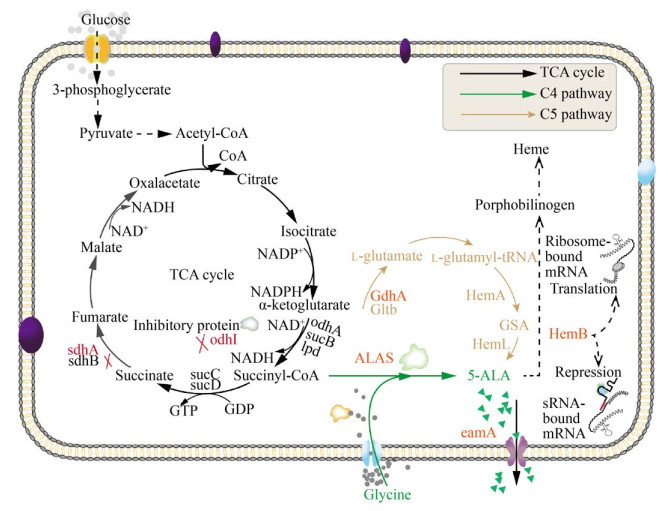
|
| 图 1 代谢工程化谷氨酸棒杆菌从葡萄糖中生物合成5-ALA的示意图 Fig. 1 Schematic illustration of the biosynthesis of 5-ALA by metabolically engineered C. glutamicum from glucose. |
| |
本研究系统地改造C. glutamicum ATCC13032来高效合成5-ALA,将荚膜红杆菌Rhodobacter capsulatus和沼泽红假单胞菌Rhodopseudomonas palustris来源的ALAS基因hemA在C. glutamicum ATCC13032中分别进行异源表达,选取酶活相对较高的RphemA作为关键合成酶;敲除TCA循环中关键基因促进5-ALA途径通量,通过sRNA[17]抑制调控5-ALA降解途径的第一个关键酶5-ALA脱水酶(ALAD,由hemB编码) 的表达,并过表达5-ALA转运蛋白eamA进一步提高5-ALA的产量。本研究为C. glutamicum 5-ALA高产菌株的工业应用奠定了基础。
1 材料与方法 1.1 材料 1.1.1 主要试剂琼脂糖凝胶DNA回收试剂盒、小量质粒提取试剂盒均购于上海捷瑞生物工程有限公司;同源重组酶克隆试剂盒(ClonExpress Ⅱ One Step Cloning Kit) 购自南京诺维赞生物科技有限公司;EcoRⅠ和Hind Ⅲ等限制性内切酶购自TaKaRa公司。另外硫酸卡那霉素(Kan) 和氯霉素(Chl) 等抗生素、溶菌酶、异丙基-β-D-硫代半乳糖苷(IPTG) 及核酸染料等试剂均购自上海生工生物工程股份有限公司;脑心肉汤培养基(Brain Heart Infusion Broth) 购自青岛海博生物技术有限公司;玉米浆购自阿拉丁公司,葡萄糖、甘氨酸、冰醋酸、高氯酸和乙酰丙酮等分析纯试剂购自国药集团化学试剂有限公司。
1.1.2 菌株、质粒和培养基野生型C. glutamicum ATCC13032、大肠杆菌E. coli BL21(DE3) 和质粒pXMJ19、pK18mobsacB (pK18) 均为实验室保藏。本研究中构建的菌株命名见表 1,质粒说明见表 2。
| Strains | Abbreviation | Illustration |
| C. glutamicum/pXMJ19 | CA0 | Harboring with plasmid of pXMJ19 |
| C. glutamicum/pXMJ19-RchemA | CA1 | Harboring with plasmid of pXMJ19-RchemA |
| C. glutamicum/pXMJ19-RphemA | CA2 | Harboring with plasmid of pXMJ19-RphemA |
| C. glutamicum/pXMJ19-RBS2RphemA | CA3 | Harboring with plasmid of pXMJ19-RBS2RphemA |
| C. glutamicum/pXMJ19-RBS3RphemA | CA4 | Harboring with plasmid of pXMJ19-RBS3RphemA |
| C. glutamicum/pXMJ19-RBS5RphemA | CA5 | Harboring with plasmid of pXMJ19-RBS5RphemA |
| C. glutamicum/pXMJ19-RBS8RphemA | CA6 | Harboring with plasmid of pXMJ19-RBS8RphemA |
| C. glutamicum-∆odhI | C7 | Knocking out odhI |
| C. glutamicum-∆gdhA | C8 | Knocking out gdhA |
| C. glutamicum-∆sdhA | C9 | Knocking out sdhA |
| C. glutamicum-∆odhI∆gdhA | C10 | Knocking out odhI and gdhA |
| C. glutamicum-∆odhI∆sdhA | C11 | Knocking out odhI and sdhA |
| C. glutamicum-∆odhI-pXMJ19-RphemA | CA7 | Harboring with plasmid of pXMJ19-RphemA in C7 |
| C. glutamicum-∆gdhA-pXMJ19-RphemA | CA8 | Harboring with plasmid of pXMJ19-RphemA in C8 |
| C. glutamicum-∆sdhA-pXMJ19-RphemA | CA9 | Harboring with plasmid of pXMJ19-RphemA in C9 |
| C. glutamicum-∆odhI∆gdhA-pXMJ19-RphemA | CA10 | Harboring with plasmid of pXMJ19-RphemA in C10 |
| C. glutamicum-∆odhI∆sdhA-pXMJ19-RphemA | CA11 | Harboring with plasmid of pXMJ19-RphemA in C11 |
| C. glutamicum/pXMJ19-RphemA-eamA | CA12 | Harboring with plasmid of pXMJ19-RphemA-eamA |
| C. glutamicum/pXMJ19-hemB | CA13 | Harboring with plasmid of pXMJ19-hemB |
| C. glutamicum/pXMJ19-sRNAhemB-RBS5RphemA | CA15 | Harboring with plasmid of pXMJ19-sRNAhemB-RBS5RphemA |
| C. glutamicum-∆odhI∆sdhA-sRNAhemB- RBS5RphemA-eamA | CA16 | Harboring with plasmid of pXMJ19-sRNAhemB-RBS5RphemA-eamA in C11 |
| Plasmids | Illustration |
| pXMJ19 | Expression plasmid; Ptac promoter |
| pK18mobsacB (pK18) | Integration plasmid of C. glutamicum |
| pXMJ19-RchemA | Express the gene of RchemA |
| pXMJ19-RphemA | Express the gene of RphemA |
| pXMJ19-RBS2RphemA | RBS2 replace the RBS sequence of pXMJ19-RphemA |
| pXMJ19-RBS3RphemA | RBS3 replace the RBS sequence of pXMJ19-RphemA |
| pXMJ19-RBS5RphemA | RBS5 replace the RBS sequence of pXMJ19-RphemA |
| pXMJ19-RBS8RphemA | RBS8 replace the RBS sequence of pXMJ19-RphemA |
| pK18-∆odhI | Knock out the gene of odhI |
| pK18-∆gdhA | Knock out the gene of gdhA |
| pK18-∆sdhA | Knock out the gene of sdhA |
| pXMJ19-RphemA-eamA | Tandem expression the gene of eamA on plasmid of pXMJ19-RphemA |
| pXMJ19-hfq | Express the gene of hfq |
| p0 | Synthetic plasmid containing micc sequence and T7 terminator |
| pXMJ19-sRNAhemB-RBS5RphemA | Inhibit the recombinant plasmid of pXMJ19-RBS5RphemA with sRNA |
| pXMJ19-hemB | Express the gene of hemB |
| pXMJ19-sRNAhemB-RBS5RphemA-eamA | Tandem expression the gene of eamA on plasmid of pXMJ19-sRNAhemB-RBS5RphemA |
LB培养基(g/L):酵母粉5,蛋白胨10,NaCl 10 (固体培养基加入1.5%–2.0%的琼脂粉)。
脑心肉汤(BHI) 培养基:称取成品粉末38.5 g,搅拌溶解于1 000 mL蒸馏水中(固体培养基加入1.5%–2.0%的琼脂粉)。
C. glutamicum感受态培养基:每100 mL BHI培养基中加入3 g甘氨酸以及100 μL吐温-80混匀。
C. glutamicum摇瓶发酵培养基(g/L):葡萄糖140,K2HPO4·3H2O 1.0,MgSO4 0.6,玉米浆5,FeSO4·7H2O 0.002,MnSO4·4H2O 0.002,尿素7.0 (pH 7.0)。
C. glutamicum 5 L发酵罐培养基(g/L):葡萄糖125,(NH4)2SO4 20,KH2PO4 2.0,MgSO4·7H2O 0.5,玉米浆50,FeSO4·7H2O 0.02,MnSO4·4H2O 0.02,尿素1.0 (pH 6.5)。
1.1.3 PCR引物设计本研究中PCR扩增所用引物由苏州金唯智生物公司合成,序列见表 3。
| Primer names | Primer sequences (5′–3′) |
| RchemA-F | ACAGAATTAATTAAGCTTAAAGGAGGGAAATCATGGACTACAACCTCGCACT |
| RchemA-R | AGCCAAGCTGAATTCTTAGTGGTGGTGGTGGTGGTGTGCGCAGCGAGCCCACAGCAGAT |
| RphemA-F | ACAGAATTAATTAAGCTTAAAGGAGGGAAATCATGGATTACACCAAGTTCTTCGC |
| RphemA-R | CAAAACAGCCAAGCTGAATTCTTAGTGGTGGTGGTGGTGGTGTTCCGCAGCGAGCGGCT |
| odhI-1 | AGGAAACAGCTATGACATGATTACGAATTCGTCCGTGAGGGAACAGGCAGCACGA |
| odhI-2 | TAACGCCCATCCACTAAACTTAAACAGAGCAGTACCGGTGCTCGACTCCATTTCCT |
| odhI-3 | CTGCTCTGTTTAAGTTTAGTGGATGGGCGTTAACCGCGAGCCACGCAACGCTCAGG |
| odhI-4 | CGTTGTAAAACGACGGCCAGTGCCAAGCTTAGGAACCAAGGATGGCGGTGACAGA |
| gdhA-1 | CACAGGAAACAGCTATGACATGATTACGAATTCCAACTACGGTCTGGAACCATCACCATC |
| gdhA-2 | GGGCTTTGAGCAGACCCATCCACTAAACTTAAACAAGACAACGGCTGCCGTTTCGTTGCT |
| gdhA-3 | CGGCAGCCGTTGTCTTGTTTAAGTTTAGTGGATGGGTCTGCTCAAAGCCCAGGAACTTCA |
| gdhA-4 | ACGACGTTGTAAAACGACGGCCAGTGCCAAGCTTTACCAATTCCATTTGAGGGCGCTCAA |
| sdhA-1 | AGGAAACAGCTATGACATGATTACGAATTCCCTACGTATTATCCTGCTGGTCGCATTGGT |
| sdhA-2 | CGCTGCCTGTGCTTCCCCATCCACTAAACTTAAACAGGTTGCCAAGGCGCCACCGTATTC |
| sdhA-3 | GTGGCGCCTTGGCAACCTGTTTAAGTTTAGTGGATGGGGAAGCACAGGCAGCGATTGCGC |
| sdhA-4 | TCACGACGTTGTAAAACGACGGCCAGTGCCAAGCTTGCGCTCTTCCTTACCCAGTGGGAG |
| RBS2RphemA-F | GAAACAGAATTAATTAAGCTTAAAGGAGGTTGTCATGGACTACACCAAGTTCTTTGCTGA |
| RBS3RphemA-F | GAAACAGAATTAATTAAGCTTGAAAGGAGAATGGACTACACCAAGTTCTTTGCTGA |
| RBS5RphemA-F | GAAACAGAATTAATTAAGCTTGGAAGGAAGGAAACATGGACTACACCAAGTTCTTTGCTG |
| RBS8RphemA-F | GAAACAGAATTAATTAAGCTTAAAGGCTGGAATTATGGACTACACCAAGTTCTTTGCTGA |
| RphemA-eamA-1 | GAAACAGAATTAATTAAGCTTAAAGGAGGGAAATCATGGACTACACCAAGTTCTTTGCTG |
| RphemA-eamA-2 | CGAGCCGATGATTAATTGTCAATTAGTGGTGGTGGTGGTGGTGTTCTGCTGCCAGTGGCT |
| eamA-F | TTGACAATTAATCATCGGCTCGTATAATGGAAAGGAGAATGTCGCGAAAAGATGGGGTG |
| eamA-R | CCGCCAAAACAGCCAAGCTGAATTCTTAACTTCCCACCTTTACCGCT |
| hemB-F | AGAATTAATTAAGCTTAAAGGAGGGAAATCATGAGCACTTCTTCTGATTACTCCC |
| hemB-R | CAAAACAGCCAAGCTGAATTCTTAAGCGTTTCGCAGTGCGCG |
| p19-hfq-F1 | AGAATTAATTAAGCTTAAAGGAGGATTAGATGGCTAAGGGGCAATCTTTACAA |
| p19-hfq-R1 | ATCCGCCAAAACAGCCAAGCTGAATTCTTATTCGGTTTCTTCGCTGTCCT |
| p19-ssRNA (hemB)-F2 | GAGCGGATTTGGGAGTAATCAGAAGAAGTGCTCATTTTCTGTTGGGCCATTGCATT |
| p19-ssRNA(hemB)-R2 | AATAAACAAAAGAGTTTGTAGAAACGCACGCAGAAAGGCCCACCCGAAGG |
| end-p19-hfq-R | CATTGCCAGCTTATGTGGAGAAAAAAACGCAAAAAGGCCATCCGTCAGGA |
| p19-H11F- sRNAhemB-F3 |
CCTTTTTGCGTTTTTTTCTCCACATAAGCTGGCAATGTTGCGACGCAACAGGTACAGTGTAATTCAGTGGAGCGGATTTGGGAGTAATCA |
| p19-hfq-H11F-miccF4 | GTAATTCAGTGGAGCGGATTTGGGAGTAATCAGAAGAAGTGCTCATTTTCTGTTGGGCCA |
| p19-hfq-H11F-micc R4 | TCCTTCCTTCCCATTATACGAGCCGATGATTAATTGTCAACGCAGAAAGGCCCACCCGAA |
| RBS5RphemA-EamA- hfq-H11F-micc-F5 |
TAATCATCGGCTCGTATAATGGGAAGGAAGGAAACATGGACTACACCAAGTTCTTTGCTG |
| RBS5RphemA-hfq- H11F-micc-R5 |
CGAGCCGATGATTAATTGTCAATTAGTGGTGGTGGTGGTGGTGTTCT |
| P19-RpeA-F | CGTAAAGCGGTAAAGGTGGGAAGTTAAGAATTCAGCTTGGCTGTTTTGGC |
| P19-RpeA-R | CGAGCCGATGATTAATTGTCAATTAGTGGTGGTGGTGGTGGTGTTCTGCT |
| Ptac-eamA-F | CCACTAATTGACAATTAATCATCGGCTCGTATAATGGAAAGGAGAATGTCGCGAAAAG |
| Ptac-eamA-R | CATCCGCCAAAACAGCCAAGCTGAATTCTTAACTTCCCACCTTTACCGCTTTAC |
| The bold sequences are the restriction sites and the underlined sequences are the different RBS sequences. The bold and italic sequences are the Ptac promoter sequences. | |
将R. capsulatus来源的基因RchemA (GenBank:KU687108.1) 序列和R. palustris来源的基因RphemA (GenBank:AY489 557.1) 序列由苏州金唯智生物有限公司进行合成。以合成含有目的基因的重组质粒为模板,分别使用引物RchemA-F、RchemA-R和RphemA-F、RphemA-R,通过PCR扩增获得目的基因片段,与经过Hind Ⅲ和EcoRⅠ双酶切载体pXMJ19后的线性化产物进行同源重组连接,并转化E. coli BL21(DE3) 感受态细胞。从转化平板挑取形态大小均一的单菌落作为模板进行菌落PCR验证,并将验证呈阳性的转化子在LB培养基(Chl终浓度为15 µg/mL) 中培养12 h后,使用小量质粒提取试剂盒提取质粒pXMJ19-RchemA、pXMJ19-RphemA送至苏州金唯智生物科技有限公司测序。测序成功后将质粒分别以电穿孔法转化C. glutamicum ATCC13032感受态细胞,并验证筛选正确的阳性转化子,送苏州金唯智生物科技有限公司测序验证。
将验证正确的菌株在BHI固体平板上分别划线活化并挑取单菌落接入含有Chl (15 µg/mL) 抗性的10 mL BHI培养基中,30 ℃、180 r/min振荡培养18 h后以1%的接种量转接入50 mL BHI培养基中继续培养4–5 h,待OD600达到0.6–0.8时加入0.5 mmol/L IPTG,16 ℃、180 r/min继续培养12 h后,于4 ℃、10 000 r/min离心20 min。菌体收集后用100 mmol/L Tris-HCl缓冲液(pH 7.5) 洗涤2次,加入终浓度10 mg/mL的溶菌酶冰上静置2 h后进行超声波破碎(工作2 s,间隔5 s,破碎30–40 min)。将破碎液于4 ℃、12 000 r/min离心30 min,取上清液即为ALAS粗酶液,之后进行酶活力测定及分析。
1.2.2 C. glutamicum基因缺失突变株的构建C. glutamicum基因敲除采用敲除质粒pK18mobsacB[18]。以C. glutamicum ATCC13032基因组为模板,分别利用odhI-1/odhI-2和odhI-3/odhI-4引物扩增完整的odhI基因及其上下游部分片段(含启动子序列) 的odhI1和odhI2基因片段。然后以odhI1和odhI2基因片段为模板,利用odhI-1/odhI-4引物扩增出缺失型△odhI基因片段。纯化回收后的△odhI基因片段与pK18质粒酶切后获得的线性化产物利用DNA连接酶连接,将连接产物使用化学转化法导入E. coli BL21感受态细胞,于Kan抗性平板培养,挑取单菌落转接至LB培养基,取菌液抽提重组质粒,并分别作为模板进行PCR扩增验证,将验证成功的重组质粒送至苏州金唯智生物科技有限公司测序以确认重组质粒pK18-△odhI构建成功。
随后将确认后的重组质粒pK18-△odhI以电穿孔法转化C. glutamicum ATCC13032感受态细胞,培养2 h后涂布于Kan抗性的BHI平板,30 ℃振荡培养36–48 h进行第一轮筛选,挑取转化子在Kan抗性BHI平板划线,30 ℃培养24 h后挑取单菌落于含有Kan (50 µg/mL) 的液体BHI培养基继续培养12 h,吸取100 μL培养液涂布于含有10%蔗糖的BHI无抗固体培养基上,进行第二轮筛选,随机挑取在蔗糖平板上生长的转化子进行PCR鉴定,筛选得到odhI缺失菌株C7,提取基因组后再次进行PCR验证并送至苏州金唯智生物科技有限公司测序以确认基因odhI被成功敲除。
其他基因敲除菌株C8、C9、C10、C11的构建与上述方法类似。将质粒pXMJ19-RphemA分别以电穿孔法转化入菌株C7-C11中,获得重组菌株CA7-CA11。
1.2.3 ALAS酶活检测ALAS酶活反应体系为100 mmol/L Tris-HCl (pH 7.5),200 mmol/L甘氨酸,0.2 mmol/L琥珀酰CoA,0.1 mmol/L磷酸吡哆醛和适量的粗酶。37 ℃温浴10 min后添加10% (V/V) 三氯乙酸终止反应,用Ehrlich’s试剂显色法(1 g对二甲氨基苯甲醛与8 mL的70%的高氯酸用冰乙酸定容至50 mL) 测定上清液5-ALA的浓度。以牛血清蛋白作为标准品,采用Bradford方法测定蛋白质浓度[19]。酶活则定义为:37 ℃下每分钟催化形成1 nmol 5-ALA的酶量为1 U。
1.2.4 摇瓶发酵验证将保存于-80 ℃的菌种在BHI平板上划线活化,从活化平板上挑取单菌落接入含有Chl (15 µg/mL) 抗性的10 mL BHI液体培养基,于180 r/min、30 ℃培养24 h,然后以1%的接种量转接入10 mL含有相同浓度抗生素的种子培养基培养,220 r/min、30 ℃过夜培养24 h,后以10%的接种量转接入50 mL发酵培养基(Chl浓度15 µg/mL) 发酵并且添加浓度为0.5 mmol/L诱导剂IPTG诱导表达。发酵控制温度为30 ℃,转速为220 r/min,并且以25%–30%的氨水维持发酵过程pH为6.5。转接之后补加10 mg/mL的甘氨酸进入发酵培养基。
1.2.5 代谢物检测方法5-ALA检测方法:400 μL稀释后的发酵液加入200 μL乙酸钠缓冲液(pH 4.6),然后加入100 μL乙酰丙酮,于沸水中煮沸15 min,冷却至室温后加700 μL Ehrlich’s试剂,显色20 min后于554 nm波长测定吸光度[20]。
葡萄糖分析方法:采用山东省科学院生产的SBA-40C生物传感分析仪进行检测。
有机酸的检测方法:高效液相色谱(HPLC)法检测琥珀酸,液相色谱仪为安捷伦1 260,色谱柱为BIO-RAD Aminex® HPX-87H Column 300 mm×7.8 mm,流动相为5 mmol/L H2SO4,流速为0.4 mL/min,柱温为65 ℃[21]。
1.2.6 通过核糖体结合位点工程优化ALAS核糖体结合位点(Ribosome-binding site,RBS) 工程可有效调节基因的表达水平。为进一步提高5-ALA的产量,本研究采用RBS工程策略调控ALAS的表达水平。筛选了4个较优的RBS序列(RBS2:5′-AAAGGAGGTTGTC-3′;RBS3:5′-GAAAGGAGA-3′;RBS5:5′-TCTTAATCATGC TGGGGAGGGTTTCTAATG-3′;RBS8:5′-AAAG GCTGGAATT-3′)[14]替代原始序列(5′-AAAGGAG GGAAATC-3′),以优化ALAS的表达水平。按照1.2.1的方法在RphemA基因前替换不同的RBS序列以构建不同的重组菌株CA3-CA6。再按照1.2.3和1.2.4的方法分别检测酶活并进行摇瓶水平验证。
使用引物RphemA-eamA-1和RphemA-eamA-2以质粒pXMJ19-RphemA为模板扩增RphemA基因片段;再使用引物eamA-F和eamA-R以E. coli BL21基因组为模板PCR扩增获得5′端含有Ptac启动子序列的eamA基因片段,将上述两个片段进行融合PCR后获得串联目的基因片段,按照1.2.1的方法构建重组菌株CA12。
1.2.7 sRNA的设计及ALAD酶活检测本研究中的sRNA结构包含H11F启动子、24 bp (hemB) mRNA靶向结合序列、骨架序列和转录终止序列4个元件[22],同时将E. coli来源的hfq蛋白与上述sRNA串联表达构建pXMJ19- sRNAhemB重组质粒来调控C. glutamicum hemB基因的表达[23],以促进5-ALA的产量。mRNA靶向结合序列经软件http://mfold.rna.albany.edu.进行脱靶率计算后生成。
使用引物p19-hfq-F1和p19-hfq-R1,以E. coli BL21基因组为模板扩增获得hfq基因,按照1.2.1的方法构建获得重组质粒pXMJ19-hfq;以重组质粒p19-hfq为模板使用引物p19-hfq-F1和end-p19-hfq-R扩增获得hfq-terminator序列(图 2 Fragment 1)。同时以合成的质粒p0为模板用引物p19-ssRNA (hemB)-F2和p19-ssRNA (hemB)-R2获得24 bp的(hemB) mRNA靶向结合序列、骨架序列以及转录终止序列;再以此目的片段为模板使用引物H11F-sRNAhemB-F3和p19-ssRNA (hemB)-R2进行扩增,在5′端添加H11F高强度启动子序列,得到图 2的Fragment 2。然后将上述两个片段进行重叠延伸PCR获得sRNA目的片段(图 2 Fragment 3),以Fragment 3为模板使用引物p19-hfq-H11F-miccF4和p19-hfq-H11F-miccR4扩增得到含同源臂的sRNA目的片段,同时用引物RBS5RphemA-hfq-H11F-micc-F5和RBS5RphemA- hfq-H11F-micc-R5以重组质粒pXMJ19-RBS5RphemA为模板扩增获得整个线性化质粒(图 2 Fragment 4)。最后将含同源臂的sRNA目的片段和pXMJ19-RBS5RphemA线性化质粒片段进行同源重组连接,将获得的产物使用化学转化法导入感受态细胞E. coli BL21,同方法1.2.1验证并确认成功后将菌株命名为BL21-pXMJ19-sRNAhemB- RBS5RphemA,将构建获得的质粒电转化C. glutamicum ATCC13032,获得验证正确的菌株即为CA15。使用引物hemB-F和hemB-R,以C. glutamicum ATCC13032基因组为模板扩增出hemB基因片段,同方法1.2.1构建pXMJ19-hemB质粒,将其电转化C. glutamicum ATCC13032获得菌株CA13。

|
| 图 2 重组菌株CA12、CA15和CA16构建过程 Fig. 2 The construction process of recombinant strains CA12, CA15 and CA16. |
| |
ALAD酶活的测定:反应体系(500 μL) 包含430 μL粗酶液,5 mmol/L 5-ALA,1 mmol/L MgCl2,50 μmol/L ZnCl2,50 mmol/L磷酸钾溶液。反应以加入终浓度5 mmol/L的5-ALA为起始。于37 ℃振荡反应10 min后,加入等体积的10% (V/V) 三氯乙酸溶液终止反应。8 000 r/min离心10 min,取上清液至干净的离心管,与等体积改良的Ehrlich’s试剂混合,静置25 min后显色稳定,于554 nm测得其吸光值,对照标准曲线计算对应产物PBG的含量[24]。
酶活定义:37 ℃条件下,每小时产生1 nmol PBG所需的酶量定义为一个酶活力单位(1 U)。
1.2.8 5 L发酵罐发酵条件使用引物P19-RpeA-F和P19-RpeA-R,以质粒pXMJ19-sRNAhemB-RBS5RphemA为模板反向PCR扩增获得线性化质粒片段,再用引物Ptac-eamA-F和Ptac-eamA-R以菌株CA12的重组质粒为模板扩增得到Ptac-eamA片段,将2个基因片段使用同源重组酶克隆试剂盒进行连接,将连接产物用化学热激转化法转化E. coli BL21感受态细胞,验证成功构建重组菌株BL21/pXMJ19-sRNAhemB- RBS5RphemA-eamA之后,对其进行培养并取菌液提取质粒pXMJ19-sRNAhemB-RBS5RphemA-eamA,电穿孔法转化基因敲除菌株C11,获得重组菌株CA16。将活化的菌株接入20 mL种子培养基,而后转入发酵培养基,30 ℃、180 r/min振荡培养20–22 h之后,将发酵液转入含有2 L发酵培养基的5 L发酵罐,30 ℃发酵,OD600为40左右时加入终浓度为0.1 mmol/L IPTG诱导,并加入终浓度为4 g/L的甘氨酸溶液,于30 ℃继续发酵72 h。具体发酵条件为:发酵前期控制30%溶氧,用25%的氨水维持pH 6.5,24 h后控制10%的溶氧,同时调整发酵罐pH使其维持在6.0。发酵过程流加200 g/L的甘氨酸和800 g/L的葡萄糖溶液。
2 结果与分析 2.1 ALAS重组菌株的构建与优化 2.1.1 不同来源的ALAS重组菌株的构建按照1.2.1的方法在C. glutamicum ATCC13032中过表达RchemA和RphemA基因,构建5-ALA生产菌株。首先在基因合成完成后通过PCR扩增获得hemA基因片段,PCR结果如图 3A和3B所示,RchemA基因片段长度为1 203 bp,RphemA基因长度为1 230 bp,目的条带大小与预期一致。PCR产物与载体pXMJ19连接转化获得转化子,并将菌落PCR验证正确的转化子提取质粒送样测序,最终确认成功构建重组质粒pXMJ19-RchemA和pXMJ19-RphemA。分别将上述重组质粒以电穿孔法转化C. glutamicum ATCC13032,挑取转化子菌落PCR验证结果如图 3C所示,送样测序验证,最终确认构建成功的菌株命名为CA1和CA2。
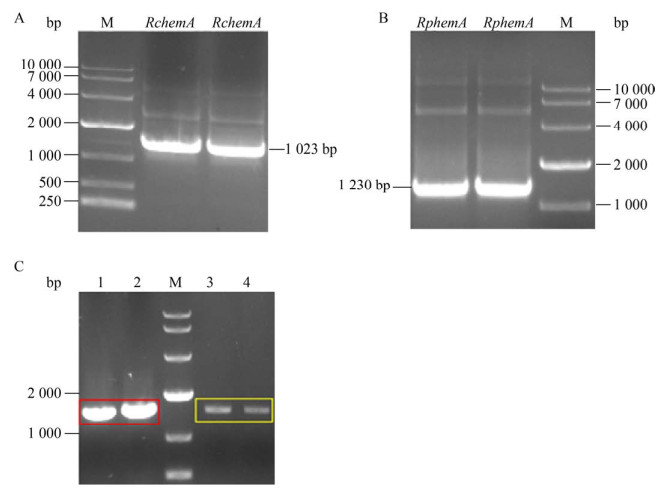
|
| 图 3 RchemA和RphemA基因的克隆(A, B) 和在CA中转化子的菌落PCR验证图(C) Fig. 3 The amplification of RchemA and RphemA gene (A, B) and colony PCR verification in C. glutamicum ATCC13032 (C). 1–2: CA1; M: DL10 000 marker; 3–4: CA2. |
| |
按照1.2.3的方法检测重组菌株CA0、CA1和CA2的酶活,结果如表 4所示,相同条件下菌株CA2比酶活可达(158.80±4.08) U/mg,有较高的酶活力。进一步进行摇瓶发酵验证结果如图 4所示,菌株CA1在66 h的5-ALA积累量最高为7.10 g/L,而CA2菌株的最大产量在66 h为7.58 g/L,相比于CA1高6.76%。在66 h,CA2也相对消耗更多的葡萄糖,剩余含量为80 g/L。而底物葡萄糖的添加量是140 g/L,即CA2在66 h内的单位糖耗产5-ALA的量为0.12 g/h。
| Recombinant strains | Specific ALAS activity (U/mg) |
| CA0 | 9.13±0.32 |
| CA1 | 131.68±2.34 |
| CA2 | 158.80±4.08 |
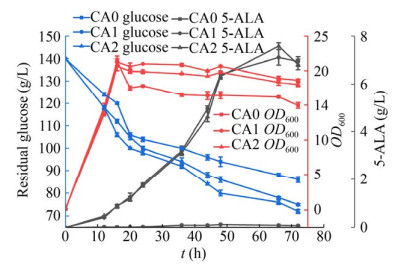
|
| 图 4 不同菌株从葡萄糖通过一步发酵生产5-ALA Fig. 4 One-step fermentative production of 5-ALA from glucose using strains CA0, CA1 and CA2. |
| |
综上,我们选择RphemA进行深入的研究,以提高生产效率并生产更多的5-ALA。
2.1.2 通过RBS工程优化ALAS表达按照1.2.6的方法构建菌株,获得4株不同的重组菌株CA3、CA4、CA5、CA6,并测定相应的ALAS酶活,结果如表 5所示,CA3、CA5、CA6三个菌株在粗提物中表现出更高的ALAS活性,菌株CA5活性最高,比酶活为(221.87±3.10) U/mg,比对照CA2菌株(158.80±4.08) U/mg提高39.71%;然而CA4菌株的比酶活较对照菌株低16.19%。同时对上述菌株进行摇瓶验证,结果如图 5所示,菌株首先主要消耗葡萄糖用于生长,在20 h左右细胞OD600值达到最高,之后葡萄糖的消耗速率下降,5-ALA也逐渐开始加速积累。如图 5B所示,CA5摇瓶发酵5-ALA积累量最高,可达9.47 g/L,比原始菌株CA2提高了24.93%。这表明核糖体结合位点(RBS) 工程可优化ALAS表达,且RBS5显示出了较高的ALAS表达水平,促进5-ALA的合成。
| Recombinant strains | Specific ALAS activity (U/mg) |
| CA2 | 158.80±4.08 |
| CA3 | 177.68±2.34 |
| CA4 | 133.09±1.10 |
| CA5 | 221.87±3.10 |
| CA6 | 186.80±1.78 |
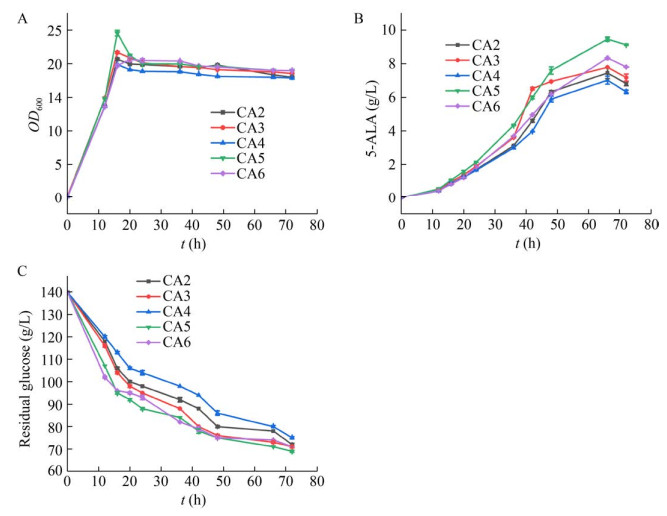
|
| 图 5 不同RBS序列重组菌株的发酵过程性能参数 Fig. 5 Fermentation profiles of recombinant strains with different RBS sequences. (A) OD600. (B) 5-ALA production. (C) Residual glucose. |
| |
按照1.2.2的方法构建敲除菌株,分别得到敲除α-酮戊二酸(α-KG) 脱氢酶抑制蛋白(odhI) 基因,敲除NADP特异性谷氨酸脱氢酶(gdhA) 基因,敲除琥珀酸脱氢酶(sdhA) 基因的菌株C7、C8、C9以及同时敲除odhI和gdhA两个基因的菌株C10和同时敲除odhI和sdhA的菌株C11。再按照1.2.1的方法分别在上述菌株中过表达基因RphemA,获得菌株CA7、CA8、CA9、CA10和CA11。对CA7、CA8、CA10三株菌株进行摇瓶发酵培养,结果如图 6所示,与对照菌株CA2相比,菌株CA7和CA8的菌体生长和5-ALA产量基本没有差异。而在菌株C10中过表达基因RphemA后,CA10菌株消耗葡萄糖的能力明显降低,生长受到抑制。推测α-KG途径的狭窄流量阻止了琥珀酰CoA的积累,双敲除gdhA和odhI后严重影响了TCA循环。菌株CA9生长较对照菌株CA2缓慢,5-ALA积累也较低,最高产量为2.07 g/L。而菌株CA11葡萄糖代谢相对较快,OD600值略高,生长良好,5-ALA积累也最多,66 h时产量达9.94 g/L,较对照菌株CA2提高了33.42%,这表明同时敲除sdhA和odhI基因能够有效增加5-ALA的积累。
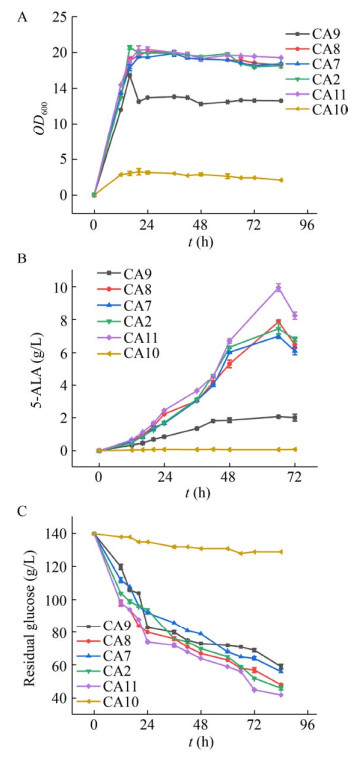
|
| 图 6 基因敲除突变株的发酵过程性能参数 Fig. 6 Fermentation profiles of gene knockout mutants. (A) OD600. (B) 5-ALA production. (C) Residual glucose. |
| |
我们构建了重组C. glutamicum工程菌株CA15且检测了部分重组C. glutamicum ALAD比活力的大小,结果如表 6所示,菌株CA13的ALAD相对酶活为9.46×103 U/mg,菌株CA5比酶活为687.42 U/mg,而菌株CA15通过sRNA弱化hemB基因后ALAD比酶活明显下降,这表明以sRNA抑制可有地效减弱hemB基因的表达。菌株CA5、CA13、CA15的摇瓶发酵结果如图 7所示,CA13几乎不产5-ALA,菌株CA15的5-ALA产量较CA5略高,65 h产量最高达9.73 g/L,表明sRNA弱化hemB基因表达可促进5-ALA合成积累。
| Recombinant strains | PBG concentration (mg/L) |
Specific ALAD activity (U/mg) |
| CA0 | 0.49±0.020 | 146.90±2.80 |
| CA13 | 7.52±1.340 | 9 469.00±780.00 |
| CA5 | 2.09±0.250 | 687.42±4.24 |
| CA15 | 0.11±0.005 | 8.35±0.32 |
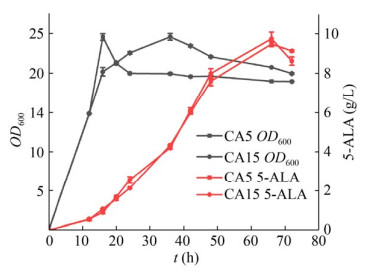
|
| 图 7 sRNA抑制hemB表达菌株发酵生产5-ALA Fig. 7 The yield of 5-ALA by strain where hemB expression was inhibited by sRNA. |
| |
通过改善5-ALA转运水平降低胞内5-ALA含量。E. coli中苏氨酸和高丝氨酸外排系统rhtA转运蛋白的过表达可加速C. glutamicum中5-ALA的转运[21]。张良程证明半胱氨酸/O-乙酰丝氨酸转运蛋白eamA是5-ALA的向外转运蛋白,过表达E. coli来源的eamA使得胞外5-ALA提高了25.43%[25]。因此本文通过构建重组菌株CA12进一步增加了5-ALA的积累。
将菌株CA2和CA12同时进行摇瓶发酵,结果如图 8所示,发酵72 h 5-ALA的最大产量从7.58 g/L提高到9.56 g/L,较对照菌株CA2提高了26.12%,这表明将5-ALA及时输送至胞外也能有效提高其胞外积累。
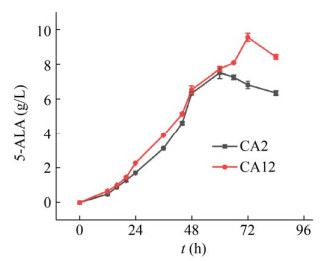
|
| 图 8 异源表达外排蛋白eamA重组菌株5-ALA产量 Fig. 8 The yield of 5-ALA in recombinant strain with heterologous expression of efflux protein eamA. |
| |
经上述代谢改造策略进行组合后获得重组菌株CA16,进行摇瓶发酵,结果如图 9所示,在72 h 5-ALA产量最高为11.90 g/L,相比于原始菌株CA2,产量得到明显提高。

|
| 图 9 重组菌株CA16的5-ALA产量 Fig. 9 Production of 5-ALA in recombinant strain CA16. |
| |
将工程菌CA16进行补料分批发酵,分别检测菌株OD600、残糖量和5-ALA的含量,结果如图 10所示,发酵前30 h内菌株迅速增长,菌株最大生物量出现在30 h,OD600值达146.80。之后细胞生物量开始降低,细胞进入稳定期,不断积累5-ALA。在48 h获得最高产量为25.05 g/L,之后由于酶活力下降以及5-ALA发生降解或其他代谢而使得5-ALA产量减少。
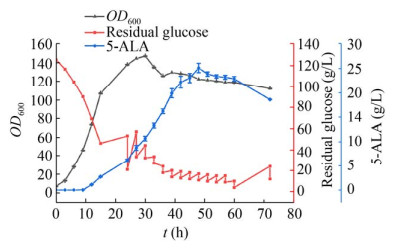
|
| 图 10 在5 L发酵罐中用于5-ALA生产的菌株CA16的补料分批发酵 Fig. 10 Fed-batch fermentation profile of strain CA16 for 5-ALA production in a 5 L fermentation tank. |
| |
5-氨基乙酰丙酸广泛应用于农业、食品、生物医药等行业,有着巨大的应用前景。
本研究通过系统代谢工程构建高产5-ALA重组C. glutamicum工程菌株。研究首先在C. glutamicum ATCC13032中异源表达R. palustris来源的高酶活ALAS基因hemA,并进行了后续深入的代谢研究。也筛选到RBS5序列优化了RphemA的表达,提高了5-ALA的合成产量。这说明筛选高效的ALAS有利于5-ALA的积累。
本研究采用提高底物积聚,平衡代谢流量的策略进一步提高5-ALA产量。敲除α-酮戊二酸脱氢酶抑制蛋白(odhI) 和琥珀酸脱氢酶编码基因sdhA可增加从琥珀酸向琥珀酰CoA方向的转换,增加前体琥珀酰CoA的积累。敲除sdhA和odhI后细胞生长得以恢复,5-ALA积累量增加,odhI和sdhA缺失菌株生长和产物合成得到更好地平衡。由于暂时缺失相关内容的深入研究和分析,我们推测敲除sdhA导致了更多的琥珀酸富集,促使琥珀酸与琥珀酰CoA的可逆反应偏向生成琥珀酰CoA的方向进行[26],而敲除odhI也增加了琥珀酰CoA积累量,进而促进了5-ALA合成。另外5-ALA由胞内分泌到胞外对提高5-ALA的积累量至关重要。结果表明,E. coli来源的eamA能够有效增加C. glutamicum工程菌中5-ALA的输出。sRNA抑制hemB活性也可有效缓解下游中间体和最终产物血红素引起的反馈抑制,从而增强ALA的积累[6]。
重组菌株CA16通过5 L发酵罐补料分批发酵,在流加甘氨酸的情况下发酵48 h合成了25.05 g/L的5-ALA,生产率为0.52 g/(L·h),糖酸转化率达16.79%,这是以葡萄糖为碳源的一步发酵5-ALA的最高产量,与此前报道的在C. glutamicum C4途径中以葡萄糖为碳源获得的最高产量16.30 g/L相比[14],提高了53.68%,未来继续研究在提高产量的同时也需要提高生产率,减少碳源的消耗。综上所述,本文所构建C4途径的高产5-ALA工程菌C. glutamicum为5-ALA的工业化生产进一步奠定了理论和实验基础,具有较大的影响力和发展潜力。
| [1] |
Li JM, Brathwaite O, Cosloy SD, et al. 5-aminolevulinic acid synthesis in Escherichia coli. J Bacteriol, 1989, 171(5): 2547-2552. DOI:10.1128/jb.171.5.2547-2552.1989
|
| [2] |
Inoue K. 5-aminolevulinic acid-mediated photodynamic therapy for bladder cancer. Int J Urol, 2017, 24(2): 97-101. DOI:10.1111/iju.13291
|
| [3] |
Akram NA, Iqbal M, Muhammad A, et al. Aminolevulinic acid and nitric oxide regulate oxidative defense and secondary metabolisms in canola (Brassica napus L.) under drought stress. Protoplasma, 2018, 255(1): 163-174. DOI:10.1007/s00709-017-1140-x
|
| [4] |
Yu XL, Jin HY, Liu WJ, et al. Engineering Corynebacterium glutamicum to produce 5-aminolevulinic acid from glucose. Microb Cell Fact, 2015, 14: 183. DOI:10.1186/s12934-015-0364-8
|
| [5] |
Hara KY, Saito M, Kato H, et al. 5-aminolevulinic acid fermentation using engineered Saccharomyces cerevisiae. Microb Cell Fact, 2019, 18(1): 194. DOI:10.1186/s12934-019-1242-6
|
| [6] |
Ding WW, Weng HJ, Du GC, et al. 5-aminolevulinic acid production from inexpensive glucose by engineering the C4 pathway in Escherichia coli. J Ind Microbiol Biot, 2017, 44(8): 1127-1135. DOI:10.1007/s10295-017-1940-1
|
| [7] |
Yang P, Liu W, Cheng X, et al. A new strategy for production of 5-aminolevulinic acid in recombinant Corynebacterium glutamicum with high yield. Appl Environ Microbiol, 2016, 82(9): 2709-2717. DOI:10.1128/AEM.00224-16
|
| [8] |
Tran NT, Pham DN, Kim CJ. Production of 5-aminolevulinic acid by recombinant Streptomyces coelicolor expressing hemA from Rhodobacter sphaeroides. Biotechnol Bioproc E, 2019, 24(3): 488-499. DOI:10.1007/s12257-018-0484-1
|
| [9] |
Zhang J, Weng H, Zhou Z, et al. Engineering of multiple modular pathways for high-yield production of 5-aminolevulinic acid in Escherichia coli. Bioresour Technol, 2019, 274: 353-360. DOI:10.1016/j.biortech.2018.12.004
|
| [10] |
Woodard SI, Dailey HA. Regulation of heme biosynthesis in Escherichia coli. Arch Biochem Biophys, 1995, 316(1): 110-115. DOI:10.1006/abbi.1995.1016
|
| [11] |
Kang Z, Wang Y, Gu P, et al. Engineering Escherichia coli for efficient production of 5-aminolevulinic acid from glucose. Metab Eng, 2011, 13(5): 492-498. DOI:10.1016/j.ymben.2011.05.003
|
| [12] |
Liu XX, Wang L, Wang YJ, et al. D-glucose enhanced 5-aminolevulinic acid production in recombinant Escherichia coli culture. Appl Biochem Biotech, 2010, 160(3): 822-830. DOI:10.1007/s12010-009-8608-x
|
| [13] |
Lin J, Fu W, Cen P. Characterization of 5-aminolevulinate synthase from Agrobacterium radiobacter, screening new inhibitors for 5-aminolevulinate dehydratase from Escherichia coli and their potential use for high 5-aminolevulinate production. Bioresour Technol, 2009, 100(7): 2293-2297. DOI:10.1016/j.biortech.2008.11.008
|
| [14] |
Chen J, Wang Y, Guo X, et al. Efficient bioproduction of 5-aminolevulinic acid, a promising biostimulant and nutrient, from renewable bioresources by engineered Corynebacterium glutamicum. Biotechnol Biofuels, 2020, 13: 41. DOI:10.1186/s13068-020-01685-0
|
| [15] |
Zhang JL, Kang Z, Chen J, et al. Optimization of the heme biosynthesis pathway for the production of 5-aminolevulinic acid in Escherichia coli. Sci Rep, 2015, 5: 8584. DOI:10.1038/srep08584
|
| [16] |
陈丹园, 沈云杰, 杨燕, 等. 关键酶基因的过表达与环境因素对大肠杆菌血红素合成的调控. 食品与发酵工业, 2018, 44(11): 7-14. Chen DY, Shen YJ, Yang Y, et al. Regulation of heme synthesis in Escherichia coli by overexpression of genes for the key enzymes and environmental factors. Food Ferment Ind, 2018, 44(11): 7-14 (in Chinese). |
| [17] |
Sun D, Chen J, Wang Y, et al. Metabolic engineering of Corynebacterium glutamicum by synthetic small regulatory RNAs. J Ind Microbiol Biot, 2019, 46(2): 203-208. DOI:10.1007/s10295-018-02128-4
|
| [18] |
Gibson DG. Enzymatic assembly of overlapping DNA fragments. Methods Enzymol, 2011, 498: 349-361.
|
| [19] |
Bradford M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Anal Biochem, 1976, 72: 248-254. DOI:10.1016/0003-2697(76)90527-3
|
| [20] |
Li F, Wang Y, Gong K, et al. Constitutive expression of RyhB regulates the heme biosynthesis pathway and increases the 5-aminolevulinic acid accumulation in Escherichia coli. FEMS Microbiol Lett, 2014, 350(2): 209-215. DOI:10.1111/1574-6968.12322
|
| [21] |
Feng LL, Zhang Y, Fu J, et al. Metabolic engineering of Corynebacterium glutamicum for efficient production of 5-aminolevulinic acid. Biotechnol Bioeng, 2016, 113(6): 1284-1293. DOI:10.1002/bit.25886
|
| [22] |
Yoo SM, Na D, Lee SY. Design and use of synthetic regulatory small RNAs to control gene expression in Escherichia coli. Nat Protoc, 2013, 8(9): 1694-1707. DOI:10.1038/nprot.2013.105
|
| [23] |
Aiba H. Mechanism of RNA silencing by Hfq-binding small RNAs. Curr Opin Microbiol, 2007, 10(2): 134-139. DOI:10.1016/j.mib.2007.03.010
|
| [24] |
杨佳倩. 5-氨基乙酰丙酸脱水酶的克隆表达与酶学性质的初步研究[D]. 杭州: 浙江大学, 2008. Yang JQ. Cloning and expression of 5-aminolevulinate dehydratase from Escherichia coli Rosetta(DE3) and primary study on its enzymatic properties[D]. Hangzhou: Zhejiang University, 2008(in Chinese). |
| [25] |
张良程. 大肠杆菌中5-氨基乙酰丙酸转运蛋白的研究[D]. 天津: 天津科技大学, 2016. Zhang LC. Study on 5-aminolevulinic acid transporters in Escherichia coli[D]. Tianjin: Tianjin University of Science & Technology, 2016(in Chinese). |
| [26] |
蒲伟, 陈久洲, 孙村民, 等. 琥珀酸脱氢酶或琥珀酰辅酶A合成酶缺失促进大肠杆菌积累5-氨基乙酰丙酸. 生物工程学报, 2013, 29(10): 1494-1503. Pu W, Chen JZ, Sun CM, et al. Deficiency of succinic dehydrogenase or succinyl-CoA synthetase enhances the production of 5-aminolevulinic acid in recombinant Escherichia coli. Chin J Biotech, 2013, 29(10): 1494-1503 (in Chinese). |
 2021, Vol. 37
2021, Vol. 37




