中国科学院微生物研究所、中国微生物学会主办
文章信息
- 陈淑萍, 杨晗, 蒋金露, 于思远, 李廷栋, 葛胜祥
- Chen Shuping, Yang Han, Jiang Jinlu, Yu Siyuan, Li Tingdong, Ge Shengxiang
- 靶向蛋白降解技术及其在疾病治疗中的研究进展
- Advances of targeted protein degradation technology and its applications in diseases therapy
- 生物工程学报, 2021, 37(11): 3915-3932
- Chinese Journal of Biotechnology, 2021, 37(11): 3915-3932
- 10.13345/j.cjb.200816
-
文章历史
- Received: December 23, 2020
- Accepted: March 12, 2021
- Published: April 14, 2021
2. 厦门大学 公共卫生学院 厦门大学分子疫苗学和分子诊断学国家重点实验室,福建 厦门 361102;
3. 医用生物制品省部共建协同创新中心,福建 厦门 361102
2. State Key Laboratory of Molecular Vaccinology and Molecular Diagnostics, School of Public Health, Xiamen University, Xiamen 361102, Fujian, China;
3. Collaborative Innovation Centers of Biological Products, Xiamen University, Xiamen 361102, Fujian, China
对致病蛋白的降解或干预已经成为目前最有效的疾病治疗策略之一[1]。传统的小分子抑制剂通过结合目标蛋白(Protein of interest,POI) 的活性位点来影响致病蛋白的生物学功能,从而达到治疗效果,至今已被成功用于癌症等多种疾病的临床治疗中[2]。然而,传统小分子药物的研发策略决定其只能针对含有“可容纳其结合的且具有明确功能”位点的蛋白如激酶、离子通道等,但对于“无成药性” (Undruggable) 的靶点蛋白如转录因子、支架蛋白却无计可施。据研究报道,目前细胞内仅有20%左右的蛋白可以作为靶点被应用于小分子药物的临床研究中[3]。与此同时,疾病相关蛋白表达水平的操控也可以在其编码基因和mRNA水平上实现。通过基因编辑技术如CRISPR (Clustered regularly interspaced short palindromic repeats)/Cas (CRISPR-associated) 系统敲除、修复致病蛋白基因或者利用RNAi技术影响其mRNA水平的方法[4-5],均可以实现对目标蛋白的调控。尽管CRISPR技术已经进入临床试验阶段,而第一种RNAi药物Onpattro也于2017年被食品药品监督管理局(Food and Drug Administration,FDA) 批准用于由遗传性甲状腺素介导的淀粉样变性引起的多发性神经疾病的治疗[6],但是它们仍然存在着如无法作用于存在翻译后修饰的功能异常蛋白以及脱靶效应较强等在内的诸多局限性[7]。为突破现有药物研发技术的诸多瓶颈,靶向蛋白降解(Targeted protein degradation,TPD) 技术应运而生,并在近些年获得突飞猛进的发展,成为极有潜力的新药开发技术之一[8-9]。
1 靶向蛋白降解技术TPD技术利用细胞内天然存在的蛋白降解系统实现对疾病相关蛋白的特异、高效降解,从而达到疾病治疗的效果[10-11]。相较于小分子抑制剂等其他治疗策略,靶向蛋白降解技术凭借如下诸多优势成为未来药物研发的主要方向之一:1) 作用靶点丰富,TPD不依赖于传统小分子抑制剂“占有驱动”的作用方式,而是采用“事件驱动”方式,使其作为靶点的蛋白限制更少[12];2) 作用速度更快,TPD通常会在几分钟到几小时内快速耗尽目标蛋白,而不是操纵mRNA或基因组DNA方法所需的几天或几周;3) 特异性更高,致病变体蛋白与正常蛋白在空间结构存在差异,TPD药物可以基于此结构差异,在不影响正常蛋白功能的前提下,选择性结合并引起某种致病变体蛋白的降解,而CRISPR及RNAi技术的作用则会影响蛋白所有变体的功能[13]。
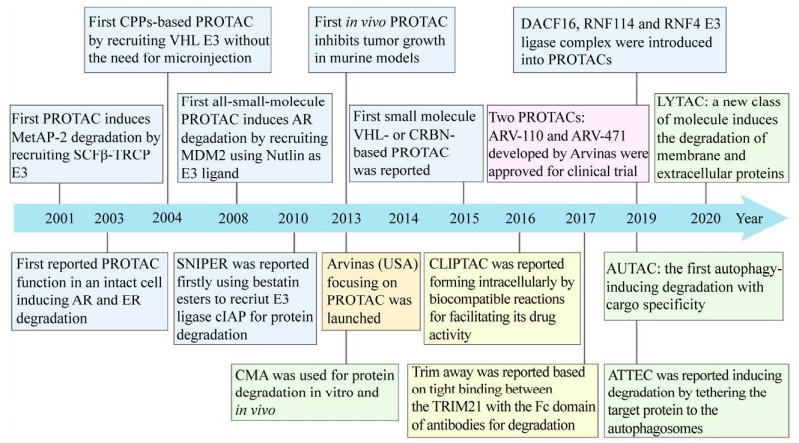
Proteinix公司研究人员于1999年递交了基于泛素机制使用小分子化合物降解特定蛋白的专利申请[14],这标志着TPD技术概念的初步形成。2008年基于TPD技术的第一种小分子药物的研究报道则标志着这一新兴药物研发领域冉冉升起,自此TPD技术便开始受到众多学术机构、大型药企以及投资机构的青睐。Arvina、C4 Therapeutics、Kymera Therapeutics (KT)、Vividion Therapeutics、Warp Drive Bio和Cullgen等生物技术公司正在如火如荼地开展蛋白质降解新药的研发,国际大型制药企业如基因泰克、辉瑞、Atlas、诺华、默沙东等也纷纷进军该领域,国内制药公司如凌科制药、成都分迪科技等也形成了我国在该领域的第一梯队企业。2019年用于治疗前列腺癌(ARV-110) 和乳腺癌(ARV-471) 的TPD药物相继进入临床阶段成为该技术领域里程碑式的事件(图 1)。在同年发表于Nature杂志的新闻评论中,作者Asher Mullard则认为“2019年到2021年将是蛋白靶向降解领域爆发的时期 (2019 through 2021 will be big years for this space)”[15]。因此,考虑到基于TPD技术的降解方法种类繁多,目前也无较为系统的分类,并且在近些年有大量相关研究成果产生,文中将对TPD技术相关的各种降解系统在疾病治疗中的研究现状以及未来的研究方向进行系统性阐述。
|
| 图 1 TPD技术发展过程中的代表性事件(2001–2020) Fig. 1 Milestones in the development of TPD technology (From 2001 to 2020). |
| |
细胞内的蛋白质处于不断的合成和降解过程中,泛素化-蛋白酶体系统(Ubiquitin-proteasome system,UPS) 与溶酶体降解途径是真核细胞内负责降解蛋白的两种主要方式。UPS主要负责错误折叠和受损蛋白的降解,更主要的是调控许多关键细胞过程的蛋白,而溶酶体途径主要是在应激状态下降解胞内蛋白。相应地,TPD技术则分别基于这两种降解途径发展起来。
2 基于UPS的靶向蛋白降解系统蛋白质被UPS降解涉及一种三级酶促联反应(E1-E2-E3)[16]。首先,基于ATP酶促反应,泛素激活酶E1结合并激活泛素,紧接着,E1将激活的泛素转移至泛素偶联酶E2上。泛素连接酶E3则可以同时结合至蛋白底物以及带有泛素的E2,并将泛素由E2转移至蛋白底物的赖氨酸残基上,而这一系列反应的多轮循环则会在底物上形成多聚泛素链。最终,带有多聚泛素链的蛋白底物从E3复合物释放,进而被26S蛋白酶体识别及降解。基于UPS的靶向蛋白降解系统正是通过“劫持”细胞内的正常运行的泛素化过程来降解目标蛋白(图 2)。
|
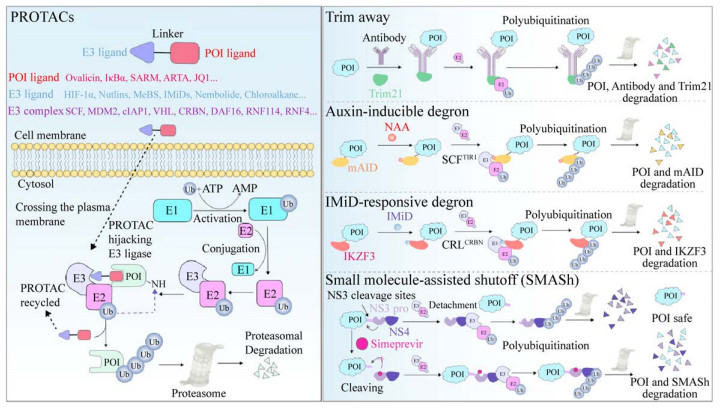
| 图 2 PROTACs和其他基于UPS的TPD技术作用机制 Fig. 2 Schematic overview of mechanisms of PROTACs and other UPS-based TPD technologies. |
| |
在基于UPS的降解方法中,蛋白水解靶向嵌合体(Proteolysis targeting chimeras,PROTACs)是研究最为成熟的系统。PROTAC是一种双官能团分子,包括用于结合目标蛋白的配体(POI ligand) 和能够招募E3复合物的配体(E3 ligand),以及将二者相连的连接子。PROTAC通过连接子连接两端配体来拉近POI与E3的距离,从而促进E3对POI的泛素化修饰[10-11]。当PROTACs进入细胞质后,其双功能团分别与POI、E3结合形成三元复合物,被E3配体招募来的E3复合物引起目标蛋白的泛素化,同时PROTAC分离。最终,多聚泛素化修饰的POI被蛋白酶体识别并降解,而脱离的PROTAC进入下一个降解循环(图 2)。
2.1.1 多肽形式的PROTACs(1) PROTAC概念初步形成
2001年耶鲁大学的Crews团队和加州理工大学的Deshaies团队首次报道了PROTAC技术(图 1),他们设计的PROTAC-1利用IκBα磷酸肽招募Skp1-Cullin-F boxβ-TRCP (SCFβ-TRCP) E3泛素连接酶来诱导甲硫氨酰氨肽酶-2 (Methionine aminopeptidase-2,METAP-2) 的降解[14]。在该研究中,靶向METAP-2的血管生成抑制剂Ovalicin与IκBα磷酸肽共价结合形成的PROTAC-1能够在胞外引起METAP-2的快速降解。但正如Crews所说的“PROTAC-1远远称不上一种药物,只能算一次科研尝试”。在胞外实验被证明有效后,这个开创性工作继续被推进,2003年Sakamoto等尝试对PROTACs在细胞水平的有效性进行验证。雌激素受体(Estrogen receptor,ER) 与雄激素受体(Androgen receptor,AR) 在乳腺癌和前列腺癌发生发展过程中起到关键作用,因此成为恶性肿瘤治疗中重要的药物靶标[17]。值得注意的是,这二者成为后来Arvinas公司进入临床的PROTAC药物的靶标。在该项研究中,Crews团队和Deshaies团队将PROTAC-1靶向METAP-2的Ovalicin分别更换成结合ER的雌二醇(Estradiol,E2) 与结合AR的二羟睾酮(Dihydroxytestosterone,DHT)。胞外结果显示PROTACs能够有效诱导AR和ER的降解,这也说明通过简单更换蛋白配体即可实现PROTACs对不同类型蛋白的降解[18]。更令人惊喜的是通过显微注射PROTACs到过表达融合绿色荧光蛋白的AR (AR-GFP) 的HEK- 293T细胞系内,Crews等发现PROTACs能够在细胞内发挥降解AR-GFP的效果,这也是关于PROTACs胞内有效性的首次报道。
(2) 基于细胞穿膜肽的PROTACs
细胞生物膜具有选择通过性的特征,而多肽形式的E3配体会导致PROTACs分子量大,无法自由穿过细胞膜,之前的胞内验证实验也仅能通过显微注射方式进行,这使得PROTACs药物的应用潜力大打折扣。利用细胞穿膜肽(Cell- penetrating peptides,CPPs) 介导生物大分子药物入胞是目前生物医药领域的研究热点之一[19]。Schneekloth等通过在PROTAC的连接子处融合多聚赖氨酸穿膜肽赋予其细胞通透性,并在细胞水平上验证了该分子能够自发进入细胞内并降解目标蛋白[20]。值得注意的是,在该研究中E3配体选用的是低氧诱导因子1α (Hypoxia-inducible factor 1α,HIF-1α) 中可被E3识别的最短肽段,它能够招募另一种E3连接酶复合物VBC-Cul2中von Hippel-Lindau (VHL) 蛋白,VHL成为后续PROTAC应用中最常用到的E3复合物之一[21]。同时,目标蛋白配体部分由小分子AP21998构成,它靶向的是与各类心血管疾病有关的FK506结合蛋白(FK506 binding protein 12 kDa,FKBP12) 的F36V突变体(FKBP12F36V) 的。但直至2013年,Hines等在小鼠模型中证实基于穿膜肽的PhosphoPROTACs分子能够通过靶向降解激酶磷脂酰肌醇-3激酶(Phosphatidylinositol 3 kinase,PI3K) 从而抑制肿瘤细胞的生长,这也成为关于PROTACs体内治疗效果的首个报道[22]。尽管CPPs在一定程度上可以促进多肽PROTACs入胞发挥作用,但CPPs体系本身也存在递送效果差的问题,因此建立高效的CPP系统将成为多肽PROTACs发展的迫切任务之一。
2.1.2 小分子形式PROTACs尽管多肽PROTACs生物相容性高、体内毒性低,即拥有安全性更高的巨大优势,但却面临着细胞通透性差、稳定性弱以及大分子量和脆弱肽键带来的合成纯化难度大等问题[23],这一系列因素都限制着多肽PROTACs作为新型治疗药物的潜力。相比之下,小分子PROTACs更容易被人体吸收,使其有希望被开发成药物。此外,小分子蛋白配体部分可以利用已获批的临床小分子药物充当,这在很大程度上减少了研发周期和研发成本,换句话说,寻找合适的小分子形式的E3配体成为PROTACs领域最为迫切的需求[24]。
(1) 首个完全小分子形式的PROTAC
小鼠双微体(Mouse double minute,MDM) E3连接酶家族在肿瘤发生、发展中具有重要作用,尤其是在p53的功能调节中扮演着重要角色,而咪唑啉衍生物Nutlins凭借其与MDM2的高亲和力、能够破坏MDM2-p53之间结合的特点,常被用于癌症的临床治疗[25]。2008年,Smith等创造性地将Nutlins与靶向AR的小分子Selective androgen receptor modular (SARM) 相连合成了首个完全小分子形式的PROTAC (图 1)[26],最终胞内实验证实其能够有效降解AR蛋白。至此,科学界才开始普遍认为PROTACs技术或许能真正被开发成为药物。但是,SARM-Nutlins的有效剂量非常高,只有毫摩尔级别才能够有效降解目标蛋白[26]。因此,筛选能够高效招募其他E3复合物的小分子配体成为之后PROTAC技术发展的重要突破口。
(2) 基于IAPs E3复合物招募的小分子PROTACs
在发现乌苯美司酯(Methylbestatin,MeBS)能够结合凋亡抑制蛋白(Inhibitor of apoptosis proteins,IAPs) E3家族中的细胞凋亡抑制蛋白1 (Cellular inhibitor of apoptosis protein 1,cIAP1) 之后,Itoh等于2010年合成了首个招募cIAP-1 E3连接酶的PROTAC,并将其命名为SNIPER (Specific and non-genetic inhibitor-of- apoptosis proteins- dependent protein eraser)[27]。SNIPER通过全反式维甲酸(All-trans retinoic acid,ARTA) 靶向细胞视黄醇和视黄酸结合蛋白(Cellular retinoic acid binding proteins-Ⅰ/Ⅱ,CRABP-Ⅰ/Ⅱ)[27],这些蛋白在急性早幼粒性白血病和神经母细胞瘤中有重要作用。在随后的细胞实验中,SNIPER能够诱导神经母细胞瘤细胞IMR-32内CRABP-Ⅰ/Ⅱ的快速降解,而且SNIPER处理过的IMR-32其迁移速度明显变慢。随后,MeBS的衍生物MV1、LCL161相继被用作IAP E3的配体,并且表现出不同的底物特异性[28]。至今,SNIPER技术已被成功用于多种与疾病相关蛋白的降解,如ER[29]、AR[30]、溴结构域包含蛋白(Bromodomain-containing protein,BRD)[29]以及TACC3 (Transforming acidic coiled-coil-3)[31]。然而由于基于IAP的PROTAC更多只能在微摩尔范围内起效,而且在诱导目标蛋白降解的同时也会引起IAP本身的降解,因此用作药物治疗可能存在较大风险,目前的研究也仅仅停留在小鼠水平[32]。
(3) 基于VHL E3招募的小分子PROTACs
基于VHL E3的PROTAC的研究工作主要集中在为HIF-1α多肽片段寻找合适的小分子替代物,这个工作于2012年取得了重要进展,Crews团队和Ciulli团队基于生物信息学的方法筛选到了第一种能够和VHL E3结合的小分子配体,而且二者之间的亲和力远远高于HIF-1α[33]。在此基础上他们甚至得到一种解离常数(Kd) 约为185 nmol/L的小分子配体[34-35]。利用该小分子配体,2015年Bondeson等便设计并合成了首个基于VHL E3招募的小分子PROTACs,实验结果表明,其在纳摩尔范围内即可降解掉几乎所有的目标蛋白(> 90%),而小鼠体内结果也显示,PROTAC-ERRα能够导致约40%左右雌激素相关受体α (Oestrogen- related receptor-α,ERRα) 的降解[36]。至今,基于VHL E3的PROTAC被用于多种疾病相关蛋白的靶向降解,如BCR-ABL[37]、间变性淋巴瘤激酶(Anaplastic lymphoma kinase,ALK)[38]以及黏着斑激酶(Focal adhesion kinase,FAK)[39]等。
(4) 基于CREN E3复合物招募的小分子PROTACs
针对Cereblon (CRBN) E3复合物的研究同样在2010年取得了重要突破,研究人员在研究免疫调节剂(Immunomodulatory drugs,IMiDs) 沙利度胺(Thalidomide) 机制的过程中发现,其主要靶标为Culin-RING泛素连接酶(Culin-RING ubiqutin ligase,CRL) 复合物中的CRBN组分[40]。随后的研究揭示除沙利度胺外,其他的邻苯二甲酰亚胺(Phthalimides) 药物,如泊马度胺(Pomalidomide)、来那度胺(Lenalidomide) 等,均是通过与CRBN结合从而降解不同的目标蛋白如Ikaros家族锌指蛋白1和3 (IKAROS family zinc finger 1 and 3、IKZF1 and 3) 等达到治疗效果[41-42]。
2015年注定成为PROTACs领域发展历程中最不平凡的一年(图 1)。除了VHL-E3小分子PROTAC的建立外,在IMiDs能够结合CREN特性的启示下,Lu等在同年构建了首个基于CRL E3复合物招募的小分子PROTAC[43]。转录共刺激因子BRD4是溴结构域和超末端结构域(Bromodomain and extraterminal domain,BET) 家族成员,因其能够调节多种原癌基因的表达水平而成为多种癌症治疗的重要靶标蛋白,而目前绝大部分PROTACs相关研究也都针对此蛋白。Crews等通过将BRD4的小分子抑制剂OTX015与pomalidomide相连合成ARV-825,实验结果显示,利用纳摩尔浓度的ARV-825处理Burkitt淋巴瘤细胞2 h左右后,即有超过50%的BRD4发生降解,最终甚至可以达到BRD4被完全降解的效果。更重要的是,相比于单独的OTX015,ARV-825能够更为有效地抑制肿瘤细胞增殖[43]。几乎同时,另外一种pan-BET抑制剂JQ1也被用于PROTAC的研究中[44],Bai等将JQ1与phthalimides相连合成dBET分子,并通过大规模蛋白质组学的方法证实dBET1能够高效且特异性地引起BRD4在内的所有BRD家族蛋白的降解,而且该过程仅在2 h内即可完成,同时在小鼠体内表现出明显的肿瘤抑制效果,延缓了白血病的进程。此后,利用CRBN E3配体与新筛选的BET小分子抑制剂合成的BETd-246[45]、QCA570[46]和BETd-260[47]等也相继被用于癌症治疗。其实在2015年稍早前,Zengerle等已将JQ1与VHL E3配体结合形成PROTAC分子MZ1,这也是首次将PROTACs用于BET家族蛋白的降解。但是,相对于ARV-825以及dBET1对BET家族的广泛抑制,MZ1对于BRD4展现出更高的特异性[48],这同时也说明了PROTAC中不同的配体搭档会影响底物特异性。有趣的是,Lai等以激酶BCR-ABL为目标,对VHL E3与CRBN配体分别相连进行平行比较,他们发现与VHL E3相比,CRBN E3的降解方式似乎更为灵活,因此在药物研发方面可能更具潜力[49]。目前,基于CRBN的PROTACs已被用于STAT3[50]、MDM2[51]、P300/CBP相关因子/通用控制蛋白5 (P300/CBP-associated factor/general control nonderepressible 5,PCAF/GCN5)[52]等多种靶标蛋白的降解。
(5) 基于共价结合E3复合物方式的PROTACs
能够共价结合E3连接酶复合物小分子的发现将更多类型的E3复合物类型如DCAF16、Ring finger protein 114 (RNF114) 以及RNF4引入到PROTACs应用中。2019年,Spradlin等在研究小分子药物印苦楝内酯(Nimbolide) 抗癌机制的过程中发现,Nimbolide能够通过与RNF114 E3连接酶复合物N末端的半胱氨酸残基形成共价结合,从而破坏RNF114对底物如肿瘤抑制因子p21和p57的识别及降解,达到治疗效果[53]。该团队随即将Nimbolide与JQ1结合发现其确实可以有效结合RNF114,并导致BET家族降解。与此同时,Zhang等利用靶向半胱氨酸的小分子库,分别筛选到了能够和CRL41 E3连接酶复合物中组分DCAF16结合的小分子KB02[54],以及同RNF4 E3复合物共价结合的小分子TRH 1-23[55]。相较于传统的E3及其配体的非共价结合方式,E3与配体之间形成不可逆的结合在动力学上似乎更为有利,因为它们在循环过程中只需要底物蛋白和配体之间的单次反应即可开始目标蛋白的降解过程。
2.1.3 新型PROTACs在不断扩大PROTACs的作用靶点类型和E3类型的同时,研究人员针对PROTACs的分子量、起效控制方式等问题进行了进一步的探索,区别于传统的多肽及小分子形式PROTACs,我们将其归类为新型PROTACs。
(1) 提高细胞通透性:CLIPTACs
合适的小分子药物大小控制应在500 Da以下[56],然而PROTAC与两种小分子配体相加后分子量可达1 000 Da甚至更大,因此小分子PROTACs仍然存在细胞膜通透性差的问题,尤其是在其需要通过血脑屏障的情况下。针对此问题,Astex制药公司开发了基于小分子前体的胞内CLIPTACs (In-cell click-formed proteolysis targeting chimeras,CLIPTACs)[57]系统。该系统中的小分子前体的分子量更小,如四嗪(Tetrazine,Tz) 标记的沙利度胺衍生物(约572 Da) 和反式环辛烯(Trans-cyclo-octene,TCO) 标记的CRBN E3配体(约609 Da)。以上两种小分子前体先后入胞后,基于Tz和TCO之间的加成反应在细胞内形成完整的PROTAC分子。实验结果显示,CLIPTACs能够在HeLa、A375以及HCT116等3种细胞系中成功诱导BRD4与ERK1/2的降解。相比于传统的PROTACs分子,CLIPTACs不仅在细胞渗透性、溶解度上有明显提升,且不需要对连接子进行优化,对不同的目标蛋白降解只需更换蛋白配体部分即可,更加灵活方便。
(2) 提高靶向性:光调控PROTACs
在PROTACs中加入操控元件,从而使其可在特定位置或特定时间起效,比如仅在癌症细胞内发挥作用,将有效提升其靶向性并降低潜在毒性。凭借无创性、起效快以及较好的时空精确度等优势,光诱导调控已在生物医学等领域广泛应用[58]。2019年Xue等建立了一种光诱导起效的PROTACs,称之为光敏笼状PROTACs (Photo-caged PROTACs,pc-PROTACs)[59]。研究显示高剂量的dBET1会导致正常细胞内几乎所有的BET家族成员蛋白被完全耗尽,存在较大的细胞毒性,这一缺陷限制了其在体内的进一步应用[44]。Xue等通过在dBET1的JQ1上引入DMNB (4, 5-dimethoxy-2-nitrobenzyl) 基团,从而将pc-PROTAC与BET的结合能力降低100倍以上。在UVA诱导后,DMNB可从pc-PROTAC上被移除,并在4 h内完全降解掉BRD4,该效果与dBET1相当。2020年,哈佛医学院和西奈山医学院研究团队则通过在E3配体Pomalidomide的关键氢键上引入一种光不稳定封闭基团Nitroveratryloxycarbonyl (NOVC) 阻断其与的CRBN结合,而在UVA激发下,opto-PROTAC中惰性Pomalidomide能恢复招募CRBN的功能[60]。Naro等同样采取了修饰E3配体的方式,即在VHL或CRBN E3配体的关键羟基上引入DEACM基团实现基于UVA的功能调控[61]。Reynders等甚至在E3配体上仅仅引入偶氮苯环作为开关,设计并合成了分子量最小的光开关PROTAC,并将其命名为PHOTAC。该分子的降解功能可以分别在390 nm和525 nm的波长条件下被打开或关闭[62]。除了在配体上引入光控元件外,Pfaff等尝试在两个功能配体之间连接子上引入带有邻-F4-偶氮苯(Ortho-F4-azobenzene),在偶氮顺式连接子的情况下,功能配体之间的距离较近,导致PROTACs无法与目标蛋白及E3结合,而在UVA刺激下使其转变成反式之后,功能配体距离合适,从而恢复各自结合活性[63]。Jin等设计的Azo-PROTAC也利用了类似的光诱导连接子[64]。但是由于UVA会对基因组DNA造成破坏,以及穿透性较差的天然缺陷,导致其目前只能在诸如皮肤癌、肺癌等疾病治疗中应用,因此寻找合适的激发光源将成为未来重要的研究方向。
(3) 提高灵活性:基于融合标签的PROTACs
PROTACs的靶向结合能力很大程度上取决于能够特异性结合目标蛋白的小分子(多肽) 配体,然而针对新的靶标蛋白的PROTAC的研究就需要重新寻找或者筛选相应配体,这严重限制了PROTACs在研究中的灵活性。因此,基于融合标签的PROTACs不断被开发出来。2015年,Crews等将Promega公司研发的HaloTag (HT) 技术引入到PROTACs的研究中,建立了HaloPROTAC系统[36]。HT是一种工程化的细菌脱卤酶,可以与氯烷烃(Chloroalkane) 之间形成共价交联。HaloPROTAC正是将E3小分子配体与氯烷烃结合用于HT融合蛋白的降解。目前,已有多种类型的基于VHL E3配体以及cIAP配体的HaloPROTAC被广泛用于各种融合HT的目标蛋白的降解[65-67]。与HaloTag工作原理类似,dTAG系统将FKBP12的工程化变体(FKBP12F36V) 作为融合标签,该突变体会在蛋白中产生野生型不具备的“空穴”,从而适合作为配体被特异性识别。Bradner等将IMiD与FKBP12F36V小分子配体如AP1867相连成功诱导了FKBP12F36V标签融合蛋白的降解,而且最重要的是其对内源性的FKBP12未产生任何影响[68]。但是,基于融合标签的PROTACs由于需要对目标蛋白引入融合蛋白,因此目前该系统更多是作为一种研究工具而非治疗手段。
随着PROTAC技术的发展,目前已有SCF、MDM2、cIAP1、VHL、CRBN以及共价结合方式的DACF16、RNF114、RNF4等8种E3复合物被引入到PROTACs药物的研发,涉及靶蛋白有50多种,并被成功用于癌症、神经退行性疾病、自身免疫病等多种疾病的治疗研究中[69],其中代表性的靶标蛋白及相关疾病总结于表 1[70]。
| Targeted proteins | Diseases | Degradation systems | References |
| PROTAC | |||
| ALK | Chromosomal translocation associated with anaplastic large cell lymphoma (ALCL); diffuse large B-cell lymphoma (DLBCL); neuroblastoma (NB) | CRBN | [71] |
| AR | Prostate cancer | VHL | [72] |
| BCR-ABL | Chronic myelogenous lymphoma (CML) | VHL/CRBN | [49] |
| BRD4 | Prostate cancer; leukemia; lymphoma | VHL/CRBN | [44] |
| CRABP-Ⅰ/Ⅱ | Neuroblastoma; acute promyelocytic leukemia | cIAP1 | [27] |
| EGFR and HER2 | Breast cancer; cervical cancer; non-small cell lung cancer (NSCLC); lung cancer | VHL | [73] |
| ER | Breast cancer | VHL | [74] |
| ERRα | Breast cancer | VHL/CRBN | [75] |
| FAK | Breast cancer; prostate cancer; hepatocellular carcinoma (HCC) | VHL/CRBN | [39] |
| FKBP12 | Acute lymphocytic leukemia (ALL); cardiovascular disease | CRBN | [44] |
| METAP-2 | Cancers | SCF | [14] |
| Mcm 4 | Lymphoma | SCF | [76] |
| MDM2 | Leukemia; acute myelocytic leukemia (AML) | CRBN | [77] |
| PI3K | Liver cancer | CRBN | [78] |
| RIPK2 | Autoinflammatory diseases | VHL | [79] |
| STAT3 | AML; anaplastic large cell lymphoma (ALCL) | CRBN | [50] |
| TACC3 | Cancers | cIAP1 | [31] |
| NS3 | Hepatitis C virus (HCV) infection | CRBN | [80] |
| IRAK4 | Autoimmune; inflammatory; oncological diseases | VHL | [81] |
| IKZF1/3 | Myeloma | CRBN | [82] |
| PCAF/GCN5 | Inflammatory diseases | CRBN | [52] |
| Tau | Neurodegenerative diseases | CRBN | [83] |
| Lysosome-based TPD | |||
| ApoE4 | Neurodegenerative disease | LYTAC | [76] |
| CD71 | Acute T-cell leukemia | LYTAC | [76] |
| DAPK1 | Cerebral ischemia | CMA | [84] |
| EGFR | Cervical cancer | LYTAC | [76] |
| METAP-2 | Cervical cancer | AUTAC | [85] |
| Mutant huntingtin | HD | ATTEC | [86] |
| PSD-95 | Neurodegenerative synucleinopathies | CMA | [84] |
| PD-L1 | Breast cancer | LYTAC | [76] |
(1) 生长素诱导的降解决定子(Auxin-inducible degron,AID)
生长素(Auxin) 如吲哚乙酸(Indole-3-acetic acid,IAA) 和萘乙酸(Naphthylacetic acid,NAA)等,能够与F-box转运抑制响应蛋白1 (Transport inhibitor response protein 1,TIR1) 结合,并促进E3泛素连接酶SCF-TIR1与AID的相互作用,最终导致AID泛素化降解(图 2)。2009年,Nishimura等首先在酵母中对AID进行了尝试,他们选用了来源于拟南芥的AID与目标蛋白融合,在加入NAA的情况下成功诱导了微小染色体维持蛋白4 (Minichromosome maintenance proteins 4,Mcm 4)的降解[87]。之后,该团队将AID体系成功应用于真核细胞蛋白的降解, 并将AID的大小降至7 kDa,提升了该体系的性能[88]。尽管所有真核生物都存在SCF,但TIR1类似物和AID只在植物物种中才会出现,因此,需要外源表达TIR1以及目标蛋白必须融合AID等前提条件成了该技术广泛应用的最大限制。同时,TIR1的异源表达是否会引起胞内蛋白非特异性的降解仍有待研究。
(2) IMiD诱导的IKZF3降解决定子(IMiD- responsive IKZF3 degron)
前文提到IMiDs如沙利度胺,能够与E3复合物中的CRBN组分结合,这会导致CRBN特异性改变从而靶向结合IKZF1和IKZF3并引起其降解。研究人员于2014年确定了在IMiDs存在下,CRBN识别IKZF3的区域为锌指2的136–180位氨基酸[82]。Koduri等于2019年将该识别区段进一步缩小至144–168位氨基酸,即最小区段IKZF3降解决定子(Minimal-IKZF3 degron),并发现IMiDs在体内外均成功诱导与minimal-IKZF3 degron融合的目标蛋白的降解[89] (图 2)。但是,随后的研究显示minimal-IKZF3 degron标记的蛋白类型中仅有50%能够被成功降解[89],而且,由于在部分物种如小鼠中CRBN存在序列变异[90],会导致其无法被IMiDs招募,因此为实现在药物研发中的广泛应用,该体系仍需要进一步优化。
(3) 小分子诱导开关(Small molecule-assisted shutoff,SMASh)
上述两种诱导降解系统,都是基于小分子对E3复合物的招募能力,而SMASh系统则采用了截然不同的操控方式。SMASh由3部分构成,依次为降解因子、来源于丙型肝炎病毒HCV的NS3蛋白酶结构域(NS3 protease domain,NS3 pro) 以及其相应酶切位点氨基酸,目标蛋白则通过融合的方式与酶切位点相邻[91]。NS3蛋白酶的切割活性可以被临床上用于控制HCV感染的小分子药物如西咪匹韦(Simeprevir) 等所抑制。因此,在没有药物的情况下,NS3 pro会在NS3酶切位点处将SMASh切断从而导致目标蛋白与降解因子NS4分离,因而无法被泛素标记并降解。相反地,在药物抑制NS3酶切活性的条件下,SMASh保持完整,降解因子则可诱导目标蛋白的泛素化降解(图 2)。但该方法同样需要融合整个SMASh复合物才能达到降解目的。
2.2.2 Trim-away降解系统Trim-away技术的核心元件是一种由英国医学研究理事会(Medical Research Council,MRC) 分子生物学实验室的Leo James研究组发现的E3泛素连接酶Trim21。它能够识别进入细胞病毒附着的抗体,并与抗体Fc结构域结合,最终在Trim21自身泛素化的过程中引起抗体/目标蛋白复合物一同被降解[92] (图 2)。由于Trim21在多种细胞类型和组织中广泛表达[93],因此只需要向细胞内提供目标蛋白的抗体分子,即可达到降解靶标蛋白的目的。但是抗体并不具有细胞渗透性,尽管可以通过显微注射或电转的方式导入,但这些物理导入方式对于细胞的伤害问题仍是无法避免的,因此高效生物大分子入胞递送系统的引入将成为推进Trim-away系统应用的重要手段。此外,由于Trim21及抗体分子会和目标蛋白一同被降解,因此目标蛋白会迅速恢复至基线水平,从而影响最终的治疗效果。
3 基于溶酶体途径的靶向蛋白降解技术近几年,不依赖于蛋白酶体的另一种胞内蛋白降解途径——溶酶体(Lysosome) 途径也逐渐进入科学家们的视野,并被应用于TPD技术的研究中。溶酶体降解途径主要包括内吞小泡(Endosome)/溶酶体和自噬(Autophagy)-溶酶体两种途径,目前,这两种途径均已被用于疾病相关蛋白降解的研究中[94]。
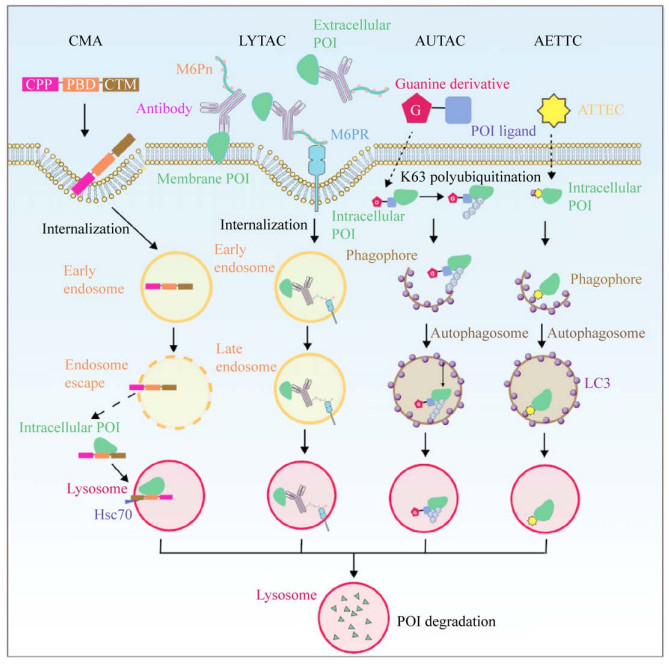
3.1 基于内吞小泡/溶酶体途径的溶酶体靶向嵌合系统虽然基于UPS的大量研究在体内外展现出良好的降解效果,但其靶标蛋白主要定位于胞内。然而,大部分细胞外蛋白和细胞膜蛋白也与癌症、自身免疫病等疾病的发生发展息息相关[95],因此这些蛋白的调控或降解对疾病治疗同样具有重要意义。2020年Banik等基于内吞小泡-溶酶体途径,利用能够同时结合细胞表面溶酶体靶向受体(Lysosome-targeting receptors,LTRs) 和跨膜蛋白胞外区段的抗体分子,建立了靶向降解胞膜蛋白和细胞质蛋白的溶酶体靶向嵌合体系统(Lysosome- targeting chimaeras,LYTACs)[76]。阳离子非依赖型甘露糖6-磷酸受体(Cation-independent mannose- 6-phosphate receptor,CI-M6PR) 是真核生物细胞内最为典型的LTR,它能将带有甘露糖6-磷酸(Mannose-6-phosphate,M6P) 残基的N-聚糖的蛋白质内源性转运至溶酶体并降解。在该研究中,Banik等将寡聚甘露糖-6-磷酸(M6Pn) 共价交联在目标蛋白的特异性抗体上。在抗体识别目标蛋白后,在其上交联的M6Pn会结合细胞表面的CI-M6PR,从而将多种胞膜蛋白如载脂蛋白E4 (Apolipoprotein E4)、表皮生长因子受体(Epidermal growth factor receptor,EGFR)、程序性死亡蛋白配体-1 (Programmed death-ligand 1,PD-L1) 以及CD71运送至溶酶体进行降解(图 3)。
|
| 图 3 基于溶酶体途径的TPD技术作用机制 Fig. 3 Schematic overview of mechanisms of lysosome degradation pathway-based TPD technologies. |
| |
细胞自噬是细胞在自噬相关基因的调控下利用溶酶体降解自身受损的大分子物质和细胞器的过程。自噬主要分为3种类型:巨自噬,也是我们常提及的“自噬” (后简称自噬),其特点为自噬过程中形成了自噬体(Autophagosomes) 这样的双侧膜细胞器;微自噬,特点为通过溶酶体膜的内陷吞噬待降解物质;由伴侣蛋白如热激关联蛋白70 (Heat shock cognate protein 70,Hsc70) 等介导的自噬(Chaperone-mediated autophagy,CMA)[96]。
3.2.1 基于CMA的蛋白降解系统CMA途径降解的蛋白底物均包含有KFERQ类似的五肽,即CMA靶向基序(CMA-targeting motif,CTM)。研究人员将CPP、CTM与蛋白结合域(Protein binding domain,PBD) 3种功能多肽融合表达,这种嵌合蛋白在原代神经元内能有效降解死亡相关蛋白激酶1 (Death associated protein kinase 1,DAPK1) 以及支架蛋白突触后致密区95 (Postsynaptic Density Protein-95,PSD-95)[84] (图 3),但CMA的研究仍依赖于较低递送效率穿膜肽TAT,因此,目前治疗效果很大程度上受限于TAT的递送效率。
3.2.2 基于自噬途径的蛋白靶向降解系统自噬是一种多步骤降解过程,起始于自噬泡(Phagophore) 的形成。之后,微管相关蛋白轻链3 (Microtubule-associated protein light chain 3,LC3) 由可溶解形式(LC3-Ⅰ) 转变为脂溶形式(LC3-Ⅱ),并与自噬泡结合形成自噬体(Autophagosome)[96]。自噬体通过表面受体LC3识别并捕获被K63位多聚泛素化(蛋白酶体多识别K48多聚泛素化)修饰的待降解分子。最终,自噬体与溶酶体结合形成自噬溶酶体(Autolysosomes),并将内容物降解。2019年,Takahashi等根据自噬过程设计了用于降解内源性蛋白的自噬靶向嵌合体(Autophagy-targeting chimera,AUTAC)[85] (图 3)。AUTAC分子主要包含两部分:降解标签鸟嘌呤类似物(如S-鸟苷酸) 和目标蛋白配体,鸟嘌呤类似物可以诱导目标蛋白的K63多聚泛素化标记,从而招募自噬体将其降解[85]。但是自噬过程步骤多且极其复杂,影响因素会非常多,因此稳定性较差。除此之外,S-鸟苷酸诱导的K63泛素化分子机制、AUTAC是否会出现脱靶效应以及在体内的效果如何等方面都还需深入研究。
基于自噬通路,Li等则采取小分子招募胞浆LC3这样一种更为直接的方式,建立了自噬体连接化合物系统(Autophagosome-tethering compound,ATTEC)[86]。亨廷顿病(Huntington’s disease,HTT dieases,HD) 是一种遗传性神经退行性疾病,目前已有研究表明突变的HTT蛋白(mHTT) 是HD的主要病因[97]。针对mHTT这个特殊靶标,该团队利用小分子微阵列的方法,筛选到能够同时结合mHTT和LC3但不与野生型相互作用的ATTEC分子。ATTEC在结合LC3后定位至自噬泡并将mHTT包入自噬体内,最终将其降解,而与此同时野生型并未受影响。值得注意的是ATTEC的工作原理类似于分子胶(Molecule glue),即由一个分子完成目标蛋白结合和降解相关蛋白的招募两个任务,因此,其灵活性不如双模块化的PROTAC、LYTAC以及AUTAC (图 3)。
4 总结与展望综上所述,TPD技术不仅为新型药物的研发提供了新的思路,也为疾病的治疗提供了新的选择,目前已被成功用于包括癌症、免疫疾病和神经退行性疾病等多种疾病的治疗性研究中(表 1)。在诸多TPD技术中,研究最为成熟的是基于UPS的PROTAC药物,前列腺癌药物ARV-110在临床试验中显示出良好的耐受性,并且在部分患者中出现了明显的前列腺表面抗原下降的现象。尽管如此,PROTAC技术还存在诸多问题亟须解决(表 2)。小分子形式的PROTACs在药物吸收效果上优势巨大,但其面临着E3连接酶复合物类型利用率低的主要问题,因此,对各种类型E3复合物的招募、降解机制的充分研究、通过高通量方法筛选可以结合其他种类E3复合物及其结合能力以及特异性更高的小分子化合物将成为小分子形式PROTACs的主要研究方向。多肽形式的PROTAC,相比于其他基于小分子配体的降解方法,在靶向结合特异性、生物相容性上存在明显的优势,但由于细胞膜选择透过性的限制,导致多肽PROTACs难以入胞,而在系统中引入细胞穿膜肽成为目前最有效的促进入胞手段。然而细胞穿膜肽如TAT、R9等本身也存在着在内吞过程中内吞小泡逃逸效率极低(据报道,TAT介导的货物分子的小泡逃逸效率不到1%[98]) 导致最终递送效果差的问题,同时,尽管小分子PROTACs在入胞能力上已经有很大提升,但目前大多数研究中所用的分子大小也达到了1 kDa左右。因此建立具有高效小泡逃逸效率的新型递送系统将极大地推动PROTAC系统及其相关药物的研发。其他基于UPS的降解方法,如小分子诱导的降解途径依赖于降解因子与目标蛋白的融合成为这些系统作为药物的最大障碍;而Trim-away需要将抗体导入的前提条件则极大地限制着其发展,与多肽PROTACs类似,高逃逸效率胞内递送系统的引入也将推进该技术的进一步应用。
| Degradation pathway | Degradation system | Advantages | Limitations |
| Ubiquitin-proteasome system | PROTAC | In vivo applications; clear mechanisms of action; relatively high selectivity | High molecular weight; uncontrollable; limited E3 complex knowledge |
| AID | No off-target effect | Need exogenous expression of TIR1 | |
| SMASh | FDA-approved HCV drug | Need modification of SMASh cassette | |
| IKZF3 | Endogenous IKZF3 not targeted; FDA-approved IMiD | Need exogenous expression of IKZF3; CREN variation | |
| Trim-away | Only need introduction of antibody | Limitation of Trim 21 level; poor cell membrane permeability | |
| Lysosomal pathway | CMA | Only five amino acid modification | Poor cellular membrane permeability |
| LYTAC | Target to extracellular and transmembrane proteins; independent of ubiquitination and proteasomal degradation | Large molecular weight and weak cellular membrane permeability; possible induction of immune response in vivo | |
| AUTAC | Potentially a broad target spectrum; proteasome independent; demonstrated ability to degrade mitochondria | Lack of key information of mechanisms; K63 ubiquitination; possible influence on natural autophagy | |
| ATTEC | Potentially a broad target spectrum; direct machinery; low molecular weight | The LC3-bound chemical moieties need to be solved; low versatility |
除此之外,近几年兴起的基于溶酶体途径的CMA、LYTAC、ATTEC、AUTAC降解方法虽然在靶蛋白的降解上为我们提供了全新的思路,进一步扩展了TPD药物的靶点蛋白范围。但是,招募溶酶体途径的相关元件的具体机制仍未完全掌握,“劫持”细胞溶酶体降解途径是否会对机体造成影响尚不明确,因此,基于溶酶体途径的TPD药物还有很长的路要走,对工作机制的了解、结构的优化都将为其未来的发展铺平道路。值得注意的是,目前对于TPD的靶向递送的研究较少,而在正常细胞内的蛋白降解很可能造成严重的体内毒性,因此,引入靶向/操控元件(如靶向肽),促使TPD药物能够递送到特定细胞或仅能在特定细胞内起效将成为TPD技术各系统未来研究必须重视的方向。尽管TPD技术的发展尚处于起步阶段,但凭借其靶标蛋白限制小、特异性高以及作用时间快等多种优势,势必将为药物研发领域开创新的纪元。
| [1] |
Wang T, Birsoy K, Hughes NW, et al. Identification and characterization of essential genes in the human genome. Science, 2015, 350(6264): 1096-1101. DOI:10.1126/science.aac7041
|
| [2] |
Bedard PL, Hyman DM, Davids MS, et al. Small molecules, big impact: 20 years of targeted therapy in oncology. Lancet, 2020, 395(10229): 1078-1088. DOI:10.1016/S0140-6736(20)30164-1
|
| [3] |
Lazo JS, Sharlow ER. Drugging undruggable molecular cancer targets. Annu Rev Pharmacol Toxicol, 2016, 56: 23-40. DOI:10.1146/annurev-pharmtox-010715-103440
|
| [4] |
Doudna JA. The promise and challenge of therapeutic genome editing. Nature, 2020, 578(7794): 229-236. DOI:10.1038/s41586-020-1978-5
|
| [5] |
Revia RA, Stephen ZR, Zhang MQ. Theranostic nanoparticles for RNA-based cancer treatment. Acc Chem Res, 2019, 52(6): 1496-1506. DOI:10.1021/acs.accounts.9b00101
|
| [6] |
Adams D, Polydefkis M, González-Duarte A, et al. Long-term safety and efficacy of patisiran for hereditary transthyretin-mediated amyloidosis with polyneuropathy: 12-month results of an open-label extension study. Lancet Neurol, 2021, 20(1): 49-59. DOI:10.1016/S1474-4422(20)30368-9
|
| [7] |
Guo JP, Liu J, Wei WY. Degrading proteins in animals: "PROTAC" tion goes in vivo. Cell Res, 2019, 29(3): 179-180. DOI:10.1038/s41422-019-0144-9
|
| [8] |
Nalawansha DA, Crews CM. PROTACs: an emerging therapeutic modality in precision medicine. Cell Chem Biol, 2020, 27(8): 998-1014. DOI:10.1016/j.chembiol.2020.07.020
|
| [9] |
Yang J, Wang QL, Feng GK, et al. Significance of selective protein degradation in the development of novel targeted drugs and its implications in cancer therapy. Adv Therapeut, 2020, 3(6): 1900210. DOI:10.1002/adtp.201900210
|
| [10] |
Burslem GM, Crews CM. Proteolysis-targeting chimeras as therapeutics and tools for biological discovery. Cell, 2020, 181(1): 102-114. DOI:10.1016/j.cell.2019.11.031
|
| [11] |
Schapira M, Calabrese MF, Bullock AN, et al. Targeted protein degradation: expanding the toolbox. Nat Rev Drug Discov, 2019, 18(12): 949-963. DOI:10.1038/s41573-019-0047-y
|
| [12] |
Pettersson M, Crews CM. Proteolysis targeting chimeras (PROTACs)-past, present and future. Drug Discov Today Technol, 2019, 31: 15-27. DOI:10.1016/j.ddtec.2019.01.002
|
| [13] |
Wu T, Yoon H, Xiong Y, et al. Targeted protein degradation as a powerful research tool in basic biology and drug target discovery. Nat Struct Mol Biol, 2020, 27(7): 605-614. DOI:10.1038/s41594-020-0438-0
|
| [14] |
Sakamoto KM, Kim KB, Kumagai A, et al. Protacs: chimeric molecules that target proteins to the Skp1-Cullin-F box complex for ubiquitination and degradation. Proc Natl Acad Sci USA, 2001, 98(15): 8554-8559. DOI:10.1073/pnas.141230798
|
| [15] |
Mullard A. First targeted protein degrader hits the clinic. Nat Rev Drug Discov, 2019. DOI:10.1038/d41573-019-00043-6
|
| [16] |
Verma R, Mohl D, Deshaies RJ. Harnessing the power of proteolysis for targeted protein inactivation. Mol Cell, 2020, 77(3): 446-460. DOI:10.1016/j.molcel.2020.01.010
|
| [17] |
Sakamoto KM, Kim KB, Verma R, et al. Development of PROTACS to target cancer-promoting proteins for ubiquitination and degradation. Mol Cell Proteomics, 2003, 2(12): 1350-1358. DOI:10.1074/mcp.T300009-MCP200
|
| [18] |
Howell A, Howell SJ, Evans DG. New approaches to the endocrine prevention and treatment of breast cancer. Cancer Chemother Pharmacol, 2003, 52(Suppl 1): S39-S44.
|
| [19] |
Zhu PP, Jin L. Cell penetrating peptides: a promising tool for the cellular uptake of macromolecular drugs. Curr Protein Pept Sci, 2018, 19(2): 211-220.
|
| [20] |
Schneekloth JS, Fonseca FN, Koldobskiy M, et al. Chemical genetic control of protein levels: selective in vivo targeted degradation. J Am Chem Soc, 2004, 126(12): 3748-3754. DOI:10.1021/ja039025z
|
| [21] |
Paiva SL, Crews CM. Targeted protein degradation: elements of PROTAC design. Curr Opin Chem Biol, 2019, 50: 111-119. DOI:10.1016/j.cbpa.2019.02.022
|
| [22] |
Hines J, Gough JD, Corson TW, et al. Posttranslational protein knockdown coupled to receptor tyrosine kinase activation with phosphoPROTACs. Proc Natl Acad Sci USA, 2013, 110(22): 8942-8947. DOI:10.1073/pnas.1217206110
|
| [23] |
Jin JM, Wu Y, Chen JJ, et al. The peptide PROTAC modality: a novel strategy for targeted protein ubiquitination. Theranostics, 2020, 10(22): 10141-10153. DOI:10.7150/thno.46985
|
| [24] |
Chamberlain PP, Hamann LG. Development of targeted protein degradation therapeutics. Nat Chem Biol, 2019, 15(10): 937-944. DOI:10.1038/s41589-019-0362-y
|
| [25] |
Vassilev LT, Vu BT, Graves B, et al. In vivo activation of the p53 pathway by small-molecule antagonists of MDM2. Science, 2004, 303(5659): 844-848. DOI:10.1126/science.1092472
|
| [26] |
Schneekloth AR, Pucheault M, Tae HS, et al. Targeted intracellular protein degradation induced by a small molecule: En route to chemical proteomics. Bioorg Med Chem Lett, 2008, 18(22): 5904-5908. DOI:10.1016/j.bmcl.2008.07.114
|
| [27] |
Itoh Y, Ishikawa M, Naito M, et al. Protein knockdown using methyl bestatin-ligand hybrid molecules: design and synthesis of inducers of ubiquitination-mediated degradation of cellular retinoic acid-binding proteins. J Am Chem Soc, 2010, 132(16): 5820-5826. DOI:10.1021/ja100691p
|
| [28] |
Yokoo H, Ohoka N, Naito M, et al. Design and synthesis of peptide-based chimeric molecules to induce degradation of the estrogen and androgen receptors. Bioorg Med Chem, 2020, 28(15): 115595. DOI:10.1016/j.bmc.2020.115595
|
| [29] |
Ohoka N, Okuhira K, Ito M, et al. In vivo knockdown of pathogenic proteins via specific and nongenetic inhibitor of apoptosis protein (IAP)-dependent protein erasers (SNIPERs). J Biol Chem, 2017, 292(11): 4556-4570. DOI:10.1074/jbc.M116.768853
|
| [30] |
Shibata N, Nagai K, Morita Y, et al. Development of protein degradation inducers of androgen receptor by conjugation of androgen receptor ligands and inhibitor of apoptosis protein ligands. J Med Chem, 2018, 61(2): 543-575. DOI:10.1021/acs.jmedchem.7b00168
|
| [31] |
Ohoka N, Nagai K, Shibata N, et al. SNIPER(TACC3) induces cytoplasmic vacuolization and sensitizes cancer cells to Bortezomib. Cancer Sci, 2017, 108(5): 1032-1041. DOI:10.1111/cas.13198
|
| [32] |
Naito M, Ohoka N, Shibata N. SNIPERs—hijacking IAP activity to induce protein degradation. Drug Discov Today Technol, 2019, 31: 35-42. DOI:10.1016/j.ddtec.2018.12.002
|
| [33] |
Buckley D L, Gustafson J L, Van Molle I, et al. Small-molecule inhibitors of the interaction between the E3 ligase VHL and HIF1α. Angew Chem Int Ed Engl, 2012, 51(46): 11463-11467. DOI:10.1002/anie.201206231
|
| [34] |
Van Molle I, Thomann A, Buckley DL, et al. Dissecting fragment-based lead discovery at the von Hippel-Lindau protein: hypoxia inducible factor 1α protein-protein interface. Chem Biol, 2012, 19(10): 1300-1312. DOI:10.1016/j.chembiol.2012.08.015
|
| [35] |
Galdeano C, Gadd MS, Soares P, et al. Structure-guided design and optimization of small molecules targeting the protein-protein interaction between the von Hippel-Lindau (VHL) E3 ubiquitin ligase and the hypoxia inducible factor (HIF) alpha subunit with in vitro nanomolar affinities. J Med Chem, 2014, 57(20): 8657-8663. DOI:10.1021/jm5011258
|
| [36] |
Bondeson DP, Mares A, Smith IED, et al. Catalytic in vivo protein knockdown by small-molecule PROTACs. Nat Chem Biol, 2015, 11: 611-617. DOI:10.1038/nchembio.1858
|
| [37] |
Zhao QJ, Ren CW, Liu LY, et al. Discovery of SIAIS178 as an effective BCR-ABL degrader by recruiting von hippel-lindau (VHL) E3 ubiquitin ligase. J Med Chem, 2019, 62(20): 9281-9298. DOI:10.1021/acs.jmedchem.9b01264
|
| [38] |
Kang CH, Lee DH, Lee CO, et al. Induced protein degradation of anaplastic lymphoma kinase (ALK) by proteolysis targeting chimera (PROTAC). Biochem Biophys Res Commun, 2018, 505(2): 542-547. DOI:10.1016/j.bbrc.2018.09.169
|
| [39] |
Cromm PM, Samarasinghe KTG, Hines J, et al. Addressing kinase-independent functions of fak via PROTAC-mediated degradation. J Am Chem Soc, 2018, 140(49): 17019-17026. DOI:10.1021/jacs.8b08008
|
| [40] |
Ito T, Ando H, Suzuki T, et al. Identification of a primary target of thalidomide teratogenicity. Science, 2010, 327(5971): 1345-1350. DOI:10.1126/science.1177319
|
| [41] |
Petzold G, Fischer ES, Thomä NH. Structural basis of lenalidomide-induced CK1α degradation by the CRL4(CRBN) ubiquitin ligase. Nature, 2016, 532(7597): 127-130. DOI:10.1038/nature16979
|
| [42] |
Sun YH, Zhao XW, Ding N, et al. PROTAC-induced BTK degradation as a novel therapy for mutated BTK C481S induced ibrutinib-resistant B-cell malignancies. Cell Res, 2018, 28(7): 779-781. DOI:10.1038/s41422-018-0055-1
|
| [43] |
Lu J, Qian YM, Altieri M, et al. Hijacking the E3 ubiquitin ligase cereblon to efficiently target BRD4. Chem Biol, 2015, 22(6): 755-763. DOI:10.1016/j.chembiol.2015.05.009
|
| [44] |
Winter GE, Buckley DL, Paulk J, et al. Phthalimide conjugation as a strategy for in vivo target protein degradation. Science, 2015, 348(6241): 1376-1381. DOI:10.1126/science.aab1433
|
| [45] |
Bai LC, Zhou B, Yang CY, et al. Targeted degradation of BET proteins in triple-negative breast cancer. Cancer Res, 2017, 77(9): 2476-2487. DOI:10.1158/0008-5472.CAN-16-2622
|
| [46] |
Qin C, Hu Y, Zhou B, et al. Discovery of QCA570 as an exceptionally potent and efficacious proteolysis targeting chimera (PROTAC) degrader of the bromodomain and extra-terminal (BET) proteins capable of inducing complete and durable tumor regression. J Med Chem, 2018, 61(15): 6685-6704. DOI:10.1021/acs.jmedchem.8b00506
|
| [47] |
Zhang HP, Li GQ, Zhang Y, et al. Targeting BET proteins with a PROTAC molecule elicits potent anticancer activity in HCC cells. Front Oncol, 2020, 9: 1471. DOI:10.3389/fonc.2019.01471
|
| [48] |
Zengerle M, Chan KH, Ciulli A. Selective small molecule induced degradation of the bet bromodomain protein BRD4. ACS Chem Biol, 2015, 10(8): 1770-1777. DOI:10.1021/acschembio.5b00216
|
| [49] |
Lai AC, Toure M, Hellerschmied D, et al. Modular PROTAC design for the degradation of oncogenic BCR-ABL. Angew Chem Int Ed Engl, 2016, 55(2): 807-810. DOI:10.1002/anie.201507634
|
| [50] |
Bai LC, Zhou HB, Xu RQ, et al. A potent and selective small-molecule degrader of STAT3 achieves complete tumor regression in vivo. Cancer Cell, 2019, 36(5): 498-511. e17. DOI:10.1016/j.ccell.2019.10.002
|
| [51] |
Li YB, Yang JL, Aguilar A, et al. Discovery of MD-224 as a first-in-class, highly potent, and efficacious proteolysis targeting chimera murine double minute 2 degrader capable of achieving complete and durable tumor regression. J Med Chem, 2019, 62(2): 448-466. DOI:10.1021/acs.jmedchem.8b00909
|
| [52] |
Bassi ZI, Fillmore MC, Miah AH, et al. Modulating PCAF/GCN5 immune cell function through a PROTAC approach. ACS Chem Biol, 2018, 13(10): 2862-2867. DOI:10.1021/acschembio.8b00705
|
| [53] |
Spradlin JN, Hu XR, Ward CC, et al. Harnessing the anti-cancer natural product nimbolide for targeted protein degradation. Nat Chem Biol, 2019, 15(7): 747-755. DOI:10.1038/s41589-019-0304-8
|
| [54] |
Zhang XY, Crowley VM, Wucherpfennig TG, et al. Electrophilic PROTACs that degrade nuclear proteins by engaging DCAF16. Nat Chem Biol, 2019, 15(7): 737-746. DOI:10.1038/s41589-019-0279-5
|
| [55] |
Ward CC, Kleinman JI, Brittain SM, et al. Covalent ligand screening uncovers a RNF4 E3 ligase recruiter for targeted protein degradation applications. ACS Chem Biol, 2019, 14(11): 2430-2440. DOI:10.1021/acschembio.8b01083
|
| [56] |
Wally V, Reisenberger M, Kitzmüller S, et al. Small molecule drug development for rare genodermatoses— evaluation of the current status in epidermolysis bullosa. Orphanet J Rare Dis, 2020, 15: 292. DOI:10.1186/s13023-020-01467-9
|
| [57] |
Lebraud H, Wright DJ, Johnson CN, et al. Protein degradation by in-cell self-assembly of proteolysis targeting chimeras. ACS Cent Sci, 2016, 2(12): 927-934. DOI:10.1021/acscentsci.6b00280
|
| [58] |
Shafirstein G, Battoo A, Harris K, et al. Photodynamic therapy of non-small cell lung cancer. Narrative review and future directions. Ann Am Thorac Soc, 2016, 13(2): 265-275.
|
| [59] |
Xue G, Wang K, Zhou DL, et al. Light-induced protein degradation with photocaged PROTACs. J Am Chem Soc, 2019, 141(46): 18370-18374. DOI:10.1021/jacs.9b06422
|
| [60] |
Liu J, Chen H, Ma LN, et al. Light-induced control of protein destruction by opto-PROTAC. Sci Adv, 2020, 6(8): eaay5154. DOI:10.1126/sciadv.aay5154
|
| [61] |
Naro Y, Darrah K, Deiters A. Optical control of small molecule-induced protein degradation. J Am Chem Soc, 2020, 142(5): 2193-2197. DOI:10.1021/jacs.9b12718
|
| [62] |
Reynders M, Matsuura BS, Bérouti M, et al. PHOTACs enable optical control of protein degradation. Sci Adv, 2020, 6(8): eaay5064. DOI:10.1126/sciadv.aay5064
|
| [63] |
Pfaff P, Samarasinghe KTG, Crews CM, et al. Reversible spatiotemporal control of induced protein degradation by bistable photoPROTACs. ACS Cent Sci, 2019, 5(10): 1682-1690. DOI:10.1021/acscentsci.9b00713
|
| [64] |
Jin YH, Lu MC, Wang Y, et al. Azo-PROTAC: novel light-controlled small-molecule tool for protein knockdown. J Med Chem, 2020, 63(9): 4644-4654. DOI:10.1021/acs.jmedchem.9b02058
|
| [65] |
Tovell H, Testa A, Maniaci C, et al. Rapid and reversible knockdown of endogenously tagged endosomal proteins via an optimized haloPROTAC degrader. ACS Chem Biol, 2019, 14(5): 882-892. DOI:10.1021/acschembio.8b01016
|
| [66] |
Tomoshige S, Hashimoto Y, Ishikawa M. Efficient protein knockdown of HaloTag-fused proteins using hybrid molecules consisting of IAP antagonist and HaloTag ligand. Bioorg Med Chem, 2016, 24(14): 3144-3148. DOI:10.1016/j.bmc.2016.05.035
|
| [67] |
Tomoshige S, Naito M, Hashimoto Y, et al. Degradation of HaloTag-fused nuclear proteins using bestatin-HaloTag ligand hybrid molecules. Org Biomol Chem, 2015, 13(38): 9746-9750. DOI:10.1039/C5OB01395J
|
| [68] |
Nabet B, Roberts JM, Buckley DL, et al. The dTAG system for immediate and target-specific protein degradation. Nat Chem Biol, 2018, 14(5): 431-441. DOI:10.1038/s41589-018-0021-8
|
| [69] |
Sun XY, Gao HY, Yang YQ, et al. PROTACs: great opportunities for academia and industry. Signal Transduct Target Ther, 2019, 4: 64. DOI:10.1038/s41392-019-0101-6
|
| [70] |
Owen D. Choosing your druggability battle. Nat Chem Biol, 2019, 15(7): 652-653. DOI:10.1038/s41589-019-0305-7
|
| [71] |
Powell CE, Gao Y, Tan L, et al. Chemically induced degradation of anaplastic lymphoma kinase (ALK). J Med Chem, 2018, 61(9): 4249-4255. DOI:10.1021/acs.jmedchem.7b01655
|
| [72] |
Salami J, Alabi S, Willard RR, et al. Androgen receptor degradation by the proteolysis-targeting chimera ARCC-4 outperforms enzalutamide in cellular models of prostate cancer drug resistance. Commun Biol, 2018, 1: 100. DOI:10.1038/s42003-018-0105-8
|
| [73] |
Burslem GM, Smith BE, Lai AC, et al. The advantages of targeted protein degradation over inhibition: an RTK case study. Cell Chem Biol, 2018, 25(1): 67-77. e3. DOI:10.1016/j.chembiol.2017.09.009
|
| [74] |
Hu JT, Hu B, Wang ML, et al. Discovery of ERD-308 as a highly potent proteolysis targeting chimera (PROTAC) degrader of estrogen receptor (ER). J Med Chem, 2019, 62(3): 1420-1442. DOI:10.1021/acs.jmedchem.8b01572
|
| [75] |
Matyskiela ME, Lu G, Ito T, et al. A novel cereblon modulator recruits GSPT1 to the CRL4CRBN ubiquitin ligase. Nature, 2016, 535(7611): 252-257. DOI:10.1038/nature18611
|
| [76] |
Banik SM, Pedram K, Wisnovsky S, et al. Lysosome-targeting chimaeras for degradation of extracellular proteins. Nature, 2020, 584(7820): 291-297. DOI:10.1038/s41586-020-2545-9
|
| [77] |
Wang B, Wu SZ, Liu J, et al. Development of selective small molecule MDM2 degraders based on nutlin. Eur J Med Chem, 2019, 176: 476-491. DOI:10.1016/j.ejmech.2019.05.046
|
| [78] |
Li WL, Gao CM, Zhao L, et al. Phthalimide conjugations for the degradation of oncogenic PI3K. Eur J Med Chem, 2018, 151: 237-247. DOI:10.1016/j.ejmech.2018.03.066
|
| [79] |
Bondeson DP, Mares A, Smith IED, et al. Catalytic in vivo protein knockdown by small-molecule PROTACs. Nat Chem Biol, 2015, 11(8): 611-617. DOI:10.1038/nchembio.1858
|
| [80] |
De Wispelaere M, Du GY, Donovan KA, et al. Small molecule degraders of the hepatitis C virus protease reduce susceptibility to resistance mutations. Nat Commun, 2019, 10(1): 3468. DOI:10.1038/s41467-019-11429-w
|
| [81] |
Nunes J, McGonagle GA, Eden J, et al. Targeting IRAK4 for degradation with PROTACs. ACS Med Chem Lett, 2019, 10(7): 1081-1085. DOI:10.1021/acsmedchemlett.9b00219
|
| [82] |
Krönke J, Udeshi ND, Narla A, et al. Lenalidomide causes selective degradation of IKZF1 and IKZF3 in multiple myeloma cells. Science, 2014, 343(6168): 301-305. DOI:10.1126/science.1244851
|
| [83] |
Silva MC, Ferguson FM, Cai Q, et al. Targeted degradation of aberrant tau in frontotemporal dementia patient-derived neuronal cell models. Elife, 2019, 8: e45457. DOI:10.7554/eLife.45457
|
| [84] |
Fan XL, Jin WY, Lu J, et al. Rapid and reversible knockdown of endogenous proteins by peptide- directed lysosomal degradation. Nat Neurosci, 2014, 17(3): 471-480. DOI:10.1038/nn.3637
|
| [85] |
Takahashi D, Moriyama J, Nakamura T, et al. AUTACs: cargo-specific degraders using selective autophagy. Mol Cell, 2019, 76(5): 797-810. e10. DOI:10.1016/j.molcel.2019.09.009
|
| [86] |
Li ZY, Wang C, Wang ZY, et al. Allele-selective lowering of mutant HTT protein by HTT-LC3 linker compounds. Nature, 2019, 575(7781): 203-209. DOI:10.1038/s41586-019-1722-1
|
| [87] |
Nishimura K, Fukagawa T, Takisawa H, et al. An auxin-based degron system for the rapid depletion of proteins in nonplant cells. Nat Methods, 2009, 6(12): 917-922. DOI:10.1038/nmeth.1401
|
| [88] |
Natsume T, Kiyomitsu T, Saga Y, et al. Rapid protein depletion in human cells by auxin-inducible degron tagging with short homology donors. Cell Rep, 2016, 15(1): 210-218. DOI:10.1016/j.celrep.2016.03.001
|
| [89] |
Koduri V, McBrayer SK, Liberzon E, et al. Peptidic degron for IMiD-induced degradation of heterologous proteins. Proc Natl Acad Sci USA, 2019, 116(7): 2539-2544. DOI:10.1073/pnas.1818109116
|
| [90] |
Fink EC, McConkey M, Adams DN, et al. CrbnI391V is sufficient to confer in vivo sensitivity to thalidomide and its derivatives in mice. Blood, 2018, 132(14): 1535-1544. DOI:10.1182/blood-2018-05-852798
|
| [91] |
Chung HK, Jacobs CL, Huo YW, et al. Tunable and reversible drug control of protein production via a self-excising degron. Nat Chem Biol, 2015, 11(9): 713-720. DOI:10.1038/nchembio.1869
|
| [92] |
Clift D, McEwan WA, Labzin LI, et al. A method for the acute and rapid degradation of endogenous proteins. Cell, 2017, 171(7): 1692-1706. e18. DOI:10.1016/j.cell.2017.10.033
|
| [93] |
Yoshimi R, Chang TH, Wang HS, et al. Gene disruption study reveals a nonredundant role for TRIM21/Ro52 in NF-kappa B-dependent cytokine expression in fibroblasts. J Immunol, 2009, 182(12): 7527-7538. DOI:10.4049/jimmunol.0804121
|
| [94] |
Ding Y, Fei YY, Lu BX. Emerging new concepts of degrader technologies. Trends Pharmacol Sci, 2020, 41(7): 464-474. DOI:10.1016/j.tips.2020.04.005
|
| [95] |
Uhlén M, Fagerberg L, Hallström BM, et al. Proteomics. Tissue-based map of the human proteome. Science, 2015, 347(6620): 1260419.
|
| [96] |
Sinha RA, Singh BK, Yen PM. Reciprocal crosstalk between autophagic and endocrine signaling in metabolic homeostasis. Endocr Rev, 2017, 38(1): 69-102. DOI:10.1210/er.2016-1103
|
| [97] |
Tabrizi SJ, Flower MD, Ross CA, et al. Huntington disease: new insights into molecular pathogenesis and therapeutic opportunities. Nat Rev Neurol, 2020, 16(10): 529-546. DOI:10.1038/s41582-020-0389-4
|
| [98] |
Lonn P, Dowdy SF. Cationic PTD/CPP-mediated macromolecular delivery: charging into the cell. Expert Opin Drug Deliv, 2015, 12(10): 1627-1636. DOI:10.1517/17425247.2015.1046431
|
 2021, Vol. 37
2021, Vol. 37