中国科学院微生物研究所、中国微生物学会主办
文章信息
- 何洁, 徐安明, 刘嘉唯, 周杰, 崔中利, 董维亮, 姜岷
- He Jie, Xu Anming, Liu Jiawei, Zhou Jie, Cui Zhongli, Dong Weiliang, Jiang Min
- 一株聚氨酯降解菌的分离及其降解特性解析
- Isolation and characterization of a polyurethane-degrading bacterium
- 生物工程学报, 2021, 37(10): 3675-3684
- Chinese Journal of Biotechnology, 2021, 37(10): 3675-3684
- 10.13345/j.cjb.210411
-
文章历史
- Received: June 1, 2021
- Accepted: August 12, 2021
2. 南京农业大学 生命科学学院,江苏 南京 210095
2. College of Life Sciences, Nanjing Agricultural University, Nanjing 210095, Jiangsu, China
聚氨酯(Polyurethane,PUR) 是一类衍生自多异氰酸酯和多元醇缩合的聚合物,主要分为聚酯型和聚醚型两种。通过改变多元醇和异氰酸酯的类型和比例,可以生产出各种性质的PUR,依据选择原料的多元醇分子的不同,PUR可被划分为聚酯型PUR和聚醚型PUR。PUR大多数是柔性或硬质泡沫聚合物,广泛应用于建筑、汽车工业、绝缘材料、纺织涂料和防腐蚀涂料等领域[1]。据统计,PUR的使用量占整个塑料使用量的7.9%,我国2019年的PUR使用量约为5.3万t,较上年增长了20%以上[2]。
对PUR的巨大需求将产生等量的固体废物,其中很大一部分目前未回收利用,最终被填埋和焚烧。由于大部分PUR是热固性聚合物,自然条件下的不易分解将导致其在环境中保存数百年以上。受环境风力、河流和洋流等外力迁移作用,PUR废物可能会不断释放环境污染物,例如4, 4′-亚甲基二苯胺(MDA)、2, 4′-甲苯二胺(TDA)和微塑料(Microplastic) 等。值得注意的是,这些二胺物质已经被证明具有潜在的“致癌、致突变和生殖毒性”。此外,相比于肉眼可见的聚氨酯塑料形成的“白色污染”,肉眼不可见的微塑料对整个地球系统具有更大的危害。微塑料污染可以通过大气和洋流进行全球循环。根据已有报道,海产品中存在着大量的微塑料,微塑料还可通过植物根际进入植物体内进行运输,并通过食物链的富集,转移到动物或人类体内,最终影响人类健康[3]。
目前,使用微生物或酶进行生物降解已成为塑料回收的一种有前景的替代方法[4]。首先,利用微生物对PUR废弃物进行生物降解的关键在于获得高催化活性的微生物菌株。目前的研究认为,只有聚酯型PUR才具备生物降解的可能性,聚醚型PUR的生物降解鲜有报道[5]。针对聚酯型PUR的生物降解菌株,已有多株来自β-变形菌门的假单胞菌Pseudomonas protegens被报道具有PUR生物降解的功能。Howard等从绿针假单胞菌Pseudomonas chlororaphis中鉴定得到了第一个具备PUR解聚的脂肪酶PueB[6]。此外,Pseudomonas protegens、恶臭假单胞菌Pseudomonas putida、食酸丛毛单胞菌Comamonas acidovorans等细菌都被发现可以利用类似脂肪酶或酯酶来参与PUR塑料的生物降解[7-9]。近期的一些研究表明,相对于细菌而言,PUR更容易受真菌侵袭。茄病镰刀菌Fusarium solani、嗜酒假丝酵母Candida ethanolica、皱褶假丝酵母Candida rugosa以及枝孢菌Cladosporium sp.都被发现具有PUR降解的功能[10-12],其中多数真菌也是通过脂肪酶和酯酶来发挥PUR的解聚功能[13]。
值得注意的是,尽管目前在PUR生物降解的研究上已经取得了明显进步,但是多数研究还仅仅依靠平板水解圈来判断菌株PUR水解活性的强弱,这种实验检测手段很容易造成结果的误判。例如,一些来源于假单胞菌和芽孢杆菌Bacillus sp.的酯酶能够在可溶性PUR平板上形成巨大水解圈,然而面对商业的PUR塑料却无法发挥降解功能[14]。因此,在筛选PUR高效降解菌株的同时更应该关注菌株对商业PUR塑料的评估。本研究从废弃塑料厂分离鉴定出1株聚氨酯高效降解细菌P10,通过对该菌株降解动力学的检测,发现该菌株能以PUR为唯一碳源进行生长。我们进一步探究了该菌株的系统发育地位及其最适降解条件,并使用商业聚酯型聚氨酯海绵考察了该菌株的降解活性。研究发现,菌株P10不仅具有非常强的PUR模式底物的降解能力,对商业PUR塑料也具有一定的降解效果,表明菌株P10在PUR塑料生物降解领域具备一定的开发价值和应用前景。
1 材料与方法 1.1 实验材料 1.1.1 菌株和PUR塑料样品本研究中使用的菌株P10分离自南通海绵制造厂附近的土壤样品,现为本实验室保存菌株。所使用的PUR塑料模式底物水性聚氨酯(Impranil DLN) 购买自(Bayer Materials Science,PA),商业PUR海绵购买自南通大工有限公司。
1.1.2 培养基和主要试剂的配置(1) LB液体培养基:NaCl 10 g/L,蛋白胨10 g/L,酵母粉5 g/L,在121 ℃条件下高压灭菌20 min。用作细菌的保存活化。
(2) 富集培养基(LD):含0.2% (V/V) Impranil DLN的50% (V/V) LB培养液。
(3) 固体筛选培养基(MDA):无机盐培养基(MSM)——硫酸铵1 g/L,十二水合磷酸二氢钾1.5 g/L,磷酸氢二钾1.5 g/L,二水合氯化钙0.01 g/L,七水合硫酸亚铁0.001 g/L,七水合硫酸镁0.2 g/L,作为基础培养基,调节pH值至7.0,加入琼脂15–20 g/L、121 ℃灭菌20 min。灭菌后注入1% (V/V) 的Impranil DLN。
(4) 液体降解培养基(MD):含0.4% (V/V) Impranil DLN的无机盐培养基。
(5) 磷酸缓冲液配置(50 mmol/L pH 7.0):A液(NaH2PO4·2H2O 3.12 g),B液(Na2HPO4·12H2O 7.17 g),分别溶于蒸馏水,定溶至100 mL。A液与B液按39︰61比例混合,并稀释成400 mL。
(6) 本研究所需主要试剂包括:2.5% (V/V) 戊二醛溶液;1 mol/L HCl;1 mol/L NaOH;水性聚氨酯(Impranil DLN)。
1.2 试验方法 1.2.1 Impranil DLN降解菌的富集与分离称取5 g采自南通海绵制造厂周边的土壤接入50 mL 0.2% (V/V) Impranil DLN的富集培养基(LD) 中,置于30 ℃、180 r/min恒温摇床振荡培养2 d。吸取5 mL富集培养液转接至50 mL新鲜富集培养液中,继续富集培养2 d。将对Impranil DLN富集的菌液按103–106倍梯度稀释后,分别涂布于筛选平板(MDA),37 ℃倒置培养4–5 d。挑选水解透明圈显著且大的菌落,另在空白的筛选平板上三区划线,单菌落即为能够利用Impranil DLN的降解菌株,命名为P10。
1.2.2 形态特征鉴定肉眼观察降解菌在1% (V/V) 的Impranil DLN固体平板上(MDA) 的菌落形状、颜色、透明度、隆起和边缘特征。
离心收集肉眼可见的细胞沉淀芝麻至绿豆大小,弃培养基后用磷酸盐缓冲液轻轻漂洗后,加2.5% (V/V) 戊二醛电镜固定液,并将细胞吹打开悬浮于固定液内,室温固定2 h,再转移至4 ℃保存。将固定好的样品经0.1 mol/L磷酸缓冲液(pH 7.0) 漂洗3次,每次15 min。再用30%、50%、70%、80%、90%、95% (V/V) 的酒精和乙酸异戊酯依次脱水15 min。将样本放入临界点干燥仪内进行干燥。处理后的样品紧贴于导电碳膜双面胶上放入离子溅射仪样品台上进行喷金30 s左右。在扫描电子显微镜下观察采图。
1.2.3 16S rDNA基因序列测定与系统发育分析采用细菌基因组DNA提取试剂盒(天根生化科技(北京) 有限公司) 法提取菌体总DNA。利用16S rDNA通用上、下游引物27F:5′-AGA GTTTGATCCTGGCTCAG-3′和1492R:5′-GGTTA CCTTGTTACGACTT-3′扩增P10菌株的16S rDNA序列,将所获得的扩增片段进行测序。测序结果与NCBI数据库进行比对,利用MEGA-X软件进行多序列比对分析并构建系统发育树。
1.2.4 菌悬液的制备将菌株接种至LB培养基中活化培养14 h后,于6 000 r/min、4 ℃的条件下离心10 min。后续用pH 7.0的无菌磷酸缓冲溶液,重悬底部菌团使其分散,再次离心去除原始培养基,重复此步骤2次,最后一次加PBS后,涡旋分散,不离心,稀释菌悬液(无菌PBS为对照),使其在600 nm处的OD值为0.7–0.8。
1.2.5 Impranil DLN标准曲线的建立采用紫外分光光度法对MSM培养基中Impranil DLN的浓度进行定量测定。以不加Impranil DLN底物的空白MSM培养基为对照品,含Impranil DLN的MSM混合溶液则为测试样品。配置浓度梯度为0.2%、0.4%、0.6%、0.8%、1.0% (V/V) 的MSM-DLN混合溶液,分别稀释5倍后,在特征吸收波长400 nm[8]下测定不同浓度的吸光度值(OD值),并以Impranil DLN溶液浓度为横坐标,吸光度值为纵坐标,绘制标准曲线。
1.2.6 Impranil DLN降解率计算将制备好的菌悬液先按初始比例10% (V/V)接入50 mL的降解培养基(MD) 中,30 ℃、180 r/min振荡培养6 d,同时设置3个实验组及3个统一空白组(无菌接入)。平均每24 h取样,10 000 r/min离心1 min后稀释5倍,在特征吸收波长400 nm下用紫外分光光度计测定Impranil DLN的吸光度值(OD值),同时以无底物无机盐培养液比色调零。根据标准曲线的拟合方程计算培养基中Impranil DLN的浓度。
1.2.7 菌株对Impranil DLN的降解研究通过单因素实验法,将制备好的菌悬液,按初始比例10% (V/V) 分别接入不同pH (4.0–9.0)的降解培养基(MD) 中,连续振荡培养36 h,分别根据在特征波长600 nm和400 nm下测定的OD值,绘制菌种的生长曲线和降解曲线,来确定Impranil DLN对菌株生长的影响,以及培养过程中菌株对Impranil DLN的降解情况。
1.2.8 菌株对聚酯型聚氨酯海绵的降解研究称取0.3 g左右的海绵,分别倒入pH 5.0、80 mL的无机盐培养基和含有5% (V/V) LB的无机盐培养基中,并同时在121 ℃下灭菌20 min。将制备好的菌悬液按10% (V/V) 的比例分别接种到已灭菌的海绵降解培养基中。在30 ℃、180 r/min培养6 d,同时设置3个实验组及3个统一空白组(无菌接入)。6 d后取出海绵,用5% (V/V) 的次氯酸钠溶液浸泡4 h并超声2 h以除去菌体,无菌水每10 min润洗一遍除去残留的次氯酸钠,之后放入60 ℃烘箱烘至恒重,称取并记录降解后的干重。利用扫描电镜观察海绵的表面形态和菌体附着情况,进一步呈现菌株P10对海绵的降解程度。
2 结果与分析 2.1 Impranil DLN降解菌株的筛选及分离为了筛选获得具有潜在PUR降解功能的菌株,我们选择了可溶性的PUR模式底物Impranil DLN作为筛选材料。对取自塑料厂附近的污染土壤经过驯化和富集后,采用如图 1A所示的筛选分离手段,获得1株有效降解Impranil DLN的菌株P10。在温度30 ℃和pH 7.0条件下,P10能利用Impranil DLN作为唯一碳源在MSM培养基中生长,并在24 h测得菌株的最大生长密度(OD600) 为3.501 (图 1B)。该菌株在MDA平板上的菌落呈乳白色,不透明,形态呈圆形,表面光滑,微微隆起,边缘整齐,肉眼可见明显的水解圈(图 1C)。扫描电镜下观察到菌体细胞呈杆状,(0.7–0.9) μm×(3–5) μm,革兰氏阳性,以周生鞭毛运动(图 1D)。

|
| 图 1 菌株P10的筛选及鉴定 Fig. 1 Screening and identification of Brevibacillus sp. P10. (A) Schematic diagram of the process of screening potential PUR-degrading strains using DLN. (B) The growth curve of strain P10. (C) The colony morphology of strain P10 on MSM plate containing 1% (V/V) Impranil DLN. (D) SEM image of strain P10 (12.4 mm×5.00 K). |
| |
通过对菌株P10的16S rDNA序列的比对分析发现,菌株P10与美丽短芽孢杆菌Brevibacillus formosus、短短芽孢杆菌Brevibacillus brevis、桥石短芽孢杆菌Brevibacillus choshinensis等多株短芽孢杆菌的同源性大于98.5%,可以推测菌株P10为短芽孢杆菌属细菌。为进一步确定菌株P10的系统发育地位,利用Geneious和MEGA软件对包括菌株P10在内的31株同源性较高的菌株构建系统发育树。从系统发育树的结果可以看出(图 2),菌株P10与短芽孢杆菌属的细菌聚在了同一分枝。由于16S rDNA的种间分辨率较低,目前还无法通过16S rDNA信息对菌株P10的种间发育地位进行确定。综合形态学、生理生化特征、16S rDNA基因序列和系统发育分析,可以确定菌株P10为短芽孢杆菌属Brevibacillus sp.。

|
| 图 2 菌株P10的16S rDNA序列系统发育树 Fig. 2 The phylogenetic tree based on the 16S rDNA sequences. |
| |
为了探究短芽孢杆菌P10对PUR模式底物(DLN) 的降解特性,我们对DLN浓度与吸光度值(400 nm) 间的标准曲线进行了测定。Impranil DLN的标准曲线拟合方程有较好的线性关系,Impranil DLN浓度与吸光度的线性方程为y=7.114 2x+ 0.083 5。由于DLN具有一定的生理毒性,我们首先评估了菌株对不同浓度DLN的耐受性。通过设置不同的DLN浓度梯度,比较了菌株P10在6 d内对DLN的降解率。图 3A中我们发现随着DLN浓度的升高,菌株P10对DLN的降解率呈现逐渐下降趋势。当DLN浓度为0.2% (V/V) 时,P10可实现对86% DLN的降解,然而对于浓度为2% (V/V) 的DLN,菌株的降解率仅为20%。当DLN的浓度为0.4% (V/V) 时,P10在6 d内即可完成对65% DLN的降解。底物浓度过低时会导致碳源不足,限制菌株生长;而浓度过高一方面会对微生物产生毒性,一方面又会使氮磷等营养元素缺乏导致营养比例失衡,也会使微生物生长受到抑制进而导致降解率下降。因此我们选取0.4% (V/V) 浓度开展了后续的实验。

|
| 图 3 菌株P10对Impranil DLN的降解特性 Fig. 3 Impranil DLN degradation of Brevibacillus sp. P10. (A) Tolerance of P10 to different concentrations of DLN. (B) pH optimization for P10 degradation of DLN. (C) Degradation kinetics of P10 to 0.4% (V/V) DLN. (D) The standard curve between DLN concentration and its absorbance. |
| |
pH是影响微生物生长的重要因素之一,我们探究了不同pH对P10降解DLN水平的影响。由图 3B可知,菌株P10的最适降解pH为5.0。在pH 4.0–6.0条件下,其对Impranil DLN的降解率显著提升,24 h就已达到32.1%。尤其是在pH为5.0时,Impranil DLN的降解率达到最高点71.4%,远远高于碱性条件下的降解率,说明菌株P10适合在酸性的自然环境中发挥高效的降解水平。后续的实验中,我们考察了菌株P10在DLN浓度为0.4% (V/V)、pH为5.0条件下的降解动力学(图 3C)。由图可知,随着培养时间的延长,菌株对DLN的降解水平逐渐减缓,大约在第6天时DLN的降解趋于最高值。
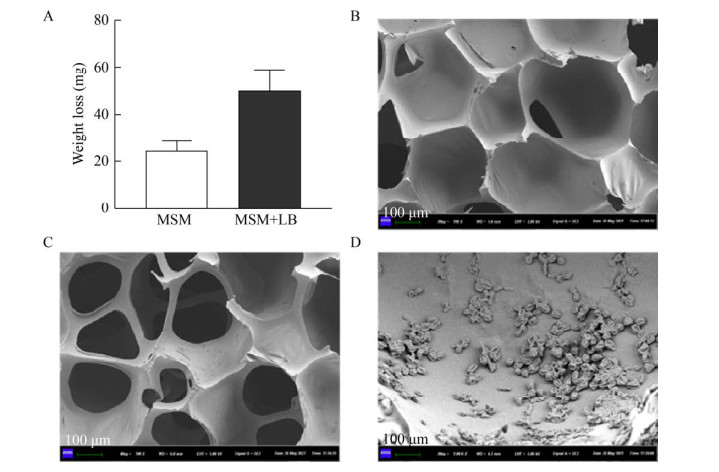
2.4 最优条件下海绵的降解效果在前面的实验中,我们探究了P10对PUR模式底物Impranil DLN的降解特性,菌株P10能够以DLN为唯一碳源进行生长,并表现出良好的降解特性。我们进一步探究了P10对商业聚酯型PUR泡沫的降解。在本研究中,根据图 4A显示,在培养的一周内实验组和对照组均有质量损失。虽然在以海绵为唯一碳源时,P10对海绵的降解率达到20 mg,但在加入5% (V/V) LB后P10对海绵的降解率得到提升,达到50 mg,表明P10能够利用海绵作为碳源进行生长,且更需要易于获得的碳源来维持其生长和触发PUR的生物降解能力。此外,经细菌处理后,在聚酯型聚氨酯海绵的微观结构中也观察到降解的证据。对照组表面平整(图 4B),与对照组相比,实验组不仅看起来被菌丝体生长的物理作用所破坏,而且网状结构更稀疏粗糙,更多孔(图 4C)。在第6天,聚酯型PUR海绵表面已经布满了菌体细胞及其导致的大小不一的侵蚀坑(图 4D),推测此为P10菌株利用该聚酯型PUR作为碳源进行生长。
|
| 图 4 菌株P10对海绵降解效果检测 Fig. 4 PUR foam degradation by Brevibacillus sp. P10. (A) The weight loss of PUR foam after 6-days degradation by strain P10. SEM images of the ultrastructure of PUR foam before (B) and after (C) degradation by strain P10. (D) SEM image of Brevibacillus sp. P10 grown on PUR foam. |
| |
由微生物介导的塑料降解技术是解决塑料环境污染的有效途径。文献已经报道了很多能降解或转化PUR的微生物[15]。Shah等的研究发现,枯草芽孢杆菌MZA-75和铜绿假单胞菌MZA-85都能以聚酯型PUR薄膜为唯一碳源生存,并在30 d内将其质量减少50 mg[16-17]。此外,很多真菌也被发现可以降解聚酯型PUR,Mathur等的研究中黄曲霉Aspergillus flavus菌株能以聚酯型PU薄膜为唯一碳源,并在30 d内,使其质量损失达到60.6%[18]。在聚酯型PUR的微生物降解方面,普遍来看真菌的降解效率高于细菌的降解效率[19]。在这项工作中,我们报告了1株能够以PUR模拟底物Impranil DLN作为生长的唯一碳源并将其高效降解的短芽孢杆菌P10。通过对DLN降解特性的研究,发现低浓度的Impranil DLN有利于该菌株生长代谢,当浓度超过0.6% (V/V) 后会显著降低P10的降解能力。目前,短芽孢杆菌作为一种新型嗜热短杆菌在微生物降解塑料废物的应用研究中逐渐被熟知。例如,Sinosh等筛选出的短芽孢杆菌系列能够在140 d内使聚乙烯和聚丙烯衍生物质量损失50%[20];Hwang等通过使用新型生物过滤器获得了短芽孢杆菌一天内对苯乙烯3 kg/m3的高消除能力[21]。但是,关于该菌株降解聚氨酯塑料的研究甚少,特别是对商业聚氨酯塑料来说。
因此,本研究又考察了菌株P10对聚酯型聚氨酯海绵的降解水平,发现该菌株也能够利用PUR海绵作为唯一碳源进行生长,并在一周内使海绵的质量损失达到20 mg。为了提高降解率,额外添加的共代谢碳源LB,使PUR海绵的质量损失高达50 mg。从单位时间内的降解效果来看,P10菌株对聚氨酯的降解能力稍强于其他研究中的降解菌株。在Gunawan等的研究中显示了在堆肥和土壤中培养的聚酯型聚氨酯泡沫每周能被藻类微生物降解5.8%左右[22]。同样地,在Álvarez-Barragán的工作中,聚醚型聚氨酯泡沫在50% (V/V) 的马铃薯葡萄糖液体培养基(PDB) 中与6种不同的枝孢菌一起培养3周后的重量损失范围为35%–65%[10]。由于聚氨酯成分复杂,加之每项研究中所使用的降解底物均不同,培养条件也不同,因此降解效率也有差别,导致不同研究间的可比性较差。
微生物对PUR塑料的降解在很大程度上受其分泌或表面结合的水解酶作用介导,例如酯酶、脲酶、蛋白酶和酰胺酶[5]。尽管Impranil DLN和PUR泡沫的组成成分不同,但氨基甲酸酯和酯基团是所有聚氨酯中最常见的成分。因此,在利用DLN为唯一碳源生长的过程中,检测上清中酯酶、脲酶和蛋白酶等相关水解酶的表达情况将进一步验证该菌株是否可以分泌水解酶对DLN进行水解。在后续的研究中,我们将进一步对菌株P10参与PUR塑料降解的关键酶和关键解聚产物的代谢途径进行解析,阐明菌株P10降解PUR塑料的分子机制。
| [1] |
Seymour RB, Kauffman GB. Polyurethanes: a class of modern versatile materials. J Chem Educ, 1992, 69(11): 909. DOI:10.1021/ed069p909
|
| [2] |
2020–2025年中国PUR热熔胶行业应用市场需求及开拓机会研究报告[EB/OL]. [2020-07-15]. https://zhuanlan.zhihu.com/p/347578922.
|
| [3] |
Černá T, Pražanová K, Beneš H, et al. Polycyclic aromatic hydrocarbon accumulation in aged and unaged polyurethane microplastics in contaminated soil. Sci Total Environ, 2021, 770: 145254. DOI:10.1016/j.scitotenv.2021.145254
|
| [4] |
Wierckx N, Prieto MA, Pomposiello P, et al. Plastic waste as a novel substrate for industrial biotechnology. Microb Biotechnol, 2015, 8(6): 900-903. DOI:10.1111/1751-7915.12312
|
| [5] |
Mahajan N, Gupta P. New insights into the microbial degradation of polyurethanes. RSC Adv, 2015, 5(52): 41839-41854. DOI:10.1039/C5RA04589D
|
| [6] |
Howard GT, Crother B, Vicknair J. Cloning, nucleotide sequencing and characterization of a polyurethanase gene (pueB) from Pseudomonas chlororaphis. Int Biodeterior Biodegrad, 2001, 47(3): 141-149. DOI:10.1016/S0964-8305(01)00042-7
|
| [7] |
Hung CS, Zingarelli S, Nadeau LJ, et al. Carbon catabolite repression and impranil polyurethane degradation in Pseudomonas protegens strain Pf-5. Appl Environ Microbiol, 2016, 82(20): 6080-6090. DOI:10.1128/AEM.01448-16
|
| [8] |
Peng YH, Shih YH, Lai YC, et al. Degradation of polyurethane by bacterium isolated from soil and assessment of polyurethanolytic activity of a Pseudomonas putida strain. Environ Sci Pollut Res, 2014, 21(16): 9529-9537. DOI:10.1007/s11356-014-2647-8
|
| [9] |
Martínez-González MA, Fernández-Jarne E, Serrano-Martinez S, et al. Mediterranean diet and reduction in the risk of a first acute myocardial infarction: an operational healthy dietary score. Eur J Nutr, 2002, 41(4): 153-160. DOI:10.1007/s00394-002-0370-6
|
| [10] |
Álvarez-Barragán J, Domínguez-Malfavón L, Vargas-Suárez M, 等. Biodegradative activities of selected environmental fungi on a polyester polyurethane varnish and polyether polyurethane foams. Appl Environ Microbiol, 2016, 82(17): 5225-5235. |
| [11] |
Zafar U, Houlden A, Robson GD. Fungal communities associated with the biodegradation of polyester polyurethane buried under compost at different temperatures. Appl Environ Microbiol, 2013, 79(23): 7313-7324. DOI:10.1128/AEM.02536-13
|
| [12] |
Russell JR, Huang J, Anand P, et al. Biodegradation of polyester polyurethane by endophytic fungi. Appl Environ Microbiol, 2011, 77(17): 6076-6084. DOI:10.1128/AEM.00521-11
|
| [13] |
Mathur G, Prasad R. Degradation of polyurethane by Aspergillus flavus (ITCC 6051) isolated from soil. Appl Biochem Biotechnol, 2012, 167(6): 1595-1602. DOI:10.1007/s12010-012-9572-4
|
| [14] |
Biffinger JC, Barlow DE, Cockrell AL, et al. The applicability of Impranil® DLN for gauging the biodegradation of polyurethanes. Polymer Degradation Stability, 2015, 120: 178-185. DOI:10.1016/j.polymdegradstab.2015.06.020
|
| [15] |
Liu J, He J, Xue R, et al. Biodegradation and up-cycling of polyurethanes: progress, challenges, and prospects. Biotechnol Adv, 2021, 48: 107730. DOI:10.1016/j.biotechadv.2021.107730
|
| [16] |
Shah Z, Hasan F, Krumholz L, et al. Degradation of polyester polyurethane by newly isolated Pseudomonas aeruginosa strain MZA-85 and analysis of degradation products by GC-MS. Int Biodeterior Biodegrad, 2013, 77: 114-122. DOI:10.1016/j.ibiod.2012.11.009
|
| [17] |
Shah Z, Krumholz L, Aktas DF, et al. Degradation of polyester polyurethane by a newly isolated soil bacterium, Bacillus subtilis strain MZA-75. Biodegradation, 2013, 24(6): 865-877. DOI:10.1007/s10532-013-9634-5
|
| [18] |
Osman M, Satti SM, Luqman A, et al. Degradation of polyester polyurethane by Aspergillus sp. strain S45 isolated from soil. J Polym Environ, 2018, 26(1): 301-310. DOI:10.1007/s10924-017-0954-0
|
| [19] |
Danso D, Chow J, Streit WR. Plastics: environmental and biotechnological perspectives on microbial degradation. Appl Environ Microbiol, 2019, 85(19): e01095-19.
|
| [20] |
Skariyachan S, Patil AA, Shankar A, et al. Enhanced polymer degradation of polyethylene and polypropylene by novel thermophilic consortia of Brevibacillus sps. and Aneurinibacillus sp. screened from waste management landfills and sewage treatment plants. Polym Degrad Stab, 2018, 149: 52-68. DOI:10.1016/j.polymdegradstab.2018.01.018
|
| [21] |
Hwang JW, Choi CY, Park S, et al. Biodegradation of gaseous styrene by Brevibacillus sp. using a novel agitating biotrickling filter. Biotechnol Lett, 2008, 30(7): 1207-1212. DOI:10.1007/s10529-008-9670-0
|
| [22] |
Gunawan NR, Tessman M, Schreiman AC, et al. Rapid biodegradation of renewable polyurethane foams with identification of associated microorganisms and decomposition products. Bioresour Technol Rep, 2020, 11: 100513. DOI:10.1016/j.biteb.2020.100513
|
 2021, Vol. 37
2021, Vol. 37




