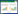中国科学院微生物研究所、中国微生物学会主办
文章信息
- 陈珏, 黄佳敏, 燕天鹤, 彭晓宇, 林峻
- Chen Jue, Huang Jiamin, Yan Tianhe, Peng Xiaoyu, Lin Jun
- 随机突变文库构建与筛选研究进展
- Progress in the construction and screening of random mutation library
- 生物工程学报, 2021, 37(1): 163-177
- Chinese Journal of Biotechnology, 2021, 37(1): 163-177
- 10.13345/j.cjb.200261
-
文章历史
- Received: May 9, 2020
- Accepted: July 29, 2020
- Published: August 12, 2020
2. 福州大学 生物科学与工程学院,福建 福州 350108;
3. 福州大学 福建省海洋酶工程重点实验室,福建 福州 350108
2. College of Biological Science and Engineering, Fuzhou University, Fuzhou 350108, Fujian, China;
3. Fujian Key Laboratory of Marine Enzyme Engineering, Fuzhou University, Fuzhou 350108, Fujian, China
定向进化(Directed evolution)是一种广泛应用的工程策略,通过反复突变和选择来提高目标分子的稳定性或生化功能。通过人工模拟进化历程,将多样化的基因文库翻译成相应的基因产物文库,根据基因型和表型之间的对应关系来筛选功能变体[1]。定向进化已成功用于蛋白质工程,其优点是:在未知蛋白质结构和作用机制时[2],能在现有的蛋白质(天然或经过工程改造)中引入突变,然后筛选出具有增强活性或另一种所需性状的子代蛋白质,持续进化以达到目标性能水平。定向进化利用连续的突变和筛选获得目标DNA,进而得到对应的蛋白质,如改良的抗体[3]、更高效的[4]或活性增强的突变蛋白[5],这些生物分子可用于基础生物学和生物技术研究。目前,实验室的定向进化如图 1所示,已被广泛用于优化或改变单个基因或基因产物的活性。
|
| 图 1 实验室定向进化中的关键步骤 Fig. 1 Key steps in the laboratory directed evolution. |
| |
定向进化的成功主要取决于两个方面:多样性突变和高通量筛选[6]。随着分子生物学技术的发展,研究人员开发出各种各样的突变方法扩大突变文库。扩大突变文库提高了获取目标分子的机率,但也增加了突变文库的筛选难度和分析成本。定向进化策略的关键因素之一是开发合适的选择和筛选技术,这将大大增加从大型突变文库中获取具有所需特性突变体的机会,并减少时间和成本。高通量筛选技术简化了传统筛选过程,减少了时间和成本,更易检测到目标性能变体。因此,采用定向进化技术,不仅需要考虑如何构建突变体文库,还要考虑如何在高通量条件下筛选突变体[7-8]。定向进化的主要局限性是必须开发一种高通量筛选方法,蛋白质半理性设计旨在构建小而精的突变体文库,比其他的随机方法更高效。
本文总结了近年来发表的最新的定向进化方法。用于生成突变文库的传统技术如易错PCR和DNA改组在不断完善中得到广泛应用。重点介绍了正交体内连续进化和3种CRISPR介导的定向进化平台:EvolvR、CRISPR-X和CasPER,还提及了近年来快速发展的蛋白质半理性设计策略。此外,介绍了近年来出现的高通量筛选方法,这些方法共同加速了定向进化领域的发展。
1 突变文库构建策略在自然界中,DNA的遗传突变源于在基因组复制过程中引入错误,紫外线或活性化学物质对DNA产生破坏。在实验室中,选择合适的突变策略是成功构建大型DNA文库的第一步。突变文库的质量对定向进化起着决定性作用。不同的突变策略应该在多样的DNA文库产生以及在突变蛋白结构和功能研究上有广泛应用。用于生成突变文库的传统技术包括易错PCR和DNA改组至今仍在沿用,而新型突变技术的出现简化了定向进化的进程。表 1总结和比较了本文论述的几种突变文库构建策略。
| Mutation method | Evolution strategy | In vivo or in vitro | Targeting | Source of mutation | References |
| Error-prone PCR | Random point mutations introduce genetic diversity through incomplete mutation response | In vitro | Single or multiple genomic loci | Random point mutation | [9-10] |
| DNA shuffling | Sequence diversity of recombinant genes based on homology | In vitro | Multiple genomic loci | Homologous recombination | [14-15] |
| OrthoRep | Error-prone replication target gene | In vivo | Orthogonal mutant plasmid | In vivo error-prone orthogonal replicase | [22] |
| EvolvR | Error-prone repair of single-strand gap produced by nCas9 | In vivo | Target sequence | Error-prone DNA polymerase in vivo | [33] |
| CRISPR-X | dCas9 targets highly active cytidine deaminase AID | In vivo | Multiple genomic loci | dCas9 targeted AID | [34] |
| CasPER | Error-prone PCR and Cas9 binding mediate mutant library | In vitro | Single or multiple genomic loci | Error-prone PCR and in vitro evolution of Cas9 | [35] |
易错PCR (Error-prone PCR)是一种应用广泛的体外随机突变技术。Leung等[9]研究表明DNA聚合酶在某些条件下保真度降低,可在目的基因中随机引入核苷酸突变。McCullum等[10]将易错PCR方法用于优化起始进化的蛋白质,以改善蛋白质的稳定性、溶解性以及结合亲和力。Arnold等[11]使用易错PCR将随机突变引入了枯草杆菌蛋白酶E。Mao等[12]采用易错PCR法,突变具有草甘膦抗性的植物内源基因EPSPS,得到5种突变体ELs-EPSPS,将抗草甘膦的能力提高5.4倍。之后,Shao等[13]开发了原位易错PCR (is-epPCR)法,可通过多轮is-epPCR获得一个序列的所有突变,以进一步提高构建定向进化突变文库的效率。该方法具有以下优点:1)封闭环形PCR产物可以直接转化大肠杆菌感受态细胞,且易于被其他抗生素筛选;2)在表达质粒中对可变DNA进行一步易错扩增,建立突变文库并进行筛选;3)通过多轮is-epPCR可以在序列中获得目标突变的积累。
DNA改组(DNA shuffling)也被广泛用于体外突变文库的产生。DNA改组是由W.P.C Stemmer发明的一种同源基因体外重组的方法[14-15]。最初,该技术用于随机重组单个基因的各种突变体,如单基因改组。随后,DNA改组也能有效重组不同微生物菌株和物种的同源基因,如家族改组(Family shuffling)[16]。Arnold等[17]开发了一种简单且可重复的方法,他们利用核酸内切酶V产生DNA片段,成功实现了DNA改组,其重组效率与DNase Ⅰ相当。Ouyang等[18]开发了一种新型的DNA改组方法,以构建重组大肠杆菌菌株中的随机突变文库。漆酶CotA在大肠杆菌中的异源表达只有较低的催化效率,不能满足工业要求,而鉴定出的突变体5E29催化活性是原来的1.21倍。
1.2 正交体内连续进化聚合酶(Polymerases)校对和复制后错配修复是确保保真度的关键因素。研究人员为此开展了大量的DNAPs (DNA polymerases)工程工作,得到了高度易错的正交DNAPs变体。在真核生物中,酵母是用于定向进化的优秀宿主菌,它能快速维持较大的种群规模。Herr等[19]研究发现酵母能够承受1 000倍的突变率。Gunge等[20]在酿酒酵母Saccharomyces cerevisiae中开发了一种特殊的DNA复制系统,该系统由一对与基因组复制正交的DNAP /质粒对组成,工程化的正交DNAP只复制正交质粒。正交复制系统是体内连续进化的平台,可以独立于宿主进行调控[21]。
以Chang C. Liu为代表的研究团队[22]在酵母中开发了一种高度易错的正交复制系统(OrthoRep),独立于宿主的体内连续进化系统,其突变率比体内宿主基因组高10万倍。OrthoRep由一个含有目标基因的末端蛋白(Terminal proteins,TP)质粒和另一个含有所有必需基因的质粒组成。DNAP靶向TP质粒,实现了正交TP-DNA聚合酶(DNAP)自主复制过程中的靶向突变,从而导致目标质粒的快速突变。OrthoRep基于一种DNA聚合酶(TP-DNAP1),它是能在酿酒酵母中自主复制的线性、高拷贝性细胞质DNA质粒p1。在p1上编码的基因连续突变速度比基因组上编码基因的突变速度快约100 000倍,而染色体基因则保持其自然突变率为每个碱基10–10个替换(s.p.b)。酵母细胞质定位的pGKL1/TP-DNAP1质粒/DNA聚合酶对形成了一个正交的DNA复制系统。在不影响基因组复制的情况下,其突变率显著提高,从而支持了体内连续进化。Ravikumar等[22]利用OrthoRep在90个独立复制实验中进化出耐药性疟疾二氢叶酸还原酶(Drug-resistant malarial dihydrofolate reductases,DHFRs),为生物分子和细胞功能的常规、高通量进化提供了一种新范例,并证明其在耐药性研究方面的效用。
在现有的进化实验中,OrthoRep能保持超过300代的高突变率。然而,OrthoRep使用一种特殊的转录系统,其组成部分(如启动子)限制了OrthoRep编码基因的表达强度。Ravikumar等[22]试图增强OrthoRep的表达水平,使其在蛋白质进化中更具通用性;他们从之前的OrthoRep连续进化实验中筛选启动子突变体,将这些启动子与基因编码的3′poly(A)尾巴结合。Zhong等[23]开发了一组OrthoRep基因表达盒,使OrthoRep编码基因超出酿酒酵母内源基因可以达到的表达水平。具体来说,各种启动子突变以及经过基因编码的poly(A)尾巴增加了OrthoRep上编码基因的表达水平。表达水平在传代过程中稳定提高,并在多个基因和具有不同突变率的OrthoRep系统中保持一致。这些表达上的改进扩展了OrthoRep的应用范围,进一步将OrthoRep的适用性扩展到蛋白质体内连续进化。
Arzumanyan等[24]开发了pGKL2/TP-DNAP2质粒/DNA聚合酶对,形成了第2个正交复制系统;他们构建了p2质粒和TP-DNAP2所需的相关遗传技术,并证明pGKL2可以编码和表达自定义基因,可以工程化易错的TP-DNAP2s。TP-DNAP2复制的pGKL2既与酿酒酵母基因组复制正交,又与TP-DNAP1复制的pGKL1正交,从而在同一细胞中形成2个相互正交的DNA复制系统。这样,一对正交复制系统可以调节2个聚合酶的错配,实现多基因在体内以不同的突变率进化。这2个相互正交的DNA复制系统为合成生物学的新应用奠定了基础。2019年,Javanpour等[25]继续研究和扩展了OrthoRep的通用性,这将促进OrthoRep在定向进化中的广泛应用;他们利用CRISPR/Cas9加速p1的遗传操作,显示至少22 kb的遗传物质可以很容易地在p1上编码,这表明OrthoRep是一种高效通用的酵母系统,广泛适用于体内目标基因连续进化。
1.3 CRISPR/Cas 介导的定向进化从化脓性链球菌Streptococcus pyogenes中获得的Ⅱ型CRISPR系统,又称CRISPR/Cas9系统。CRISPR/Cas9可在使用者选择的DNA序列上产生一个位点特异性双链断裂(DSB)。这种DSB可以通过精确的同源定向修复(HDR)或易出错的非同源末端连接(NHEJ)修复机制进行修复,达到定点敲除某种基因的目的。化脓性链球菌Cas9 (SpCas9)需要原型间隔区相邻基序(PAM)序列(NGG)用于靶识别,从而限制可靶向的基因组位点[26]。
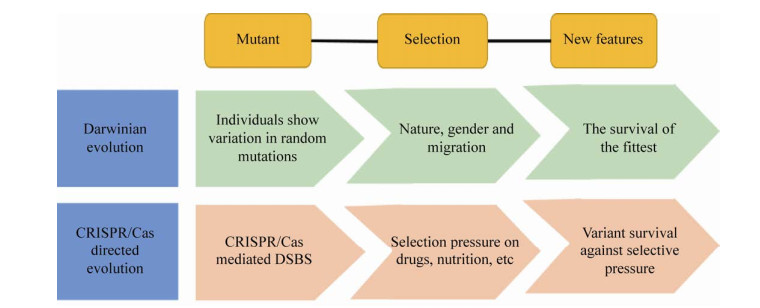
CRISPR技术的不断发展,为定向进化的研究带来了机遇和挑战。CRISPR/Cas9已可用于靶向几乎任何基因组位点并执行定向进化。CRISPR/Cas定向进化(CDE)平台采用CRISPR/ Cas9在目的基因所有可能的编码序列位点产生DSB,CRISPR/Cas9要在NGG PAM序列进行切割。CDE模仿达尔文进化(图 2),该方法高效简便且高度灵活。CDE通过设计多个具有不同目标序列的sgRNA同时进行多位点编辑[27],而在新的靶位点只需要设计新的sgRNA,这极大地简化了基因编辑过程并增加了目标位点的选择性[28]。CDE平台正成为植物科学研究的有力工具[29-31]。Butt等[32]使用CDE进化水稻SF3B1剪接体蛋白,展示了CRISPR/Cas9在定向进化上的应用;他们设计sgRNA靶向文库,克隆到二元载体中,然后转化到根瘤土壤杆菌中培养,在选择压力下再生以加速进化。
|
| 图 2 CRISPR/Cas定向进化模仿了达尔文进化 Fig. 2 CRISPR/Cas directed evolution mimics Darwinian evolution. |
| |
为了提高进化能力,可以通过多种策略来扩大目标突变范围。科学家已开发多种CRISPR/Cas介导的定向进化平台,包括EvlovR[33]、CRISPR-X[34]和CasPER[35],能在未来的CDE平台中发挥作用。
1.3.1 EvolvR传统的连续定向进化要求严格的培养条件[36]且需在用户定义的范围内突变目标核苷酸[37]。目前,还没有一种高效的方法可以连续地使目标区域的所有核苷酸多样化。新的正向遗传学需要能够在目标区域不断地使所有核苷酸多样化的靶向突变体。
Halperin等[33]开发了一种名为EvolvR (图 3)的新型诱变工具。使用EvolvR在大肠杆菌中靶向2个基因:核糖体蛋白亚基rspE和rspL。两者都有已知的突变,分别针对大观霉素和链霉素的耐药性。EvolvR分别靶向rspE和rspL,同时进化这两个基因,以此产生对大观霉素和链霉素耐药的菌株。此外,研究发现rspE的新突变也赋予了大观霉素抗性,这证实EvolvR可用于产生新的蛋白质变体。
|
| 图 3 EvolvR突变过程示意图 Fig. 3 EvolvR mutation process diagram. |
| |
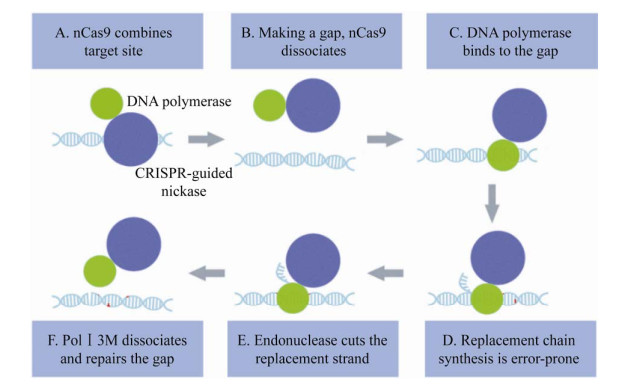
EvolvR系统巧妙地将CRISPR/Cas9的特异性与易错DNA聚合酶的突变能力结合。它通过用户定义的20 nt间隔序列的向导RNA (gRNA)来实现精准定位。EvolvR有两个重要元件,包括变体Cas9 (nickase Cas9或nCas9)[38],其中RuvC核酸酶结构域发生了突变,仅发生单链断裂。还有1个直接与nCas9融合的易错聚合酶(PolⅠ3M)[39]。PolⅠ3M包含3个点突变,这些突变会增加其错误率并消除其校对活性。研究证明了这两种元件的融合使PolⅠ3M的诱变活性指向nCas9靶向基因组位点,而不是基因组中任何切口DNA。
从机制上讲,nCas9首先定位目标基因组位点,并在与gRNA互补的单链DNA上产生1个切口(图 3A)。nCas9分离后(图 3B),PolⅠ3M在该切口处结合(图 3C)。PolⅠ3M聚合1条新的DNA链,偶尔引入错误碱基,并置换旧的DNA链(图 3D)。在解离前,PolⅠ3M的瓣状内切核酸酶结构域切割原来的置换链,留下了1个可连接的缺口(图 3E)。PolⅠ3M解离后,在宿主细胞内完成缺口的修复工作(图 3F)。在任何可被CRISPR/Cas9靶向的基因组或质粒位点下游的小区域引入半随机突变,EvolvR可以重新定位并连续进化相同的位点。
在第1次迭代中,EvolvR在目标位点的突变率是野生型大肠杆菌的24 500倍。为了进一步提高靶向突变率,将3个点突变(K848A,K1003A,R1060A)引入到nCas9 (增强的nCas9,enCas9)[40]中以提高其解离率,增加了PolⅠ3M与DNA结合的机会,从而提高了目标位点的突变率。此外,考虑到PolⅠ3M的持续合成能力(15–20 nt)是一个限制因素,将PolⅠ3M与T7噬菌体的硫氧还蛋白结合域(Thioredoxin-bingding domain,TBD)融合[41],用于提高PolⅠ的持续合成能力。基于这些改进,研究人员利用EvolvR测试到的突变率是野生型的212 000倍,且合成长度达56 nt。
EvolvR有很大的发展空间,拓展PAM范围是其能被更广泛应用的一个关键,可借鉴扩大CRISPR靶向范围的研究。Hu等[42]利用一种噬菌体辅助的连续进化系统(Phage-assisted continuous evolution,PACE)进化出一种能够识别多种PAM序列的SpCas9变体(xCas9)。xCas9可以广泛识别多种PAM序列(包括NG、GAA和GAT),这支持了它在人类细胞中的应用,包括靶向转录激活,核酸酶介导的基因破坏以及胞苷和腺嘌呤碱基编辑。xCas9具有更广泛的PAM兼容性,但与SpCas9相比,它具有更大的DNA特异性。在NGG目标位点,它的脱靶全基因组活性较低,当使用非NGG PAM作为靶标时,其脱靶活性较低。这一发现扩大了CRISPR系统DNA的靶向范围,并证明在Cas9的编辑效率,PAM兼容性和DNA特异性之间没有必要的联系。Anders等[43]开发了一种SpCas9突变体(SpCas9-NG),用于识别NG而不是NGG。先前的研究表明,SpCas9的Arg1 333和Arg1 335分别识别NGG-PAM中第2和第3个碱基。通过消除Arg1 335和第3个G之间的碱基特异性相互作用来减少PAM约束。新引入的非碱基特异性相互作用弥补了与第3个G的碱基特异性相互作用的损失,从而能够识别NG PAM。研究者发现SpCas9-NG在NG PAM靶点诱导插入缺失。此外,SpCas9-NG与活化诱导的胞苷脱氨酶(Activation-induced cytidine deaminase,AID)的融合可以介导NG PAM在靶位点产生C-T的转换。
EvolvR系统已成为CRISPR工具箱中的一种最新的独创性工具。EvolvR可以在任何细菌菌株中使用,但目前只测试了其在大肠杆菌中的用途,可能需要进一步优化其在致病性或肠道共生细菌中的用途。最后,EvolvR能否应用于真核细胞尚待证实,其复杂的基因组结构和DNA复制/修复途径可能需要进一步的工程优化[44]。
1.3.2 CRISPR-X定向进化需要引入特定大小的DNA文库。随着CRISPR/Cas9系统的出现,活性Cas9突变能有效引入插入和缺失,但可能会破坏功能元件[45],使蛋白功能失活。而催化失活的Cas9 (dCas9)[28]能将功能蛋白定位到特定的基因组位点,这扩展了活性Cas9进行蛋白质工程的研究范围。
Hess等开发的CRISPR-X[34],通过dCas9靶向高活性胞苷脱氨酶AID[46]以诱导局部、多样的点突变(图 4)。活性Cas9引入突变主要通过产生插入和缺失,或者通过Cas9切割后的同源重组生成寡核苷酸突变库。相比之下,CRISPR-X产生局部序列多样化,可同时突变多个基因组位点,特异性诱变内源性靶标。测试后发现,只要简单地电穿孔一个sgRNA和靶向AID,GFP就可以很容易地进化成EGFP[34]。利用该技术成功突变了GFP并筛选出光谱转移变体EGFP,同时利用一种活性显著增加的AID变体可以突变转录起始位点上游和下游的内源性位点。此外,对癌症治疗药物硼替佐米的靶点PSMB5进行突变,揭示已知的和新型的耐药机制,并为PSMB5的功能研究和未来的药物研发提供参考[34]。
通过电穿孔引入CRISPR-X,同时靶向多个sgRNAs,可以诱发多个基因组位点突变。CRISPR-X是一种强大的工具,允许在CRISPR靶点引入不同的点突变,连续突变和进化出新的蛋白质功能。可以设想,该系统能使两种相互作用的内源性表达蛋白共同进化,并为在自然环境下突变选择活性增强的抗体和酶蛋白提供一种简化的策略。
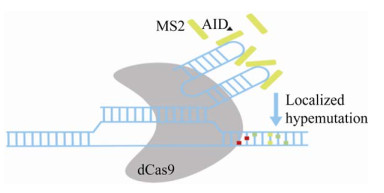
1.3.3 CasPER同源重组能高效地靶向和整合异源DNA片段。在酵母中能将同源末端的线性片段整合到染色体[47-48]。通过Cas9能将较大的DNA片段组装并整合到酿酒酵母基因组中。但目前,大多数CRISPR/Cas9定向进化方法倾向于整合相对较小(80–120 bp)的DNA片段[49-50],可能需要耗时构建供体变体文库或合成相对昂贵的多样化寡核苷酸DNA[51]。在此基础上,Jakočiūnas等扩展了现有的CRISPR技术,采用易错PCR和Cas9介导的基因组整合方法,将大型供体突变体文库整合到单个或多个基因组位点,成功率达到98%–99%[35]。该方法称为CasPER,是Cas9介导蛋白质进化反应的简称。
CasPER包括以下主要步骤(图 5):首先,选择合适的DNA突变片段。其次,在目标靶点设计gRNA。之后,在宿主基因组中合成或扩增缺乏PAM的DNA模板,通过多轮易错PCR (epPCR)进行突变。epPCR利用Cas9介导双链断裂(DSB)上下游基因组位点同源的52 bp引物产生突变文库。最后,通过电穿孔将突变的线性片段与携带gRNA的质粒共同转化到目标受体中。
CasPER在同源基因组中能促进数百个核苷酸的有效突变。通过多种方式将大型突变DNA片段整合到一起,其效率几乎达到100%,从而产生数百万个突变体。研究员利用CRISPR/Cas9在基因组靶点引入DSB,并通过天然同源定向修复整合300–600 bp线性DNA供体,提高酶在自然环境中的定向进化效率。该方法适用于以相同的效率靶向单基因组和双基因组基因座,使基因组整合子的突变分布保持均匀。应用CasPER对酿酒酵母甲羟戊酸途径中的2种关键代谢酶进行定向进化,可将异戊二烯产量提高11倍。
CasPER通过同源重组支持异源DNA基因组整合,可以在供体整个片段上实现多重基因组工程,从而实现高效率和高通量的进化研究。CasPER已在酵母中得到应用,未来它还能够应用于其他的具有高效的同源重组机制的有机体。
1.4 蛋白质半理性设计理论上,定向进化首先通过随机突变和体外重组产生分子多样性,然后通过高通量筛选或选择来鉴定突变体。并不是所有的酶活性都适用高通量筛选方法,也不是所有的筛选方法都易于实现。定向进化存在局限性,即使文库量达数百万个,对一个蛋白质来说,仍然只能从庞大的文库中抽取小部分样本。实验方法的偏差性和遗传密码的简并性进一步限制了文库的设计[52-54]。研究人员不是通过构建更大的文库和进行更多的筛选或选择来解决这些问题,而是通过超越传统的定向进化。Lutz等[55]开发出更小、更高质量文库的新策略,形成半理性设计(Semi-rational design)策略。半理性设计主要借助生物信息学方法,基于蛋白结构、功能、序列同源性以及计算预测法来预先选择合适的目标位置,再以有限的氨基酸多样性进行集中突变。该方法理性选取多个氨基酸残基进行突变,选用有效的密码子,通过构建高质量突变体文库,有针对性地对蛋白质进行改造[56]。
为了减少定向进化过程中的筛选工作,O’Maille等依据结构和序列信息开发了一些半理性“文库设计”策略,如基于结构的组合蛋白质工程(Structure-based combinatorial protein engineering,SCOPE)[57]、组合位点饱和突变(Combinatorial site-saturation mutagenesis,CSSM)[58]及其他方法,旨在生成更小和功能更丰富的突变库。随着计算机技术的发展,半理性设计可以进一步缩小筛选的突变库容量,在尽可能小的文库中,产生更多有益突变。半理性设计越来越多地被用于设计“小而精”的突变体文库[59-60]。除了基于序列和结构的设计策略外,量子化学(Quantum mechanics,QM)和分子动力学模拟(Molecular dynamics,MD)计算以及机器学习算法能有效地探索氨基酸取代对蛋白质结构和稳定性影响。当针对选择性和活性的进化时,首选组合活性位点饱和突变(Combinatorial active-site saturation test,CAST)及迭代饱和突变(Iterative saturation mutagenesis,ISM)策略,这2种方法都在结合口袋附近的位点进行饱和突变(Saturation mutagenesis,SM)[61]。随着半理性设计策略的快速发展,Fox等[62]基于统计学方法开发的ProSAR (Protein sequence activity relationships)和Gonzalez等[63]基于序列同源性开发的MORPHING (Mutagenic organized recombination process by homologous in vivo grouping)工具也广泛用于蛋白酶的设计和改造。Chen等[64]采用3种半理性策略,CSSM和2种基于结构的计算设计算法Corbit[65]和CRAM构建了野生型P450-BM3血红素结构域的突变文库,获得了丙烷和乙烷羟基化的P450-BM3变体。在半理性设计的基础上,采用蛋白质工程技术,Jamil等[66]成功地改善了单胞菌PpADI (Pseudomonas plecoglossicida)的精氨酸脱亚氨酶(Arginine deiminase,ADI)的疏水性以提高其结构稳定性和催化效率。Shin等[67]首次采用蛋白半理性设计改善人参皂苷的β-糖苷酶(β-glycosidase)水解活性。这些研究表明,半理性设计使研究人员加强对酶的结构和功能的理解。
2 突变体的选择和筛选方法随着分子生物学的发展,已开发出多种突变方法来引入足够的遗传多样性。定向进化实验的主要限制是从庞大的突变体文库中鉴定出目标变体。常规的突变文库筛选方法无法适应较大突变体文库的筛选,人们开发了新的筛选和选择方法。成功的定向进化需要高通量的筛选或选择方法,这大大增加了获得所需特性的机会,并减少了时间和成本。表 2给出了一些定向进化实验中文库选择和筛选方法的比较,包括适用范围和优缺点。
| Strategy | Library quantity | Advantages | Disadvantages | References |
| Phage Display | 109 | Multiple proteins can be displayed | Not suitable for selecting multiple enzymatic properties; not suitable for selecting eukaryotic proteins | [68-70] |
| PACE | 109 | Fast and high throughput | Need phage | [42] |
| FACS | > 107 | High sensitivity and extremely high throughput | The expression level of fluorescent protein is related to enzyme activity | [80-81] |
| FADS | > 109 | Large library | Need fluorescent display | [82] |
定向进化联合体外筛选技术是一种获取具有所需功能特性的核酸和蛋白质的强有力方法。噬菌体展示技术由Smith教授[68]于1985年首次引入,它是一种生产多肽(多达1010种)和蛋白质以及筛选特定功能分子非常有效的方法[69-70]。1990年,McCafferty等[71]将噬菌体展示(Phage display)技术成功地从多肽的展示与筛选扩展到抗体的展示与筛选,人类从此进入了不经过免疫也能获得特异性抗体的时代。噬菌体展示具有高通量、快速、易用性等特点,可用于改造人类抗体[72-73]。截至2016年初,利用噬菌体展示研发的6种人类抗体已获批准并用于治疗[74]。2018年,“多肽和抗体的噬菌体展示”技术荣获诺贝尔化学奖。
噬菌体展示技术与时俱进。Frei等[75]总结了噬菌体文库的合成路径,强调设计人性化的抗体文库和组合扫描诱变文库的重要性。Cung等[76]还开发了一种微流体芯片,该芯片能够单轮识别无需细菌感染的高亲和力噬菌体展示肽,应用领域覆盖了蛋白质、肽和分子相互作用的基础研究,以及全自动蛋白质组学筛选。
2.2 噬菌体辅助连续进化对基因反复突变和功能性选择,定向进化取得了非凡的成功。现有的定向进化方法费力耗时难以扩展,需要一个能在体内连续进化的平台。噬菌体辅助连续进化(Phage-assisted continuous evolution,PACE)系统[36]将目的蛋白与噬菌体生命周期耦联,通过自动化连续培养实现目标分子连续进化。噬菌体生命周期短,仅10 min即可产生子代噬菌体。该系统利用M13噬菌体的增殖能力,依赖于诱导蛋白质Ⅲ (pⅢ蛋白)的特性,构建了一个包含目的基因而pⅢ蛋白缺陷型的选择噬菌体。与其他系统不同,目标基因在M13噬菌体中编码,而突变(致突变质粒)和选择性(辅助质粒)因子则位于大肠杆菌中。当噬菌体感染大肠杆菌时,大肠杆菌突变质粒触发噬菌体的突变,仅释放那些诱导蛋白质Ⅲ (pⅢ)产生的突变噬菌体,从而实现筛选。PACE系统一天中可以进行多代噬菌体选择性复制和数十轮进化,无需人工干预。
PACE代表了生命系统中许多蛋白质和核酸的整合,快速生成具有新活性的生物分子。PACE已经用于优化一个扩展的PAM SpCas9突变体(xCas9)[42]。PACE可在8 d执行多达200轮的蛋白质进化,为多种蛋白质或酶的改造提供了可能。David Liu研究团队[42]利用PACE成功进化了T7 RNA聚合酶,该酶识别独特的启动子。PACE在一周内进化出3种目标活性的酶,这3种酶活性均超过其野生型T7 RNAP,提高了实验室进化速度。
David Liu研究团队不断优化PACE,开发了可溶性表达噬菌体辅助的连续进化(Soluble expression phage-assisted continuous evolution,SE-PACE)[77]。SE-PACE是一种快速进化蛋白质的系统,以快速增加其可溶性表达。实验表明,在短短3 d内,SE-PACE进化出了几个抗体片段,表达量提高了7倍,同时保持了其结合活性。另外还开发了一种具有独立活性的SE-PACE来纠正麦芽糖结合蛋白(Maltose-binding protein,MBP)的折叠缺陷变体,并进化出具有改进表达特性的真核细胞胞苷脱氨酶APOBEC1变体。当这些进化的APOBEC1变体取代野生型胞苷脱氨酶时,可以改善Cas9衍生碱基编辑的表达活性。这些结果表明,SE-PACE可以应用于多种蛋白质并快速提高它们的可溶性表达。
2019年,David Liu研究团队又开发了一种利用噬菌体辅助连续进化的碱基编辑器(Phage-assisted continuous evolution of base editors,BE-PACE),以提高其编辑效率和目标序列兼容性[78]。碱基编辑器利用DNA修饰酶和催化缺陷的CRISPR蛋白精确地引入点突变。迄今为止,已经开发出两类碱基编辑器:胞嘧啶碱基编辑器(CBE)可将C•G转换为T•A,腺嘌呤碱基编辑器(ABE)可将A•T转换为G•C[79]。BE-PACE每天可进行数十代的突变、选择和复制,从而产生具有提高目标序列兼容性和更高活性的碱基编辑器。该团队利用BE-PACE系统,克服传统CBE的靶序列限制的缺陷,进化产生了3种类型的胞嘧啶碱基编辑器。进化的EvoAPOBEC1-BE4max与野生APOBEC1脱氨酶相比,在GC背景下编辑胞嘧啶的效率提高26倍,并且在所有测试序列上都能有效编辑。该团队还进化出一种CDA1脱氨酶的CBE (CBE-EvoCDA1-BE4max)变体,对于较难编辑的靶位点有较高的编辑效率。BE-PACE系统不但可以丰富碱基编辑性能,还可以扩大碱基编辑的应用范围,为碱基编辑系统的优化提供强大工具。这些发现为碱基编辑的快速发展建立了一个系统,并为其使用和改进提供帮助。
2.3 流式细胞荧光分选技术定向进化的主要瓶颈是缺乏快速和通用的高通量筛选(High-throughput screening,HTS)方法。流式细胞荧光分选(Fluorescence activated cell sorting,FACS)能筛选分离出活性显著改善、底物特异性改变的酶变体[80]。FACS因其高灵敏度和高分析量(每天多达108个突变体)的特性从而成为筛选突变文库的强大工具。Tan等[81]开发了一种荧光活化细胞分选系统,建立了首个针对岩藻糖基化酶的单细胞超高通量筛选技术平台,能够对这些酶进行超高通量筛选(> 107个突变体/h),并设计一个配套策略来快速定量评估单细胞高通量筛选效率;在这项研究中,他们设计和合成了几个用于岩藻糖基化反应的荧光标记底物衍生物。这些岩藻糖基化酶产生了易于保留在细胞中的荧光寡糖产物,从而促进一种新的基于流式细胞仪的岩藻糖基化酶定向进化系统的开发。研究证明这一系统可作为一种通用的筛选工具,用于α-1, 3-岩藻糖基转移酶、α-1, 2-岩藻糖基转移酶,以及岩藻糖苷酶的转糖苷活性等糖基化反应的定向进化。FACS提高了筛选通量,但并非所有酶都适用FACS。今后,如何保持基因型和表型之间的联系是FACS要面临的主要挑战。
2.4 液滴微流控技术Baret等[82]将FACS技术与液滴技术相结合,开发了一种荧光活化液滴分选(Fluorescence activated droplet sorting,FADS)系统,可以高通量筛选含氟底物的酶库。Agresti等[83]提出了一种通用的超高通量筛选平台,该平台基于液滴的微流控技术,改变了筛选的规模和速度。该技术已经成为酶定向进化的有力工具,利用超高通量进行初始纯化选择以去除无活性的突变体,以每秒数千次的速度进行分类,确定了辣根过氧化物酶(Horseradish peroxidase,HRP)的突变体,该突变体的催化速率比亲本快10倍以上。与最先进的机器人筛选系统相比,该系统能够在10 h内筛选出108个HRP突变体,使用的试剂总量小于150 μL,整个检测速度提高了1 000倍,成本降低了约100万倍。
筛选庞大的突变文库以实现酶对映体选择性的定向进化一直极具挑战性。以前,改进酶对映体选择性是在微量滴定板上进行的,并且此类方法的通量被限制为每天少于104个菌落[84]。基于超高通量液滴的微流控技术可显著提高筛选菌株的能力。之后,Ma等[85]提出“超高通量双通道微流液滴筛选(Dual-channel microfluidic droplet screening,DMDS)”技术,可每天筛选多达107种酶变体。使用该系统筛选酯酶的对映选择性能获得性质优良的消炎药洛芬烯的对映异构体,已从500万个突变体中鉴定出一种对映选择性增加700倍的突变体,证明了该筛选平台可用于复杂酶特性(如区域选择性、化学选择性和对映选择性)的定向进化。
在以前的研究工作中,Ma等基于微滴反应器建立了酶的超高通量筛选系统。筛选速度可以高达108个克隆/d,比常规筛选高出1 000倍以上,同时大大减少了昂贵的荧光底物的消耗。然而,由于许多商业荧光底物不能适当地保留在微滴反应器中,导致荧光信号在微反应之间扩散,降低了筛选精度和效率,限制了系统的应用范围。Ma等[86]提出了一种酶底物设计新策略,称作“荧光液滴捕获(Fluorescence droplet entrapment,FDE)”。利用2种不同的底物工程策略,设计了几种FDE底物,用于检测磷酸三酯酶、羧酸酯酶和糖苷酶活性。
3 总结与展望传统突变技术易错PCR和DNA改组不断引入创新,而新技术的涌现为定向进化的未来发展提供了一个新视角。本文主要介绍以下几个新的突变技术:1)正交DNA聚合酶-质粒OrthoRep是一种简化的连续进化系统,加速了定向进化的发展。它能够使目标基因在宿主酵母繁殖时快速稳定地突变和进化。2)传统的定向进化通常不可能在多个位点进行独立的突变,而CRISPR介导的定向进化可以利用不同的sgRNAs定位多个位点,扩大了突变文库,并将其应用于动物、植物、微生物等不同物种中,具有广阔的发展前景。3)蛋白质半理性设计快速发展,有针对性地构建高质量的小型突变文库,进一步降低了筛选工作量。
稳定的高通量筛选方法至关重要。如何使高通量筛选方法适用更广泛的酶功能,是未来研究的重点。科学家们还需不断地改进现有的技术手段,研发和优化更高效的文库构建和筛选方法,为定向进化创造更加诱人的前景。
| [1] |
Packer MS, Liu DR. Methods for the directed evolution of proteins. Nat Rev Genet, 2015, 16(7): 379-394. DOI:10.1038/nrg3927 |
| [2] |
Tobin MB, Gustafsson C, Huisman GW. Directed evolution: the 'rational' basis for 'irrational' design. Curr Opin Struct Biol, 2000, 10(4): 421-427. DOI:10.1016/S0959-440X(00)00109-3 |
| [3] |
Doerner A, Rhiel L, Zielonka S, et al. Therapeutic antibody engineering by high efficiency cell screening. FEBS Lett, 2014, 588(2): 278-287. DOI:10.1016/j.febslet.2013.11.025 |
| [4] |
Bornscheuer UT, Huisman GW, Kazlauskas RJ, et al. Engineering the third wave of biocatalysis. Nature, 2012, 485(7397): 185-194. DOI:10.1038/nature11117 |
| [5] |
Soskine M, Tawfik DS. Mutational effects and the evolution of new protein functions. Nat Rev Genet, 2010, 11(8): 572-582. DOI:10.1038/nrg2808 |
| [6] |
Wong TS, Roccatano D, Zacharias M, et al. A statistical analysis of random mutagenesis methods used for directed protein evolution. J Mol Biol, 2006, 355(4): 858-871. DOI:10.1016/j.jmb.2005.10.082 |
| [7] |
Wong TS, Tee KL, Hauer B, et al. Sequence saturation mutagenesis (SeSaM): a novel method for directed evolution. Nucleic Acids Res, 2004, 32(3): e26. DOI:10.1093/nar/gnh028 |
| [8] |
Körfer G, Pitzler C, Vojcic L, et al. In vitro flow cytometry-based screening platform for cellulase engineering. Sci Rep, 2016, 6: 26128. DOI:10.1038/srep26128 |
| [9] |
Leung DW, Chen E, Goeddel DV. A method for random mutagenesis of a defined DNA segment using a modified polymerase chain reaction. Technique, 1989, 1: 11-15. |
| [10] |
McCullum EO, Williams BAR, Zhang JL, et al. Random mutagenesis by error-prone PCR//Braman J, Ed. In vitro Mutagenesis Protocols. Totowa, NJ: Humana Press, 2010, 634: 103-109.
|
| [11] |
Chen K, Arnold FH. Tuning the activity of an enzyme for unusual environments: sequential random mutagenesis of subtilisin E for catalysis in dimethylformamide. Proc Natl Acad Sci USA, 1993, 90(12): 5618-5622. DOI:10.1073/pnas.90.12.5618 |
| [12] |
Mao CJ, Xie HJ, Chen SG, et al. Error-prone PCR mutation of Ls-EPSPS gene from Liriope spicata conferring to its enhanced glyphosate-resistance. Pestic Biochem Physiol, 2017, 141: 90-95. DOI:10.1016/j.pestbp.2016.12.004 |
| [13] |
Shao WL, Ma KS, Le YL, et al. Development and use of a novel random mutagenesis method: in situ error-prone PCR (is-epPCR)//Reeves A, Ed. In vitro Mutagenesis. New York, NY: Humana Press, 2017: 497-506.
|
| [14] |
Yuan L, Kurek I, English J, et al. Laboratory-directed protein evolution. Microbiol Mol Biol Rev, 2005, 69(3): 373-392. DOI:10.1128/MMBR.69.3.373-392.2005 |
| [15] |
Porter JL, Rusli RA, Ollis DL. Directed evolution of enzymes for industrial biocatalysis. ChemBioChem, 2016, 17(3): 197-203. DOI:10.1002/cbic.201500280 |
| [16] |
Patel PH, Loeb LA. DNA polymerase active site is highly mutable: evolutionary consequences. Proc Natl Acad Sci USA, 2000, 97(10): 5095-5100. DOI:10.1073/pnas.97.10.5095 |
| [17] |
Chen KQ, Arnold FH. Enzyme engineering for nonaqueous solvents: random mutagenesis to enhance activity of subtilisin E in polar organic media. Bio/Technology, 1991, 9(11): 1073-1077. DOI:10.1038/nbt1191-1073 |
| [18] |
Ouyang FJ, Zhao M. Enhanced catalytic efficiency of CotA-laccase by DNA shuffling. Bioengineered, 2019, 10(1): 182-189. DOI:10.1080/21655979.2019.1621134 |
| [19] |
Herr AJ, Ogawa M, Lawrence NA, et al. Mutator suppression and escape from replication error-induced extinction in yeast. PLoS Genet, 2011, 7(10): e1002282. DOI:10.1371/journal.pgen.1002282 |
| [20] |
Gunge N, Sakaguchi K. Intergeneric transfer of deoxyribonucleic acid killer plasmids, pGKl1 and pGKl2, from Kluyveromyces lactis into Saccharomyces cerevisiae by cell fusion. J Bacteriol, 1981, 147(1): 155-160. DOI:10.1128/JB.147.1.155-160.1981 |
| [21] |
Ravikumar A, Arrieta A, Liu CC. An orthogonal DNA replication system in yeast. Nat Chem Biol, 2014, 10(3): 175-177. DOI:10.1038/nchembio.1439 |
| [22] |
Ravikumar A, Arzumanyan GA, Obadi MKA, et al. Scalable, continuous evolution of genes at mutation rates above genomic error thresholds. Cell, 2018, 175(7): 1946-1957. DOI:10.1016/j.cell.2018.10.021 |
| [23] |
Zhong ZW, Ravikumar A, Liu CC. Tunable expression systems for orthogonal DNA replication. ACS Synth Biol, 2018, 7(12): 2930-2934. DOI:10.1021/acssynbio.8b00400 |
| [24] |
Arzumanyan GA, Gabriel KN, Ravikumar A, et al. Mutually orthogonal DNA replication systems in vivo. ACS Synth Biol, 2018, 7(7): 1722-1729. DOI:10.1021/acssynbio.8b00195 |
| [25] |
Javanpour AA, Liu CC. Genetic compatibility and extensibility of orthogonal replication. ACS Synth Biol, 2019, 8(6): 1249-1256. DOI:10.1021/acssynbio.9b00122 |
| [26] |
Sternberg SH, Redding S, Jinek M, et al. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature, 2014, 507(7490): 62-67. DOI:10.1038/nature13011 |
| [27] |
Ma XL, Zhang QY, Zhu QL, et al. A robust CRISPR/Cas9 system for convenient, high-efficiency multiplex genome editing in monocot and dicot plants. Mol Plant, 2015, 8(8): 1274-1284. DOI:10.1016/j.molp.2015.04.007 |
| [28] |
Gaj T, Gersbach CA, Barbas CR. ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering. Trends Biotechnol, 2013, 31(7): 397-405. DOI:10.1016/j.tibtech.2013.04.004 |
| [29] |
Zhang YL, Ma XL, Xie XR, et al. CRISPR/Cas9-based genome editing in plants. Prog Mol Biol Transl Sci, 2017, 149: 133-150. |
| [30] |
Feng ZY, Zhang BT, Ding WN, et al. Efficient genome editing in plants using a CRISPR/Cas system. Cell Res, 2013, 23(10): 1229-1232. DOI:10.1038/cr.2013.114 |
| [31] |
Lu YM, Zhu JK. Precise editing of a target base in the rice genome using a modified CRISPR/Cas9 system. Mol Plant, 2017, 10(3): 523-525. DOI:10.1016/j.molp.2016.11.013 |
| [32] |
Butt H, Eid A, Momin AA, et al. CRISPR directed evolution of the spliceosome for resistance to splicing inhibitors. Genome Biol, 2019, 20: 73. DOI:10.1186/s13059-019-1680-9 |
| [33] |
Halperin SO, Tou CJ, Wong EB, et al. CRISPR-guided DNA polymerases enable diversification of all nucleotides in a tunable window. Nature, 2018, 560(7717): 248-252. DOI:10.1038/s41586-018-0384-8 |
| [34] |
Hess GT, Frésard L, Han K, et al. Directed evolution using dCas9-targeted somatic hypermutation in mammalian cells. Nat Methods, 2016, 13(12): 1036-1042. DOI:10.1038/nmeth.4038 |
| [35] |
Jakočiūnas T, Pedersen LE, Lis AV, et al. CasPER, a method for directed evolution in genomic contexts using mutagenesis and CRISPR/Cas9. Metab Eng, 2018, 48: 288-296. DOI:10.1016/j.ymben.2018.07.001 |
| [36] |
Esvelt KM, Carlson JC, Liu DR. A system for the continuous directed evolution of biomolecules. Nature, 2011, 472(7344): 499-503. DOI:10.1038/nature09929 |
| [37] |
Ma YQ, Zhang JY, Yin WJ, et al. Targeted AID-mediated mutagenesis (TAM) enables efficient genomic diversification in mammalian cells. Nat Methods, 2016, 13(12): 1029-1035. DOI:10.1038/nmeth.4027 |
| [38] |
Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337(6096): 816-821. DOI:10.1126/science.1225829 |
| [39] |
Camps M, Naukkarinen J, Johnson BP, et al. Targeted gene evolution in Escherichia coli using a highly error-prone DNA polymerase Ⅰ. Proc Natl Acad Sci USA, 2003, 100(17): 9727-9732. DOI:10.1073/pnas.1333928100 |
| [40] |
Slaymaker IM, Gao LY, Zetsche B, et al. Rationally engineered Cas9 nucleases with improved specificity. Science, 2016, 351(6268): 84-88. DOI:10.1126/science.aad5227 |
| [41] |
Wang Y, Prosen DE, Mei L, et al. A novel strategy to engineer DNA polymerases for enhanced processivity and improved performance in vitro. Nucleic Acids Res, 2004, 32(3): 1197-1207. DOI:10.1093/nar/gkh271 |
| [42] |
Hu JH, Miller SM, Geurts MH, et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature, 2018, 556(7699): 57-63. DOI:10.1038/nature26155 |
| [43] |
Anders C, Niewoehner O, Duerst A, et al. Structural basis of PAM-dependent target DNA recognition by the Cas9 endonuclease. Nature, 2014, 513(7519): 569-573. DOI:10.1038/nature13579 |
| [44] |
Nishimasu H, Shi X, Ishiguro S, et al. Engineered CRISPR-Cas9 nuclease with expanded targeting space. Science, 2018, 361(6408): 1259-1262. DOI:10.1126/science.aas9129 |
| [45] |
Korkmaz G, Lopes R, Ugalde AP, et al. Functional genetic screens for enhancer elements in the human genome using CRISPR-Cas9. Nat Biotechnol, 2016, 34(2): 192-198. DOI:10.1038/nbt.3450 |
| [46] |
Ma YW, Yu L, Zhang X, et al. Highly efficient and precise base editing by engineered dCas9-guide tRNA adenosine deaminase in rats. Cell Discov, 2018, 4: 39. |
| [47] |
Orr-Weaver TL, Szostak JW, Rothstein RJ. Yeast transformation: a model system for the study of recombination. Proc Natl Acad Sci USA, 78(10): 6354-6358. DOI:10.1073/pnas.78.10.6354 |
| [48] |
Szostak JW, Orr-Weaver TL, Rothstein RJ, et al. The double-strand-break repair model for recombination. Cell, 1983, 33(1): 25-35. DOI:10.1016/0092-8674(83)90331-8 |
| [49] |
Guo XG, Chavez A, Tung A, et al. High-throughput creation and functional profiling of DNA sequence variant libraries using CRISPR-Cas9 in yeast. Nat Biotechnol, 2018, 36(6): 540-546. DOI:10.1038/nbt.4147 |
| [50] |
Barbieri EM, Muir P, Akhuetie-Oni BO, et al. Precise editing at DNA replication forks enables multiplex genome engineering in eukaryotes. Cell, 2017, 171(6): 1453-1467. DOI:10.1016/j.cell.2017.10.034 |
| [51] |
Nyerges Á, Csörgő B, Draskovits G, et al. Directed evolution of multiple genomic loci allows the prediction of antibiotic resistance. Proc Natl Acad Sci USA, 2018, 115(25): E5726-E5735. DOI:10.1073/pnas.1801646115 |
| [52] |
Wong TS, Zhurina D, Schwaneberg U. The diversity challenge in directed protein evolution. Comb Chem High Throughput Screen, 2006, 9(4): 271-288. DOI:10.2174/138620706776843192 |
| [53] |
Qu G, Zhao J, Zheng P, et al. Recent advances in directed evolution. Chin J Biotech, 2018, 34(1): 1-11 (in Chinese). 曲戈, 赵晶, 郑平, 等. 定向进化技术的最新进展. 生物工程学报, 2018, 34(1): 1-11. |
| [54] |
Qu G, Zhang K, Jiang YY, et al. The Nobel Prize in Chemistry 2018: the directed evolution of enzymes and the phage display technologies. J Biol, 2019, 36(1): 1-6, 19 (in Chinese). 曲戈, 张锟, 蒋迎迎, 等. 2018诺贝尔化学奖:酶定向进化与噬菌体展示技术. 生物学杂志, 2019, 36(1): 1-6, 19. |
| [55] |
Lutz S. Beyond directed evolution-semi-rational protein engineering and design. Curr Opin Biotechnol, 2010, 21(6): 734-743. DOI:10.1016/j.copbio.2010.08.011 |
| [56] |
Cheng F, Zhu LL, Schwaneberg U. Directed evolution 2.0: improving and deciphering enzyme properties. Chem Commun (Camb), 2015, 51(48): 9760-9772. DOI:10.1039/C5CC01594D |
| [57] |
O'Maille PE, Bakhtina M, Tsai MD. Structure-based combinatorial protein engineering (SCOPE). J Mol Biol, 2002, 321(4): 677-691. DOI:10.1016/S0022-2836(02)00675-7 |
| [58] |
Widersten M, Hansson LO, Tronstad L, et al. Use of phage display and transition-state analogs to select enzyme variants with altered catalytic properties: glutathione transferase as an example. Methods Enzymol, 2000, 328: 389-404. |
| [59] |
Qu G, Zhu T, Jiang YY, et al. Protein engineering: from directed evolution to computational design. Chin J Biotech, 2019, 35(10): 1843-1856 (in Chinese). 曲戈, 朱彤, 蒋迎迎, 等. 蛋白质工程:从定向进化到计算设计. 生物工程学报, 2019, 35(10): 1843-1856. |
| [60] |
Lassila JK. Conformational diversity and computational enzyme design. Curr Opin Chem Biol, 2010, 14(5): 676-682. DOI:10.1016/j.cbpa.2010.08.010 |
| [61] |
Qu G, Li AT, Acevedo-Rocha CG, et al. The crucial role of methodology development in directed evolution of selective enzymes. Angew Chem Int Ed Engl, 2020, 59. DOI:10.1002/anie.201901491 |
| [62] |
Fox RJ, Davis SC, Mundorff EC, et al. Improving catalytic function by ProSAR-driven enzyme evolution. Nat Biotechnol, 2007, 25(3): 338-344. DOI:10.1038/nbt1286 |
| [63] |
Gonzalez-Perez D, Molina-Espeja P, Garcia-Ruiz E, et al. Mutagenic organized recombination process by homologous in vivo grouping (MORPHING) for directed enzyme evolution. PLoS ONE, 2014, 9(3): e90919. DOI:10.1371/journal.pone.0090919 |
| [64] |
Chen MMY, Snow CD, Vizcarra CL, et al. Comparison of random mutagenesis and semi-rational designed libraries for improved cytochrome P450 BM3-catalyzed hydroxylation of small alkanes. Protein Eng Des Sel, 2012, 25(4): 171-178. DOI:10.1093/protein/gzs004 |
| [65] |
Treynor TP, Vizcarra CL, Nedelcu D, et al. Computationally designed libraries of fluorescent proteins evaluated by preservation and diversity of function. Proc Natl Acad Sci USA, 2007, 104(1): 48-53. DOI:10.1073/pnas.0609647103 |
| [66] |
Jamil S, Liu MH, Liu YM, et al. Hydrophobic mutagenesis and semi-rational engineering of arginine deiminase for markedly enhanced stability and catalytic efficiency. Appl Biochem Biotechnol, 2015, 176(5): 1335-1350. DOI:10.1007/s12010-015-1649-4 |
| [67] |
Shin KC, Choi HY, Seo MJ, et al. Improved conversion of ginsenoside Rb1 to compound K by semi-rational design of Sulfolobus solfataricus β-glycosidase. AMB Express, 2017, 7: 186. DOI:10.1186/s13568-017-0487-x |
| [68] |
Smith GP. Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface. Science, 1985, 228(4705): 1315-1317. DOI:10.1126/science.4001944 |
| [69] |
Bradbury A. Recent advances in phage display: the report of the Phage Club first meeting. Immunotechnology, 1997, 3(3): 227-231. DOI:10.1016/S1380-2933(97)00017-1 |
| [70] |
Siegel DL. Research and clinical applications of antibody phage display in transfusion medicine. Transfus Med Rev, 2001, 15(1): 35-52. DOI:10.1053/tmrv.2001.19950 |
| [71] |
McCafferty J, Griffiths AD, Winter G, et al. Phage antibodies: filamentous phage displaying antibody variable domains. Nature, 1990, 348(6301): 552-554. DOI:10.1038/348552a0 |
| [72] |
Smothers JF, Henikoff S, Carter P. Tech. Sight. Phage display. Affinity selection from biological libraries. Science, 2002, 298(5593): 621-622. DOI:10.1126/science.298.5593.621 |
| [73] |
Azzazy HME, Highsmith WE Jr. Phage display technology: clinical applications and recent innovations. Clin Biochem, 2002, 35(6): 425-445. DOI:10.1016/S0009-9120(02)00343-0 |
| [74] |
Frenzel A, Schirrmann T, Hust M. Phage display-derived human antibodies in clinical development and therapy. mAbs, 2016, 8(7): 1177-1194. DOI:10.1080/19420862.2016.1212149 |
| [75] |
Frei JC, Lai JR. Protein and antibody engineering by phage display. Methods Enzymol, 2016, 580: 45-87. |
| [76] |
Cung K, Slater RL, Cui Y, et al. Rapid, multiplexed microfluidic phage display. Lab Chip, 2012, 12(3): 562-565. DOI:10.1039/C2LC21129G |
| [77] |
Wang TN, Badran AH, Huang TP, et al. Continuous directed evolution of proteins with improved soluble expression. Nat Chem Biol, 2018, 14(10): 972-980. DOI:10.1038/s41589-018-0121-5 |
| [78] |
Thuronyi BW, Koblan LW, Levy JM, et al. Continuous evolution of base editors with expanded target compatibility and improved activity. Nat Biotechnol, 2019, 37(9): 1070-1079. DOI:10.1038/s41587-019-0193-0 |
| [79] |
Rees HA, Liu DR. Base editing: precision chemistry on the genome and transcriptome of living cells. Nat Rev Genet, 2018, 19(12): 770-788. DOI:10.1038/s41576-018-0059-1 |
| [80] |
Yang GY, Withers SG. Ultrahigh-throughput FACS-based screening for directed enzyme evolution. Chem Bio Chem, 2009, 10(17): 2704-2715. DOI:10.1002/cbic.200900384 |
| [81] |
Tan YM, Zhang Y, Han YB, et al. Directed evolution of an α1, 3-fucosyltransferase using a single-cell ultrahigh-throughput screening method. Sci Adv, 2019, 5(10): w8451. DOI:10.1126/sciadv.aaw8451 |
| [82] |
Baret JC, Miller OJ, Taly V, et al. Fluorescence-activated droplet sorting (FADS): efficient microfluidic cell sorting based on enzymatic activity. Lab Chip, 2009, 9(13): 1850-1858. DOI:10.1039/b902504a |
| [83] |
Agresti JJ, Antipov E, Abate AR, et al. Ultrahigh-throughput screening in drop-based microfluidics for directed evolution. Proc Natl Acad Sci USA, 2010, 107(9): 4004-4009. DOI:10.1073/pnas.0910781107 |
| [84] |
Liebeton K, Zonta A, Schimossek K, et al. Directed evolution of an enantioselective lipase. Chem Biol, 2000, 7(9): 709-718. DOI:10.1016/S1074-5521(00)00015-6 |
| [85] |
Ma FQ, Chung MT, Yao YB, et al. Efficient molecular evolution to generate enantioselective enzymes using a dual-channel microfluidic droplet screening platform. Nat Commun, 2018, 9: 1030. DOI:10.1038/s41467-018-03492-6 |
| [86] |
Ma FQ, Fischer M, Han Y, et al. Substrate engineering enabling fluorescence droplet entrapment for IVC-FACS-based Ultrahigh-throughput screening. Anal Chem, 2016, 88(17): 8587-8595. DOI:10.1021/acs.analchem.6b01712 |
 2021, Vol. 37
2021, Vol. 37