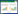中国科学院微生物研究所、中国微生物学会主办
文章信息
- 田苗, 张俊, 罗文, 王治元, 付俊鹰, 黄少伟, 吕鹏梅
- Tian Miao, Zhang Jun, Luo Wen, Wang Zhiyuan, Fu Junying, Huang Shaowei, Lü Pengmei
- 前导肽介导的蛋白折叠机制及其对脂肪酶影响的研究进展
- Propeptide-mediated protein folding: mechanism and its impact on lipase
- 生物工程学报, 2021, 37(1): 88-99
- Chinese Journal of Biotechnology, 2021, 37(1): 88-99
- 10.13345/j.cjb.200206
-
文章历史
- Received: April 14, 2020
- Accepted: September 23, 2020
2. 中国科学院大学, 北京 100049;
3. 华南农业大学 林学与风景园林学院, 广东 广州 510642
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. College of Forestry and Landscape Architecture, South China Agricultural University, Guangzhou 510642, Guangdong, China
原核生物和真核生物的分泌蛋白通常具有前体形式, 在一个或多个水解酶的作用下被加工为成熟蛋白。1973年, 关于核糖核酸酶的开创性著作中, Anfinsen提出每一种蛋白质都有特定的氨基酸组成和排列顺序, 这些氨基酸的排列顺序决定了蛋白质的空间结构, 也就是说, 蛋白质的空间结构由其氨基酸的一级结构决定, 不需要额外的因素[1]。然而, 随着蛋白质折叠研究的发展, 开始出现了与自然折叠模型相冲突的数据。1987年, 枯草杆菌蛋白酶E (Subtilisin E)被首次证明蛋白质不能自发地形成活性构象[2]。随后, 在纳豆激酶[3]、羧肽酶Y (Carboxypeptidase Y)[4]及来自于米黑根毛霉Rhizomucor miehei[5]和米根霉Rhizopus oryzae[6]等的脂肪酶折叠研究中也发现了这一现象, 这证明脂肪酶也不能自发折叠形成活性构象, 它需要前导肽的协助才能折叠成具有生物活性的酶。由于前导肽的功能类似于一个大的热休克蛋白家族, 所以它们被广泛地归类为分子伴侣, 但是前导肽和分子伴侣之间存在着显著的差异, 因此前导肽被称为分子内分子伴侣[7]。分子伴侣在某些情况下可以帮助蛋白质组装成寡聚体结构, 但它们不是这些结构的最终组成部分, 它的折叠符合Anfinsen规则。然而以含前导肽(Propeptide)的前体形式合成的蛋白酶, 它的正确折叠与成熟必须有前导肽的存在才能完成。在缺乏前导肽的情况下, 蛋白质不能自发形成具有活性的酶, 其折叠不完全遵守Anfinsen规则[3]。
脂肪酶, 又称三酯酰甘油酰基水解酶(EC3.1.1.3), 属于α/β水解酶超家族中的一员。脂肪酶的催化中心由Ser-His-Asp或Ser-His-Glu组成, 且隐藏在结构内部。由于对硝基苯磷酸二乙酯对脂肪酶有抑制作用, 因此脂肪酶属于丝氨酸水解酶[8]。脂肪酶能在油水界面水解三酰基甘油生成脂肪酸、甘油和单、二酰基甘油, 并能逆转非水介质中的反应, 是生物体进行脂质代谢必不可少的水解酶, 也是一种在非水溶剂中进行脂质可逆反应的酶[9], 在食品加工、污水处理、洗涤剂和新能源等领域具有很大的应用空间[10-15]。脂肪酶与许多其他蛋白酶类似, 在折叠过程中包含一个特别的区域, 该区域随后会自动或被外部蛋白酶裂解, 这个区域称为前导肽。近年来, 前导肽的研究已成为脂肪酶研究中一个重要方向, 主要集中在前导肽对脂肪酶分泌表达、活性、稳定性及折叠方面的影响。米黑根毛霉脂肪酶是第一个结构被解析的脂肪酶, 但其最终的结构只有成熟肽[16]。Boel等[5]发现, 来源于米黑根毛霉的脂肪酶(Rhizomucor miehei Lipase, RML)会先合成为一个前体酶, 这个前体酶包含由70个氨基酸组成的前导肽, 在产生活性的RML后, 它在信号肽的帮助下穿过胞质膜, 最后前导肽会被降解除去。目前已有研究报道了有(PDB:6QPR)和没有(PDB:3TGL)前导肽的RML晶体结构, 因为RML的最终表达产物只有成熟肽, 所以有前导肽的晶体结构是通过同源建模构建的(图 1)。王珏等[17]通过克隆带有前导肽和不带前导肽的RML, 发现没有前导肽的RML在大肠杆菌中不表达。Satomura等[18]对米根霉脂肪酶(Rhizopus oryzae lipase, ROL)的前导肽进行突变, 也发现ROL前导肽的突变可以动态地诱导酶活性的变化。Samuelson等[19]研究猪葡萄球菌脂肪酶(Staphylococcus hyicus lipase, SHL)在肉毒杆菌的表面展示, 发现前导肽对肉毒杆菌中猪葡萄球菌脂肪酶的转位有积极的影响。笔者课题组[20]先前对RML的前导肽进行易错PCR和组合诱变, 获得了7个活性和稳定性得到改善的前导肽突变酶, 发现上述现象的产生可能与蛋白质折叠过程中保守区域的突变以及前导肽与功能区域的相互作用有关。这些研究都证明了前导肽影响脂肪酶的折叠、表达、产生功能性等整个过程, 对前导肽上的氨基酸进行基因工程操作, 会改变脂肪酶的结构与功能。迄今为止, 已知的真菌脂肪酶至少有33种, 而我们的研究对象主要集中于嗜热单胞菌脂肪酶(Thermomyces lanuginosus lipase, TLL)、华根霉脂肪酶(Rhizopus chinonsis lipase, RCL)、RML、ROL、SHL等这几种脂肪酶。此外, 脂肪酶前导肽的研究处于起步阶段, 研究主要集中在前导肽对脂肪酶性质、折叠与分泌表达的影响, 关于机理方面的研究较少, 尤其是前导肽介导脂肪酶折叠的机制。

|
| 图 1 RML带有和不带有前导肽的三维模型图(A:不带有前导肽的RML (PDB:3TGL);B:带有前导肽的RML (PDB:6QPR)) Fig. 1 Three-dimensional model diagrams of RML with and without propeptide. (A) Without propeptide (PDB:3TGL). (B) With propeptide (PDB:6QPR). |
| |
大多数分泌的水解酶都是由前体酶合成的, 其中前体酶主要由信号肽、前导肽和成熟肽构成。信号肽是胞外蛋白表达必不可少的结构, 它的作用是与异源系统产生的蛋白结合, 引导其从胞内分泌到胞外, 一般位于初生蛋白的N端, 也有少部分位于蛋白内部或C端[21]。成熟肽的作用是执行蛋白生物学功能。前导肽的作用是辅助蛋白酶折叠, 避免蛋白酶错误折叠或形成聚集体。此外, 它还可以在蛋白酶从细胞释放之前使蛋白酶保持在不活跃状态, 或者将蛋白酶固定在细胞膜上[22-23]。前导肽在长度和位置上存在广泛的差异:一些位于成熟区域的N端, 如枯草杆菌蛋白酶E[24];一些位于成熟区域的C端, 如IgA蛋白酶[25];还有一些在成熟区域的N端和C端都有, 如丝氨酸蛋白酶[26]。前导肽在协助蛋白质折叠完成后, 一般自身会被降解, 只有少部分脂肪酶的前导肽不会被完全降解。虽然分泌后的成熟蛋白酶没有前导肽, 但是前导肽在成熟蛋白酶合成过程中已经调节了它的折叠、结构和功能[27]。前导肽结构的改变, 可能极大地改变生物体的蛋白质功能, 而且它可以在不改变高度保守的催化结构域的情况下改变蛋白质的生物学特性[28-29]。由于前导肽的性质并非水解酶所独有, 因此前导肽介导的进化似乎是一种普遍的生物学机制。
1.1 辅助折叠机制前导肽对蛋白质的折叠机制最早是在枯草杆菌蛋白酶E中发现的。枯草杆菌蛋白酶E是枯草芽孢杆菌Bacillus subtilis 168的碱性丝氨酸蛋白水解酶, 最早作为前体产生。对于枯草杆菌蛋白酶E, 它的前体酶有折叠能力, 而稳定的成熟酶则没有, 这说明折叠途径不是稳定的自然状态的必然结果, 而是对蛋白质进化的额外约束。Baker等[30]通过α-lytic蛋白酶的体外复性研究实验发现, 在天然状态同完全变性状态之间, 存在一种中间态。通过实验发现, 这种中间态类似熔球状, 它的二级结构与天然态的二级结构很相似, 可是不存在三级结构。这种中间态的出现, 使得它的构象更加稳定, 具有更低的能量。从变性状态的复性过程中, 需要克服一个具有很高活化能(30 kcal/mol)的过渡状态。没有前导肽时需要克服这个折叠屏障, 折叠速率非常缓慢(t1/2约2 000年)。前导肽的加入降低了折叠反应的活化能, 使得折叠过程变得容易进行, 加速了成熟肽的折叠, 折叠速率常数为3×109。此外, 天然性质的展开速度似乎也很慢(t1/2约1年), 这表明一旦正确折叠并不能有效地展开。前导肽使成熟结构域稳定, 实际上是成熟酶的有效抑制剂(Ki约为10–10 mol/L)。
脂肪酶是水解甘油三酸酯酯键的α/β水解酶超家族的成员, 活性位点由经典催化三联体Ser-His-Asp或者Ser-His-Glu组成, 与丝氨酸蛋白酶相反, 其活性位点埋在结构内部。丝氨酸(Ser)是一种亲核氨基酸, 主要参与水解的过程[31]。脂肪酶的活性中心是一个类似“盖子”的结构, 在其内表面, 对应催化中心的疏水区域, 表现为疏水性;在其外表面, 表现为亲水性。由于盖子结构的这种双亲性, 当脂肪酶处于油水界面时, 脂肪酶与油水界面的缔合, 盖子结构被打开, 疏水的催化中心被暴露, 此时疏水的底物进入与活性中心接触, 发生催化反应, 这就是脂肪酶的界面激活, 此时脂肪酶处于“盖子”开放的活性形式。“盖子”结构能加强对脂肪酶自身的保护作用, 保护脂肪酶的催化中心, 保证脂肪酶的稳定, 当其闭合时, 脂肪酶处于阻止底物进入活性中心的闭合状态[32]。“盖子”结构的形成需要前导肽的帮助, Yan等[33]发现, 假单胞菌Pseudomonas protegens Pf-5中脂肪酶A的N端肽段是前导肽, 位于脂肪酶A的结构内部, 协助活性中心的“盖子” (由α螺旋构成)由“关闭状态”转变为“打开状态”, 这是第一个在脂肪酶亚家族Ⅰ.1中发现的由前导肽协助折叠的实例。前导肽介导脂肪酶的折叠, 首先需要与成熟肽作用, 形成亚稳态的中间复合体, 它会保护蛋白不被降解, 不会对细胞产生毒性, 但此时这个中间复合体形成的酶原没有活性, 前导肽能使成熟结构域稳定, 实际上是成熟酶的有效抑制剂。只有当前导肽被降解后, 成熟酶才会有活性[27, 34]。前导肽被降解的方式至少有3种:通过蛋白水解切割去除前肽、自动催化去除前导肽、结合前导肽的自动加工以及随后由另一种蛋白酶介导的切割[32]。前导肽的功能与其疏水性有关, 它通过和前体蛋白的某些区域相互作用, 有助于疏水蛋白核心的形成, 从而促进折叠, 进而提高表达和功能。Feng等[34]构建了嗜热单胞菌脂肪酶(TLL)模型, 发现TLL前导肽疏水性的增加是脂肪酶TLL表达增强的主要原因, 通过作为疏水支架辅助蛋白质成核, 从而加速蛋白折叠的形成。其中, TLL前导肽上“SPIRR”五肽的结构可以增强其表达, 并辅助成熟脂肪酶的结构折叠。Hans等[35]提出了一种模型证明了前导肽对米根霉脂肪酶折叠及活性的影响。米根霉脂肪酶会先合成一个具有N端前导肽和成熟肽的前体, 随后N端前导肽被裂解产生一个具有活性的成熟酶。其中, 前导肽主要有两个功能:第一是调节脂肪酶的酶活性, 使这种酶能够以非破坏性的形式合成, 因为在细胞中直接合成成熟形式的脂肪酶有毒性的后果, 至少部分是由于磷脂酶的活性抑制了原蛋白。第二是它支持脂肪酶的折叠, 且其途径受68位半胱氨酸残基的影响, 其中Cys68充当分子内硫代二硫化物试剂, 在酶的折叠中起催化作用。正常情况下, 米根霉菌脂肪酶ROL在培养基中的表达产物是带有前导肽的, 即ROL32, 但将其在0 ℃和6 ℃下放置几个月或几天, 发现其前导肽自动降解, 得到去除N端28个氨基酸的成熟肽ROL29。通过对两种ROL进行分析, 发现ROL的前导肽可以作为分子内分子伴侣在不与成熟脂肪酶共价结合的情况下, 或在其他重组宿主如酿酒酵母中, 帮助蛋白质折叠[36]。我们的研究也发现这一现象, 即在培养基中的表达产物是带有前导肽的RML, 将其在4 ℃冰箱放置5个月后, 前导肽自动降解, 得到只有成熟肽的RML (数据未发表)。Ueda等[37]发现米根霉菌脂肪酶的前导肽可以生成α-螺旋的二级结构, 它可能以部分成熟酶作为相对保守的结构域形成亚稳态结构, 产生保守性表面。在前导肽与成熟区域相互作用时, 这种二级结构形成一种支架, 有利于降低活化ROL所需的能量, 克服蛋白质折叠途径中的能量障碍, 促进对ROL的正确折叠。前导肽除了介导自身的折叠外, 研究发现葡萄球菌脂肪酶的前导肽不仅可以辅助其他一些脂肪酶甚至是其他种类的酶的折叠, 如人β-1, 4半乳糖基转移酶1[38]。
以上研究表明, 前导肽介导脂肪酶的折叠机制可能与其他丝氨酸蛋白水解酶类似。前导肽辅助折叠形成成熟酶的过程主要包括4个步骤:(1)前导肽介导的酶同步折叠;(2)共价结合前导肽的裂解, 形成非共价结合的前导肽-成熟蛋白复合物;(3)前导肽的降解, 使酶形成稳定构象;(4)酶的激活。前导肽通过作为水解酶结构域的模板来促进折叠, 但它并不构成该结构域的一部分。
1.2 二硫键作用在蛋白质折叠研究中, 我们发现某些蛋白质折叠伴随二硫键的形成, 其中最具特征的是牛胰蛋白酶抑制剂(Bovine pancreatic trypsin inhibitor, BPTI)的体外折叠。Weissman和Kim[39]在牛胰蛋白酶抑制剂的合成过程中发现, 3个二硫化物桥的形成是这种小分子蛋白质折叠途径的限制因素。前导肽中的半胱氨酸残基, 通过柔性连接物连接到BPTI的C末端的单个半胱氨酸残基, 协助二硫化物的形成, 促进蛋白折叠。这些半胱氨酸残基是通过向复性体系中加入低分子量硫醇及氧化态的方法获得, 常用试剂有谷胱甘肽还原对、半胱氨酸等。然而, Creighton等[40]解释说, 在生理条件下, 由前导肽导致的重排事件率小幅增加并不显著。已有研究发现, 二硫键在前导肽介导脂肪酶的折叠中发挥着重要的作用。Hans等[35]将米根霉脂肪酶(Rhizopus oryzae lipase, ROL)前导肽上第68位的半胱氨酸(Cys)突变为丝氨酸(Ser), 邻近的69位精氨酸(Arg)残基突变为丙氨酸(Ala), 在该模型中, 通过对序列分析发现, 第69位的Arg增强了第68位Cys的离去基特征, 这可能是因为ProROL前导肽中的半胱氨酸残基是作为一种分子内的硫代二硫化物试剂在酶的催化中起催化作用, 这不仅证明了半胱氨酸残基的关键作用, 而且也证明了邻近氨基酸的重要性。然而, 与BPTI的模型相比, Hans等认为Cys68在ProROL中的关键功能在很大程度上取决于其多肽链的直接环境;如果没有适当的环境, 这些残留物将无法在折叠过程中发挥催化作用;这可能意味着蛋白质结构可以通过前导肽中半胱氨酸残基的环境对折叠过程进行相当微妙的控制。此外, 以上的事实也证明了天然构象并不能到达能量的最低状态, 只是在前导肽的帮助下处于动力学上不可逆的能量状态中[30]。
1.3 加速折叠机理一个有效的折叠路径意味着生产的中间产物比周围展开和错误折叠的构象要稳定得多。Philip等[41]通过对前导肽-枯草杆菌蛋白酶E折叠复合体的2.0 Å分辨率结构和催化折叠动力学的分析, 提出了前导肽加速折叠机理:前导肽通过稳定枯草杆菌蛋白酶E中αβα的中心亚结构来促进折叠。前导肽本身基本上是没有结构的, 但当前导肽结合在枯草杆菌蛋白酶E的两个平行表面α-螺旋上时, 会向两个螺旋的N末端提供帽状结构, 形成前导肽-枯草杆菌蛋白酶复合物这一中间体。相对于其他未展开的状态, 前导肽可以稳定这一中间体。在缺乏前导肽的情况下, αβα子结构没有足够的独立稳定性来非常频繁地引发折叠。在TLL的前导肽研究中, 我们发现前导肽的疏水性有助于疏水核心的形成, 这个疏水核心能加速蛋白的折叠[34]。前导肽除了能辅助蛋白质的折叠外, 还能加快蛋白质的折叠, 这为我们接下来的研究提供了一种思路, 我们可以在脂肪酶等其他蛋白酶的研究中, 通过替换前导肽中的关键氨基酸为疏水性氨基酸, 从而构建能够加速折叠的中间体来促进折叠。
2 前导肽对脂肪酶的影响脂肪酶的全基因序列一般由信号肽、前导肽和成熟肽序列组成。通过枯草杆菌蛋白酶前导肽的研究发现, 前导肽可以作为一个模板, 向蛋白酶结构域传递结构信息[42-43]。即使前导肽的突变不是最终形成的蛋白酶域的一部分, 但是前导肽中的点突变依然可以改变蛋白酶的结构和活性。脂肪酶前导肽首次出现在来源于真菌的米黑根毛霉脂肪酶中, 在大肠杆菌中构建的互补DNA文库分析显示, 最初的基因产物是由24个氨基酸残基信号肽、70个残基N端前导肽和269个残基成熟酶对应的结构域组成[5]。研究报道发现, 脂肪酶中的前导肽可以作为分泌信号, 有助于翻译后修饰酶的识别;辅助成熟肽折叠, 保护成熟肽不被降解;中和成熟肽的毒性以及影响脂肪酶的稳定性和底物特异性等酶学性质[37, 44]。其中前导肽作为翻译后修饰酶的识别基序, 不仅有助于合成结构独特的活性产物, 而且可在此基础上进行人工改造, 使获得的产物性能更优。综述脂肪酶前导肽的研究, 发现前导肽对脂肪酶的影响主要集中在空间结构、分泌表达、生化性质和对成熟区域的抑制作用等方面。
2.1 空间结构前导肽的主要功能之一是引导蛋白酶结构域的适当折叠。折叠完成后, 前导肽区域自动嵌入, 产生活性蛋白酶。当前导肽与成熟结构域分离时, 它就没有二级和三级结构[32]。Ueda等[6]利用对前导肽突变体的构建, 产生了一种新型的米根霉脂肪酶(Rhizopus oryzae lipase, ROL), 该酶对中长链脂肪酸具有更好的亲和力。与野生型ROL相比, 新型ROL酶蛋白质的结构发生变化, 导致其生物活性及动力学参数发生改变。随后通过对ROL的前导肽氨基酸序列分段研究发现, ROL前导肽由97个氨基酸组成, 其中第28–57个氨基酸功能区域对活性ROL的正确折叠至关重要。这说明, 前导肽上的不同位置是调控脂肪酶性质的“开关”, 也如先前的研究, 在整个蛋白酶全基因序列中, 前导肽处于多肽链的不同位置, 影响蛋白酶的不同功能。
2.2 分泌表达许多研究发现脂肪酶的前导肽影响成熟蛋白的分泌表达。王珏等[17]研究发现, 在大肠杆菌中, 米黑根毛霉脂肪酶的前导肽没有被蛋白酶水解。此外, 如果人工去除前导肽, 则米黑根毛霉脂肪酶不表达。Takahashi等[6, 45]将ROL的成熟区域(mROL)和pre-α-factor信号肽融合, 并没有观察到活性mROL的分泌;然而, 当mROL与前导肽反式作用时, 则观察到活性mROL的分泌;这表明ROL的前导肽有助于纠正mROL折叠及其在酿酒酵母中的分泌, 但是前导肽与成熟区域的物理连接不是先决条件。
2.3 生化特性通过对根霉脂肪酶全基因序列研究发现, 由于前导肽的作用, 带有前导肽的脂肪酶和只有成熟肽的脂肪酶在酶的性能方面存在很大差异。当脂肪酶的成熟肽基因单独在异源系统中表达时, 无表达活性, 而当脂肪酶的前导肽和成熟肽在异源系统中共表达时, 则有表达活性, 这说明前导肽对脂肪酶的活性有很重要的影响。Takahashi等[45]将ROL的前导肽序列分成7段进行研究, 发现前导肽上20–37个氨基酸残基是酶分泌表达的功能区, 且前导肽氨基酸序列的完整性对酶的热稳定性以及最适温度也十分重要。喻晓蔚等[46]对ROL各种不同表达策略和发酵技术进行了优化, 酶活在毕赤酵母中最高可达1 334 U/mL, 但将ROL的前导肽用RC的前导肽代替后, 酶活性可达4 050 U/mL, 是原始酶的11倍。Niu等[47]通过在不同温度下孵育无根根霉脂肪酶(Rhizopus arrhizus lipases, RAL) r28ProRAL和rRAL 30 min, 考察其热稳定性, 发现r28ProRAL的酶活性略高于rRAL。在4 ℃下, 将r28ProRAL和rRAL在不同pH值的指定缓冲液中孵化16 h, 发现在酸性条件下(pH 4.0–7.0), rPro28RAL的稳定性仅略高于rRAL, 但在碱性条件下(pH 7.0–10.0), 稳定性明显降低。两种结构均在pH 7.0时达到最佳稳定性, 这说明前导肽对RAL的稳定性有着重要影响。Yang等对RML的底物特异性分析发现, 前导肽对pRML底物特异性和位置特异性无影响[48]。以上事实证明, 前导肽对脂肪酶的生化性质影响较大, 尤其当对前导肽的保守区域进行突变时, 脂肪酶的生化性质变化更明显。
2.4 成熟区域的抑制作用一些前导肽在细胞运输过程中作为抑制剂, 可以暂时抑制成熟蛋白酶的活性, 使这种酶能够以非破坏性的形式合成。由于一部分磷脂酶的活性被抑制在前体蛋白中, 在细胞中直接合成成熟形式的脂肪酶会产生毒性[35]。Olga等[32]将RML前导肽上第95和96位点删除, 利用X射线晶体衍射分析了野生型和缺失两个残基的ProRmL样品的三维结构, 发现大多数前导肽的结构在前导肽和成熟区域之间有一个小的无序区域。在前导肽-成熟区域复合结构中, 由于前导肽位于活性位点的顶部, 阻止了活性位点的“盖子”打开, 使得成熟区域处于封闭的非活性状态, 而前导肽的其余部分位于酶的表面。证明了前导肽对成熟区域的抑制作用, 也证实了前导肽在抑制脂肪酶表达过程中的关键作用。黄瓜素也是丝氨酸蛋白酶中的一种, 对其前导肽的研究也证实了前导肽对成熟区域有抑制作用[49]。
2.5 前导肽的糖基化及其对脂肪酶的影响糖基化是蛋白质进行翻译后修饰的重要加工过程, 对蛋白质的空间构象有着重要的修饰作用, 这个过程影响蛋白质的折叠、蛋白质与配体间的相互作用以及蛋白质的生物学功能与结构。微生物脂肪酶一般进行N-糖基化修饰, 通过对N-糖基化反应进行调控, 可以修饰酶的催化性能、底物特异性、热稳定性以及调节酶的分泌表达。相比于未经糖基化修饰的蛋白质, 经过不同糖基化修饰后的蛋白质, 其功能和内在性质均发生改变, 这说明糖链对糖蛋白整体的生物学功能至关重要[50-51]。研究表明, 脂肪酶结构中的糖基化位点通常位于前导肽上, 一般有1–4个糖基化位点, 发生糖基化的位置以及糖基化位点的数目都会影响脂肪酶的酶学性质与功能[52]。RML前导肽上的第8和58位是两个糖基化位点, 刘悦等[45, 53]通过基因突变技术构建了3种米黑根毛霉脂肪酶(RML)突变菌株M1 (N8A)、M2 (N58A)、M3 (N8A和N58A), 发现前导肽的糖基化是Kex2蛋白酶去除前导肽所必需的。对比野生型和突变酶M1、M2、M3的酶活, 发现野生型活性最低, M1、M2、M3活性依次提高, 其中M3的活性大约是野生型的2倍, 这也说明前导肽的去糖基化能显著提高脂肪酶的活性。华根霉脂肪酶基因和米根霉脂肪酶基因在毕赤酵母中表达时, 其酶学性质有很大差异, 主要是因为华根霉脂肪酶基因的前导肽序列有3个糖基化位点, 米根霉脂肪酶基因有1个糖基化位点。利用毕赤酵母异源系统表达脂肪酶时, 存在过度糖基化的现象, 这对重组蛋白的合成与分泌、功能以及后续的蛋白质提纯十分不利。糖基化现象对蛋白质的酶学性质和功能都有着重要影响, 鉴于大多数脂肪酶糖基化位点位于其前导序列中, 因此研究前导肽糖基化对脂肪酶性能的影响机制对后续脂肪酶的改善十分重要。
不同蛋白质和前导肽中不同位置会导致前导肽作用有所差异, 但它们有着共同的作用, 即作为分子伴侣参与酶的折叠以及影响分泌后的酶的稳定性、酶活大小和底物特异性等性质。因此, 未来的研究也将主要围绕如何提高其稳定性和酶活, 这也是脂肪酶能否进一步在工业上大规模应用的前提。
3 脂肪酶前导肽的研究方法通过X射线晶体衍射技术, 确定了大多数蛋白酶的前体酶结构, 所以前导肽对蛋白酶的具体作用及作用机制的研究已经很成熟, 然而前导肽在脂肪酶中的确切作用及作用机制还不完全清楚。之前为了阐明米根霉脂肪酶前导肽的功能, 对其进行结构研究, 发现只有成熟酶的结构被报道(PDB ID: 1TIC)[54], 所以无法确定前导肽对它的作用。不久之后, 有研究报道了前导肽突变对米黑根毛霉脂肪酶活性的影响, 以及对来自TLL的另一类脂肪酶的影响, 但是由于很难获得前导肽结构, 前体酶的结构和前导肽的抑制作用机理都未曾报道[34, 49, 55]。近年来, 关于前导肽中间体的研究不胜枚举, 主要有两大类:一类是通过分子生物学技术改造前导肽的核酸序列或氨基酸序列, 从而改善脂肪酶的酶学性质和应用状况, 这是当前主要的研究方向;另一类是利用分子动力学模拟的方法来研究前导肽对脂肪酶的影响。
3.1 酶分子改造通过酶分子改造前导肽主要从核酸和蛋白质两个方向进行。核酸方面主要是通过改变碱基序列从而改变遗传学信息。蛋白质方面主要是通过改变蛋白质一级结构的氨基酸序列从而改变其结构与功能。最常用的酶分子改造方法就是定向进化。虽然前导肽的修饰会影响蛋白质的折叠和功能, 但对蛋白质前导肽的定向进化的研究很少, 大多数都聚焦于前体的功能区域。Jafar等[56]通过SEOing PCR技术, 将前导肽上14、13或12位的氨基酸残基替换为等效的凝血酶原氨基酸, 提高了维生素K依赖的(Vitamin K-dependent, VKD)蛋白的活性和表达量。随后, 王珏等[17]对RML的前导肽进行定向进化, 发现前导肽的关键突变促进了酶活性。除了定向进化, 还可以通过前导肽的替换来研究前导肽的功能。本课题组[57]将华根霉脂肪酶和米根霉脂肪酶的前导肽置换, 在毕赤酵母中成功地表达了这两种具有新前导肽区域的脂肪酶, 发现由于前导肽区域的改变, 相同的多肽可以折叠成不同的构象。定向进化和前导肽区域替换等酶分子改造方法操作简单, 且为蛋白酶中前导肽功能的研究提供了新的思路。在接下来的研究中, 我们可以利用酶分子改造方法研究前导肽和催化区域对酶活性的协同作用, 这可以为今后的工作提供一种定向进化策略的新方法。
3.2 分子动力学模拟对于生物分子系统的模拟, 最常用的技术是分子动力学(Molecular dynamics, MD)模拟, 这是用数值方法逐步迭代地求解经典牛顿运动方程, 通过在由分子体系不同状态构成的系统中抽取样本, 从而计算体系的构型积分, 并以构型积分的结果为基础进一步计算体系的热力学量和其他宏观性质。MD模拟的起点通常是通过实验确定蛋白质结构, 这些结构信息一般来自X射线晶体学或溶液核磁共振。蛋白质结构预测的计算方法, 例如同源性模型, 可以用来为没有确定结构的蛋白质建立构象[58]。MD不论是在时空上还是在几何性、灵活性以及分子间相互作用的结构和构象的预测上都具有很高的精确度。Olga等[32]通过MD模拟了RML成熟酶(mature RML)和前体酶(ProRML)的三维结构模型, 证明了前导肽在标准脂肪酶检测中表现出对脂肪酶活性的抑制;同时他们提出前导肽具有确保酶在原宿主表达途径中不被激活的作用。本课题组[59]先前利用分子动力学模拟及分子对接技术建立了米黑根毛霉脂肪酶的前导肽模型、前导肽与成熟肽复合模型以及突变酶的结构模型, 获得了RML前导肽的关键氨基酸残基位点, 初步明晰了RML前导肽对成熟肽的别构调控机理。酶分子改造方法是当前蛋白酶等领域主要的研究方法, 它操作简单, 但是工作量较大, 对于实验结果的理论研究需要借助核磁共振等技术, 这些技术实验要求高、难度高, 对于大分子物质很难测得;分子动力学模拟的方法对于计算机能力要求较高, 难度也较大, 但是结合实验结果, 利于理论计算研究。此外, 将分子动力学模拟与酶分子改造等方法相结合, 不仅可以大大地减少实验的工作量, 还可以解释一些实验无法测得的理论。如糖蛋白的研究, 王小艳等[60]利用分子动力学模拟来分析糖链与蛋白相互作用的结合构象, 并且模拟了完整糖链与多亚基大分子蛋白结合的稳定性, 得到了“糖基发卡”与“糖基垫片”结构, 这为理性设计提高酶稳定性提供理论基础。
4 总结与展望脂肪酶是重要的工业酶制剂, 在制药、化工和新能源等领域应用广泛。其在工业上的进一步扩大利用, 主要取决于它的性质和功能。目前已有多种蛋白酶研究出了前导肽辅助蛋白酶折叠的机制, 这些蛋白酶大多是丝氨酸蛋白酶。由于脂肪酶也属于丝氨酸蛋白酶, 因此蛋白酶辅助蛋白质的折叠机制可以指导脂肪酶折叠机制的研究。此外, 我们发现前导肽影响脂肪酶在异源宿主中的分泌表达, 以及表达后脂肪酶的空间结构、活性和稳定性等生化性质。对前导肽的某些特殊位点进行突变, 如糖基化位点和催化中心等, 发现脂肪酶的性质变化很大, 但是关于前导肽突变导致脂肪酶性质变化的机制依然不够清晰, 这主要是因为目前的研究手段较为单一。因此, 接下来的研究可以考虑从以下3个方面进行:(1)研究前导肽介导脂肪酶折叠的机制及前导肽发挥的作用, 从而可通过改变前导肽辅助折叠的途径调控脂肪酶的活性与功能;(2)将分子动力学模拟与定点突变技术相结合, 研究前导肽上糖基化位点对脂肪酶活性的影响及机理;(3)目前前导肽的研究方法较为单一, 主要是通过随机突变和定点突变等酶分子改造方法, 未来的研究可以探索更多的前导肽研究方法。随着前导肽对脂肪酶影响研究的进步, 不仅会丰富蛋白质的折叠理论, 还会拓展蛋白质工程, 为未来的蛋白质研究提供更多可能。
| [1] |
Anfinsen CB. Principles that govern the folding of protein chains. Science, 1973, 181(4096): 223-230. DOI:10.1126/science.181.4096.223 |
| [2] |
Ikemura H, Takagi H, Inouye M. Requirement of pro-sequence for the production of active subtilisin E in Escherichia coli. J Biol Chem, 1987, 262(16): 7859-7864. DOI:10.1016/S0021-9258(18)47646-6 |
| [3] |
Jia Y, Cao XH, Deng Y, et al. Four residues of propeptide are essential for precursor folding of nattokinase. Acta Bioch Biophys Sin, 2014, 46(11): 957-964. DOI:10.1093/abbs/gmu093 |
| [4] |
Nagayama M, Kuroda K, Ueda M. Identification of interaction site of propeptide toward mature carboxypeptidase Y (mCPY) based on the similarity between propeptide and CPY inhibitor (IC). Biosci Biotech Bioch, 2012, 76(1): 153-156. DOI:10.1271/bbb.110668 |
| [5] |
Boel E, Huge-Jensen B, Christensen M, et al. Rhizomucor miehei triglyceride lipase is synthesized as a precursor. Lipids, 1988, 23(7): 701-706. DOI:10.1007/BF02535672 |
| [6] |
Ueda M, Takahashi S, Washida M, et al. Expression of Rhizopus oryzae lipase gene in Saccharomyces cerevisiae. J Mol Catal B Enzym, 2002, 17(3/5): 113-124. |
| [7] |
Shinde U, Inouye M. Intramolecular chaperones and protein folding. Trends Biochem Sci, 1993, 18(11): 442-446. DOI:10.1016/0968-0004(93)90146-E |
| [8] |
Brzozowski AM, Derewenda U, Derewenda ZS, et al. A model for interfacial activation in lipases from the structure of a fungal lipase-inhibitor complex. Nature, 1992, 351(6326): 491-494. |
| [9] |
van Leusden P, Den Hartog GJM, Bast A, et al. Lipase diffusion in oil-filled, alginate micro- and macrobeads. Food Hydrocolloid, 2018, 85: 242-247. DOI:10.1016/j.foodhyd.2018.07.028 |
| [10] |
Maldonado RR, Lopes DB, Aguiar-Oliveira E, et al. A review on geotrichum lipases: production, purification, immobilization and applications. Chem Biochem Eng, 2016, 30(4): 439-454. |
| [11] |
Lee HS, Oh Y, Kim MJ, et al. Molecular basis of aqueous-like activity of lipase treated with glucose-headed surfactant in organic solvent. J Phys Chem B, 2018, 122(47): 10659-10668. DOI:10.1021/acs.jpcb.8b07686 |
| [12] |
Huang ZG, Brennan CS, Zheng HT, et al. The effects of fungal lipase-treated milk lipids on bread making. LWT, 2020, 128: 109455. DOI:10.1016/j.lwt.2020.109455 |
| [13] |
Singh R, Kumar M, Mittal A, et al. Microbial enzymes: Industrial progress in 21st century. 3 Biotech, 2016, 6(2): 174. DOI:10.1007/s13205-016-0485-8 |
| [14] |
Radha P, Prabhu K, Jayakumar A, et al. Biochemical and kinetic evaluation of lipase and biosurfactant assisted ex novo synthesis of microbial oil for biodiesel production by Yarrowia lipolytica utilizing chicken tallow. Process Biochem, 2020, 95: 17-29. DOI:10.1016/j.procbio.2020.05.009 |
| [15] |
Lu XL, Zhang WH, Wang YD, et al. A high-detergent-performance, cold-adapted lipase from Pseudomonas stutzeri PS59 suitable for detergent formulation. J Mol Catal B Enzym, 2014, 102: 16-24. DOI:10.1016/j.molcatb.2014.01.006 |
| [16] |
Brady L, Brzozowski AM, Derewenda ZS, et al. A serine protease triad forms the catalytic centre of a triacylglycerol lipase. Nature, 1990, 343(6260): 767-770. DOI:10.1038/343767a0 |
| [17] |
Wang J, Wang D, Wang B, et al. Enhanced activity of Rhizomucor miehei lipase by directed evolution with simultaneous evolution of the propeptide. Appl Microbiol Biotechnol, 2013, 96(2): 443-450. |
| [18] |
Satomura A, Kuroda K, Ueda M. Generation of a functionally distinct Rhizopus oryzae lipase through protein folding memory. PLoS ONE, 2015, 10(5): e0124545. DOI:10.1371/journal.pone.0124545 |
| [19] |
Samuelson P, Cano F, Robert A, et al. Engineering of a Staphylococcus carnosus surface display system by substitution or deletion of a Staphylococcus hyicus lipase propeptide. FEMS Microbiol Lett, 1999, 179(1): 131-139. DOI:10.1111/j.1574-6968.1999.tb08718.x |
| [20] |
Luo W, He D, Fu JY, et al. Effect of propeptide variation on properties of Rhizomucor miehei lipase. J Biobased Mater Bio, 2018, 12(4): 330-338. DOI:10.1166/jbmb.2018.1786 |
| [21] |
Shi YP, Zhou X, Hu MR, et al. Effect of signal peptides on the expression of laccase in Pichia pastoris. Acta Microbiol Sin, 2014, 54(12): 1446-1452 (in Chinese). 石艺平, 周雪, 胡美荣, 等. 不同信号肽对毕赤酵母表达漆酶的影响. 微生物学报, 2014, 54(12): 1446-1452. |
| [22] |
González-Páez GE, Roncase EJ, Wolan DW. X-ray structure of an inactive zymogen clostripain-like protease from Parabacteroides distasonis. Acta Cryst D, 2019, 75(3): 325-332. DOI:10.1107/S2059798319000809 |
| [23] |
Wandersman C. Secretion, processing and activation of bacterial extracellular proteases. Mol Microbiol, 1989, 3(12): 1825-1831. DOI:10.1111/j.1365-2958.1989.tb00169.x |
| [24] |
Liu YH, Lin S, Zhang XQ, et al. A novel approach for improving the yield of Bacillus subtilis transglutaminase in heterologous strains. J Ind Microbiol Biotechnol, 2014, 41(8): 1227-1235. DOI:10.1007/s10295-014-1468-6 |
| [25] |
Ayalew S, Murdock BK, Snider TA, et al. Mannheimia haemolytica IgA-specific proteases. Vet Microbiol, 2019, 239: 108487. DOI:10.1016/j.vetmic.2019.108487 |
| [26] |
Óskarsson KR, Kristjánsson MM. Improved expression, purification and characterization of VPR, a cold active subtilisin-like serine proteinase and the effects of calcium on expression and stability. Biochim Biophys Acta Proteins Proteom, 2019, 1867(2): 152-162. DOI:10.1016/j.bbapap.2018.11.010 |
| [27] |
Boon L, Ugarte-Berzal E, Vandooren J, et al. Protease propeptide structures, mechanisms of activation, and functions. Crit Rev Biochem Mol Biol, 2020, 55(2): 111-165. DOI:10.1080/10409238.2020.1742090 |
| [28] |
Demidyuk IV, Shubin AV, Gasanov EV, et al. Propeptides as modulators of functional activity of proteases. Biomol Concepts, 2010, 1(3/4): 305-322. |
| [29] |
Nabiabad HS, Amini M, Kianersi F. Ipomoea batatas: papain propeptide inhibits cysteine protease in main plant parasites and enhances resistance of transgenic tomato to parasites. Physiol Mol Biol Plants, 2019, 25(4): 933-943. DOI:10.1007/s12298-019-00675-3 |
| [30] |
Baker D, Sohl JL, Agard DA. A protein-folding reaction under kinetic control. Nature, 1992, 356(6366): 263-265. DOI:10.1038/356263a0 |
| [31] |
Derewenda ZS, Derewenda U, Dodson GG. The crystal and molecular structure of the Rhizomucor Miehei Triacylglyceride lipase at 1.9 a resolution. J Mol Biol, 1992, 227(3): 818-839. DOI:10.1016/0022-2836(92)90225-9 |
| [32] |
Moroz OV, Blagova E, Reiser V, et al. Novel inhibitory function of the Rhizomucor miehei lipase propeptide and three-dimensional structures of its complexes with the enzyme. ACS Omega, 2019, 4(6): 9964-9975. DOI:10.1021/acsomega.9b00612 |
| [33] |
Zha DM, Zhang HD, Zhang HJ, et al. N-terminal transmembrane domain of lipase LipA from Pseudomonas protegens Pf-5: A must for its efficient folding into an active conformation. Biochimie, 2014, 105: 165-171. DOI:10.1016/j.biochi.2014.07.007 |
| [34] |
Cai HY, Zhao MJ, Li Y, et al. Pentapeptide prosequence enhances expression and structure folding of recombinant Thermomyces lanuginosus lipase in Pichia pastoris. Protein Pept Lett, 2017, 24(7): 676-681. |
| [35] |
Beer HD, Wohlfahrt G, Schmid RD, et al. The folding and activity of the extracellular lipase of Rhizopus oryzae are modulated by a prosequence. Biochem J, 1996, 319(2): 351-359. DOI:10.1042/bj3190351 |
| [36] |
Sayari A, Frikha F, Miled N, et al. N-terminal peptide of Rhizopus oryzae lipase is important for its catalytic properties. FEBS Lett, 2005, 579(5): 976-982. DOI:10.1016/j.febslet.2004.12.068 |
| [37] |
Takahashi S, Ueda M, Tanaka A. Function of the prosequence for in vivo folding and secretion of active Rhizopus oryzae lipase in Saccharomyces cerevisiae. Appl Microbiol Biotechnol, 2001, 55(4): 454-462. DOI:10.1007/s002530000537 |
| [38] |
Sauerzapfe B, Namdjou DJ, Schumacher T, et al. Characterization of recombinant fusion constructs of human β-1, 4-galactosyltransferase 1 and the lipase pre-propeptide from Staphylococcus hyicus. J Mol Catal B Enzym, 2008, 50(2/4): 128-140. |
| [39] |
Weissman JS, Kim PS. The pro region of BPTI facilitates folding. Cell, 1992, 71(5): 841-851. DOI:10.1016/0092-8674(92)90559-U |
| [40] |
Creighton TE, Bagley CJ, Cooper L, et al. On the biosynthesis of bovine pancreatic trypsin inhibitor (BPTI): Structure, processing, folding and disulphide bond formation of the precursor in vitro and in microsomes. J Mol Biol, 1993, 232(4): 1176-1196. DOI:10.1006/jmbi.1993.1470 |
| [41] |
Bryan P, Wang L, Hoskins J, et al. Catalysis of a protein folding reaction: Mechanistic implications of the 2.0. ANG. structure of the subtilisin-prodomain complex. Biochemistry, 1995, 34(32): 10310-10318. DOI:10.1021/bi00032a026 |
| [42] |
Shinde UP, Liu JJ, Inouye M. Protein memory through altered folding mediated by intramolecular chaperones. Nature, 1997, 389(6650): 520-522. DOI:10.1038/39097 |
| [43] |
Bjerga GEK, Larsen Ø, Arsın H, et al. Mutational analysis of the pro-peptide of a marine intracellular subtilisin protease supports its role in inhibition. Proteins, 2018, 86(9): 965-977. DOI:10.1002/prot.25528 |
| [44] |
Yan XX. Cloning, codon optimization and expression of Rhizopus oryzae lipase gene in Pichia pastoris[D]. Wuhan: Huazhong University of Science & Technology, 2009 (in Chinese). 严翔翔.米根霉脂肪酶基因的克隆、密码子优化及在毕赤酵母中的分泌表达[D].武汉: 华中科技大学, 2009. |
| [45] |
Liu Y, Xie WP, Liu HW. Enhanced activity of Rhizomucor miehei lipase by deglycosylation of its propeptide in Pichia pastoris. Curr Microbiol, 2014, 68(2): 186-191. DOI:10.1007/s00284-013-0460-0 |
| [46] |
Yu XW, Sha C, Guo YL, et al. High-level expression and characterization of a chimeric lipase from Rhizopus oryzae for biodiesel production. Biotechnol Biofuels, 2013, 6: 29. DOI:10.1186/1754-6834-6-29 |
| [47] |
Niu WN, Li ZP, Tan TW. Secretion of pro-and mature Rhizopus arrhizus lipases by Pichia pastoris and properties of the proteins. Mol Biotechnol, 2006, 32(1): 73-81. DOI:10.1385/MB:32:1:073 |
| [48] |
Huang JJ, Zhao QY, Chen LX, et al. Improved production of recombinant Rhizomucor miehei lipase by coexpressing protein folding chaperones in Pichia pastoris, which triggered ER stress. Bioengineered, 2020, 11(1): 375-385. DOI:10.1080/21655979.2020.1738127 |
| [49] |
Nakagawa M, Ueyama M, Tsuruta H, et al. Functional analysis of the cucumisin propeptide as a potent inhibitor of its mature enzyme. J Biol Chem, 2010, 285(39): 29797-29807. DOI:10.1074/jbc.M109.083162 |
| [50] |
Wang N, Wang KY, Xu FF, et al. The effect of N-glycosylation on the expression of the tetanus toxin fragment C in Pichia pastoris. Protein Express Purif, 2020, 166: 105503. DOI:10.1016/j.pep.2019.105503 |
| [51] |
Kajiwara S, Yamada R, Matsumoto T, et al. N-linked glycosylation of thermostable lipase from Bacillus thermocatenulatus to improve organic solvent stability. Enzyme Microb Tech, 2020, 132: 109416. DOI:10.1016/j.enzmictec.2019.109416 |
| [52] |
Yang M, Yu XW, Zheng HY, et al. Role of N-linked glycosylation in the secretion and enzymatic properties of Rhizopus chinensis lipase expressed in Pichia pastoris. Microb Cell Fact, 2015, 14: 40. DOI:10.1186/s12934-015-0225-5 |
| [53] |
Liu Y. Expression and molecular modification of Rhizomucor miehei Lipase in Pichia pastoris[D]. Hangzhou: Zhejiang University, 2014 (in Chinese). 刘悦.米黑霉脂肪酶在毕赤酵母中的表达及分子改造[D].杭州: 浙江大学, 2014. |
| [54] |
Derewenda U, Swenson L, Wei Y, et al. Conformational lability of lipases observed in the absence of an oil-water interface: crystallographic studies of enzymes from the fungi Humicola lanuginosa and Rhizopus delemar. J Lipid Res, 1994, 35(3): 524-534. DOI:10.1016/S0022-2275(20)41203-9 |
| [55] |
Falzon L, Patel S, Chen YJ, et al. Autotomic behavior of the propeptide in propeptide-mediated folding of prosubtilisin E. J Mol Biol, 2007, 366(2): 494-503. DOI:10.1016/j.jmb.2006.11.019 |
| [56] |
Vatandoost J, Bos MHA. Improved activity and expression of recombinant human factor IX by propeptide engineering. DARU J Pharm Sci, 2019, 27(2): 653-660. DOI:10.1007/s40199-019-00299-8 |
| [57] |
Wang ZY, Lv PM, Luo W, et al. Expression in Pichia pastoris and characterization of Rhizomucor miehei lipases containing a new propeptide region. J Gen Appl Microbiol, 2016, 62(1): 25-30. DOI:10.2323/jgam.62.25 |
| [58] |
Cheung DL, Samantray S. Molecular dynamics simulation of protein biosurfactants. Colloids Interfaces, 2018, 2(3): 39. DOI:10.3390/colloids2030039 |
| [59] |
Luo W. The allosteric regulation mechanism of Rhizomucormiehei lipase via modification of the propeptide[D]. Guangzhou: South China Agricultural University, 2018 (in Chinese). 罗文.前导肽对米黑根毛霉脂肪酶的别构调控及其影响机理探究[D].广州: 华南农业大学, 2018. |
| [60] |
Wang XY. The effect of N-glycosylation design on the structure and properties of β-glucuronidase[D]. Tianjin: Tianjin University, 2018 (in Chinese). 王小艳.糖基化设计对β-葡萄糖醛酸苷酶结构和性能的影响[D].天津: 天津大学, 2016. |
 2021, Vol. 37
2021, Vol. 37