中国科学院微生物研究所、中国微生物学会主办
文章信息
- 宋小平, 王雅洁, 蔡晶晶, 余银, 管利, 张宇亭
- Song Xiaoping, Wang Yajie, Cai Jingjing, Yu Yin, Guan Li, Zhang Yuting
- 基因拷贝数对重组毕赤酵母产谷氨酰胺转胺酶的影响
- Impact of gene dosage on recombinant transglutaminase production of Pichia pastoris
- 生物工程学报, 2020, 36(8): 1679-1688
- Chinese Journal of Biotechnology, 2020, 36(8): 1679-1688
- 10.13345/j.cjb.190531
-
文章历史
- Received: November 27, 2019
- Accepted: March 8, 2020
- Published: April 7, 2020
2. 安徽省重组蛋白药物工程技术研究中心,安徽 合肥 230022
2. Anhui Engineering Research Center of Recombinant Protein Pharmaceutical Biotechnology, Hefei 230022, Anhui, China
微生物的谷氨酰胺转胺酶(Microbial transglutaminase,MTG)由于具有低蛋白分子量、非Ca2+依赖性、较广的底物范围等较优的酶学性质,被广泛用于食品工业、生物医药[1-3]、组织工程[4-5]、定点蛋白交联[1, 5]、同源抗体偶联药物的构建[5]等多个领域。
目前,由于微生物发酵法可用于大规模生产谷氨酰胺转胺酶,所以最终实现了该酶的商业化生产[5-6],但MTG价格较高。鉴于茂源链霉菌特有的催化性能及活化机制使MTG在异源宿主中的高效表达遇到诸多问题[7-9]。在酵母中单独表达MTG成熟酶,无法获得活性MTG[8, 10]。通过将该酶原的前导肽(Pro-peptide)和成熟酶MTG作为两个独立元件共表达,可直接表达活性MTG,200 mL摇瓶发酵上清中MTG酶活最高达1.83 U/mL,是目前报道的最高水平[11-12],然而其酶活水平需要进一步提高。
外源基因的拷贝数通常成为蛋白表达量的限制性因素,增加外源基因的拷贝数能提高外源蛋白的表达量[13]。根据酵母菌较易发生同源重组的特点,使用重复序列作为载体同源重组位点是提高外源基因在酵母基因组中拷贝数的有效途径[13-14]。核糖体DNA (Ribosomal DNA,rDNA)是指编码核糖体RNA的一段高度重复的DNA序列,重复序列由转录区和非转录区组成[14]。毕赤酵母GS115基因组的4种rRNA基因包括18S、5.8S、25S和5S [14-15],其中18S、5.8S和25S rRNA基因是串联在一起的,存在100–120个重复序列,被rDNA的非转录区(Non-transcribed intergenic spacer,NTS)隔开[16]。如果以毕赤酵母非转录rDNA序列(rDNA)为目的基因整合位点,理论上可以得到100–120个目的基因拷贝数[13-14, 17-18]。
本研究基于毕赤酵母非转录rDNA序列构建mtg拷贝数定向放大的表达载体pPICZα-rDNA- mtg,并被转化到表达前导肽(Pro-peptide)的重组菌株pGAP9-pro/GS115,构建得到共表达菌株pro/rDNA-mtg,分析了基因剂量对重组毕赤酵母表达MTG的影响,并研究了较高表达菌株在发酵罐中高密度发酵。
1 材料与方法 1.1 试验菌株、试剂和培养基 1.1.1 菌株和质粒质粒载体pGAP9-pro和pPICZa-mtg及pGAP9-pro/GS115菌株为本实验室构建。
1.1.2 试剂N-carboxybenzoyl-L-glutaminyl-glycine (N- CBZ-Gln-Gly)和L-谷氨酸-γ-单羟胺酸购自Sigma-Aldrich公司;还原型谷胱甘肽购自华美生物工程有限公司;anti-MTG购自Eurogentec公司。荧光定量PCR仪购自Applied Biosystems公司。AKTA Pure蛋白质层析纯化系统和镍柱购自GE公司。其他试剂均为国产分析纯。
1.1.3 培养基大肠杆菌Escherichia coli培养基:LLB;毕赤酵母培养基:YPD、MD、BMGY、BMMY,配方参照Invitrogen公司的操作手册,毕赤酵母菌培养温度28 ℃,诱导温度25 ℃[12, 14]。
1.2 方法 1.2.1 基因、引物合成和DNA序列测定引物均由南京金斯瑞生物科技有限公司合成,DNA测序由通用生物系统(安徽)有限公司完成。
1.2.2 多拷贝重组表达菌株的构建多拷贝基因(mtg)及其前导肽基因(pro)共表达菌株的构建原理如图 1所示。将谷氨酰胺转胺酶的前导肽基因(pro)与成熟酶基因(mtg)的编码序列分别插入到pGAP9和pPICZα的XhoⅠ、NotⅠ位点,构建得到表达质粒pGAP9-pro和pPICZα- rDNA-mtg。质粒pGAP9-pro和pPICZα-rDNA-mtg分别含有组氨酸脱氢酶(HIS4)和rDNA重复片段基因,分别同源重组到P. pastoris (GS115)的基因组his4区和非编码rDNA序列(图 1)。理论上避免了pro和mtg基因在P. pastoris同一位点同源重组的概率,确保mtg定向整合到P. pastoris的rDNA序列,保证mtg的多拷贝[2, 14]。
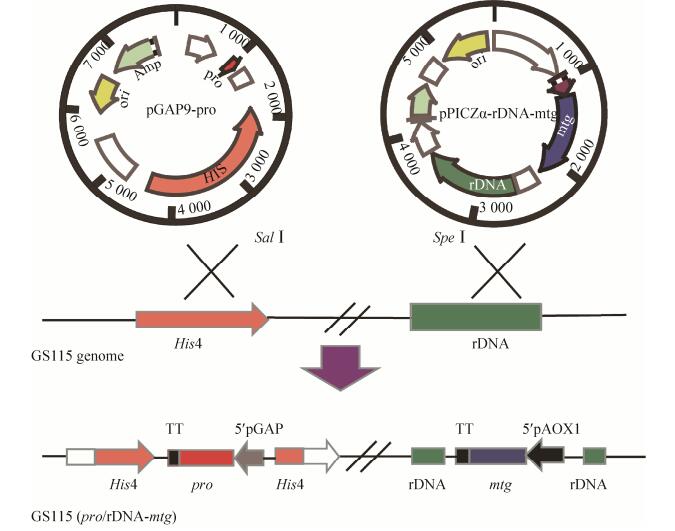
|
| 图 1 基于rDNA的多拷贝基因(rDNA-mtg)及其前导肽基因(pro)共表达菌株的构建 Fig. 1 Principle of co-expression of multicopy gene (rDNA-mtg) and its precursor peptide gene (pro). |
| |
构建过程分两步:
第一步,构建rDNA介导的多拷贝谷氨酰胺转氨酶基因(mtg)表达载体。首先,重叠PCR扩增AOX1-rDNA融合基因:分别扩增AOX1- terminator (表 1:引物P1和P2)和rDNA片段(表 1:引物P3和P4),其中AOX1-terminator 5′端引入NotⅠ酶切位点,3′端引入rDNA overlap片段,用于与rDNA基因的拼接,大小410 bp。rDNA 5′端引入AOX1 terminator重叠序列,3′端引入BamHⅠ酶切位点,含有SpeⅠ单酶切位点,大小为1.2 kb。扩增产物采用核酸凝胶回收试剂盒回收,得到预期为1.6 kb的产物,此基因产物两端分别带上NotⅠ和BamHⅠ酶切位点,命名为AOX1-rDNA。然后,将获得的1.6 kb AOX1-rDNA以及载体pPICZa-mtg (实验室保存)均使用限制性内切酶NotⅠ和BamHⅠ双酶切。酶切产物采用PCR试剂盒回收。回收所得的产物采用T4 DNA连接酶连接,并转至E. coli TOP10感受态细胞,均匀涂布到含50 μg/mL Zeocin抗性的LLB平板上,37 ℃培养过夜。通过菌液PCR法鉴定阳性克隆,鉴定正确的阳性克隆送测序,测序正确的克隆命名为pPICZα-rDNA-mtg。
| Primer | Primer sequence (5′–3′) |
| P1 | TATGCGGCCGCCAGCTTTCTAGAACAAAAACTCATCTC |
| P2 |  TTATCTCACTTAATCTTCTGTACTCTG
TTATCTCACTTAATCTTCTGTACTCTG |
| P3 |  TAGTTAGGTTACCGTTTTTCCTAATATTTAAG
TAGTTAGGTTACCGTTTTTCCTAATATTTAAG |
| P4 P5 P6 P7 P8 |
CGC  CTTCCACCAACAGTCAACCAC CTTCCACCAACAGTCAACCACCAGTC GGTATTAACGGTTTCGGACGTATTG GATGTTGACAGGGTCTCTCTCTTGG TGAAGAAAGAATTGGCTAACGG AGCTGGTCTGAAAGCATCTGG |
Remarks: The underlined sites (__) and the double corrugated underlined sites(
 )are thoses for the digestion of restriction enzymes XhoⅠ and BamHⅠ, respectively. The overlapping segments of the AOX1 terminal-rDNA fusion gene is marked with the bold underlining(
)are thoses for the digestion of restriction enzymes XhoⅠ and BamHⅠ, respectively. The overlapping segments of the AOX1 terminal-rDNA fusion gene is marked with the bold underlining(
 ).
).
|
|
第二步,将构建成功的表达载体pPICZα-rDNA-mtg用SpeⅠ线性化并电转化到pGAP9-pro/GS115 (实验室保存)[2],电转液均匀涂布于MD+Zeocin (200 μg/mL)平板上,同时设置阴性对照,28 ℃培养3–4 d,直至长出直径1 mm左右克隆,选择8个克隆进行直接PCR鉴定。
鉴定正确的克隆GS115 (pro/rDNA-mtg)接入24孔板筛选阳性表达克隆,BMGY (pH 6.0)培养体系2 mL,28 ℃、200 r/min过夜培养,24 h后测OD600=5–6,离心收集菌体,用2 mL BMMY (1%甲醇,pH 6.0)中重悬菌体后诱导表达,25 ℃诱导、每隔24 h补加一次甲醇至终浓度为1% (V/V),诱导48 h,发酵液12 000×g离心10 min,滤液进行Western blotting检测,抗体为Anti-MTG,阳性表达克隆保存于−20 ℃。
1.2.3 重组菌株中mtg拷贝数的检测选择蛋白表达较高的4个阳性克隆,实时荧光定量PCR (qPCR)技术测定mtg基因在毕赤酵母基因组中的拷贝数,用管家基因gap的拷贝数表征模板中基因组的起始拷贝数[19]。首先,用含有gap和mtg的标准质粒制备gap和mtg的标准曲线。然后,荧光定量PCR获得每个样品gap和mtg的Ct值,将得到的Ct值分别代入gap和mtg标准曲线中,求出每个样品中gap基因和mtg基因的起始模板拷贝数[19-20]。mtg基因在毕赤酵母基因组中的拷贝数可以通过mtg基因起始模板拷贝数与gap基因起始模板拷贝数的比值求出[19, 21]。具体方法:提取4个样品中毕赤酵母菌株基因组DNA作为模板,分别用引物P5/P6和P7/P8 (表 1)进行荧光定量PCR,每个样品重复检测3次,每次进行独立的质粒抽提和荧光定量PCR过程[21]。反应体系:10 μL,其中包括SYRB Green Realtime PCR Master 5 μL,DNA模板1 μL,引物各0.5 μL (10 μmol/L),dd H2O 3 μL。反应条件:37 ℃保温2 min,95 ℃变性10 min,95 ℃退火10 s,60 ℃延伸30 s,72 ℃ 1 min,共40个循环[21]。
1.2.4 拷贝数对酶活和蛋白表达的影响将上述实验获得的不同拷贝数的菌株接种于YPDZ (Z:200 μg/mL)固体培养基中,28 ℃培养2–3 d,直至长出单克隆。挑取单菌落于5 mL YPD液体培养基中,摇瓶过夜培养,全部转接到50 mL BMGY (pH 6.0)的摇瓶中,28 ℃、200 r/min培养过夜,24 h后测OD600,当OD600达到6时,低速离心收集菌体,用200 mL的BMMY (甲醇1%,pH 6.0)重悬菌体后诱导表达,初始OD600= 1.0–1.5,诱导温度25 ℃,每隔24 h补加一次甲醇至终浓度为1% (V/V),并取样,诱导72 h。
将72 h发酵上清pH调至7.2,12 000×g离心10 min,去除菌体沉淀,留上清。将样品进样,注入AKTA蛋白纯化仪的镍柱。按照流程:平衡→上样→平衡→Wash→洗脱。A液平衡(50 mmol/L PBS,0.3 mol/L NaCl,pH 7.2),B液冲洗(50 mmol/L PBS,0.3 mol/L NaCl,20 mmol/L咪唑,pH 7.2),C液洗脱(20 mmol/L PBS,0.3 mol/L NaCl,200 mmol/L咪唑,pH 7.2);收集洗脱液。洗脱液用0.2 mol/L PBS (pH 7.2)透析去除咪唑,透析样品采用非还原SDS-PAGE检测,胶浓度10%,每孔上样量20 µL,Western blotting鉴定目标蛋白,鉴定用抗体Anti-MTG。
异羟肟酸比色法测定MTG酶活,按照参考文献[2, 7, 11, 12]的方法进行。MTG酶活定义为:在37 ℃条件下,MTG每分钟催化底物(N-CBZ- Gln-Gly,Sigma公司)生成1 µmol谷氨酸-单羟胺酸(氧肟酸)为1个酶活单位。蛋白浓度测定采用Brandford方法,标准蛋白为牛血清蛋白(Sigma)[2, 11]。
1.2.5 mtg-3c、mtg-6c的高密度发酵摇瓶发酵初步考察了拷贝数对MTG酶活和蛋白表达的影响,为了进一步考察拷贝数对菌体细胞产酶和细胞生长过程的影响,分别选择其中较低和较高拷贝数的两株菌(mtg-3c、mtg-6c)进行高密度发酵。在保证发酵条件一致的前提下,比较发酵过程的变化情况。将100 mL YPD种子液接到1.2 L BMGY (pH 6.0)发酵培养基,初始培养基组成:1.2 L BMGY (4%甘油) +150 mL YNB+150 mL PPB,发酵参数设定为:pH 6.0,生长温度28 ℃,搅拌转速600–800 r/min,通气量10 L/min。当罐内基础甘油用完后,溶氧会在短时间内上升,此时开始补甘油,50%甘油(含12 mL/L PTM1))恒速补料25 mL/(L·h)。当菌体湿重达到35–40 g/L时停止补甘油,开始补加100%甲醇诱导(甲醇含12 mL/L PTM1)。甲醇诱导采用三步补料(根据甲醇电极判断补料时间点),第一步补甲醇速率3.6 mL/(L·h) (大约持续4–6 h),第二步补甲醇速率7.2 mL/(L·h),第三步补甲醇10.8 ml/(L·h),持续到下罐。诱导温度调整为25 ℃。整个发酵过程中控制溶氧水平为25%–30%[14]。每12 h取样测定菌体湿重和酶活,诱导72 h。发酵液预处理及MTG纯化过程参照1.2.4。
2 结果与分析 2.1 不同拷贝数重组毕赤酵母菌株的构建提取GS115 (pro/rDNA-mtg) 8个克隆的基因组,分别采用目的基因pro和mtg的上、下游引物进行PCR鉴定,分别扩增出100–200 bp和1 000 bp左右的条带(图 2A)。PCR结果显示pro和mtg基因分别整合到毕赤酵母染色体的不同位点,所有克隆构建正确(图 2A)。

|
| 图 2 不同拷贝数重组毕赤酵母菌株pro/rDNA-mtg的建立 Fig. 2 Establishment of pro/rDNA-mtg with different copy number. (A) Detection of GS115(pro/rDNA-mtg) expression strains by PCR. M: DNA marker; 1–8: the 8 clones of pro/rDNA-mtg were amplified by PCR with the primers of pro and mtg, and the products were 1 000 bp and 150 bp, respectively. (B) The transformed clones of pro/rDNA-mtg were screened by Western blotting (Anti-MTG). |
| |
选择构建正确的8个克隆,用24孔板初步筛选阳性克隆,对诱导48 h的发酵上清进行检测,结果如图 2B所示。经检测的8个克隆均有阳性表达,其中克隆#3、#4、#6、#7表达较高。
2.2 重组菌株中mtg基因拷贝数将MTG表达量较高的阳性表达克隆(#3、#4、#6、#7)进行实时荧光定量PCR (qPCR)检测,mtg基因在毕赤酵母基因组中的拷贝数结果见图 3和表 2。gap和mtg基因标准曲线为:y= –3.754 6x+39.663 (R2=0.998 5),y=–3.363 2x+37.131 (R2=0.997 8) (图 3A、B)。标准曲线的反应效率即引物扩增效率,用E表示,通过公式E=10 (–1/斜斜率)–1计算求出gap和mtg基因的扩增效率分别为86.66%和94.88%,说明两者的扩增反应有效,因而该标准曲线可用于计算被测菌株中mtg基因拷贝数。

|
| 图 3 gap (A)和mtg (B)基因的标准曲线 Fig. 3 Standard curves for gap (A) and mtg genes (B). |
| |
| Strain | Value Ct | Gene copy (10n) | Number of copies of mtg gene in P. pichia genome (mtg copy number/gap copy number) | ||||
| mtg gene | gap gene | mtg gene | gap gene | ||||
| 1 | 14.14±0.27 | 16.49±0.17 | 5.58±1.02 | 1.66±0.19 | 3.36±0.24 | ||
| 2 | 14.93±0.28 | 16.67±0.22 | 3.29±0.50 | 1.48±0.18 | 2.21±0.11 | ||
| 3 | 14.97±0.30 | 18.23±0.23 | 3.20±0.63 | 5.60±0.71 | 5.72±0.35 | ||
| 4 | 15.30±0.41 | 19.05±0.17 | 2.56±0.55 | 3.36±0.31 | 7.62±1.11 | ||
| Datas are presented as x±s of triplicate observations. strain 1: the clone with the highest enzyme activity. strain 2, 3, 4: another three positive clones tested. | |||||||
被检测的4个共表达菌株基因组中mtg拷贝数如表 2[2]所示,结果显示酶活最高的菌株中mtg基因以3.36个拷贝的形式存在于酵母基因组中,命名为mtg-3c。其他3个菌株中mtg基因拷贝数分别为2.21、5.72和7.62 (命名为mtg-2c、mtg-6c、mtg-8c)。实验结果表明,不同拷贝的mtg基因定向到P. pastoris的重复rDNA位点,被检测菌株的最高拷贝数为mtg-8c,但是表达蛋白和酶活都表现出最低。mtg-3c菌株产MTG酶活最高。
2.3 基因拷贝数影响蛋白表达和酶活为了考察基因的拷贝数对蛋白质表达的影响,通过对发酵过程中诱导阶段不同时间点取样的测定,结果如图 4、5和表 3所示。
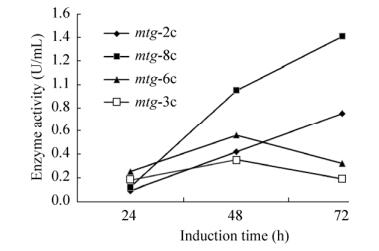
|
| 图 4 不同拷贝数mtg对谷氨酰胺转胺酶活力的影响 Fig. 4 Effect of different gene copy number on transaminase activity. |
| |

|
| 图 5 不同拷贝数的菌体表达蛋白的纯化结果 Fig. 5 Purified protein expressed by different copy number |
| |
| Strain (pro/rDNA-mtg) |
Enzyme activity (U/mL) |
Protein concentration (µg/ mL) |
| mtg-2c mtg-3c mtg-6c mtg-8c |
0.75±0.04b 1.41±0.08a 0.32±0.03c 0.19±0.03c |
51.3±3.24b 89.13±5.85a 21.18±3.21c 16.35±2.51c |
| Datas are presented as x±s of triplicate observations. Values with different superscripts are significantly different (P < 0.05). | ||
从图 4看出,诱导24 h,谷氨酰胺转胺酶酶活随基因拷贝数的增加而增加, 即呈正效应(拷贝数不超过6);诱导48 h,mtg-6c和mtg-8c菌株表达谷氨酰胺转胺酶的酶活急剧下降;诱导72 h,mtg-3c菌株表达的谷氨酰胺转胺酶活力最高,可达到1.41 U/mL;拷贝数为2、6、8 (mtg-2c,mtg-6c,mtg-8c)菌株均表现出较低的酶活(酶活分别为0.75、0.32、0.19 U/mL)。由此推测, 在一定拷贝数范围内(拷贝数不超过6),毕赤酵母表达重组谷氨酰胺转胺酶的能力先随目的基因拷贝数的增加而增加,呈递增趋势;当拷贝数超过一定数值时(拷贝数为3),则呈下降趋势。
由于发酵液中总蛋白含有杂蛋白,总蛋白浓度并不能真实反映拷贝数对目的蛋白的影响,因此,对诱导72 h后发酵液纯化,纯化蛋白的电泳结果见图 5。从图 5可见,mtg-3c、mtg-2c和mtg-6c在43 kDa处均有两条特异性蛋白带,大小分别为38 kDa和40 kDa,分别为非糖基化条带和糖基化条带(非糖基化条带有活性,糖基化条带是否有活性需要进一步实验验证),与文献报道出现1–3条带的结果相一致[2, 11],说明目的蛋白纯度高。
不同拷贝数菌体表达的蛋白结果为:mtg-3c > mtg-2c > mtg-6c > mtg-8c (表 3),结果表明,mtg-3c表达最高;高拷贝数(mtg-8c、mtg-6c)随着诱导时间的延长,目的蛋白表达显著减少或降解加剧。目的蛋白浓度的水平也从侧面验证了谷氨酰胺转胺酶活力的高低。这与诱导72 h后谷氨酰胺转胺酶的活力显著下降是相一致的(图 4)。
2.4 pro/rDNA-mtg (mtg-3c,mtg-6c)的高密度表达在保证发酵条件一致的前提下,mtg-3c、mtg-6c的发酵结果见图 6,整个诱导过程持续72 h。结果表明,mtg-3c诱导前60 h,发酵上清酶活呈现上升趋势,诱导60 h后酶活力不再继续升高,此时最高酶活为3.12 U/mL,发酵菌体浓度可以达到59.4 g/L,mtg-3c的单位菌体酶活为52.1 U/g湿重。而mtg-6c在诱导前48 h,发酵上清酶活呈现上升趋势,48 h的最高酶活达到2.07 U/mL(最高水平),此时菌体浓度可以达到56.7 g/L,单位菌体酶活为36.5 U/g湿重;48 h后,酶活快速降至1.12 U/mL。从图 6可以看出,mtg-3c和mtg-6c最高酶活时的菌体浓度接近,但是mtg-3c的单位菌体酶活是mtg-6c的1.4倍,与表 3中拷贝数影响谷氨酰胺转胺酶蛋白浓度的结果一致,推测mtg-3c的高酶活与菌株较高分泌蛋白有关。
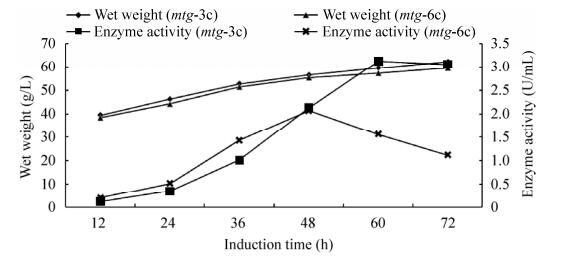
|
| 图 6 不同拷贝数菌株(mtg-3c、mtg-6c)的生长和产酶分析 Fig. 6 Analysis of growth and enzyme production of strains with different copy numbers. |
| |
对mtg-3c的72 h发酵上清进行纯化,检测纯化酶的酶活最高达到7.21 U/mL,蛋白浓度为437.2 µg/mL。
3 讨论毕赤酵母作为外源蛋白表达的宿主具有强效可调控启动子AOX1、表达量高、遗传性状稳定、培养基成分简单等突出的优点,已高效表达了干扰素、乙型肝炎病毒表面抗原、肌醇六磷酸酶、白蛋白、抗体、胶原蛋白等多种外源蛋白[1, 3-5]。尽管已经有研究人员尝试利用毕赤酵母来表达重组链霉菌MTG,但是MTG的产量还达不到工业化要求[9, 11]。本文基于rDNA构建多拷贝的谷氨酰胺转胺酶菌株,获得基因拷贝数分别为2、3、6和8的工程菌,并通过摇瓶发酵初步考察了拷贝数(2、3、6和8)对MTG酶活和蛋白表达的影响,在此基础上,对3拷贝和6拷贝菌株进行高密度发酵,进一步考察了拷贝数对菌体细胞产酶和细胞生长过程的影响。研究结果显示mtg-3c和mtg-6c最高酶活时的发酵菌体浓度相近,但是mtg-3c的最高单位菌体酶活是mtg-6c的1.4倍,与拷贝数影响谷氨酰胺转胺酶蛋白浓度的结果一致,推测mtg-3c的高酶活与菌株较高分泌表达有关。mtg-3c的纯化酶酶活最高达到7.21 U/mL,蛋白浓度为437.2 µg/mL。
研究表明,外源基因的拷贝数是影响外源蛋白高效表达的重要因素之一,通过增加基因的拷贝数可以有效提高外源蛋白的表达[13, 17-18, 22]。对于分泌表达的蛋白,当基因拷贝数为4–9左右,基因拷贝数与蛋白量呈正相关,一旦拷贝数超出这个范围,表达水平反而降低,可能是由于高拷贝外源基因的表达影响菌体生长,从而影响目的蛋白前体的合成和所需能量的供给[18, 23]。因此,适合毕赤酵母高表达的拷贝数,需要通过实验筛选,并不是拷贝数越多越好[18, 23]。本研究构建了表达载体pPICZα-rDNA-mtg,这个载体包括目的基因mtg以及在其两端连上来自宿主GS115 rDNA的非转录基因间隔区(NTS),基于rDNA定向增加mtg基因拷贝数,构建了mtg不同拷贝数的重组菌株[13, 17-18]。运用绝对定量PCR技术检测重组菌株mtg拷贝数,在被检测的阳性克隆中,mtg基因分别以2、3、6、8拷贝的形式存在于毕赤酵母基因组中。在相同培养条件下(200 mL BMMY),不同拷贝mtg对蛋白表达和酶活性的影响为:mtg-3c > mtg-2c > mtg-6c (表 3和图 4)。结果表明,在mtg基因拷贝数不超过3的情况下,重组菌株产生酶的速度较快,表明基因拷贝数对酶的产生有积极的影响。但是当基因拷贝数大于3时,菌株产酶活性明显下降。因此,对于毕赤酵母分泌表达MTG,3拷贝最适合pro/rDNA-mtg中pro和mtg的共表达,一旦超过这个3拷贝,此时基因拷贝数已经不再是MTG分泌表达的主要限制性因素,而蛋白翻译、翻译后修饰及蛋白质前体的靶向输送等蛋白表达环节可能成为影响其产量的主要瓶颈[24-25]。
最后,对3拷贝和6拷贝菌株进行高密度发酵,在相同培养条件下(pH、温度、溶解氧、甲醇三步补料),mtg-3c和mtg-6c发酵上清的最高酶活和单位菌体酶活分别为3.12 U/mL、52.1 U/g湿重和2.07 U/mL、36.5 U/g湿重。结果表明mtg-3c的单位菌体酶活是mtg-6c的1.4倍,mtg-3c生产的纯化酶酶活最高达到7.21 U/mL,蛋白浓度为437.2 µg/mL。在高密度发酵过程中,通过严格控制pH、温度、溶氧及甲醇分步补料,可以较大幅度提高MTG的产量。
研究表明,在mtg基因拷贝数不超过3的情况下,基因拷贝数对酶的产生有积极的影响,然而通过单纯增加基因的剂量提高蛋白表达而提高酶活是有限的。若结合分子进化改造获得突变酶可大幅度提高酶活及其热稳定性[25-27],是目前比较有效的方法,这有待后续研究。
| [1] |
de Souza CFV, Venzke JG, Flôres SH, et al. Enzymatic properties of transglutaminase produced by a new strain of Bacillus circulans BL32 and its action over food proteins. LWT-Food Sci Technol, 2011, 44(2): 443-450. |
| [2] |
Song XP, Shao CS, Guo YG, et al. Improved the expression level of active transglutaminase by directional increasing copy of mtg gene in Pichia pastoris. BMC Biotechnol, 2019, 19: 54. DOI:10.1186/s12896-019-0542-6 |
| [3] |
Garcia Y, Wilkins B, Collighan RJ, et al. Towards development of a dermal rudiment for enhanced wound healing response. Biomaterials, 2008, 29(7): 857-868. |
| [4] |
Martins IM, Matos M, Costa R, et al. Transglutaminases: recent achievements and new sources. Appl Microbiol Biotechnol, 2014, 98(16): 6957-6964. |
| [5] |
Dennler P, Chiotellis A, Fischer E, et al. Transglutaminase-based chemo-enzymatic conjugation approach yields homogeneous antibody- drug conjugates. Bioconjugate Chem, 2014, 25(3): 569-578. |
| [6] |
Huang L, Liu YH, Li MJ, et al. Expression of the transglutaminase from Bacillus subtilis and optimization of fermentation conditions for Corynebacterium glutamicum. J Tianjin Univ Sci Technol, 2016, 31(6): 11-16 (in Chinese). 黄琳, 刘逸寒, 李明杰, 等. 枯草芽胞杆菌谷氨酰胺转氨酶在谷氨酸棒杆菌中的分泌表达及诱导条件的优化. 天津科技大学学报, 2016, 31(6): 11-16. |
| [7] |
Ando H, Adachi M, Umeda K, et al. Purification and characteristics of a novel transglutaminase derived from microorganisms. Agri Biol Chem, 1989, 53(10): 2613-2617. |
| [8] |
Javitt G, Ben-Barak-Zelas Z, Jerabek-Willemsen M, et al. Constitutive expression of active microbial transglutaminase in Escherichia coli and comparative characterization to a known variant. BMC Biotechnol, 2017, 17: 23. |
| [9] |
Liu S, Zhang DX, Du GC, et al. Progress in expression and molecular modification of microbial transglutaminase. Chin J Biotech, 2011, 27(12): 1681-1689 (in Chinese). 刘松, 张东旭, 堵国成, 等. 微生物谷氨酰胺转胺酶的表达及分子改造研究进展. 生物工程学报, 2011, 27(12): 1681-1889. |
| [10] |
Salis B, Spinetti G, Scaramuzza S, et al. High-level expression of a recombinant active microbial transglutaminase in Escherichia coli. BMC Biotechnol, 2015, 15: 84. |
| [11] |
Yurimoto H, Yamane M, Kikuchi Y, et al. The pro-peptide of Streptomyces mobaraensis transglutaminase functions in cis and in trans to mediate efficient secretion of active enzyme from methylotrophic yeasts. Biosci Biotechnol Biochem, 2004, 68(10): 2058-2069. |
| [12] |
Li PF, Sun HB, You LJ, et al. Direct secretory expression of active microbial transglutaminase in Pichia pastoris. Chin J Biotech, 2013, 29(2): 180-188 (in Chinese). 李鹏飞, 孙红兵, 游丽金, 等. 利用毕赤酵母系统直接分泌表达具有活性的谷氨酰胺转胺酶. 生物工程学报, 2013, 29(2): 180-188. |
| [13] |
Marx H, Mecklenbräuker A, Gasser B, et al. Directed gene copy number amplification in Pichia pastoris by vector integration into the ribosomal DNA locus. FEMS Yeast Res, 2009, 9(8): 1260-1270. |
| [14] |
Guo YG, Wu J, Jia H, et al. Balancing the expression and production of a heterodimeric protein: recombinant agkisacutacin as a novel antithrombotic drug candidate. Sci Rep, 2015, 25: 11730. |
| [15] |
Zhi XH, Wang LN, Zhu P, et al. Construction and application of Saccharomyces cerevisiae integration vector based on rDNA sequence. Chin Med Biotechnol, 2011, 6(5): 330-335 (in Chinese). 支晓慧, 王丽娜, 朱平, 等. 基于rDNA序列的酵母整合载体的构建及应用. 中国医药生物技术, 2011, 6(5): 330-335. |
| [16] |
Li H, Fan H, Li Y, et al. Construction and application of multi-host integrative vector system for xylose-fermenting yeast. Fems Yeast Res, 2017, 17(6), fox055: 1-10.
|
| [17] |
Sun HY, Zang XN, Zhang XC. Optimization of expression and safety evaluation of the multi-copy human/salmon chimeric calcitonin in Saccharomyces cerevisiae. J Wuhan Univ, 2014, 43(1): 37-44 (in Chinese). 孙恒一, 臧晓南, 张学成. 酿酒酵母rDNA的结构及其介导整合影响因素研究进展. 武汉大学学报, 2014, 43(1): 37-44. |
| [18] |
Lin XQ. iTRAQ-based quantitative proteomic analysis of high-level expression of xylanase in Pichia pastoris[D]. Guangzhou: South China University of Teachnology, 2013(in Chinese). 林小琼.基于iTRAQ技术的高效表达木聚糖酶重组毕赤酵母细胞的蛋白质组学研究[D].广州: 华南理工大学, 2013. http://cdmd.cnki.com.cn/Article/CDMD-10561-1014153353.htm |
| [19] |
Johnson MR, Wang K, Smith JB, et al. Quantitation of dihydropyrimidine dehydrogenase expression by real-time reverse transcription polymerase chain reaction. Anal Biochem, 2000, 278(2): 175-184. |
| [20] |
Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCt method. Methods, 2001, 25(4): 402-408. |
| [21] |
Xuan YJ, Zhou XS, Zhang YX. Determination of copy number of foreign gene in genome of Pichia pastoris by real-time fluorescent quantitative PCR. Chin J Biologicals, 2009, 22(12): 1236-1243 (in Chinese). 宣姚吉, 周祥山, 张元兴. 实时荧光定量PCR检测毕赤酵母基因组中外源基因拷贝数. 中国生物制品学杂志, 2009, 22(12): 1236-1243. |
| [22] |
Salim D, Bradford WD, Freeland A, et al. DNA replication stress restricts ribosomal DNA copy number. PLoS Genet, 2017, 13(9): e1007006. |
| [23] |
Juturu V, Wu JC. Heterologous protein expression in Pichia pastoris: latest research progress and applications. ChemBioChem, 2017, 19(1): 7-21. |
| [24] |
Li F, Yu XW, Sha C, et al. Impact of gene dosage and methanol concentration on Rhizopus chinensis recombinant lipase production in Pichia pastoris. Microb China, 2011, 38(3): 301-309 (in Chinese). 李飞, 喻晓蔚, 沙冲, 等. 基因拷贝数和甲醇浓度对重组毕赤酵母产华根霉脂肪酶的影响. 微生物学通报, 2011, 38(3): 301-309. |
| [25] |
Li AT, Ilie A, Sun ZT, et al. Whole-cell-catalyzed multiple regio- and stereoselective functionalizations in cascade reactions enabled by directed evolution. Angew Chem Int Ed Engl, 2016, 55(39): 12026-12029. |
| [26] |
Yu D, Wang JB, Reetz MT. Exploiting designed oxidase-peroxygenase mutual benefit system for asymmetric cascade reactions. J Am Chem Soc, 2019, 141(14): 5655-5658. |
| [27] |
Qu G, Zhu T, Jiang YY, et al. Protein engineering: from directed evolution to computational design. Chin J Biotech, 2019, 35(10): 1843-1856 (in Chinese). 曲戈, 朱彤, 蒋迎迎, 等. 蛋白质工程:从定向进化到计算设计. 生物工程学报, 2019, 35(10): 1843-1856. |
 2020, Vol. 36
2020, Vol. 36




