中国科学院微生物研究所、中国微生物学会主办
文章信息
- 贾云耀, 方芳
- Jia Yunyao, Fang Fang
- 定点突变提高解淀粉芽孢杆菌JP-21脲酶应用特性
- Improving applicability of urease from Bacillus amyloliquefaciens JP-21 by site-directed mutagenesis
- 生物工程学报, 2020, 36(8): 1640-1649
- Chinese Journal of Biotechnology, 2020, 36(8): 1640-1649
- 10.13345/j.cjb.190566
-
文章历史
- Received: December 18, 2019
- Accepted: March 5, 2020
2. 江南大学 食品科学与技术国家重点实验室,江苏 无锡 214122
2. State Key Laboratory of Food Science and Technology, Jiangnan University, Wuxi 214122, Jiangsu, China
氨基甲酸乙酯(Ethyl carbamate,EC)是一种在传统发酵食品中检测出的氨类危害物[1-3]。2007年EC被国际癌症研究机构(IARC)正式归类为2A类致癌物质,即对人类很可能有致癌性[4]。在酒精饮料中,EC主要由尿素和乙醇反应生成。因此消除酒精饮料中EC的思路分为降低EC前体物质尿素的含量和直接降低EC的含量[5]。脲酶(EC 3.5.1.5)具有尿素酶和氨基甲酸乙酯水解酶两种酶的活性,利用脲酶同时降解EC及其前体尿素被认为是最有希望彻底消除酒精饮料中EC的方法[5-7]。目前,来源于发酵乳杆菌Lactobacillus fermentum和运动节杆菌Arthrobacter mobilis的脲酶已应用于降解酒精饮料中的尿素。发酵乳杆菌L. fermentum的脲酶在15 ℃条件下反应2 d (或在30 ℃条件下反应15 h),可使清酒(pH 4.4,17%±1% (V/V)乙醇)中的尿素降至1 μg/L以下[8]。运动节杆菌A. mobilis的脲酶(90 U/L),在15 ℃条件下反应13 d可彻底消除日本清酒中的尿素[9]。罗伊氏乳杆菌Lacobacillus reuteri 100-23脲酶(500 U/L),可在20 ℃条件下反应2 h后消除黄酒模拟体系(pH 4.8,16% (V/V)乙醇,48 mg/L尿素,106 μg/L EC)中的尿素,但是对EC没有明显的降解效果。向黄酒中添加6 000 U/L的副地衣芽孢杆菌Bacillus paralicheniformis脲酶,可在37 ℃下、50 h内高效降解尿素,但是对EC并无明显降解效果。副地衣芽孢杆菌脲酶突变体(10 000 U/L)于20 ℃反应50 h可使黄酒中EC从500 μg/L降至320 μg/L[10-11]。
本研究以一株可降解白酒酒醅中尿素和EC的解淀粉芽孢杆菌B. amyloliquefaciens JP-21脲酶为研究对象,通过模拟脲酶与EC分子空间对接寻找与酶催化活性相关的突变位点,以期获得对EC催化能力提高的突变体,为促进酶法高效降解发酵食品饮料中的EC及其前体提供参考。
1 材料与方法 1.1 菌株与质粒本研究所用细菌菌株及质粒均为本实验室保藏(表 1)。
| Name | Description | Source |
| Plasmids | ||
| pRSF-Duet-1 | Expression vector, kanR, T7 promotor | Lab stock |
| pRSF-Duet-UreABC | pRSF-Duet-1 containing UreABC | This work |
| pRSF-Duet-UreCAB | pRSF-Duet-1 containing UreCAB | This work |
| Strains | ||
| E. coli BL21(DE3) | Expression host | Lab stock |
| Bacillus amyloliquefaciens JP-21 | Strain with capability of degrading urea and EC | [13] |
| E. coli BL21-p-UreCAB | E. coli harboring pRSF-Duet-UreCAB | This work |
试剂:D5-氨基甲酸乙酯、9-羟基占吨购于西格玛奥德里奇(上海)贸易有限公司;异丙基-β-D-硫代吡喃半乳糖苷(IPTG)、质粒提取试剂盒、购于生工生物工程(上海)股份有限公司;细菌基因组提取试剂盒购自天根生化(北京)有限公司;胶回收试剂盒购于美国Thermo Scientific公司;尿素购自上海麦克林生化科技有限公司;氨基甲酸乙酯购自上海阿拉丁生化科技股份有限公司;DNA聚合酶、无缝克隆试剂盒购自诺唯赞生物科技有限公司;其他药品均购于国药集团化学试剂有限公司。
仪器:氮吹仪、酶标仪购自美国Thermo Fisher公司;GC/MS-QP2010购自日本岛津公司;安捷伦1260高效液相色谱(HPLC)购自美国安捷伦公司。
1.3 脲酶的克隆表达与纯化 1.3.1 表达脲酶重组菌的构建本研究所用引物如表 2所示。以解淀粉芽孢杆菌JP-21的基因组为模板,用引物UreABC-F/ UreABC-R扩增脲酶基因。以质粒pRSF-Duet-1为模板,用引物P1-F/P1-R利用PCR进行质粒线性化,PCR产物经胶回收后用限制性核酸内切酶DpnⅠ消化模板。基因片段和线性化质粒经胶回收后利用无缝克隆试剂盒进行连接,并转化至E. coli JM109,获得质粒pRSF-Duet-UreABC。为强化催化亚基ureC的表达,将ureC置于ureA和ureB的上游进行表达[12]。具体操作为:以质粒pRSF-Duet-UreABC为模板,用引物UreC-F/ UreC-R、UreAB-F/UreAB-F分别进行扩增,胶回收后用无缝克隆试剂盒将这两个片段与线性化质粒pRSF-Duet-1进行连接,并转化至E. coli JM109。提取含有正确序列转化子的重组质粒,转化至E. coli BL21(DE3),构建的表达脲酶的重组菌命名为E. coli BL21-p-UreCAB。
| Primer name | Primer sequence (5′–3′) |
| P1-F | TTAACCTAGGCTGCTGCCAC |
| P1-R | GGTATATCTCCTTATTAAAGTTAAACAAAATTA |
| UreABC-F | CTTTAATAAGGAGATATACCATGAAACTGACACCGGTTGAACA |
| UreABC-R | GTGGCAGCAGCCTAGGTTAATTAAAATAAGAAATAACGCTGTCCTAGC |
| UreC-F | CTTTAATAAGGAGATATACCATGCACCACCACCACCACCACAAAATGTCGCGTGAGCAA |
| UreC-R | CAACCGGTGTCAGTTTCATTTAAAATAAGAAATAACGCTGTCCTAGC |
| UreAB-F | AATGAAACTGACACCGGTTGAACA |
| UreAB-F | GTGGCAGCAGCCTAGGTTAATCATCGGATCGCCTCCTCC |
| M326-F | TGATATGNNKATGGTCTGCCATC |
| M326-R | ACCATMNNCATATCAAGATGCTCG |
| M374-F | GCAGGCGNNKGGCAG |
| M374-R | CTGCCMNNCGCCTGC |
| Homologous sequences used for one-step seamless cloning were underlined. Codons used for mutation were indicated in bold. | |
用于表达脲酶的重组大肠杆菌在LB培养基(含50 mg/L卡那霉素),37 ℃、220 r/min条件下过夜培养,然后按1% (V/V)的接种量转接至TB培养基(含50 mg/L卡那霉素),37 ℃、220 r/min培养至OD600为0.6–0.8,加入6 mmol/L的Ni2SO4和0.1 mmol/L IPTG,在30 ℃、220 r/min的条件下继续培养15 h。所获菌液于4 ℃、8 000 r/min条件下离心5 min收集菌体,用20 mmol/L PBS缓冲液(pH 7.4)洗涤2次后,重悬于结合缓冲液(20 mmol/L pH 7.4 PBS缓冲液、0.5 mol/L NaCl),冰水浴超声破碎后于4 ℃、12 000 r/min离心20 min,收集上清液即为脲酶粗酶液。重组脲酶采用镍柱亲和层析进行纯化,用洗脱缓冲液(20 mmol/L pH 7.4 PBS、0.5 mol/L NaCl、0.5 mol/L咪唑)进行梯度洗脱,收集有活性的组分进行透析脱盐用于后续分析和研究。
1.4 脲酶活力测定采用Berthelot比色法测定脲酶活力[14]:脲酶可以降解尿素或EC生成氨,氨与苯酚-次氯酸钠反应形成靛酚蓝,通过检测该显色反应OD625的变化来表征酶活。
具体操作为:取经稀释的酶液200 μL,添加到200 μL含有30 g/L尿素或EC的50 mmol/L柠檬酸-柠檬酸钠缓冲液(pH 6.0)中,混匀后在37 ℃条件下反应15 min,立即加入200 μL终止剂(10%三氯乙酸),振荡混匀后,再依次加入200 μL显色剂Ⅰ和200 μL显色剂Ⅱ。再次混匀后,于37 ℃继续反应20 min。反应液经过稀释后,测定其OD625的变化,以灭活的酶液作为空白对照。利用0、0.1、0.2、0.3、0.4 mmol/L NH4Cl溶液作为铵离子标准曲线,根据氯化铵标准曲线即可计算反应体系生成铵的总量,以此来计算酶活。
酶活单位定义:在常压、37 ℃及pH 6.0的条件下,每分钟分解1 μmol EC或尿素所需要的酶量为一个酶活力单位。
1.5 同源建模及蛋白结构分析方法脲酶的UreC亚基为催化亚基,包含了脲酶的催化活性口袋及底物结合口袋[15-16],通过氨基酸序列比对,选择巴氏芽孢杆菌Bacillus pasteurii脲酶UreC的晶体结构(PDB ID:2UBPc)为模板,利用Swiss Model数据库对解淀粉芽孢杆菌B. amyloliquefaciens JP-21 UreC进行3D结构模拟[17]。利用Discovery Studio 2017 (Accelrys, San Diego,CA,USA)软件对UreC与底物EC进行分子对接,并用Pymol 2.1软件(https://pymol.org/2/)对蛋白质结构进行分析。
1.6 单点饱和突变库的构建及突变库筛选方法采用重叠延伸的方法构建单点饱和突变文库,在建库过程中,通过引入简并密码子NNK (N:A、T、G、C;K:G、C)的方式在突变位点引入随机突变。简并密码子NNK可以代表 32种密码子,编码20种氨基酸,因而可以保证构建突变体文库的覆盖性。以质粒pRSF-Duet-UreCAB为模板,用表 2中的引物M326-F/M326R、M374-F/M374-R分别进行PCR,获得环形的PCR产物,经限制性核酸内切酶DpnⅠ消化模板质粒及胶回收后,转化E. coli BL21(DE3)。
将含有突变体的转化子分别在96孔板和摇瓶中培养,通过比较EC酶活性进行初筛和复筛,挑选EC酶酶活高的突变体进行测序。
1.7 脲酶突变体酶学性质研究 1.7.1 脲酶温度稳定性测定将脲酶纯酶(1 000–1 200 U/L)置于不同温度下(30 ℃、40 ℃、50 ℃、60 ℃、70 ℃)保温30 min后测定酶活。以未经保温处理的酶活为100%,计算不同温度下的相对酶活。
1.7.2 脲酶最适pH及pH稳定性测定测定最适pH时,使用50 mmol/L、pH 3.0–7.0的柠檬酸-Na2HPO4缓冲液配制底物溶液,在不同pH条件下测定酶活,以测得的最高酶活为100%,分别计算其他pH条件下酶活的相对值。
测定pH稳定性时,将脲酶纯酶置于pH 3.0–7.0的缓冲液中,4 ℃放置6 h后测定其残余酶活力。以测得的最高酶活为100%,分别计算其他pH条件下酶活的相对值。
1.7.3 脲酶对乙醇的耐受性测定将脲酶与乙醇(终浓度为0–40% (V/V))混合,在37 ℃条件下放置2 h后测定酶活。以不含乙醇的酶液所测酶活力为100%,分别计算不同乙醇浓度下的相对酶活。
1.7.4 脲酶酶学动力学参数测定分别测定不同底物浓度下的催化反应速率,利用GraphPad Prism 7对结果进行非线性回归曲线拟合,以此计算Km和Vmax。以尿素为底物时的浓度范围为2–80 mmol/L,以EC为底物时的浓度范围为100–1 800 mmo/L。
1.7.5 脲酶半衰期测定脲酶半衰期的测定利用Arrhenius方程的原理进行测定和计算[18]。将稀释的脲酶纯酶在70 ℃条件下保温,每隔2 min取样测定其残余酶活力。取残余酶活力最高的为100%。记脲酶残余酶活力的百分比为Ar,通过绘制保温时间与lnAr的线性拟合,所得直线斜率的相反数为Kd,即热失活速率常数。脲酶半衰期(t1/2)的计算公式为t1/2=ln2/Kd。
1.7.6 脲酶降解黄酒中尿素及EC为考察解淀粉芽孢杆菌JP-21脲酶对黄酒中EC和尿素的降解并比较其他脲酶的降解效果[10],将市售黄酒中尿素和EC含量分别调整至51.90 mg/L和513.25 μg/L (经测定,所购市售黄酒含35.27 mg/L尿素,320.43 μg/L EC)。向此黄酒中添加终浓度为6 000 U/L的脲酶,37 ℃下反应50 h,取样测定样品中尿素及EC的含量。
尿素测定采用二乙酰单氧化反应法用高效液相色谱进行测定[19],EC的测定采用固相微萃取结合GC-MS的方法[20-22]。
尿素与乙醇可自发反应生成EC,因此黄酒存放一段时间后EC含量会逐渐增加。为考察脲酶对黄酒中EC的全面减控效果,采用加热黄酒的方法(90 ℃保温30 min)加速EC生成过程,并评估脲酶对EC的减控效果[23]。
1.7.7 统计分析方法运用SPSS 19.0软件对野生脲酶及脲酶突变体降解黄酒中EC含量的结果进行显著性差异分析。
2 结果与分析 2.1 酶催化中心氨基酸位点分析解淀粉芽孢杆菌JP-21脲酶为脱辅基脲酶,由ureA、ureB和ureC三个基因编码,具有尿素酶和EC酶两种活性。但是其对EC的亲和力较差,因此需要改造以提高它对EC的亲和力。据报道,位于活性位点周围的甲硫氨酸易被氧化,会使支链变大[24],从而影响酶活性位点空间,阻碍酶与底物的完美结合,使得酶活力降低甚至失活[25]。而脲酶催化亚基UreC活性中心的Ni2+作为路易斯酸,具有氧化剂的作用[26]。为减小甲硫氨酸被氧化引起的空间位阻效应,选择在脲酶活性中心周围寻找甲硫氨酸进行突变。脲酶催化亚基UreC模拟结构与底物EC对接的结果展示了其活性中心附近氨基酸残基的分布状况,距离EC分子2.541 Å的M326及距离EC分子3.776 Å的M374这两个甲硫氨酸是最靠近脲酶活性中心的,易受Ni2+氧化性的影响,于是选择这两个甲硫氨酸作为改造位点(图 1)。
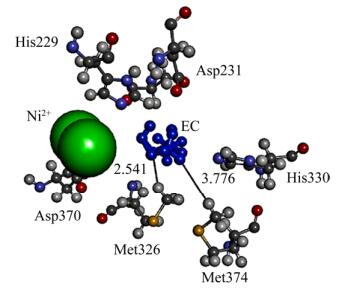
|
| 图 1 脲酶alpha亚基活性中心氨基酸分布示意图 Fig. 1 Schematic diagram of amino acid distribution of UreC active center. |
| |
为了提高解淀粉芽孢杆菌JP-21脲酶对EC的催化效率,根据酶催化中心氨基酸位点分析,选择了M326和M374两个位点进行点饱和突变。通过初筛和复筛,获得了EC酶活性提高的正向突变体M326V、M374A及M374T,EC酶活性分别为378.28 U/L (比酶活2.71 U/mg,蛋白量139.59 mg/L)、328.19 U/L (比酶活2.60 U/mg,蛋白量126.23 mg/L)、508.71 U/L (比酶活3.38 U/mg,蛋白量150.51 mg/L),分别是野生脲酶的1.67、1.14、2.23倍(图 2)。
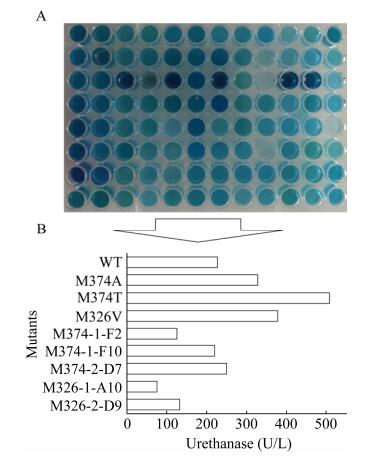
|
| 图 2 氨基甲酸乙酯水解酶活性提高的脲酶突变体筛选(A:以EC为底物进行96孔板筛选;B:脲酶突变体摇瓶发酵水平的EC酶活力,其中M374-1-F2至M326-2-D9为突变体编号) Fig. 2 Screening of urease mutants with increased urethanase activity. (A) 96-well plate screening with EC as substrate. (B) Urethanase activity of urease mutants in shake flask fermentation levels, where M374-1-F2 to M326-2-D9 represent urease mutants. EC: Docking with EC as substrate. |
| |
通过6×His标签介导的亲和层析,对WT和上述3个正向脲酶突变体M326V、M374A和M374T进行了纯化,获得了较纯的B. amyloliquefaciens JP-21脲酶纯酶,由UreA、UreB和UreC三个亚基组成(图 3)。
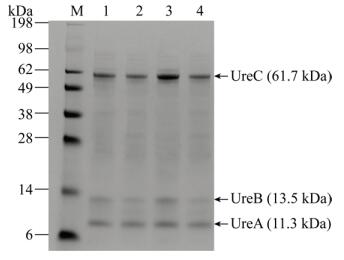
|
| 图 3 脲酶及其突变体的表达与纯化 Fig. 3 Expression and purification of urease and its mutants. M: standard protein marker; 1–4: purified wild type urease and its mutants M374A, M374T, M326V. |
| |
通过对脲酶热稳定性的研究,发现野生脲酶和脲酶突变体在低于50 ℃的条件下均保持了近100%的酶活。M326V在70 ℃条件下,尿素酶和EC酶热稳定性明显高于野生脲酶和其他两个突变体,比野生脲酶的相对酶活分别提高了9.34%和7.12% (图 4)。
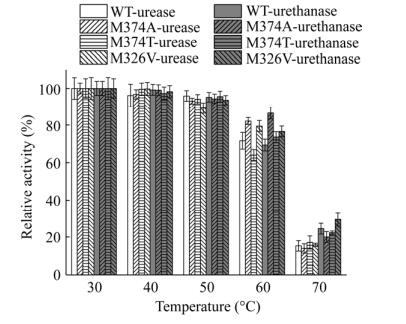
|
| 图 4 脲酶及其突变体的温度稳定性 Fig. 4 Thermostability of urease and its mutants. |
| |
发酵食品一般为酸性体系,如黄酒的pH为3.8–4.9[27]。研究脲酶最适pH及pH稳定性对于评价脲酶在黄酒中的应用至关重要。由图 5可知,野生脲酶和脲酶突变体的最适pH均为6.0。虽然最适反应pH没有改变,但M374A和野生脲酶及其他突变体相比,具有更宽的pH稳定性范围,尤其是在pH 3.0–5.5时,M374A的尿素酶和EC酶活性分别比野生脲酶高30%和20%。此外,野生脲酶在pH 6.0时稳定性最好,而突变体在pH 5.5时稳定性最佳,并且在pH 4.5时,M374A的脲酶相对酶活相比野生脲酶提高了39% (图 6)。

|
| 图 5 脲酶及其突变体的最适反应pH Fig. 5 Detection of the optimum reaction pH of urease and its mutants. |
| |

|
| 图 6 脲酶及其突变体的pH稳定性 Fig. 6 pH stability of urease and its mutants. |
| |
黄酒的酒精度为14%–20%[28],探究脲酶在不同酒精浓度下的耐受性可以更好地表征其实际应用性能。由图 7可以看出,来源于JP-21的脲酶及其突变体在20%乙醇条件下保存2 h,其尿素酶和EC酶活力仍有60%和50%以上。这说明,此酶对的乙醇耐受性较好,具有降解酒精饮料中EC及尿素的潜力。
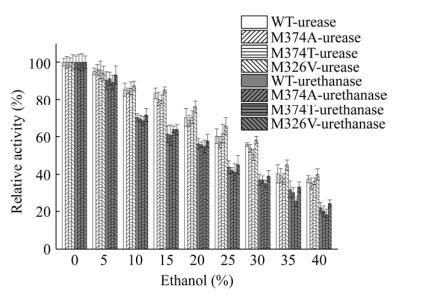
|
| 图 7 脲酶及其突变体对乙醇的耐受性 Fig. 7 Tolerance of urease and its mutants to ethanol. |
| |
为进一步比较脲酶及其突变体的稳定性,分析并比较了它们的半衰期。结果表明,野生脲酶和M374位点突变体的半衰期没有显著差异,而突变体M326V的尿素酶和EC酶半衰期与野生脲酶相比略有延长,分别是野生脲酶的1.10倍和1.15倍(表 3)。
| Name | Urease | Urethanase | ||||
| Kd | t1/2 (min) | Kd | t1/2 (min) | |||
| WT | 0.086 9 | 7.97 | 0.142 0 | 4.88 | ||
| M374A | 0.086 1 | 8.05 | 0.139 1 | 4.98 | ||
| M374T | 0.087 0 | 7.96 | 0.154 3 | 4.49 | ||
| M326V | 0.079 3 | 8.74 | 0.122 6 | 5.65 | ||
为比较脲酶及其突变体的催化特性,测定了它们的酶反应动力学参数。由表 4可以看出,突变体M374A、M374T和M326V以EC为底物的Km比野生脲酶分别降低了50.82%、37.47%和41.24%,酶的比活力分别是野生脲酶的1.03倍、1.09倍和1.05倍。这说明,通过改造由模拟结构确定的影响酶与底物结合的关键位点M374和M326,显著提高了解淀粉芽孢杆菌JP-21脲酶对EC的亲和力。M374A对尿素和EC的Km分别降低到3.05 mmol/L和101.84 mmol/L,显著低于来源于副地衣芽孢杆菌B. paralicheniformis的脲酶(尿素酶Km为23.7 mmol/L,EC酶Km为1 018 mmol/L)。突变体对尿素的Km基本没有发生变化,M374A、M374T和M326V三个突变体酶的比活力分别是野生型的1.15倍、1.29倍和1.19倍。本研究是以EC作为底物分子与脲酶催化亚基UreC进行分子对接来选择突变位点的。由于EC与尿素在结构和分子大小上存在差异,改造策略对EC Km的提高优于对尿素Km的提高。
| Name | Urease | Urethanase | |||||||
| Km (mmol/L) | Vmax (U/L) | Activity (U/mg) | Km (mmol/L) | Vmax (U/L) | Activity (U/mg) | ||||
| WT | 3.41±0.27 | 880.71±10.28 | 35.63±1.25 | 207.07±5.27 | 596.81±54.31 | 17.71±1.57 | |||
| M374A | 3.05±0.36 | 574.23±7.21 | 39.22±2.18 | 101.84±10.53 | 337.84±10.43 | 18.26±1.29 | |||
| M374T | 3.83±0.18 | 847.17±9.77 | 39.35±1.79 | 129.49±8.73 | 499.23±8.47 | 19.27±1.84 | |||
| M326V | 3.30±0.17 | 732.52±9.54 | 38.54±2.51 | 121.67±5.24 | 454.47±10.37 | 18.66±1.73 | |||
为了考察脲酶突变体的应用性能是否得到了提高,考察了脲酶对黄酒中尿素和EC的降解能力。结果显示,添加脲酶反应50 h后,黄酒中的尿素基本被完全降解,尿素含量为1.6–2.5 mg/L,去除率大于97% (图 8A)。在野生脲酶作用下,黄酒中的EC由513.90 μg/L降到了452.21 μg/L,降解率达12.00%。脲酶突变体M374A、M374T、M326V可将黄酒中的EC分别降至420.68 μg/L、393.57 μg/L、412.11 μg/L,降解率分别是野生脲酶的1.52倍、1.97倍和1.66倍。此外,经酶处理的酒加热后,对照中的EC由513.90 μg/L增至595.07 μg/L,而经脲酶及其突变体M374A、M374T、M326V处理过的黄酒中的EC分别为428.68 μg/L、397.77 μg/L、419.62 μg/L,含量基本没有增加(图 8B)。说明通过具有双酶活性(尿素酶和EC酶)的脲酶处理,基本消除了尿素并显著减少了EC。这对于减少黄酒的贮存过程中EC含量的增加具有重要意义。

|
| 图 8 脲酶降解黄酒中尿素和氨基甲酸乙酯 Fig. 8 Reduction of urea and EC in rice wine by urease. Urea+ and EC+: urea and EC detected in rice wine after heat treatment; * and ** represent significant differences (P < 0.05) and extremely significant (P < 0.01) differences. |
| |
利用酶法降解EC及其前体尿素被认为是最有希望彻底消除酒精饮料中EC的方法[5-7]。目前脲酶的工业化应用还存在EC酶活力差、对EC亲和力低等问题。本研究通过在线数据库查找和比对,模拟了解淀粉芽孢杆菌B. amyloliquefaciens JP-21催化亚基UreC的3D结构,通过模拟UreC与底物EC分子对接的结构,确定了影响酶与底物结合的两个关键位点M326和M374。通过对这两个位点进行突变,成功获得3株EC酶活性提高的突变体M374A、M374T和M326V。它们在pH稳定性、对EC的亲和力、对黄酒中的EC降解率等方面的特性均有不同程度提升。突变体M374A具有更宽的pH稳定性范围,在pH 3.0–5.5时,M374A的尿素酶和EC酶活性分别比野生脲酶高30%和20%,在pH 4.5时的尿素酶相对酶活比野生尿素酶提高了39%。以EC为底物时,脲酶突变体M374A、M374T和M326V的Km分别为101.84 mmol/L、129.49 mmol/L和121.67 mmol/L,比野生脲酶分别降低了50.82%、37.47%和41.24%。来源于解淀粉芽孢杆菌B. amyloliquefaciens JP-21的脲酶及其突变体可基本消除黄酒中的尿素。脲酶突变体M374A、M374T、M326V对黄酒中EC的降解率比野生型有显著提高,分别是野生型的1.52倍、1.97倍和1.66倍。降解效果最好的脲酶突变体M374T可以将黄酒中的EC从513.90 μg/L降至393.90 μg/L,并显著减少黄酒加热后EC的生成。本研究结果对今后改造脲酶催化特性和改善其应用特性具有重要意义,可为开发减控发酵食品中的微生物代谢氨(胺)类危害物策略提供参考。
| [1] |
Baan R, Straif K, Grosse Y, et al. Carcinogenicity of alcoholic beverages. Lancet Oncol, 2007, 8(4): 292-293. DOI:10.1016/S1470-2045(07)70099-2 |
| [2] |
Schlatter J, Lutz WK. The carcinogenic potential of ethyl carbamate (urethane): Risk assessment at human dietary exposure levels. Food Chem Toxicol, 1990, 28(3): 205-211. |
| [3] |
Weber JV, Sharypov VI. Ethyl carbamate in foods and beverages: a review. Environ Chem Lett, 2009, 7(3): 233-247. |
| [4] |
Lachenmeier DW. Consequences of IARC re-evaluation of alcoholic beverage consumption and ethyl carbamate on food control. Deutsche Lebensmittel-Rundschau: Zeitschrift für Lebensmittelkunde und Lebensmittelrecht, 2007, 103(7): 307-311. |
| [5] |
Zhao XR, Du GC, Zou HJ, et al. Progress in preventing the accumulation of ethyl carbamate in alcoholic beverages. Trends Food Sci Technol, 2013, 32(2): 97-107. |
| [6] |
Fidaleo M, Esti M, Moresi M. Assessment of urea degradation rate in model wine solutions by acid urease from Lactobacillus fermentum. J Agric Food Chem, 2006, 54(17): 6226-6235. |
| [7] |
Yang YQ, Kang Z, Zhou JL, et al. High-level expression and characterization of recombinant acid urease for enzymatic degradation of urea in rice wine. Appl Microbiol Biot, 2015, 99(1): 301-308. |
| [8] |
Kakimoto S, Sumino Y, Kawahara K, et al. Purification and characterization of acid urease from Lactobacillus fermentum. Appl Microbiol Biot, 1990, 32(5): 538-543. |
| [9] |
Miyagawa K, Sumida M, Nakao M, et al. Purification, characterization, and application of an acid urease from Arthrobacter mobilis. J Biotechnol, 1999, 68(2/3): 227-236. |
| [10] |
Liu QT, Chen YQ, Yuan ML, et al. A Bacillus paralicheniformis iron-containing urease reduces urea concentrations in rice wine. Appl Environ Microbiol, 2017, 83(17): 01258-17, 1-11. |
| [11] |
Liu QT, Yao XH, Liang QX, et al. Molecular engineering of Bacillus paralicheniformis acid urease to degrade urea and ethyl carbamate in model Chinese rice wine. J Agr Food Chem, 2018, 66(49): 13011-13019. |
| [12] |
Liu QT, Jin XR, Fang F, et al. Food-grade expression of an iron-containing acid urease in Bacillus subtilis. J Biotechnol, 2019, 293: 66-71. |
| [13] |
Ding X, Li QY, Liu F, et al. Isolation of microbial strains for degradation of ethyl carbamate in Luzhouflavour Baijiu and characterization of corresponding enzymes. Food Ferment Ind, 2018, 44(7): 29-36 (in Chinese). 丁霞, 李巧玉, 刘凡, 等. 降解浓香型白酒中氨基甲酸乙酯菌株的筛选及产酶特性研究. 食品与发酵工业, 2018, 44(7): 29-36. |
| [14] |
Weatherburn MW. Phenol-Hypochlorite reaction for determination of ammonia. Anal Chem, 1967, 39(8): 971-974. DOI:10.1021/ac60252a045 |
| [15] |
Carter EL, Flugga N, Boer JL, et al. Interplay of metal ions and urease. Metallomics, 2009, 1(3): 207-221. |
| [16] |
Kappaun Karine K, Piovesan Angela RP, Carlini Celia RC, et al. Ureases: Historical aspects, catalytic, and non-catalytic properties — A review. J Adv Res, 2018, 13: 3-17S1141461269. |
| [17] |
Waterhouse A, Bertoni M, Bienert S, et al. SWISS-MODEL: homology modelling of protein structures and complexes. Nucleic Acids Res, 2018, 46(W1eb Server issue): 296-303. |
| [18] |
Malikkides CO, Weiland RH. On the thermal denaturation of glucose oxidase. Biotechnol Bioeng, 2010, 24(8): 1911-1914. DOI:10.1002/bit.260240820 |
| [19] |
Clark Shona C, Francis PS, Conlan XA, et al. Determination of urea using high-performance liquid chromatography with fluorescence detection after automated derivatisation with xanthydrol. J Chromatogr A, 2007, 1161(1/2): 207-213. |
| [20] |
Xia Q, Yuan H, Wu C, et al. An improved and validated sample cleanup method for analysis of ethyl carbamate in chinese liquor. J Food Sci, 2014, 79(9): 1854-1860. |
| [21] |
Zhong QD, Yao L, Xiong ZH. Determination of EC content in rice wine by GC/MS and HPLC-FLD. Food Ferment Ind, 2007, 33(3): 115-119 (in Chinese). 钟其顶, 姚亮, 熊正河. 采用GC/MS和HPLC-FLD2种方法测定黄酒中的EC含量. 食品与发酵工业, 2007, 33(3): 115-119. |
| [22] |
Battaglia R, Conacher HBS, Page BD. Ethyl carbamate (urethane) in alcoholic beverages and foods: A review. Food Additives & Contaminants, 1990, 7(4): 477-496. |
| [23] |
Gu GX. Brewing Technology. 2nd ed. Beijing: China Light Industry Press, 1996: 506-508(in Chinese). 顾国贤.酿造酒工艺学. 2版.北京: 中国轻工业出版社, 1996: 506-508. |
| [24] |
Vogt W. Oxidation of methionyl residues in proteins: tools, targets, and reversal. Free Radical Biology & Medicine, 1995, 18(1): 93-105. |
| [25] |
Khemakhem B, Ali MB, Aghajari N, et al. Engineering of the α-amylase from Geobacillus stearothermophilus US100 for detergent incorporation. Biochemical & Biophysical Research Communications, 2009, 102(2): 380-389. |
| [26] |
Maroney MJ, Ciurli S. Nonredox nickel enzymes. Chem Rev, 2014, 114(8): 4206-4228. |
| [27] |
Yu GS. Probe into pH range of rice wine and prevention and control of exceeding standards. Food Ferment Ind, 2002, 28(1): 76-78 (in Chinese). 俞关松. 黄酒pH值指标范围的探讨及超标的预防控制. 食品与发酵工业, 2002, 28(1): 76-78. |
| [28] |
Li Y. Discussion on rice wine acidity and its related indexes. China Brew, 2009, 28(6): 179-181 (in Chinese). 李颖. 黄酒酸度与其他指标的相关性探讨. 中国酿造, 2009, 28(6): 179-181. |
 2020, Vol. 36
2020, Vol. 36




