中国科学院微生物研究所、中国微生物学会主办
文章信息
- 孙伟康, 张娟, 堵国成
- Sun Weikang, Zhang Juan, Du Guocheng
- 乳酸乳球菌NZ9000基因组规模代谢网络模型的构建与验证
- Construction and verification of Lactococcus lactis NZ9000 genome-scale metabolic model
- 生物工程学报, 2020, 36(8): 1629-1639
- Chinese Journal of Biotechnology, 2020, 36(8): 1629-1639
- 10.13345/j.cjb.190552
-
文章历史
- Received: December 11, 2019
- Accepted: February 14, 2020
- Published: March 26, 2020
2. 江南大学 生物工程学院,江苏 无锡 214122;
3. 江南大学协同创新中心,江苏 无锡 214122
2. School of Biotechnology, Jiangnan University, Wuxi 214122, Jiangsu, China;
3. Synergetic Innovation Center, Jiangnan University, Wuxi 214122, Jiangsu, China
乳酸菌发酵可以产生有机酸、抑菌素、挥发性风味物质,具有重要的益生功能,普遍应用于食品、医药、饲料等多种领域[1]。在食品领域中乳酸菌应用最为常见,主要作为直投式发酵剂制备酸奶、奶酪等发酵制品,可以改善产品的风味和口感。同时发酵产生的抑菌素会抑制病原菌的繁殖,可作为生物防腐剂来保证食品的安全与品质[2-3];在医药领域,乳酸菌联合给药有助于疾病的预防、治疗和术后修复[4];饲料领域中添加乳酸菌可以有效增强动物胃肠道对营养成分的消化和吸收、提高动物机体的免疫力、减少疾病的发生,并减弱粪便的恶臭味[2, 5-6]。作为乳酸菌属的重要分支,乳酸乳球菌在工业发酵中占比超过20%,是乳品发酵的重要菌株之一,在乳制品、肉制品、果蔬制品以及豆制品中应用广泛,是公认的食品安全级工业微生物[7],而L. lactis NZ90000作为乳酸菌属的模式菌株,由于其生长迅速、遗传背景清晰,且几乎不分泌蛋白质,是基因工程改造以及外源蛋白表达的理想菌株[8]。
随着基因组测序变得越来越经济和快速,系统生物学领域对于工业微生物细胞的研究也越来越深入。基因组规模代谢网络模型(GSMM)作为系统生物学的重要组成部分,其本质是将一个生物体所有与代谢相关的基因、酶、生物反应及代谢数据有效地组装到一起[9],并通过不同的算法计算和预测生物体的各种代谢特性,系统地表征基因型和表型之间的关系[10]。GSMM被广泛应用于生长表型预测[11]、代谢工程指导[12]、种间进化差异分析[13]以及多组学数据整合[14]等4个方面。而工业微生物由于其胞内代谢活动的复杂性,难以通过单一的研究手段对其内部调控机制进行系统的分析,且无法高效获得所需表型[15]。近年来,越来越多的研究团队通过构建GSMM来全局分析工业微生物的代谢网络,特别在筛选代谢工程改造靶点、指导代谢工程改造方面取得了一定的进展。例如,Lee等利用线性规划分析方法筛选出丙酮酸合成酶基因(ptsG、pykF、pykA)是大肠杆菌过量生产琥珀酸的基因敲除靶点,实验证实ptsG、pykF和pykA的敲除使琥珀酸的产量提高了7倍[16-17]。Brochad等在GSMM的指导下,通过过量表达酿酒酵母中的hsOMT基因使其香草醛产量提高了5倍[18]。然而,对于乳酸菌这类与人类健康和生活紧密相连的微生物其关注度却很少,现在为数不多已建立GSMM的乳酸菌有乳酸乳球菌IL1403 (Lactococcus lactis IL1403)[19]、乳酸乳球菌MG1363 (Lactococcus lactis MG1363)[20]、植物乳杆菌WCFS1 (Lactobacillus plantarum WCFS1)[21]、干酪乳杆菌LC2W (Lactobacillus casei LC2W)[22]。虽同为乳酸菌属,但菌种之间个体差异较大,菌种胞内代谢调控复杂,无法直接套用。遗憾的是,L. lactis NZ9000作为乳酸菌的模式菌株,现在仍没有GSMM供其对胞内代谢调控进行全局分析。
为系统地理解乳酸乳球菌的胞内代谢调控机制,本研究以L. lactis NZ9000作为研究对象,基于基因组功能注释和比较基因组学构建了L. lactis NZ9000的第一个基因组规模代谢网络模型iWK557。并进一步在定性和定量两个层次验证了iWK557的准确性,以期为全局理解L. lactis NZ9000的胞内代谢机制和理性指导代谢工程改造提供新工具。
1 材料与方法 1.1 菌株、培养基与培养条件目标菌株为Lactococcus lactis NZ9000 (保藏编号:LLN,CP002094;荷兰“NIZO Food Research”奶制品研究所)[23]。L. lactis NZ9000于GM17 (M17培养基中添加5 g/L的D-葡萄糖)、Chemically defined medium (CDM)[24]以及Minimal medium (MS15)[25]中培养,培养温度为30 ℃。
1.2 实验方法 1.2.1 定性实验将保藏在甘油管中的菌株于GM17固体培养基中划线活化,将单菌落接种至GM17液体培养基中,于30 ℃静置培养12 h得到种子液,随后将种子液以1% (V/V)的接种量转接到MS15培养基中培养8 h,取样测定吸光度(Optical density,OD) OD600。更换18种底物作为唯一碳源,底物为:葡萄糖、淀粉、纤维二糖、果糖、半乳糖、乳糖、麦芽糖、甘露糖、蔗糖、海藻糖、半乳糖醇、肌醇、甘露醇、山梨糖醇、丙酮酸、木糖、核糖、半乳糖,每种底物进行6个梯度实验(底物浓度分别为:1、5、10、15、20、30 g/L),每个梯度3个平行样品。
1.2.2 定量实验按1.2.1所述获得的种子液以1% (V/V)的接种量转接到CDM培养基中,于30 ℃静置培养24 h,每2 h取样。测量其生物量、细胞干重以及底物消耗。
1.3 主要仪器与分析平台实验仪器:恒温培养箱,购于上海医疗器械研究所;M-100生物传感器分析仪,购于深圳市西尔曼科技有限公司;Agilent 1260高效液相色谱仪,购于安捷伦科技(中国)有限公司;多功能酶标仪,购于Thermo Scientific公司;紫外可见分光光度计(UV2450),购于日本Hitachi公司。
软件:MATLAB R2018a,COBRA Toolbox[26],Origin 2018,Gurobi 8.0.1 optimizer。
1.4 草图模型的构建在本研究中,使用半自动方法构建草图模型。从NCBI或文献中下载L. lactis NZ9000的基因组序列并以FASTA格式保存到本地。通过同源比对法和手动构建法相结合的方式来进行草图模型的构建[27-28]。(1)同源比对法:使用本地BLASTp,基于L. lactis NZ9000的系统发育树,下载亲缘相近菌株的基因组序列于本地建库比对。序列比对的筛选条件为:e-value < 1×10–6、amino acid sequence identity > 40%、sequence matching length > 70%[29]。依据比对结果于亲缘相近菌株模型中进行代谢信息抓取,得到草图模型一。(2)手动构建法:将L. lactis NZ9000的蛋白序列上传到KEGG Automatic Annotation Server (KAAS)[30]上进行基因组功能注释,并结合KEGG PATHWAY Database进行代谢信息填充,得到草图模型二。
将两种方法得到的草图模型进行整合:基于EC号对草图模型间的基因进行比对,若两者之间有相同的基因,则看其功能注释是否一致,若一致则保留草图模型一的结果,若不一致则需通过文献和相关数据库进一步查证,若无法查证,则以草图模型一为准(同源比对法较KAAS注释更准确[31]);若存在差异基因,则将草图模型二中的基因-蛋白-反应(Gene-protein-reaction,GPR)补充进草图模型一中。通过此方法循环往复最终得到内容完整的草图模型。
1.5 模型精细化草图模型构建完成后,需结合各大数据库并依据L. lactis NZ9000的实际情况分别在基因水平、代谢物水平以及反应水平对其代谢信息进行填充与查证。模型精细化具体内容如下。
1.5.1 在代谢物水平进行模型精细化代谢物格式统一:将草图模型中的代谢物缩写形式进行统一,格式参照BIGG[32]及文献中多数模型采用的小写缩写的形式。
质量电荷平衡:使用COBRA Toolbox中的CheckMassChargeBalance算法计算GSMM中反应的质量电荷是否平衡,并根据输出结果,对代谢物的化学式和电荷进行修正,对不平衡的反应进行配平。
1.5.2 在基因水平进行模型精细化亚细胞定位:对基因编码蛋白质的作用区间进行亚细胞定位,将L. lactis NZ9000的基因组序列上传到CELLO[33],依据预测结果和实际代谢情况进行确认。
多基因连接性区分:对于多个基因编码一个蛋白的情况进行区分,分为多酶复合体以及同工酶,并使用布尔逻辑关系中的“and”和“or”进行区分[34]。
1.5.3 在反应水平进行模型精细化生物量方程:依据构成L. lactis NZ9000所有前体物质及其所占的比例构建生物量方程,并依据算法‘minimum inconsistency under parsimony’(MIP)[35],基于元素及质量守恒原则并结合L. lactis NZ9000的实际生理情况,对其生物量方程的化学式、电荷、分子量进行计算并对其分子量进行标准化。
反应可逆性:通过反应对应的EC号在MetaCyc[36]和MetaNetX[37]进行手动搜索确定反应方向性;对于没有EC号的反应,通过反应名称结合BIGG或结合乳酸菌相应文献进行确认,对于无法查证的反应一律设为可逆反应[31]。
转运反应和交换反应的添加:在Transporter Classification Database (TCDB)[38]下载所有的转运蛋白序列与L. lactis NZ9000的蛋白序列进行序列比对,依据序列比对结果并结合基因组功能注释和文献进行转运反应的添加;结合添加的转运反应以及“湿实验”中L. lactis NZ9000所需营养条件进行交换反应的添加。
代谢漏洞的填补:使用gapFind以及gapAnalysis算法进行代谢漏洞查找,结合基因组功能注释结果进行代谢漏洞填补。
1.6 转化为数学模型上述1.4得到的草图模型以及1.5的模型精细化都是在EXCEL中进行,在进行模型的计算模拟之前需要将其转化为计算机可读的SBML格式。将EXCEL格式的模型导入MATLAB后使用xls2model算法将其转化为数学模型,随后使用writeCbModel算法即可将其导出为SBML格式。
2 结果与分析 2.1 基因组功能注释与草图模型的获得在本研究中,基于基因组功能注释,使用半自动构建法进行草图模型的构建。
同源比对法:基于比较基因组学进行基因组功能注释,通过L. lactis NZ9000的系统发育树,选择了已有GSMM且亲缘相近的菌株L. lactis MG1363、L. plantarum WCFS1、L. casei LC2W作为参照菌株,进行序列比对。依据1.4所述的筛选条件进行双向比对,L. lactis NZ9000与参照菌株之间的基因匹配数分别是2 275个、1 294个和1 153个。而L. lactis MG1363、L. plantarum WCFS1、L. casei LC2W的序列基因数分别是2 563个、3 009个和3 003个,由此可以发现基因匹配数的多少与基因组大小不呈正相关,与其系统发育树的亲缘远近相关。将筛选得到的结果使用自行编写的程序整合入已有GSMM的菌株模型中,将以上得到的代谢信息进行整合,得到整合模型,包含548个基因、1 290个反应,随后通过筛选,删除重复的反应和功能注释相同的基因,保留不重复的部分,从而获得草图模型一,包含455个基因、739个代谢物、874个反应。
手动构建法:将目标菌株的蛋白序列上传到KAAS后,依据返回的结果,2 314个基因中826个有具体的功能注释,其中仅有69个有明确的EC号,对应475个基因。依据EC号结合KAAS返回的注释链接在KEGG中查找对应的反应,得到草图模型二,包含475个基因、69个反应。
依据1.4所述的方法将以上得到的草图模型进行整合得到最初完整的草图模型,含有516个基因、739个代谢物、876个反应。
2.2 模型精细化分别在代谢物水平、基因水平以及反应水平对1.4得到的草图模型进行了模型代谢信息填充与修正。
(1) 在代谢物水平进行模型精细化
采用不同基因组功能注释方法得到了两种不同形式的草图模型,需要对其所有的代谢物格式进行统一。不同方法中代谢物的表现形式有所差异,以6-磷酸葡萄糖为例,在模型L. casei LC2W中代谢物格式为“g6p”,而在模型L. plantarum WCFS1中采用的则是大写缩写的格式,为“G6P[c]”。本研究基于代谢物全称对代谢物格式进行统一,将其统一为小写缩写的形式,如“g6p[c]”,同时对于不能匹配的代谢物,按照其原有形式保留在模型中。
(2) 在基因水平进行模型精细化
对基因编码蛋白的作用区间进行亚细胞定位,依据CELLO返回的结果并结合L. lactis NZ9000为原核细菌的实际情况,将蛋白作用区间分为胞内(Cytoplasmic)与胞外(Extracellular)。其中胞内2 126个、胞外188个,总共2 314个基因。随后对基因关联性进行分类,分为多酶复合体与同工酶体系,并使用布尔逻辑关系中的”and”和”or”来进行区分,其中多酶复合体催化了21个反应,同工酶催化151个反应。
(3) 在反应水平进行模型精细化
生物量方程:是人为添加用于使用模型计算细胞生长情况的虚拟反应。在拟稳态的假设下,以生物量方程作为目标函数进行优化问题求解时,模型能够预测细胞的最大比生长速率。其表现形式如下:

|
SiR是生物量前体代谢物Ri的化学计量系数。SjB是副产物Bj的化学计量系数。由于所有生物量前体代谢物系数均基于1 g DCW,基于质量守恒原则,生物量方程的分子量计算如下:

|

在本研究中,生物量方程参考框架模型iNF518[20],生长相关维持能(GAM)和非生长相关维持能(NGAM)分别确立为39.4 mmol ATP/ (g DCW·h)和0.92 mmol ATP/(g DCW·h),依据算法‘minimum inconsistency under parsimony’ (MIP),基于质量守恒原则并结合目标菌株的实际生理情况,对其生物量方程的分子量、化学式以及各前体的化学计量系数进行计算和标准化,生物量方程的分子量由原本的825.05 g/mmol标准化为1 000.00 g/mmol,化学式由C33.697509H53.495976N8.358924O13.75044P0.68944S0.248079标准化为C40.6465338215H64.0995743231N9.9076885333O16.9635199515 P0.8322218004S0.350164004。
反应可逆性:依据1.5.3所述方法对反应的方向性进行核实区分,标准化后GSMM中含有349个可逆反应、491个不可逆反应。
转运和交换反应:依据1.5.3所述方法,依据序列比对结果和文献挖掘进行转运反应添加,共添加了119个转运反应。交换反应依据L. lactis NZ9000常用培养基中的营养物质,并保证基础营养元素C、H、O、N、P、S的吸收,共添加了112个交换反应。
代谢漏洞填补:依据1.5.3所述方法进行漏洞查找和填补,共填补了105个gaps。
2.3 数学模型的转换将模型精炼得到的EXCEL格式的模型导入MATLAB依据1.6的方法转换为S(m×n)矩阵,矩阵中m行代表代谢物的数量,n列代表反应的数量,矩阵由“1”、“–1”和“0”组成,其中“1”代表该代谢物在此反应中作为底物,“–1”代表该代谢物作为产物,“0”代表该代谢物不参与该反应。这样S(m×n)矩阵就将代谢物与反应巧妙地联系起来变为数学矩阵。S(668×840)表示该GSMM含有668个代谢物和840个反应。模型中有557个基因,依据命名规则将构建完成的GSMM命名为iWK557。
2.4 模型验证iWK557构建完成后,在应用之前为进一步确认其计算预测的精准性,本研究从定性和定量两个层次对其进行验证。
2.4.1 定性验证L. lactis NZ9000能够代谢利用多种糖类和其他碳源以获得能量、氧化还原力和合成生物大分子的前体物质[39]。参考文献[19, 22, 40]报道,乳酸菌属发酵可利用的主要碳源有葡萄糖、纤维二糖、果糖、半乳糖、乳糖、麦芽糖、甘露糖、蔗糖、海藻糖、甘露醇、山梨醇。此外为了进一步考察L. lactis NZ9000的底物利用谱,参照了大肠杆菌、酵母、谷氨酸棒状杆菌及放线菌等实验室常见菌株的可利用碳源进行了碳源类型的补充,从而确定18种底物作为唯一碳源进行定性实验[19, 40]。
使用iWK557利用流平衡(Flux balance analysis, FBA)算法对18种唯一碳源的利用能力及主要产物乳酸的生成进行预测,并依据1.2.1的实验方法进行定性实验,通过模型的预测和“湿实验”的验证,进一步对模型的准确性进行评估(表 1)。其中,木糖在原文献中可被大肠杆菌利用,作为本研究的备选碳源,经实验证实不能被L. lactis NZ9000作为碳源利用。比对结果显示模型预测结果与实验结果一致,表明模型iWK557能够准确预测菌体利用不同碳源进行代谢生长和产物生成。
| Carbon source | Experimental results | Simulation results | |||
| Cell growth | Lactate production | Cell growth | Lactate production | ||
| Glucose | + | + | + | + | |
| Starch | + | + | + | + | |
| Cellose | + | + | + | + | |
| Fructose | + | + | + | + | |
| Galactose | + | + | + | + | |
| Lactose | + | + | + | + | |
| Maltcose | + | + | + | + | |
| Mannose | + | + | + | + | |
| Sucrose | + | + | + | + | |
| Trehalose | + | + | + | + | |
| Galactitol | + | + | + | + | |
| Inositol | + | + | + | + | |
| Mannitol | + | + | + | + | |
| Sorbitol | + | + | + | + | |
| Pyruvate | + | + | + | + | |
| Xylose | – | – | – | – | |
| Ribose | + | + | + | + | |
| Melibiose | + | + | + | + | |
为进一步验证iWK557预测的生长值的准确性。在本研究中使用分批发酵的实验数据进行验证,随后通过恒化实验数据进一步验证模型的预测准确性。
分批发酵验证:使用CDM培养基进行分批培养,将实验数据导入Origin 2018以获取L. lactis NZ9000的细胞生长、葡萄糖消耗以及乳酸生成曲线(图 1A),随后通过Origin 2018“Analysis”工具栏中的“Nonelinear Curve fit”进行非线性拟合获得比生长速率、葡萄糖比消耗速率以及乳酸比生成速率曲线(图 1B)。L. lactis NZ9000在分批发酵中的最大比生长速率为0.017 004 h–1,葡萄糖比消耗速率为0.55 mmol/(g DCW·h),乳酸比合成速率为0.066 mmol/(g DCW·h)。在MATLAB中使用FBA进行模拟,约束葡萄糖的吸收速率,计算得到的最大比生长速率为0.017 1 h–1,乳酸比合成速率为0.064 3 mmol/(g DCW·h)与实验结果的误差率分别为0.5%和2.57%,表明该模型可以准确预测L. lactis NZ9000的比生长速率和主要代谢产物的比合成速率。
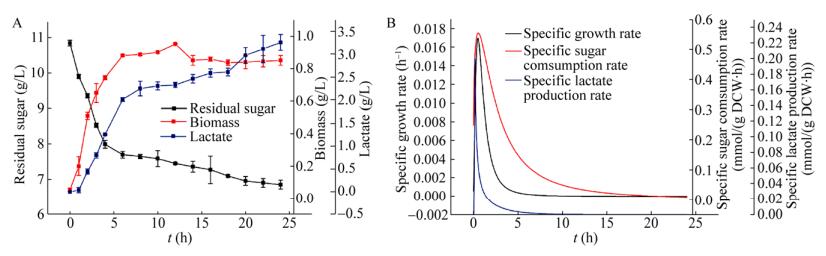
|
| 图 1 定性湿实验图(A:L. lactis NZ9000的生物量、葡萄糖消耗和乳酸生成曲线;B:L. lactis NZ9000的比生长速率、葡萄糖比消耗速率和乳酸比合成速率曲线) Fig. 1 Qualitative experiment chart. (A) Cell growth, glucose consumption and lactic acid production curve of L. lactis NZ9000. (B) Specific growth rate, glucose specific consumption rate and lactate specific production rate curve of L. lactis NZ9000. |
| |
恒化实验验证:在恒定稀释率的条件下,细胞的生长状态保持恒定,此时,测量发酵液中各种营养物的形成和消耗,可以量化特定稀释率下菌株的代谢状态。将获得的恒化实验数据进行单位转换,然后导入到模型中进行约束,从而验证模型预测的准确性。本研究使用Nicolas等[20]和Petri-Jaan等[41]基于CDM培养基的恒化实验数据进行验证,通过比较4种稀释率(0.05 h–1、0.25 h–1、0.4 h–1、0.45 h–1)条件下细胞的比生长速率及主要产物乳酸和乙酸的比合成速率和模型表型预测结果之间的差异来进行定量验证。在4种稀释率条件下,碳源输入速率分别约束为1.20 mmol/(g DCW·h)、6.20 mmol/(g DCW·h)、11.53 mmol/(g DCW·h)、16.36 mmol/(g DCW·h);氮源输入速率分别约束为0.13 mmol/(g DCW·h)、0.55 mmol/(g DCW·h)、1.26 mmol/(g DCW·h)、1.44 mmol/(g DCW·h)。如表 2所示,比生长速率(Specific growth rate,μ)、乳酸合成速率(Lactate production rate,LPR)以及乙酸合成速率(Acetate production rate,APR)的平均误差率分别为4.08%、4.21%、5.47%,最大偏差不超过9%,普遍低于10%[42],说明iWK557不仅能够准确反映细胞的生长状态,也能准确预测细胞主要产物的生成,真实反映细胞的生理代谢情况。
| Project | 0.05 (h–1) | 0.25 (h–1) | 0.40 (h–1) | 0.45 (h–1) | ||||||||||
| In silico | D****(%) | In silico | D(%) | In silico | D(%) | In silico | D(%) | |||||||
| μ* | 0.050 4 | 0.80 | 0.23 | 6.16 | 0.36 | 7.70 | 0.457 6 | 1.68 | ||||||
| LPR** | 0.920 0 | 4.16 | 4.20 | 6.87 | 18.32 | 0.43 | 27.600 0 | 5.39 | ||||||
| APR*** | 1.430 0 | 4.24 | 4.32 | 7.51 | 2.48 | 8.87 | 1.390 0 | 1.27 | ||||||
| *: μ represents specific growth rate; **: LPR represents lactate production rate; ***: APR represents acetate production rate;****: D represents deviation. | ||||||||||||||
模型iWK557包含557个基因、840个反应(609个细胞质反应、119个转运反应和112个交换反应),基因覆盖率达到23.89%。有76%的反应有明确的基因关联。参考KEGG和RAST server,将iWK557的反应分为10个代谢亚系统,各代谢亚系统的反应数目如图 2所示。在代谢亚系统中,氨基酸代谢(19.523%)所占比例最高,其次是核苷酸代谢(14.40%),最后是转运(14.16%)和交换反应(13.33%),这4个代谢亚系统所占比例超过60%。
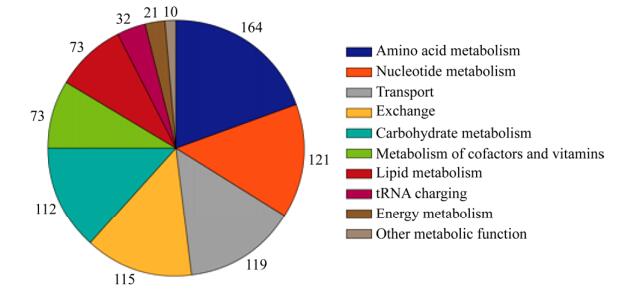
|
| 图 2 iWK557代谢亚系统分布 Fig. 2 Metabolic subsystems distribution of iWK557. |
| |
为进一步了解iWK557的详细特征,将模型iWK557与L. lactis MG1363、L. lactis Il1403的GSMM进行比较(表 3),比较结果如下所示:1) 3个模型的基因覆盖率分别为23.89%、22.23%、15.72%,3个菌株都属于乳酸乳球菌,细胞分区都分为胞内和胞外两个区间。2) 3个模型的共有反应数为319个,其中氨基酸代谢(29.15%)、核苷酸代谢(22.88%)、碳水化合物代谢(17.24%)以及维生素与辅因子代谢(10.65%)所占比例为79.93%,表明3个模型在以上4种代谢亚系统中有较高的相似性。iWK557特有207个代谢反应,主要集中于氨基酸代谢、转运反应、碳水化合物代谢以及核苷酸代谢,特有反应的代谢亚系统分布如图 3所示。3) 3个模型中,L. lactis Il1403的代谢物是另外两个菌株模型的子集,其中,L. lactis MG1363含有特有代谢物67个,iWK557含有特有代谢物86个。
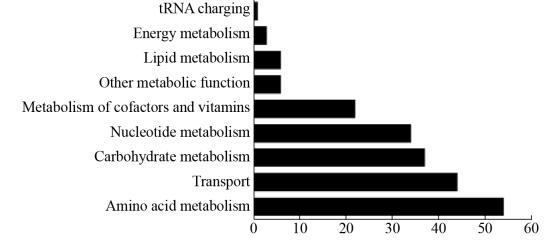
|
| 图 3 iWK557特有反应代谢亚系统分布 Fig. 3 Metabolic subsystems distribution for specific reactions in iWK557. |
| |
| Parameter | L. lactis NZ9000 | L. lactis MG1363 | L. lactis Il1403 |
| Genomic information | |||
| Total length (Mb) | 2.53 | 2.52 | 2.36 |
| Protein count | 2 314.00 | 2 563.00 | 2 310.00 |
| GC (%) | 35.70 | 35.70 | 35.30 |
| Model contents | |||
| Total reactions | 840.00 | 754.00 | 621.00 |
| Internal reactions | 601.00 | 530.00 | 492.00 |
| Transport reactions | 116.00 | 119.00 | 129.00 |
| Metabolites | 668.00 | 650.00 | 422.00 |
| Gene | 557.00 | 518.00 | 358.00 |
| Gene coverage (%) | 23.89 | 22.23 | 15.72 |
| Compartment | (c, e) | (c, e) | (c, e) |
在本研究中,基于基因组功能注释和比较基因组学构建了L. lactis NZ9000的首个全基因组规模代谢网络模型,将其命名为iWK557。通过定性和定量分析对iWK557进行了验证。定性验证结果显示,模型iWK557对不同碳源的模拟预测结果与实验结果一致,说明iWK557能够准确表征菌体生长和产物合成这两个最为重要的细胞特征;随后在分批发酵和恒化实验中对iWK557对菌体生长和产物合成预测的准确性进行定量验证,总平均偏差不超过5%,偏差率普遍低于10%,说明iWK557能够准确预测L. lactis NZ9000的生长和产物生成,反映细胞的真实生理状态。
在后续的研究中,将利用iWK557对L. lactis NZ9000的内部代谢调控机制进行全局分析,并以此为基础,通过整合多组学数据,构建条件特异性模型进行代谢流量分析和代谢靶点预测用以理性指导代谢工程改造。
| [1] |
Zhu ZM, Ji XM, Wu ZM, et al. Improved acid-stress tolerance of Lactococcus lactis NZ9000 and Escherichia coli BL21 by overexpression of the anti-acid component recT. J Ind Microbiol Biotechnol, 2018, 45(12): 1091-1101. |
| [2] |
Zhang YW. Regulation of aspartate metabolic pathways for improving acid stress resistance in Lactococcus lactis NZ9000[D]. Wuxi: Jiangnan University, 2018(in Chinese). 张彦位.调控天冬氨酸代谢途径提高Lactococcus lactis NZ9000的酸胁迫抗性[D].无锡: 江南大学, 2018. http://cdmd.cnki.com.cn/Article/CDMD-10295-1018252347.htm |
| [3] |
Zanirati DF, Abatemarco M Jr, De Cicco Sandes SH, et al. Selection of lactic acid bacteria from Brazilian kefir grains for potential use as starter or probiotic cultures. Anaerobe, 2015, 32: 70-76. |
| [4] |
Aragón F, Carino S, Perdigón G, et al. The administration of milk fermented by the probiotic Lactobacillus casei CRL 431 exerts an immunomodulatory effect against a breast tumour in a mouse model. Immunobiology, 2014, 219(6): 457-464. |
| [5] |
Matusiak K, Oleksy M, Borowski S, et al. The use of Yucca schidigera and microbial preparation for poultry manure deodorization and hygienization. J Environ Manag, 2016, 170: 50-59. |
| [6] |
Kim JD, Park KM. Effectiveness of Lactobacillus plantarum strain KJ-10311 to remove characteristic malodorous gases in piggery slurry. Asian-Australasian J Anim Sci, 2006, 19(1): 144-152. |
| [7] |
Cavanagh D, Fitzgerald GF, McAuliffe O. From field to fermentation: The origins of Lactococcus lactis and its domestication to the dairy environment. Food Microbiol, 2015, 47: 45-61. |
| [8] |
Wu CD, Huang J, Zhou RQ. Genomics of lactic acid bacteria: current status and potential applications. Crit Rev Microbiol, 2017, 43(4): 393-404. |
| [9] |
Dong FQ. Reconstruction and verification of the genome-scale metabolic models of Bacillus Coagulans 36D1[D]. Shanghai: East China University of Science and Technology, 2017(in Chinese). 董风晴.凝结芽孢杆菌36D1全基因组代谢网络模型的构建和验证[D].上海: 华东理工大学, 2017. http://cdmd.cnki.com.cn/Article/CDMD-10251-1017151448.htm |
| [10] |
Feist AM, Palsson BØ. The growing scope of applications of genome-scale metabolic reconstructions using Escherichia coli. Nat Biotechnol, 2008, 26(6): 659-667. |
| [11] |
Motamedian E, Saeidi M, Shojaosadati SA. Reconstruction of a charge balanced genome-scale metabolic model to study the energy-uncoupled growth of Zymomonas mobilis ZM1. Mol Biosyst, 2016, 12(4): 1241-1249. |
| [12] |
Oberhardt MA, Palsson BØ, Papin JA. Applications of genome-scale metabolic reconstructions. Mol Syst Biol, 2009, 5: 320. DOI:10.1038/msb.2009.77 |
| [13] |
Pál C, Papp B, Lercher MJ, et al. Chance and necessity in the evolution of minimal metabolic networks. Nature, 2006, 440(7084): 667-670. |
| [14] |
O'Brien EJ, Palsson BO. Computing the functional proteome: recent progress and future prospects for genome-scale models. Curr Opin Biotechnol, 2015, 34: 125-134. |
| [15] |
Ye C, Xu N, Chen XL, et al. Application of metabolic network model to analyze intracellular metabolism of industrial microorganisms. Chin J Biotech, 2019, 35(10): 1901-1913 (in Chinese). 叶超, 徐楠, 陈修来, 等. 应用代谢网络模型解析工业微生物胞内代谢. 生物工程学报, 2019, 35(10): 1901-1913. |
| [16] |
Lee SJ, Lee DY, Kim TY, et al. Metabolic engineering of Escherichia coli for enhanced production of succinic acid, based on genome comparison and in silico gene knockout simulation. Appl Environ Microbiol, 2005, 71(12): 7880-7887. |
| [17] |
Wang QZ, Zhao XM. The research progress of succinic acid fermentation strains. Chin J Biotechnol, 2007, 23(4): 570-576 (in Chinese). 王庆昭, 赵学明. 琥珀酸发酵菌种研究进展. 生物工程学报, 2007, 23(4): 570-576. |
| [18] |
Brochado AR, Patil KR. Overexpression of O-methyltransferase leads to improved vanillin production in baker's yeast only when complemented with model-guided network engineering. Biotechnol Bioeng, 2013, 110(2): 656-659. |
| [19] |
Oliveira AP, Nielsen J, Förster J. Modeling Lactococcus lactis using a genome-scale flux model. Bmc Microbiol, 2005, 5: 39. |
| [20] |
Flahaut NAL, Wiersma A, Van De Bunt B, et al. Genome-scale metabolic model for Lactococcus lactis MG1363 and its application to the analysis of flavor formation. Appl Microbiol Biotechnol, 2013, 97(19): 8729-8739. |
| [21] |
Teusink B, Wiersma A, Molenaar D, et al. Analysis of growth of Lactobacillus plantarum WCFS1 on a complex medium using a genome-scale metabolic model. J Biol Chem, 2006, 281(52): 40041-40048. |
| [22] |
Xu N, Liu J, Ai LZ, et al. Reconstruction and analysis of the genome-scale metabolic model of Lactobacillus casei LC2W. Gene, 2015, 554(2): 140-147. |
| [23] |
Shi WJ, Li Y, Gao XL, et al. Improvement of the respiration efficiency of Lactococcus lactis by decreasing the culture pH. Biotechnol Lett, 2016, 38(3): 495-501. |
| [24] |
Poolman B, Konings WN. Relation of growth of Streptococcus lactis and Streptococcus cremoris to amino acid transport. J Bacteriol, 1988, 170(2): 700-707. |
| [25] |
Cocaign-Bousquet M, Garrigues C, Novak L, et al. Rational development of a simple synthetic medium for the sustained growth of lactococcus-lactis. J Appld Bacteriol, 1995, 79(1): 108-116. |
| [26] |
Heirendt L, Arreckx S, Pfau T, et al. Creation and analysis of biochemical constraint-based models using the COBRA Toolbox v.3.0. Nat Protocols, 2019, 14(3): 639-702. |
| [27] |
Liu T, Zou W, Liu LM, et al. A constraint-based model of Scheffersomyces stipitis for improved ethanol production. Biotechnol Biofuels, 2012, 5: 72. |
| [28] |
Ma ZB, Ye C, Deng WW, et al. Reconstruction and analysis of a genome-scale metabolic model of ganoderma lucidum for improved extracellular polysaccharide production. Front Endocrinol, 2018, 9: 12. DOI:10.3389/fmicb.2018.03076 |
| [29] |
Tian WD, Skolnick J. How well is enzyme function conserved as a function of pairwise sequence identity?. J Mol Biol, 2003, 333(4): 863-882. |
| [30] |
Moriya Y, Itoh M, Okuda S, et al. KAAS: an automatic genome annotation and pathway reconstruction server. Nucleic Acids Res, 2007, 35(S2): W182-W185. |
| [31] |
Liu T. Reconstruction and application of Scheffersomyces stipits genome-scale metabolic model[D]. Jiangsu: Jiangnan University, 2013(in Chinese). 刘婷.树干毕赤酵母基因组规模代谢网络模型构建及应用[D].无锡: 江南大学, 2013. http://cdmd.cnki.com.cn/Article/CDMD-10295-1013309498.htm |
| [32] |
King ZA, Lu J, Dräeger A, et al. BiGG models: a platform for integrating, standardizing and sharing genome-scale models. Nucleic Acids Res, 2016, 44(D1): D515-D522. |
| [33] |
Yu CS, Lin CJ, Hwang JK. Predicting subcellular localization of proteins for Gram-negative bacteria by support vector machines based on n-peptide compositions. Protein Sci, 2004, 13(5): 1402-1406. |
| [34] |
Thiele I, Palsson BO. A protocol for generating a high-quality genome-scale metabolic reconstruction. Nat Protoc, 2010, 5(1): 93-121. |
| [35] |
Chan SHJ, Cai JY, Wang L, et al. Standardizing biomass reactions and ensuring complete mass balance in genome-scale metabolic models. Bioinformatics, 2017, 33(22): 3603-3609. |
| [36] |
Caspi R, Billington R, Fulcher CA, et al. The MetaCyc database of metabolic pathways and enzymes. Nucleic Acids Res, 2018, 46(D1): D633-D639. DOI:10.1093/nar/gkm900 |
| [37] |
Moretti S, Martin O, Van Du Tran T, et al. MetaNetX/MNXref - reconciliation of metabolites and biochemical reactions to bring together genome-scale metabolic networks. Nucleic Acids Res, 2016, 44(D1): D523-D526. DOI:10.1093/nar/gkv1117 |
| [38] |
Saier MH Jr, Reddy VS, Tsu BV, et al. The transporter classification database (TCDB): recent advances. Nucleic Acids Res, 2016, 44(D1): D372-D379. |
| [39] |
De Vos WM. Metabolic engineering of sugar catabolism in lactic acid bacteria. Anton Van Leeuwenh, 1996, 70(2): 223-242. |
| [40] |
Castro R, Neves AR, Fonseca LL, et al. Characterization of the individual glucose uptake systems of Lactococcus lactis: mannose-PTS, cellobiose-PTS and the novel GlcU permease. Mol Microbiol, 2009, 71(3): 795-806. |
| [41] |
Lahtvee PJ, Adamberg K, Arike L, et al. Multi-omics approach to study the growth efficiency and amino acid metabolism in Lactococcus lactis at various specific growth rates. Microb Fact, 2011, 10: 12. |
| [42] |
Wang YL, Xu N, Ye C, et al. Reconstruction and in silico analysis of an Actinoplanes sp. SE50/110 genome-scale metabolic model for acarbose production. Front Microbiol, 2015, 6: 632. |
 2020, Vol. 36
2020, Vol. 36




