中国科学院微生物研究所、中国微生物学会主办
文章信息
- 王甜忆, 程中一, 郭军玲, 夏媛媛, 刘中美, 周哲敏
- Wang Tianyi, Cheng Zhongyi, Guo Junling, Xia Yuanyuan, Liu Zhongmei, Zhou Zhemin
- 红球菌低分子量型腈水合酶的异源激活及激活子的结构域功能
- Activation of low-molecular-mass nitrile hydratase from Rhodococcus rhodochrous J1 by heterologous activators
- 生物工程学报, 2020, 36(8): 1578-1589
- Chinese Journal of Biotechnology, 2020, 36(8): 1578-1589
- 10.13345/j.cjb.190562
-
文章历史
- Received: December 17, 2019
- Accepted: February 17, 2020
腈水合酶(Nitrile hydratase,简称NHase,EC 4.2.1.84)是一类能将腈类化合物通过水合作用生成相应酰胺化合物的金属酶[1]。与化学法合成酰胺相比,酶法合成酰胺具有反应条件温和、转化率高、产量高等优势,因此NHase作为生物催化剂,在工业上应用于大规模生产丙烯酰胺、烟酰胺和5-氰基戊酰胺等产品[2]。晶体结构分析表明NHase由α亚基和β亚基等比例组成[3]。α亚基具有一段高度保守的序列VC (S/T) LCSC (T/Y),该序列构成NHase的活性中心[4-5],根据活性中心结合的金属离子(一个非卟啉铁离子或一个非咕啉钴离子)不同,可将NHase分为铁型腈水合酶(Fe-NHase)[6]和钴型腈水合酶(Co- NHase)[7]。NHase通常表现为α2β2四聚体[8-10],此外也有八聚体、二十四聚体等四级结构形式。
NHase最早由Asano等在节杆菌Arthrobacter sp. J-1中发现[11],随后在假单胞菌[12-13]、芽胞杆菌[9]、假诺卡氏菌[8]等原核生物中得到报道,随着基因组数据的不断增长,近年来在真核生物中也发现了NHase的存在[14]。在原核生物的基因组中,编码α亚基和β亚基的结构基因下游通常具有一个开放阅读框,其编码的蛋白产物被称为激活子。NHase获取金属离子,完成翻译后修饰,最终形成有功能的成熟酶,一般需要激活子的协助[15]。尽管Fe-NHase与Co-NHase的序列相似性较高,但两者激活子的序列相似性却很低,在大小上也存在很大差异。也有报道睾丸酮丛毛单胞菌Comamonas testosteroni Ni1来源的Fe-NHase和真核生物领鞭毛虫Monosiga brevicollis来源的Co-NHase不需要激活子[16-17]。
虽然激活子对于NHase的活性表达具有至关重要的作用,但与NHase本身的研究相比,关于激活子的研究较少,尚未有结构、功能方面的全面报道。尤其是Co-NHase激活子,由于分子量小、不稳定等因素,有时难以检测到其表达。Liu等通过N端序列改造在大肠杆菌中成功表达了来源于恶臭假单胞菌Pseudomonas putida NRRL- 18668的NHase激活子P14K[18]。
目前认为Fe-NHase激活子作为金属离子伴随子,主要参与Fe-NHase生物合成过程中的铁离子输送。通过序列比对发现Fe-NHase激活子具有一段保守基序CXCC,可能构成金属离子结合位点,对于该基序中3个保守的半胱氨酸残基进行突变,将影响Fe-NHase的正常表达[19]。马红球菌Rhodococcus equi TG328-2来源的Fe-NHase激活子属于GTPase的一个亚家族,具有将GTP水解为GDP的能力[20]。
对玫瑰色红球菌Rhodococcus rhodochrous J1来源的Co-NHase进行研究时发现,它的成熟遵循一种“亚基自身交换”机制[21]。在体内,α亚基与β亚基结合形成不含钴离子的apo-α2β2,与此同时,α亚基与激活子结合形成含钴离子的蛋白复合物holo-αe2,在亚基自身交换驱动力的作用下,apo-α2β2与holo-αe2之间发生α亚基的交换,最终形成有活性的NHase。这种机制在P. putida NRRL-18668来源的Co-NHase中也得到验证[22]。另外,Xia等在对Co-NHase进行亚基融合时发现,在不能发生亚基自身交换的情况下,激活子同样能激活Co-NHase[23]。上述研究说明Co-NHase激活子可以作为亚基自身交换伴随子,也可能作为金属离子伴随子。Xia等通过研究提出Co-NHase激活子的C端区域可能具有潜在的钴离子结合位点[24];Pei等通过二级结构预测和实验验证提出Co-NHase激活子连续的α螺旋结构对于功能的发挥非常重要[25]。
不同来源的Co-NHase激活子的序列保守性较低,但激活子对于NHase的成熟十分重要,因此分析其激活作用的特点,探索其结构与功能之间的关系,对于深入了解Co-NHase的成熟机制具有重要意义。本研究将R. rhodochrous J1来源的低分子量型腈水合酶(L-NHase)分别与R. rhodochrous J1高分子量型腈水合酶(RrH-NHase)、P. putida NRRL-18668腈水合酶(PpNHase)、温泉热碱芽胞杆菌Caldalkalibacillus thermarum TA2.A1腈水合酶(CtNHase)和嗜热假诺卡氏菌Pseudonocardia thermophila JCM3095腈水合酶(PtNHase)的激活子进行组合,研究不同来源激活子对L-NHase的激活作用,对激活子的一级序列与三级结构进行分析,并进一步探索了相关结构域的功能。结果表明4种异源激活子均能激活L-NHase,但激活后L-NHase的比酶活存在差异;激活子的保守结构域是其行使激活功能的关键,同时N端和C端结构域对于激活作用也具有重要影响。
1 材料与方法 1.1 实验材料 1.1.1 菌株、质粒及培养基克隆宿主大肠杆菌Escherichia coli DH5α、表达宿主E. coli BL21 (DE3)由本实验室保藏。实验所用质粒如表 1所示。种子培养为LB培养基(g/L):胰蛋白胨10.0,酵母提取物5.0,NaCl 10.0;蛋白表达为2×YT培养基(g/L):胰蛋白胨16.0,酵母提取物10.0,NaCl 5.0;培养时添加卡那霉素至终浓度为50 μg/mL;固体培养基添加1.5%的琼脂粉。
| Plasmid | Description | Source |
| pET-24a(+) | E. coli expression vector, T7 promoter, Kanr | Lab stock |
| pRrH-NHase | pET-24a(+) with H-NHase from Rhodococcus rhodochrous J1 | Lab stock |
| pPpNHase | pET-24a(+) with NHase from Pseudomonas putida NRRL-18668 | Lab stock |
| pCtNHase | pET-24a(+) with NHase from Caldalkalibacillus thermarum TA2.A1 | Lab stock |
| pPtNHase | pET-24a(+) with NHase from Pseudonocardia thermophila JCM3095 | Lab stock |
| pBAL-NHaseE | pET-24a(+) with β subunit, α subunit, activator E | Lab stock |
| pBAL-NHase | pET-24a(+) with β subunit, α subunit | Lab stock |
| pBAL-NHaseG | pET-24a(+) with β subunit, α subunit, activator G | This study |
| pBAL-NHaseP | pET-24a(+) with β subunit, α subunit, activator P | This study |
| pBAL-NHaseC | pET-24a(+) with β subunit, α subunit, activator C | This study |
| pBAL-NHaseA | pET-24a(+) with β subunit, α subunit, activator A | This study |
| pBAL-NHaseENΔ35 | Derived from pBAL-NHaseE by deleting 35 N-terminal amino acids of activator E | This study |
| pBAL-NHaseECΔ27 | Derived from pBAL-NHaseE by deleting 27 C-terminal amino acids of activator E | This study |
| pBAL-NHaseEΔWE-YY | Derived from pBAL-NHaseE by deleting 47 conserved domain amino acids of activator E | This study |
| pBAL-NHaseGNex | Derived from pBAL-NHaseG by exchanging N-terminal amino acids of activator G with activator E | This study |
| pBAL-NHaseGCadd | Derived from pBAL-NHaseG by adding 27 C-terminal amino acids of activator E to activator G | This study |
| pBAL-NHaseGNexCadd | Derived from pBAL-NHaseGNex by adding 27 C-terminal amino acids of activator E to activator G | This study |
| pBAL-NHaseGΔWE-YY | Derived from pBAL-NHaseG by deleting 41 conserved domain amino acids of activator G | This study |
DNA聚合酶PrimeSTARTM Max DNA Polymerase、限制性内切酶Quickcut Dpn Ⅰ、蛋白质定量试剂盒TaKaRa Bradford Protein Assay Kit、反转录试剂盒PrimeScriptTM RT reagent Kit和实时荧光定量PCR (qPCR)试剂盒TB Green® Premix Ex TaqTM Ⅱ购于宝生物工程(大连)有限公司;琼脂糖凝胶DNA回收试剂盒和细菌总RNA提取试剂盒购于天根生化科技(北京)有限公司;重组试剂盒2×GenRec Assembly Master Mix Kit购于通用生物系统(安徽)有限公司;脱硫生物素d-Desthiobiotin购于西格玛奥德里奇中国公司;3-氰基吡啶(烟腈)和烟酰胺购于上海阿拉丁生化科技股份有限公司。
PCR仪、电泳仪、CFX96实时荧光定量PCR仪,美国Bio-Rad公司;ÄKTA pure蛋白纯化仪、StrepTrap HP 1 mL蛋白纯化柱,美国通用电气医疗集团;C18色谱柱,美国沃特世公司;高效液相色谱仪、U-3900紫外可见分光光度计,日本日立株式会社。
1.2 实验方法 1.2.1 异源激活子L-NHase的构建采用定向克隆的方法构建异源激活子L-NHase的表达质粒pBAL-NHaseG、pBAL-NHaseP、pBAL-NHaseC和pBAL-NHaseA,重组质粒的基因结构及构建所用引物分别如图 1和表 2所示。以pBAL-NHaseE为模板,通过反向PCR扩增得到删除激活子E基因的线性化载体,同时分别以pRrH-NHase、pPpNHase、pCtNHase、pPtNHase为模板,PCR扩增得到两端带有线性化载体同源序列的激活子G、P、C和A的基因片段。将上述线性化载体与各激活子基因片段进行琼脂糖凝胶电泳并切胶回收,然后使用2×GenRec重组试剂盒进行重组反应。将重组产物转化E. coli DH5α感受态细胞,涂布于含有卡那霉素的LB平板,37 ℃培养20 h,阳性转化子由天霖生物科技(无锡)有限公司进行测序验证。

|
| 图 1 出发酶及异源激活子L-NHase的基因结构 Fig. 1 Gene structures of the original L-NHase and the heterologous activator L-NHases. |
| |
分别以质粒pBAL-NHaseE和pBAL-NHaseG为模板,采用全质粒PCR的方法构建结构域突变激活子L-NHase的表达质粒pBAL-NHaseENΔ35、pBAL-NHaseECΔ27、pBAL-NHaseEΔWE-YY、pBAL-NHaseGNex、pBAL-NHaseGCadd、pBAL-NHaseGNexCadd和pBAL-NHase GΔWE-YY,构建所用引物见表 2。将PCR产物回收后,用Dpn Ⅰ进行酶切,消化模板质粒,然后转化E. coli DH5α感受态细胞并测序验证。
| Primer name | Primer sequence (5′–3′) | Size (bp) |
| BA-F | AGCTCCGTCGACAAGCTTGC | 20 |
| BA-R | TCAGGCCTTGCTGGGTGTGG | 20 |
| RrG-F | CCCCACCACACCCAGCAAGGCCTGAAAGGAGATATAGATATGAGCGAAGATACCCTGAC | 59 |
| RrG-R | CGGCCGCAAGCTTGTCGACGGAGCTTTAATCAATAATGGCCATACTTTCCATACGTTC | 58 |
| PpP-F | CCCCACCACACCCAGCAAGGCCTGAAAGGAGATATAGATATGAAAGACGAACGGTTTCC | 59 |
| PpP-R | CGGCCGCAAGCTTGTCGACGGAGCTTCAAGCCATTGCGGCAACGAC | 46 |
| CtC-F | CCCCACCACACCCAGCAAGGCCTGAAAGGAGATATAGATATGGAGAAAAATTGCGTGAGCCAG | 63 |
| CtC-R | CGGCCGCAAGCTTGTCGACGGAGCTTTAAAAAAACTCATCGCGTTCGCCG | 50 |
| PtA-F | CCCCACCACACCCAGCAAGGCCTGAAAGGAGATATAGATATGAGCGCTGAAGCCAAAGTG | 60 |
| PtA-R | CGGCCGCAAGCTTGTCGACGGAGCTTTAGCTGCGAACTGCCGGATG | 46 |
| ENdele35-F | CTGAAAGGAGATATAGATATGGCCTTCGATCAGGCCTGGGAG | 42 |
| ENdele35-R | CATATCTATATCTCCTTTCAGGCCTTGCTGGGTGTGG | 37 |
| ECdele27-F | GGTGCTGGCAACGCCCTGAAGCTCCGTCGACAAGCTTGC | 39 |
| ECdele27-R | GGGCGTTGCCAGCACCTGCTCGGTAC | 26 |
| EdeleWE- YY-F | TTCGATCAGGCCGAGCGTTGGATGCTCGCACTCGAA | 36 |
| EdeleWE- YY-R | CATCCAACGCTCGGCCTGATCGAAGGCGACCTCGCC | 36 |
| GNex-F | ACCCAGCAAGGCCTGAAAGGAGATATAGATATGCCCCGACTCAACGAACAACCCCACCCGG GTCTCGAAGCCAACCTCGGCGACCTGGTACAGAATCTGCCGTTCAACGAACGAATCCCCCGC CGCTCCGGTGAACTGGTTTTTACCGAACCGTGG | 156 |
| GNex-R | CTGATCGCTCAGTGCAATGGC | 21 |
| GCadd-F | GAAAGTATGGCCATTATTGATGCCGGCGCCCATCACCAGCACGCCGT GCGTGATCCCATCGCCGTGCACGCCATCGGCACACGCACCACTGACTCCGACGGGTAAAGCTCCGTCGACAAGCTTGCGG | 127 |
| GCadd-R | TTTGTTAGCAGCCGGATCTCAGTG | 24 |
| GdeleWE- YY-F | GGTGAACTGGTTTTTACCGAACCGGAAAGTTGGACCAAAGCACTGG | 46 |
| GdeleWE- YY-R | CGGTTCGGTAAAAACCAGTTCACCATTATCACGAGGAGGTGC | 42 |
| Pq-16S-F | TAATACCTTTGCTCATTG | 18 |
| Pq-16S-R | CCAGTAATTCCGATTAAC | 18 |
| Pq-B-F | TTCAATCTCGACCAGTTC | 18 |
| Pq-B-R | ATCCAGTGCTCGTAGTAT | 18 |
| Pq-A-F | CAGGGCGAAGAAATGGTC | 18 |
| Pq-A-R | ATAGCACGAGCACAAGGTA | 19 |
| Pq-G-F | CGTCTGATTCATAGCATT | 18 |
| Pq-G-R | CCACTATCAACAACACTT | 18 |
| Pq-P-F | TGTGTTTGACGAGCCTTG | 18 |
| Pq-P-R | TTCCACTGAAAGAGACCG | 18 |
将重组质粒转化E. coli BL21 (DE3)感受态细胞,挑取单菌落至5 mL LB培养基,37 ℃、200 r/min培养8 h左右。将种子液按1% (V/V)转接至100 mL 2×YT培养基,37 ℃、200 r/min培养至OD600为0.6–0.8,加入终浓度为0.4 mmol/L的异丙基硫代半乳糖苷(IPTG)以及0.1 g/L的CoCl2·6H2O,在24 ℃条件下诱导表达16 h。
L-NHase的β亚基C端融合了Strep tag (WSHPQFEK),因此采用亲和层析的方法对其进行纯化。首先离心收集菌体细胞(10 000 r/min,3min),弃培养基上清。然后用20 mL结合缓冲液(20 mmol/L Na2HPO4·12H2O,280 mmol/L NaCl,6 mmol/L KCl,pH 7.4)重悬细胞,于冰水混合物中超声破碎(150 W,15 min)。将破碎后的粗酶液离心(4 ℃,12 000 r/min,40 min),去除细胞碎片和不可溶蛋白,取上清液过0.22 μm微孔滤膜,以备上样。
纯化操作在4 ℃下进行,首先以1 mL/min的流速,用结合缓冲液平衡StrepTrap HP 1 mL蛋白纯化预装柱。然后进行上样,完成上样后用结合缓冲液洗去未结合蛋白,直至穿透峰回到基线。最后用100%的洗脱缓冲液(20 mmol/L Na2HPO4·12H2O,280 mmol/L NaCl,6 mmol/L KCl,2.5 mmol/L d-Desthiobiotin,pH 7.4)进行目的蛋白的洗脱,收集洗脱峰对应的蛋白样品。收集的目的蛋白于10 mmol/L磷酸氢二钾-磷酸二氢钾缓冲液(pH 7.4)中透析脱盐。蛋白浓度通过Bradford蛋白质定量试剂盒进行测定。目的蛋白的表达与纯化情况通过SDS-PAGE检测。
1.2.4 激活子表达水平的测定将BAL-NHaseG和BAL-NHaseP诱导表达16 h后,按照细菌总RNA提取试剂盒,分别提取其总RNA,进一步按照反转录试剂盒合成cDNA。分别设计大肠杆菌16S rRNA、L-NHase的β亚基、L-NHase的α亚基、激活子G和激活子P的基因引物,按照qPCR试剂盒进行反应,检测各基因的表达水平,记录PCR过程中达到一定荧光强度阈值时所需要的循环数(Ct值)。
1.2.5 L-NHase酶活力的测定用10 mmol/L磷酸氢二钾-磷酸二氢钾缓冲液(pH 7.4)将纯酶的浓度稀释为0.5 mg/mL,取10 μL至1.5 mL离心管中,置于25 ℃金属浴上。向离心管中加入490 μL 200 mmol/L烟腈溶液作为底物,充分涡旋混匀,25 ℃下反应10 min,然后加入500 μL 100%乙腈溶液终止反应。将反应液离心,上清液用100%乙腈溶液稀释适当倍数,过0.22 μm滤膜。
液相检测方法:检测波长为215 nm,柱温为40 ℃,流动相组成为乙腈︰水=1︰2 (V/V),流速为0.6 mL/min,测定反应体系中产物烟酰胺的生成量。一个酶活力单位(U)定义为25 ℃下,每分钟催化生成1 μmol烟酰胺所需要的酶量。比酶活(U/mg)定义为每毫克L-NHase纯酶(α2β2四聚体)所具有的酶活力单位数。
1.2.6 L-NHase钴离子特征吸收峰的测定采用紫外-可见光扫描的方法检测L-NHase中钴离子的特征吸收峰,定性反映钴离子含量。将纯酶的浓度用10 mmol/L磷酸氢二钾-磷酸二氢钾缓冲液(pH 7.4)稀释为0.5 mg/mL,首先以空白缓冲液校正基线,然后设置波长扫描范围为300–450 nm,扫描速度为300 nm/min,取300 μL蛋白样品至微量石英比色皿中进行测定。根据不同波长下样品的吸收值绘制曲线图。
1.2.7 激活子的序列分析与结构预测激活子E、G、P、C和A的保守结构域分析基于NCBI的Conserved Domain Database (https://www.ncbi.nlm.nih.gov/cdd)[26];激活子E、G、P、C、A与L-NHase的β亚基的一级序列比对使用在线软件Clustal Omega (http://www.ebi.ac.uk/Tools/msa/clustalo);激活子E和G的三级结构模拟使用在线软件C-QUARK (https://zhanglab.ccmb.med.umich.edu/C-QUARK/);蛋白质模型评估使用网站SAVES v5.0 (https://servicesn.mbi.ucla.edu/SAVES/)中的Verify 3D[27]和PROCHECK程序。
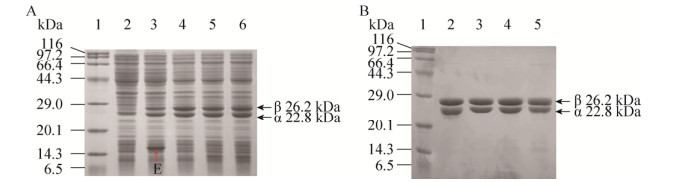
2 结果与分析 2.1 异源激活子对L-NHase的激活作用为了研究异源激活子对L-NHase的激活作用,以缺失激活子E的BAL-NHase作为对照,并将出发酶BAL-NHaseE的激活子E分别替换为RrH-NHase、PpNHase、CtNHase以及PtNHase的激活子G、P、C和A,构建了BAL-NHaseG、BAL-NHaseP、BAL-NHaseC和BAL-NHaseA。将出发酶与异源激活子L-NHase在大肠杆菌中表达,并通过β亚基C端带有的Strep亲和层析标签,进行L-NHase的纯化,结果如图 2所示。激活子的替换没有对L-NHase的表达产生较大影响,可以观察到激活子E、C和A的条带,但激活子G和P的表达情况难以检测。

|
| 图 2 SDS-PAGE分析异源激活子L-NHase的表达和纯化 Fig. 2 SDS-PAGE analysis of the heterologous activator L-NHases. (A) SDS-PAGE of the cell-free extracts. 1: marker; 2: control; 3: BAL-NHase; 4: BAL-NHaseE; 5: BAL-NHaseG; 6: BAL-NHaseP; 7: BAL-NHaseC; 8: BAL-NHaseA. (B) SDS-PAGE of the purified enzymes. 1: marker; 2: BAL-NHase; 3: BAL-NHaseE; 4: BAL-NHaseG; 5: BAL-NHaseP; 6: BAL-NHaseC; 7: BAL-NHaseA. The red arrows indicate the bands of the activators. |
| |
为了检测激活子G和P的表达水平,对BAL-NHaseG和BAL-NHaseP进行qPCR实验,分别测定了BAL-NHaseG中16S rRNA、β亚基、α亚基和激活子G的表达水平,以及BAL-NHaseP中16S rRNA、β亚基、α亚基和激活子P的表达水平。结果如图 3所示,β亚基、α亚基和激活子G (或P)的Ct值均低于大肠杆菌内源的16S rRNA,说明β亚基、α亚基和激活子G (或P)均实现了过表达;激活子G (或P)的Ct值与β亚基和α亚基基本一致,说明激活子G (或P)在转录水平上与β亚基和α亚基并无差异。根据前期研究[18],激活子G和P无法在SDS-PAGE上进行检测,可能主要与其性质不稳定、半衰期短有关。

|
| 图 3 BAL-NHaseG和BAL-NHaseP的实时荧光定量PCR Fig. 3 qPCR of BAL-NHaseG and BAL-NHaseP. (A) Ct value of 16S rRNA, β subunit, α subunit and activator G in BAL-NHaseG. (B) Ct value of 16S rRNA, β subunit, α subunit and activator P in BAL-NHaseP. |
| |
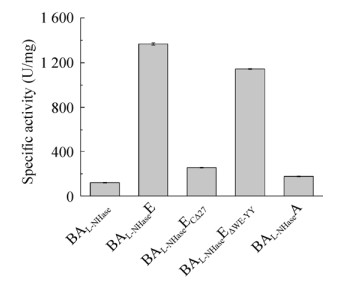
通过高效液相色谱法测定了出发酶及异源激活子L-NHase的比酶活,以此表征不同来源的激活子对L-NHase的激活作用。如图 4所示,在共表达激活子的情况下,L-NHase的比酶活均高于对照BAL-NHase的比酶活(120.10±2.13) U/mg,说明激活子对于L-NHase的激活作用具有普遍性;其中BAL-NHaseA的比酶活为(1 336.62±16.77) U/mg,达到出发酶比酶活(1 366.88±10.34) U/mg的97.79%,BAL-NHaseG的比酶活最低,为(327.24± 11.00) U/mg,是出发酶比酶活的23.94%,说明激活子对于L-NHase的激活作用具有特异性,激活子A对L-NHase的激活程度与激活子E相近,而激活子G对L-NHase的激活程度最低。

|
| 图 4 异源激活子L-NHase的比酶活 Fig. 4 Specific activities of the heterologous activator L-NHases. |
| |
钴离子对于Co-NHase的活性必不可少,Co-NHase激活子在细胞体内作为亚基自身交换伴随子发挥作用,协助α亚基摄取钴离子。推测不同来源的激活子通过影响L-NHase的钴离子含量,从而影响其活性。由于活性中心半胱氨酸残基S原子向配体Co3+的电荷转移,Co-NHase在300–350 nm波长区域存在吸收[28],吸收峰的高低可以定性反映Co-NHase中钴离子含量的多少。为了验证上述推测,对出发酶及异源激活子L-NHase进行紫外-可见光扫描。如图 5所示,出发酶及各异源激活子L-NHase在300–350 nm波长区域的吸收峰均高于对照BAL-NHase,且吸收峰值的高低与比酶活的高低趋势一致,说明不同来源的激活子均能通过协助α亚基摄取钴离子从而激活L-NHase,激活效果的差异是由于最终L-NHase的α亚基中钴离子的含量不同导致的。
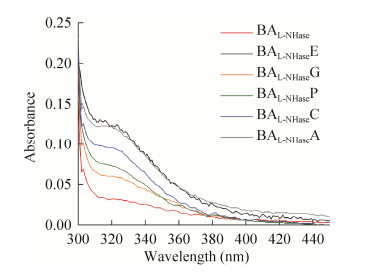
|
| 图 5 异源激活子L-NHase的紫外-可见光扫描 Fig. 5 UV-Vis absorption spectra of the heterologous activator L-NHases. |
| |
尽管来源各不相同,在表达过程中的稳定性也存在差异,上述5个激活子均能对L-NHase产生激活作用,为了研究激活子结构与功能上的关系,对其一级序列进行保守性分析。蛋白质保守结构域是蛋白质在进化过程中或者一个蛋白质家族所具有的相同的结构域,一般具有重要的功能。将激活子E、G、P、C和A的氨基酸序列提交至NCBI的保守结构域数据库(Conserved domain database),结果显示5条氨基酸序列均包含激活子保守结构域(登录号:TIGR03889),该结构域属于NHase的β亚基超家族。说明激活子E、G、P、C和A都具有结构相似的功能区域,因此均能激活L-NHase。另外,激活子与NHase的β亚基可能在序列上存在关联,因此将激活子和L-NHase的β亚基的氨基酸序列进行比对,结果如图 6所示,5条激活子序列与β亚基的N端序列存在一定的相似性。

|
| 图 6 激活子E、G、P、C、A和L-NHase的β亚基的一级序列比对 Fig. 6 Multiple sequence alignment of activator E, G, P, C, A and β subunit of L-NHase. The conserved domain (TIGR03889) is highlighted in red and the black boxes represent partial sequences of the conserved domain. |
| |
获得激活子的三维结构可以更加直观地研究其性质和功能。尽管PDB数据库中已有数十个与NHase相关的晶体结构,目前仍未有激活子的结构得到解析。随着生物信息学的快速发展,已有许多方法可以对结构未知的蛋白质进行模拟,对于没有建模模板的蛋白质,也可以通过从头计算的方式预测结构。C-QUARK是Zhang Lab建立的一种性能较好的从头预测蛋白质结构的计算方法[29],因此适用于尚无同源模板的激活子。在上述激活子中,由激活子E激活后的L-NHase的比酶活最高,而由激活子G激活后的L-NHase的比酶活最低,为了探究其中的原因,对激活子E和G的结构进行进一步研究。将两者的氨基酸序列提交至C-QUARK网站建模,然后通过SAVES v5.0网站中的Verify 3D和PROCHECK评分系统对建立好的模型进行质量评估。综合评分最高的激活子E和G的三维结构模型如图 7所示,发现两者的结构在N端存在差异,激活子E的N端是一段由α螺旋和loop构成的臂状结构,而激活子G的N端主要是loop结构;激活子G的序列短于激活子E,在模型上体现为激活子E的C端比激活子G多出一段α螺旋结构;其他部分的结构非常相似。结合一级序列分析,推测激活子的保守结构域决定了其激活作用,而对L-NHase激活能力的差异主要是由其N端和C端的结构不同导致的。

|
| 图 7 激活子E (绿色)和激活子G (蓝色)的模拟结构 Fig. 7 Three-dimensional models of activator E (green) and activator G (blue). |
| |
为了探究激活子的N端结构域、C端结构域和保守结构域对激活作用的影响,构建了BAL-NHaseENΔ35、BAL-NHaseECΔ27和BAL-NHaseEΔWE-YY。其中BAL-NHaseENΔ35代表将激活子E的N端序列截短35个氨基酸,BAL-NHaseECΔ27代表将激活子E的C端序列截短27个氨基酸,BAL-NHaseEΔWE-YY代表将激活子E的保守结构域中Trp42至Tyr88的氨基酸序列(图 6中黑色框线内氨基酸序列)删除。出发酶及结构域突变激活子(E) L-NHase的表达和纯化情况如图 8所示,当对激活子E进行结构域突变后,激活子的表达量难以检测,说明其稳定性可能受到了影响。测定出发酶与结构域突变激活子(E) L-NHase的比酶活,如图 9所示,BAL-NHaseENΔ35、BAL-NHaseECΔ27和BAL-NHase EΔWE-YY的比酶活分别为(255.04±3.01) U/mg、(1 144.62±3.75) U/mg和(177.12±2.74) U/mg。激活子E的N端35个氨基酸在三维模型中构成臂状结构,当其缺失后,L-NHase的比酶活下降了81.34%,表明N端结构域对于激活子的激活作用非常重要。由于激活子E与β亚基的N端序列具有一定的相似性,且在晶体结构已知的NHase中,β亚基的N端会形成一段环绕α亚基的loop,激活子E的N端可能也与α亚基具有相互作用,因此N端结构域的缺失会显著影响激活作用。当激活子E的C端27个氨基酸缺失时,L-NHase的比酶活下降了16.26%,说明C端结构域对于激活子的激活作用也有一定影响。激活子E的Trp42至Tyr88的氨基酸序列属于保守结构域的一部分,在三维模型中构成两个连续的螺旋结构,当其缺失时,L-NHase的比酶活接近对照BAL-NHase,说明突变后的激活子基本失去激活作用,该保守结构域对于激活子E非常关键。
|
| 图 8 SDS-PAGE分析结构域突变激活子(E) L-NHase的表达和纯化 Fig. 8 SDS-PAGE analysis of the domain-mutated activator (E) L-NHases. (A) SDS-PAGE of the cell-free extracts. 1: marker; 2: control; 3: BAL-NHaseE; 4: BAL-NHaseENΔ35; 5: BAL-NHaseECΔ27; 6: BAL-NHaseEΔWE-YY. (B) SDS-PAGE of the purified enzymes. 1: marker; 2: BAL-NHaseE; 3: BAL-NHaseENΔ35; 4: BAL-NHaseECΔ27; 5: BAL-NHaseEΔWE-YY. The red arrow indicates the band of the activator. |
| |
|
| 图 9 结构域突变激活子(E) L-NHase的比酶活 Fig. 9 Specific activities of the domain-mutated activator (E) L-NHases. |
| |
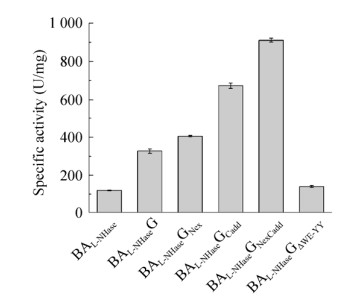
为了进一步验证激活子的N端结构域、C端结构域和保守结构域对激活作用的影响,在BAL-NHaseG的基础上,构建了BAL-NHaseGNex、BAL-NHaseGCadd、BAL-NHaseGNexCadd和BAL-NHaseGΔWE-YY。其中BAL-NHaseGNex代表用激活子E的N端32个氨基酸替换激活子G的N端21个氨基酸,BAL-NHaseGCadd代表将激活子E的C端27个氨基酸添加至激活子G的C端,BAL-NHaseGNexCadd代表在BAL-NHaseGNex的基础上,将激活子E的C端27个氨基酸添加至激活子G的C端,BAL-NHaseGΔWE-YY代表将激活子G的保守结构域中Trp31至Tyr71的氨基酸序列(图 6中黑色框线内氨基酸序列)删除。结构域突变激活子(G) L-NHase的表达和纯化情况如图 10所示,可见BAL-NHaseGCadd和BAL-NHaseGNexCadd的激活子表达量有比较明显的增加,表明添加激活子E的C端结构域可以使激活子G在表达时更加稳定。结构域突变激活子(G) L-NHase的比酶活如图 11所示,BAL-NHaseGNex、BAL-NHaseGCadd和BAL-NHaseGNexCadd的比酶活分别为(405.53±4.09) U/mg、(673.70±13.96) U/mg和(911.03±9.87) U/mg,分别比BAL-NHaseG的比酶活提高了23.85%、105.87%和178.40%。其中N端结构域替换对激活子G的激活能力提高不明显,而C端结构域延长能够使激活子G的激活能力明显提升,将N端结构域替换和C端结构域延长进行组合突变,能够使L-NHase的比酶活得到进一步提高,原因可能是激活子G的稳定性增加有利于其更好地发挥激活作用,同时说明N端结构域和C端结构域共同影响了激活子的激活能力。与BAL-NHaseEΔWE-YY的结果类似,缺失保守结构域部分氨基酸序列的BAL-NHaseGΔWE-YY的比酶活为(140.84±5.47) U/mg,接近对照BAL-NHase的比酶活,说明突变后的激活子基本失去激活作用,同样验证了保守结构域的关键作用。

|
| 图 10 SDS-PAGE分析结构域突变激活子(G) L-NHase的表达和纯化 Fig. 10 SDS-PAGE analysis of the domain-mutated activator (G) L-NHases. (A) SDS-PAGE of the cell-free extracts. 1: marker; 2: control; 3: BAL-NHaseG; 4: BAL-NHaseGNex; 5: BAL-NHaseGCadd; 6: BAL-NHaseGNexCadd; 7: BAL-NHaseGΔWE-YY. (B) SDS-PAGE of the purified enzymes. 1: marker; 2: BAL-NHaseG; 3: BAL-NHaseGNex; 4: BAL-NHaseGCadd; 5: BAL-NHaseGNexCadd; 6: BAL-NHaseGΔWE-YY. The red arrows indicate the bands of the activators. |
| |
|
| 图 11 结构域突变激活子(G) L-NHase的比酶活 Fig. 11 Specific activities of the domain-mutated activator (G) L-NHases. |
| |
本研究将R. rhodochrous J1来源L-NHase的激活子替换为其他4种异源激活子,研究它们对L-NHase的激活作用,并对激活子进行一级序列和三级结构分析,探究激活子中重要结构域的功能。四种异源激活子均能激活L-NHase,在功能上具有普遍性,但它们对于L-NHase的激活程度存在差异,在功能上也具有特异性。原因是各激活子均能协助α亚基摄取钴离子,但最终L-NHase的钴离子含量存在差异。PtNHase的激活子A对L-NHase的激活程度最高,激活后的L-NHase比酶活为出发酶的97.79%;RrH-NHase的激活子G对L-NHase的激活程度最低,激活后的L-NHase比酶活为出发酶的23.94%。激活子保守结构域对其激活作用非常关键,缺失部分序列将导致激活作用基本丧失。N端结构域和C端结构域对于激活子的激活作用也具有重要影响,将激活子G的N端序列替换为激活子E的N端序列,并将激活子E的C端序列添加至激活子G的C端,能够使L-NHase的比酶活提高178.40%。上述结果对于认识Co-NHase激活子结构与功能之间的关系、进一步研究Co-NHase的成熟机制具有重要意义。
| [1] |
Kobayashi M, Shimizu S. Metalloenzyme nitrile hydratase: structure, regulation, and application to biotechnology. Nat Biotechnol, 1998, 16(8): 733-736. DOI:10.1038/nbt0898-733 |
| [2] |
Prasad S, Bhalla TC. Nitrile hydratases (NHases): at the interface of academia and industry. Biotechnol Adv, 2010, 28(6): 725-741. DOI:10.1016/j.biotechadv.2010.05.020 |
| [3] |
Supreetha K, Rao SN, Srividya D, et al. Advances in cloning, structural and bioremediation aspects of nitrile hydratases. Mol Biol Rep, 2019, 46(4): 4661-4673. |
| [4] |
Miyanaga A, Fushinobu S, Ito K, et al. Mutational and structural analysis of cobalt‐containing nitrile hydratase on substrate and metal binding. Eur J Biochem, 2004, 271(2): 429-438. DOI:10.1046/j.1432-1033.2003.03943.x |
| [5] |
Hashimoto Y, Sasaki S, Herai S, et al. Site-directed mutagenesis for cysteine residues of cobalt-containing nitrile hydratase. J Inorg Biochem, 2002, 91(1): 70-77. DOI:10.1016/S0162-0134(02)00373-2 |
| [6] |
Sugiura Y, Kuwahara J, Nagasawa T, et al. Nitrile hydratase: the first non-heme iron enzyme with a typical low-spin iron(Ⅲ)-active center. J Am Chem Soc, 1987, 109(19): 5848-5850. DOI:10.1021/ja00253a046 |
| [7] |
Brennan BA, Alms G, Nelso MJ, et al. Nitrile hydratase from Rhodococcus rhodochrous J1 contains a non-corrin cobalt ion with two sulfur ligands. J Am Chem Soc, 1996, 118(38): 9194-9195. DOI:10.1021/ja961920d |
| [8] |
Miyanaga A, Fushinobu S, Ito K, et al. Crystal structure of cobalt-containing nitrile hydratase. Biochem Biophys Res Commun, 2001, 288(5): 1169-1174. DOI:10.1006/bbrc.2001.5897 |
| [9] |
Hourai S, Miki M, Takashima Y, et al. Crystal structure of nitrile hydratase from a thermophilic Bacillus smithii. Biochem Biophys Res Commun, 2003, 312(2): 340-345. DOI:10.1016/j.bbrc.2003.10.124 |
| [10] |
Huang WJ, Jia J, Cummings J, et al. Crystal structure of nitrile hydratase reveals a novel iron centre in a novel fold. Structure, 1997, 5(5): 691-699. DOI:10.1016/S0969-2126(97)00223-2 |
| [11] |
Asano Y, Tani Y, Yamada H. A new enzyme "nitrile hydratase" which degrades acetonitrile in combination with amidase. Agric Biol Chem, 1980, 44(9): 2251-2252. |
| [12] |
Payne MS, Wu SJ, Fallon RD, et al. A stereoselective cobalt-containing nitrile hydratase. Biochemistry, 1997, 36(18): 5447-5454. DOI:10.1021/bi962794t |
| [13] |
Masutomo S, Inoue A, Kumagai K, et al. Enantioselective hydrolysis of (RS)-2-Isopropyl-4'- chlorophenylacetonitrile by Pseudomonas sp. B21C9. Biosci Biotechnol Biochem, 1995, 59(4): 720-722. DOI:10.1271/bbb.59.720 |
| [14] |
Marron AO, Akam M, Walker G. Nitrile hydratase genes are present in multiple eukaryotic supergroups. PLoS ONE, 2012, 7(4): e32867. DOI:10.1371/journal.pone.0032867 |
| [15] |
Nojiri M, Yohda M, Odaka M, et al. Functional expression of nitrile hydratase in Escherichia coli: requirement of a nitrile hydratase activator and post-translational modification of a ligand cysteine. J Biochem, 1999, 125(4): 696-704. |
| [16] |
Martinez S, Yang XH, Bennett B, et al. A cobalt-containing eukaryotic nitrile hydratase. Biochim Biophys Acta Prot Proteom, 2017, 1865(1): 107-112. DOI:10.1016/j.bbapap.2016.09.013 |
| [17] |
Kuhn ML, Martinez S, Gumataotao N, et al. The Fe-type nitrile hydratase from Comamonas testosteroni Ni1 does not require an activator accessory protein for expression in Escherichia coli. Biochem Biophys Res Commun, 2012, 424(3): 365-370. DOI:10.1016/j.bbrc.2012.06.036 |
| [18] |
Liu Y, Cui WJ, Fang YQ, et al. Strategy for successful expression of the Pseudomonas putida nitrile hydratase activator P14K in Escherichia coli. BMC Biotechnol, 2013, 13: 48. DOI:10.1186/1472-6750-13-48 |
| [19] |
Lu J, Zheng YJ, Yamagishi H, et al. Motif CXCC in nitrile hydratase activator is critical for NHase biogenesis in vivo. FEBS Lett, 2003, 553(3): 391-396. DOI:10.1016/S0014-5793(03)01070-6 |
| [20] |
Gumataotao N, Lankathilaka KPW, Bennett B, et al. The iron-type nitrile hydratase activator protein is a GTPase. Biochem J, 2017, 474(2): 247-258. DOI:10.1042/BCJ20160884 |
| [21] |
Zhou ZM, Hashimoto Y, Shiraki K, et al. Discovery of posttranslational maturation by self-subunit swapping. Proc Natl Acad Sci USA, 2008, 105(39): 14849-14854. DOI:10.1073/pnas.0803428105 |
| [22] |
Liu Y, Cui WJ, Xia YY, et al. Self-subunit swapping occurs in another gene type of cobalt nitrile hydratase. PLoS One, 2012, 7(11): e50829. DOI:10.1371/journal.pone.0050829 |
| [23] |
Xia YY, Cui WJ, Liu ZM, et al. Construction of a subunit-fusion nitrile hydratase and discovery of an innovative metal ion transfer pattern. Sci Rep, 2016, 6: 19183. DOI:10.1038/srep19183 |
| [24] |
Xia YY, Peplowski L, Cheng ZY, et al. Metallochaperone function of the self-subunit swapping chaperone involved in the maturation of subunit-fused cobalt-type nitrile hydratase. Biotechnol Bioeng, 2019, 116(3): 481-489. DOI:10.1002/bit.26865 |
| [25] |
Pei XL, Yang ZF, Wang AM, et al. Identification and functional analysis of the activator gene involved in the biosynthesis of Co-type nitrile hydratase from Aurantimonas manganoxydans. J Biotechnol, 2017, 251: 38-46. DOI:10.1016/j.jbiotec.2017.03.016 |
| [26] |
Marchler-Bauer A, Derbyshire MK, Gonzales NR, et al. CDD: NCBI's conserved domain database. Nucleic Acids Res, 2015, 43(D1): D222-D226. DOI:10.1093/nar/gku1221 |
| [27] |
Eisenberg D, Lüthy R, Bowie JU. VERIFY3D: assessment of protein models with three-dimensional profiles. Methods Enzymol, 1997, 277: 396-404. DOI:10.1016/S0076-6879(97)77022-8 |
| [28] |
Nojiri M, Nakayama H, Odaka M, et al. Cobalt- substituted Fe-type nitrile hydratase of Rhodococcus sp. N-771. FEBS Lett, 2000, 465(2/3): 173-177. |
| [29] |
Zheng W, Li Y, Zhang CX, et al. Deep-learning contact-map guided protein structure prediction in CASP13. Proteins, 2019, 87: 1149-1164. DOI:10.1002/prot.25792 |
 2020, Vol. 36
2020, Vol. 36




