中国科学院微生物研究所、中国微生物学会主办
文章信息
- 孙圳, 卢慧, 肖伟弟, 李衍常, 徐平
- Sun Zhen, Lu Hui, Xiao Weidi, Li Yanchang, Xu Ping
- K27泛素化修饰研究进展
- Progress in K27 ubiquitin modification
- 生物工程学报, 2020, 36(8): 1484-1492
- Chinese Journal of Biotechnology, 2020, 36(8): 1484-1492
- 10.13345/j.cjb.190574
-
文章历史
- Received: December 23, 2019
- Accepted: February 26, 2020
- Published: March 26, 2020
2. 东北大学 生命科学与健康学院 微生物药物研究所, 辽宁 沈阳 110819;
3. 中国医学科学院蛋白组学与新药研发创新单元, 北京 102206
2. Institute of Microbial Pharmaceuticals, College of Life and Health Sciences, Northeastern University, Shenyang 110819, Liaoning, China;
3. Research Unit of Proteomics&Research and Development of New Drug, Chinese Academy of Medical Sciences, Beijing 102206, China
泛素(Ubiquitin)是一个含有76个氨基酸且序列高度保守的小分子蛋白。在真核生物中,泛素分子在泛素激活酶(E1)、泛素结合酶(E2)和泛素连接酶(E3)的级联酶促反应下,共价修饰到底物蛋白赖氨酸侧链的ε氨基上,从而影响被修饰底物的活性、细胞定位、与相互作用蛋白的结合等多种生物学功能[1]。早期研究发现,发生泛素化修饰的蛋白能够靶向到26S蛋白酶体中被特异性降解。但随着研究的深入和泛素化富集、鉴定技术的发展[2],人们发现泛素化修饰还具有诸多非蛋白降解的生物学功能。同时,泛素化修饰为动态可逆的翻译后修饰,去泛素化酶(Deubiquitinase,DUB)对修饰底物的泛素链进行修剪、移除等调控过程保证了泛素化的可逆性,维持了泛素-蛋白酶体系统(Ubiquitin-proteasome system,UPS)的动态平衡[3]。泛素分子本身的7个赖氨酸和N端的甲硫氨酸均可被后续的泛素继续修饰,进而形成8种不同类型的同质泛素链(K6、K11、K27、K29、K33、K48、K63和M1线性链)[4-5]。泛素链的不同连接形式具有不同的空间拓扑结构,从而携带不同的结构信息,发挥不同的生物学作用。这些结构各异的泛素链可被含有特定类型泛素结合蛋白结构域(Ubiquitin binding domain,UBD)的效应蛋白所识别,进而将泛素化信号传递下去,介导了不同的生物学功能,构成了“泛素密码(Ubiquitin code)”的精细调控网络[6]。
每一种泛素链在生物体中含量各不相同。如在酵母细胞中,7种泛素链K6︰K11︰K27︰K29︰K33︰K48︰K63的百分比约为11︰28︰9︰3︰4︰29︰16[7]。其中K48泛素链和K63泛素链被称为经典泛素链,前者主要作为介导泛素化蛋白的特异性降解信号,后者主要介导非蛋白质降解信号[8]。另外,其他泛素链类型被称为非经典泛素链,其功能和调控机制也逐步被报道。K11泛素链参与到细胞分裂过程和转录因子活性调控[9-10];K6泛素链和K33泛素链在DNA损伤后含量明显增加,说明可能参与DNA损伤修复过程[11];K29泛素链可以缓解蛋白酶体应激反应[12];M1泛素链可以修饰在NF-Kappa-B必需的调制器(NF-Kappa-B essential modulator,NEMO)蛋白上激活NF-κB (核因子κB,Nuclear factor kappa-B)通路[13]。越来越多证据表明,非经典泛素链具有重要的生物学功能,是解析和深刻理解“泛素密码”的关键突破口。
K27泛素链是研究最少的泛素链之一。目前,K27泛素链的泛素结合酶、泛素连接酶和去泛素化酶尚属未知,使得K27泛素链还无法通过酶促组装(Enzymatic reconstitution)技术进行体外合成,客观上阻碍了K27泛素链结构的解析和生物学功能机制的研究。K27泛素链的功能包括溶酶体定位、线粒体自噬、免疫过程调节和蛋白质稳态维持等诸多方面。本文就K27泛素链的结构、信号生成、传递和消除及其生物学功能等方面进行综述,并结合笔者所在实验室现有工作介绍K27泛素链相关方面的研究进展和发展趋势。
1 K27泛素链体外化学合成和结构解析泛素是一种具有球状结构且通过氢键紧密折叠的蛋白,包含了5个β折叠和1个α螺旋,构成了SSHSSS结构[14]。底物蛋白上不同类型的泛素链会产生不同的空间构象,从而被下游识别蛋白识别以传递多种多样的生物学信号。因此,想要了解生物体内泛素化修饰的分子机理,必须要合成高纯度的泛素链用于生化结构的表征。目前,通过泛素激活酶、特异性泛素结合酶和泛素连接酶进行体外反应,获得泛素链[15]。相较于其他泛素链修饰位点,K27本身在泛素分子的空间位置具有特殊性。K27、K29、K33三个赖氨酸处于泛素分子的α螺旋内,其他5个修饰位置处于β折叠等相对自由的状态;更重要的是K27的侧链氨基被包埋于泛素分子内部,而K29和K33的侧链氨基则暴露在泛素分子外部,这样的空间构象导致K27泛素链中泛素分子的空间结构发生较大变化,因此可能赋予K27泛素链功能的独特性。另外,由于缺少K27泛素链特异性的泛素结合酶和泛素连接酶,酶促组装K27泛素链的技术还不够成熟。所以在所有类型的泛素链中,解析K27泛素链结构特征成为最具挑战性的工作[16-17]。近些年,泛素链的化学合成法成为体外合成K27泛素链的主要方法。Fushman实验室通过GOPAL法(基因编码正交保护和活化连接,Genetically encoded orthogonal protection and activated ligation)合成K27二聚泛素链,并捕获了K27泛素链的核磁共振结构,发现K27泛素二聚体的异肽键是包埋在受体泛素分子中的,呈现对称的形式[15]。刘磊实验室利用化学方法合成了二聚和三聚的K27泛素链,并使用准外消旋X射线技术获得了这两种K27泛素链的X射线结构[18]。随后该实验改进了合成方法,利用单分子荧光共振能量转移技术(Single-molecule Fö rster resonance energy transfer,smFRET)和X射线晶体衍射技术(X-ray crystallography)对二聚K27泛素链进行检测,证明了二聚K27泛素链紧密的空间结构。但与泛素羧基末端水解酶L3 (Ubiqutin C-terminal hydrolase-L3,UCHL3)蛋白结合时,K27链采取了开放的空间结构[19]。
2 K27泛素链特异性的UBD和DUB泛素在E1、E2和E3的级联反应作用下,形成8种结构特异的同质泛素链。这8种泛素链会和不同的受体蛋白结合,从而行使不同的生物学功能。这些受体蛋白上都至少含有一个能特异性识别特定泛素链的泛素识别结构域。因此,K27泛素链特异性UBD的发现对寻找K27泛素化修饰底物的鉴定具有重要意义。根据K27泛素链的结构,二聚K27泛素链可能利用自身泛素上的疏水域以双配位基的方式与受体蛋白上的泛素结合结构域结合,这种结合方式类似于K48泛素链和UBA2结构域结合的方式[15, 20]。目前系统研究K27泛素链特异性UBD的方法较少[21]。Zhang实验室建立了泛素互作亲和富集质谱(Ubiquitin interactor affinity enrichment-mass spectrometry,UbIA-MS)技术。该技术通过在哺乳动物细胞全裂解液中孵育能够抵抗去泛素化酶作用的K27泛素链二聚体,再对泛素链富集的蛋白进行液相质谱检测,最终找出与K27泛素链相互作用的结合蛋白[22]。同年,Zhao实验室将单一变性氨基酸(Unatural amino acids,UAAs)合并入泛素中,合成了同样抵抗去泛素化酶作用的多聚K27泛素链,并将K27泛素链以固定基质的状态结合在链霉亲和素结合琼脂糖上,经过HEK293T细胞孵育和质谱检测,鉴定出高可信的互作蛋白[23]。两种方法所鉴定的蛋白存在重合的部分,也有特异性的部分,说明含有UBD的受体蛋白特异性结合K27泛素链的状态可能受到K27泛素链长度的影响[24]。此外,K27泛素链与特异性UBD的结合不仅需要结构上提供存在相互作用的理论基础,更需要空间上的自然接触。所以这两种方法都可以寻找到细胞中与K27泛素链特异性结合的互作蛋白,但打破了泛素链与UBD结合的空间共定位特性,所以存在一定的假阳性。
泛素化修饰是一个动态可逆的过程,不同的去泛素化酶可以修剪特定的泛素链,保证泛素系统的平衡以及正常功能的发挥[25]。虽然K27泛素链特异性的去泛素化酶到目前还没有报道,但研究人员通过一种基于芳基偶氮化合物的光亲和性二聚体泛素(diUb)探针,来寻找K27泛素链可能的特异性去泛素化酶[26]。此外,去泛素化酶USP13可以修剪干扰素基因刺激蛋白(Stimulator of interferon genes,STING)上的K27泛素链从而介导免疫反应,去泛素化酶Ataxin3能够水解Parkin蛋白上的K27泛素链调节自噬过程[27-28]。
3 生物学功能 3.1 免疫过程越来越多的证据表明,K27泛素链所介导的自噬过程对维持固有免疫动态平衡具有非常重要的作用。当细胞受到病毒侵染时,TRIM23蛋白能够在自身ARF结构域形成K27泛素链,激活自身GTP水解活性,从而促进TANK结合激酶1 (TANK binding kinase 1,TBK1)对P62的磷酸化,最终引发自噬抵抗病毒[29]。除了通过自噬促进免疫,K27泛素链还可以通过选择性自噬负调控免疫应答。当细胞产生免疫反应后,分泌的人骨髓基质干细胞抗原2 (Bone marrow stromal antigen 2,BST2)蛋白不仅可以束缚被感染细胞中的病毒颗粒,还可以招募泛素连接酶MARCH8在线粒体抗病毒信号蛋白(Mitochondrial antiviral- signaling protein,MAVS)蛋白的第7 (K7)位赖氨酸上发生K27泛素链修饰,货物受体蛋白核点蛋白52 (Nuclear dot protein 52,NDP52)蛋白能够识别MAVS上K27泛素链并介导MAVS靶向到自噬体中引发自噬,最终抑制免疫过程[30]。近期研究表明,MAVS蛋白上赖氨酸第325 (K325)位点在泛素连接酶TRIM21作用下也可以形成K27泛素链,却起到激活下游免疫的作用[31-32]。此外,K27泛素链引起的免疫负调控也出现在未被感染的细胞中[33]。
当宿主细胞受到胞内细菌感染时,泛素连接酶NEDD4会在BECN1蛋白上形成K6/K27泛素链来维持BECN1蛋白的稳定性。作者猜测BECN1蛋白上的K6/K27泛素链能够竞争性抑制自身K48链的生成,减少螺旋肌球蛋白样BCL2结合蛋白(Coiled-coil myosin-like BCL2-interacting protein,Beclin-1)蛋白的蛋白酶体降解,从而保证后续Beclin-1蛋白所介导的自噬能顺利发生[34]。当细胞受到沙门氏杆菌感染时,泛素连接酶LRSAM1通过自身LRR (Leucine-rich)结构域和RING结构域识别细菌,在细菌表面形成泛素链并可能自身形成K6/K27泛素链,从而招募货物受体蛋白NDP52介导细菌靶向到自噬体中[35-36]。除此之外,K27泛素链还可能间接参与了其他免疫应答的过程[37-38]。
K27泛素链不仅可以通过增强自噬程度对抗病毒和细菌的感染,也可以通过刺激其他免疫途径来对抗感染。Airmoto实验室发现,宿主细胞的泛素连接酶TRIM23能够在NEMO上形成K27泛素链,从而激活下游抗病毒通路并促进宿主细胞产生抗病毒干扰素β[39]。但NEMO上的K27泛素链可以和含偏菱形结构域蛋白3 (Rhomboid domain-containing protein 3,Rhbdd3)蛋白结合,招募A20介导NEMO蛋白上K63泛素链的去泛素化过程,从而抑制NF-κB活化[40]。细胞受到HSV感染时,泛素连接酶RNF185在cCAS上形成K27泛素链增加cCAS活性,激活下游STING通路[41]。而下游的STING可以被泛素连接酶AMFR进行K27泛素化修饰,在招募TBK1后激活IRF-3的产生[42]。在抗真菌免疫过程中,泛素连接酶TRIM62在胱天蛋白酶募集域蛋白9 (Caspase recruitment domain-containing protein 9,CARD9)上形成K27泛素链,激活下游免疫应答[43]。K27泛素链既促进免疫应激,也可能导致个体自身发生免疫过激。Avram实验室发现K27泛素链可以被泛素连接酶Hectd3修饰在信号传导转录活化蛋白3 (Signal transducer and activator of transcription 3,Stat3)和粘膜相关淋巴瘤转位基因(Mucosa-associated lymphoid tissue lymphoma translocation protein 1,Malt1)上,促进T辅助细胞17 (Th17)的分化,加重了脑脊髓炎(Encephalomyelitis,EAE)等自身免疫过激反应[44]。
K27泛素链除了帮助宿主细胞促进免疫过程,还能被细菌利用,减弱宿主细胞的免疫能力[45]。登革病毒侵染细胞时,非结构蛋白3 (Non-structural protein 3,NS3)蛋白上发生的K27泛素化修饰会协助非结构蛋白2B (Non-structural protein 2B,NS2B)招募NS3形成复合体蛋白酶NS2B3,形成的NS2B3能够切割STING进而抑制宿主细胞的免疫应答[46]。
3.2 DNA损伤电离辐射、紫外线等环境因素会诱发细胞发生DNA双链断裂(DNA double-strand breaks,DSBs),从而引发DNA损伤应答(DNA damage response,DDR)。以往研究表明,泛素连接酶RNF8会和泛素结合酶UBC13在DNA损伤处形成K63泛素链进行DNA损伤修复。随后Gatti等发现,细胞受到DNA损伤后,泛素连接酶RNF168会在染色质组蛋白H2A和H2A.X上形成K27泛素链。组蛋白上形成的K27泛素链能够招募DDR蛋白,对DNA损伤进行修复[47-48]。这些数据表明,K27泛素链在DNA损伤应答过程中起到招募蛋白的作用。
3.3 帕金森疾病帕金森病(Parkinson’s disease,PD)是一种中老年人群中常见的神经系统退行性疾病,遗传学上已经发现多种基因的突变可能导致该疾病的发生。而编码这些突变基因的蛋白涉及多种细胞信号通路,如线粒体质量控制、错误折叠蛋白调控、跨膜运输以及囊泡释放等[49]。在这些与PD密切相关的基因中,DJ-1基因突变与常染色体隐性遗传型帕金森病有一定的关系。Zucchelli实验室发现,泛素连接酶-肿瘤坏死因子相关受体因子(Tumor necrosis factor-receptor associated factor 6,TRAF6)能够介导突变的DJ-1蛋白和aSYN蛋白上发生K6/K27/K29泛素化修饰,并引发突变DJ-1蛋白的聚集[50]。此实验室还发现,TRAF6还能在亨廷顿蛋白的N段发生K27泛素化修饰,参与到另一种神经性退行性疾病过程中[51]。随后研究者鉴定到,泛素连接酶WSB1能够在突变的富亮氨酸重复激酶2 (Leucine-rich repeat serine/threonine- protein kinase 2,LRRK2)蛋白上形成K27/K29泛素链,促进突变LRRK2蛋白聚集,减少细胞毒性,从而保护神经元细胞[52]。此外,氧化应激引起的线粒体功能障碍导致细胞内氧依赖的蛋白失活,引发细胞死亡并促进帕金森病的产生。氧化应激状态会造成细胞线粒体损伤,并引起线粒体去极化。此时正常细胞中的PTEN诱导激酶1 (PTEN-Induced putative kinase 1,PINK1)蛋白识别受损线粒体并诱导泛素连接酶Parkin在受损线粒体上发生泛素化修饰,减轻能量代谢受损所带来的伤害。Geisler等发现Parkin蛋白可以在电压依赖性阴离子通道蛋白1 (Voltage-dependent anion channel 1,VDAC1)上形成K27泛素链。K27泛素链可以被自噬受体P62识别并形成自噬体,最终引发线粒体自噬,缓解氧化应激状态[53-55]。随后研究发现,线粒体外膜上的线粒体转运蛋白线粒体Rho GTP酶(Mitochondrial Rho GTPase 1,Miro1)在应激条件下,同样可以在泛素连接酶Parkin作用下发生K27泛素链修饰,隔离受损线粒体,防止其继续参与氧化代谢调控网络[56]。
3.4 癌症与其他功能癌症的发生是一个多级多因素的过程。和正常细胞相比,癌细胞的生长状态会发生巨大变化。其中,细胞能量代谢的异常调控、癌细胞组织浸润和转移、肿瘤炎症的发生、细胞死亡抗性等癌细胞特征成为探究癌症发生机制和治疗癌症的关键[57-58]。近些年,K27泛素化修饰在细胞增殖、癌细胞转移中可能的调控作用也逐渐被人们发现。纤维连接蛋白(Fibronectin,FN)是一种糖蛋白,对癌细胞的生长、转移具有重要的调控作用[59]。黑色素瘤细胞中的泛素连接酶HACE1能够在纤维连接蛋白上发生K27泛素化修饰,调控纤维连接蛋白的分泌,从而调节了黑色素瘤细胞中ITGAV和ITGB1的表达,并改变黑色素瘤细胞的粘附和迁移能力[60]。Kong等发现,细胞内泛素连接酶CUL7在Caspase-8发生K27等多泛素化修饰,从而抑制了Caspase的活化以及TRAIL诱导的凋亡,该过程可能促进了癌细胞存活[61]。同样,Singin实验室发现泛素连接酶TRAF4能够促进受体酪氨酸激酶TrkA上发生K27/K29泛素化修饰,提高了TrkA的激酶活性,最终促进癌细胞扩散[62]。
此外,K27泛素链还参与了转化生长因子-β (Transforming growth factor-β,TGF-β)通路[63]、YB-1蛋白的分泌[64]以及溶酶体定位等过程的调控[65-66]。
4 总结与展望近些年来,K27泛素链在DNA损伤、免疫应答、蛋白质稳态调控等生物学领域的作用日益凸显(图 1)。K27泛素链在生物体内不仅可以通过招募结合蛋白来传递信号,还可能在蛋白质的泛素化修饰位点进行“占位”,防止其他泛素链的生成。K27泛素链特异性的去泛素化酶至今没有被发现,其原因可能并不是受限于技术的发展,而是K27泛素链自身紧密包埋的泛素连接结构能够抵抗大部分去泛素化酶的“进攻”[15]。也正是这种稳定的“占位”方式,保证底物上K27泛素链不容易被切割,也防止了其他泛素链的生成,最终维持了底物的稳定。另外,能够特异性识别K27泛素链的泛素结合结构域目前报道较少,K27泛素链生成后如何识别,进而将所携带的生物学信息往下游传递,同样是该领域的热点和挑战。
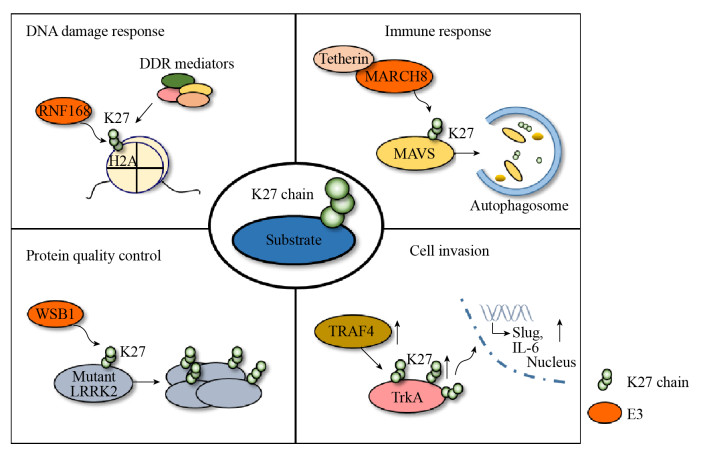
|
| 图 1 K27泛素链的生物学功能 Fig. 1 Biological function of K27 ubiquitin chain. |
| |
泛素修饰的多样化体现了泛素系统精密复杂的调控机制,但对研究者来说,却是一个巨大的挑战。随着泛素化底物富集技术以及高覆盖定量蛋白质组学技术的发展,使得富集并鉴定低丰度泛素化修饰底物成为可能[2]。目前,泛素化蛋白的纯化技术主要有泛素偶联标签法、泛素亲和介质法和diGly富集法。Gao等利用不同UBD的组合,构建出串联杂合UBD (Tandem hybrid UBD,ThUBD),能对不同类型泛素链的泛素化蛋白进行高效、无偏性富集[67]。因此,我们推测发展能够特异性识别K27泛素链的富集材料,对于系统筛选K27泛素链修饰底物,发现其参与的调控过程,进而揭示更多的K27泛素链新功能具有重要意义。高分辨率质谱的研发以及定量技术的发展,使蛋白质组学的目的从鉴定转化为定量。以质谱技术为基础的泛素绝对定量(Absolute quantification,AQUA)和多个去泛素化酶进行的去泛素化酶限制酶切分析(DUB restriction analysis,UbiCRest)使鉴定不同类型的泛素链甚至是分支链成为可能[2]。但由于K27泛素链在生物体内较低的含量以及特殊的链结构和组装方式,人们对K27泛素链的研究还处于起跑阶段[7, 19],很多问题还亟需解决。比如,K27泛素链在E1、E2和E3作用下进行组装的机制;K27泛素链特异性去泛素化酶的发现;高效系统富集K27泛素链修饰底物的筛选策略的建立;与K27泛素链特异性结合UBD结构的预测等。通过特异性结合K27泛素链UBD的结构,总结UBD结构域识别K27泛素链的序列特性,可以为预测结合K27泛素链的UBD序列以及结合机制提供理论基础,从而拓展非经典泛素链在泛素调控系统中的重要作用。
| [1] |
Kulathu YD. Komander. Atypical ubiquitylation the unexplored world of polyubiquitin beyond Lys48 and Lys63 linkages. Nat Rev Mol Cell Biol, 2012, 13(8): 508-523. DOI:10.1038/nrm3394 |
| [2] |
Ordureau A, Münch C, Harper JW. Quantifying ubiquitin signaling. Mol Cell, 2015, 58(4): 660-676. DOI:10.1016/j.molcel.2015.02.020 |
| [3] |
Mevissen TET, Komander D. Mechanisms of deubiquitinase specificity and regulation. Annu Rev Biochem, 2017, 86: 159-192. DOI:10.1146/annurev-biochem-061516-044916 |
| [4] |
Kwon YT, Ciechanover A. The ubiquitin code in the ubiquitin-proteasome system and autophagy. Trends Biochem Sci, 2017, 42(11): 873-886. DOI:10.1016/j.tibs.2017.09.002 |
| [5] |
Hershko A, Ciechanover A. The ubiquitin system. Annu Rev Biochem, 1998, 67: 425-479. DOI:10.1146/annurev.biochem.67.1.425 |
| [6] |
Mattern M, Sutherland J, Kadimisetty K, et al. Using ubiquitin binders to decipher the ubiquitin code. Trends Biochem Sci, 2019, 44(7): 599-615. DOI:10.1016/j.tibs.2019.01.011 |
| [7] |
Xu P, Duong DM, Seyfried NT, et al. Quantitative proteomics reveals the function of unconventional ubiquitin chains in proteasomal degradation. Cell, 2009, 137(1): 133-145. DOI:10.1016/j.cell.2009.01.041 |
| [8] |
Zinngrebe J, Montinaro A, Peltzer N, et al. Ubiquitin in the immune system. EMBO Rep, 2014, 15(1): 28-45. DOI:10.1002/embr.201338025 |
| [9] |
Wickliffe KE, Williamson A, Meyer HJ, et al. K11-linked ubiquitin chains as novel regulators of cell division. Trends Cell Biol, 2011, 21(11): 656-663. DOI:10.1016/j.tcb.2011.08.008 |
| [10] |
Li YC, Dammer EB, Gao Y, et al. Proteomics links ubiquitin chain topology change to transcription factor activation. Mol Cell, 2019, 76(1): 126-137. DOI:10.1016/j.molcel.2019.07.001 |
| [11] |
Elia AEH, Boardman AP, Wang DC, et al. Quantitative proteomic atlas of ubiquitination and acetylation in the DNA damage response. Mol Cell, 2015, 59(5): 867-881. DOI:10.1016/j.molcel.2015.05.006 |
| [12] |
Besche HC, Sha Z, Kukushkin NV, et al. Autoubiquitination of the 26S proteasome on Rpn13 regulates breakdown of ubiquitin conjugates. EMBO J, 2014, 33(10): 1159-1176. DOI:10.1002/embj.201386906 |
| [13] |
Tokunaga F, Sakata SI, Saeki Y, et al. Involvement of linear polyubiquitylation of NEMO in NF-κB activation. Nat Cell Biol, 2009, 11(2): 123-132. DOI:10.1038/ncb1821 |
| [14] |
Alfano C, Faggiano S, Pastore A. The ball and chain of polyubiquitin structures. Trends Biochem Sci, 2016, 41(4): 371-385. DOI:10.1016/j.tibs.2016.01.006 |
| [15] |
Castañ eda CA, Dixon EK, Walker O, et al. Linkage via K27 bestows ubiquitin chains with unique properties among polyubiquitins. Structure, 2016, 24(3): 423-436. DOI:10.1016/j.str.2016.01.007 |
| [16] |
Faggiano S, Alfano C, Pastore A. The missing links to link ubiquitin: Methods for the enzymatic production of polyubiquitin chains. Anal Biochem, 2016, 492: 82-90. DOI:10.1016/j.ab.2015.09.013 |
| [17] |
Kumar KSA, Spasser L, Erlich LA, et al. Total chemical synthesis of di-ubiquitin chains. Angew Chem Int Ed Engl, 2010, 49(48): 9126-9131. DOI:10.1002/anie.201003763 |
| [18] |
Pan M, Gao S, Zheng Y, et al. Quasi-racemic X-ray structures of K27-linked ubiquitin chains prepared by total chemical synthesis. J Am Chem Soc, 2016, 138(23): 7429-7435. DOI:10.1021/jacs.6b04031 |
| [19] |
Pan M, Zheng QY, Ding S, et al. Chemical protein synthesis enabled mechanistic studies on the molecular recognition of K27-linked ubiquitin chains. Angew Chem Int Ed, 2019, 58(9): 2627-2631. DOI:10.1002/anie.201810814 |
| [20] |
Varadan R, Assfalg M, Raasi S, et al. Structural determinants for selective recognition of a Lys48-linked polyubiquitin chain by a UBA domain. Mol Cell, 2005, 18(6): 687-698. DOI:10.1016/j.molcel.2005.05.013 |
| [21] |
Husnjak K, Dikic I. Ubiquitin-binding proteins: decoders of ubiquitin-mediated cellular functions. Annu Rev Biochem, 2012, 81: 291-322. DOI:10.1146/annurev-biochem-051810-094654 |
| [22] |
Zhang XF, Smits AH, Van Tilburg GBA, et al. An interaction landscape of ubiquitin signaling. Mol Cell, 2017, 65(5): 941-955. DOI:10.1016/j.molcel.2017.01.004 |
| [23] |
Zhao XH, Lutz J, Hö llmüller E, et al. Identification of proteins interacting with ubiquitin chains. Angew Chem Int Ed, 2017, 56(49): 15764-15768. DOI:10.1002/anie.201705898 |
| [24] |
Fushman D, Wilkinson KD. Structure and recognition of polyubiquitin chains of different lengths and linkage. F1000 Biol Rep, 2011, 3: 26. |
| [25] |
Clague MJS Urbe, Komander D. Breaking the chains: deubiquitylating enzyme specificity begets function. Nat Rev Mol Cell Biol, 2019, 20(6): 338-352. DOI:10.1038/s41580-019-0099-1 |
| [26] |
Tan XD, Pan M, Gao S, et al. A diubiquitin-based photoaffinity probe for profiling K27-linkage targeting deubiquitinases. Chem Commun (Camb), 2017, 53(73): 10208-10211. DOI:10.1039/C7CC05504H |
| [27] |
Durcan TM, Kontogiannea M, Thorarinsdottir T, et al. The Machado-Joseph disease-associated mutant form of ataxin-3 regulates parkin ubiquitination and stability. Hum Mol Genet, 2011, 20(1): 141-154. DOI:10.1093/hmg/ddq452 |
| [28] |
Sun H, Zhang Q, Jing YY, et al. USP13 negatively regulates antiviral responses by deubiquitinating STING. Nat Commun, 2017, 8: 15534. DOI:10.1038/ncomms15534 |
| [29] |
Sparrer KMJ, Gableske S, Zurenski MA, et al. TRIM23 mediates virus-induced autophagy via activation of TBK1. Nat Microbiol, 2017, 2(11): 1543-1557. DOI:10.1038/s41564-017-0017-2 |
| [30] |
Jin SH, Tian S, Luo M, et al. Tetherin suppresses Type I interferon signaling by targeting MAVS for NDP52-mediated selective autophagic degradation in human cells. Mol Cell, 2017, 68(2): 308-322. DOI:10.1016/j.molcel.2017.09.005 |
| [31] |
Xue BB, Li HY, Guo MM, et al. TRIM21 promotes innate immune response to RNA viral infection through Lys27-linked polyubiquitination of MAVS. J Virol, 2018, 92(14): e00321-18. DOI:10.1128/JVI.00321-18 |
| [32] |
Liu H, Li M, Song YH, et al. TRIM21 restricts coxsackievirus B3 replication, cardiac and pancreatic injury via interacting with mavs and positively regulating IRF3-mediated type-i interferon production. Front Immunol, 2018, 9: 2479. DOI:10.3389/fimmu.2018.02479 |
| [33] |
Zhao CY, Jia MT, Song H, et al. The E3 ubiquitin ligase TRIM40 attenuates antiviral immune responses by targeting MDA5 and RIG-I. Cell Rep, 2017, 21(6): 1613-1623. DOI:10.1016/j.celrep.2017.10.020 |
| [34] |
Pei G, Buijze H, Liu HP, et al. The E3 ubiquitin ligase NEDD4 enhances killing of membrane-perturbing intracellular bacteria by promoting autophagy. Autophagy, 2017, 13(12): 2041-2055. DOI:10.1080/15548627.2017.1376160 |
| [35] |
Huett A, Heath RJ, Begun J, et al. The LRR and RING domain protein LRSAM1 is an E3 ligase crucial for ubiquitin-dependent autophagy of intracellular Salmonella Typhimurium. Cell Host Microb, 2012, 12(6): 778-790. DOI:10.1016/j.chom.2012.10.019 |
| [36] |
Celli J. LRSAM1, an E3 Ubiquitin ligase with a sense for bacteria. Cell Host Microbe, 2012, 12(6): 735-736. DOI:10.1016/j.chom.2012.11.007 |
| [37] |
Sheng XP, You Q, Zhu HN, et al. Bacterial effector NleL promotes enterohemorrhagic E. coli-induced attaching and effacing lesions by ubiquitylating and inactivating JNK. PLoS Pathog, 2017, 13(7): e1006534. DOI:10.1371/journal.ppat.1006534 |
| [38] |
Wang CY, Fu JQ, Wang M, et al. Bartonella quintana type IV secretion effector BepE-induced selective autophagy by conjugation with K63 polyubiquitin chain. Cell Microbiol, 2019, 21(4): e12984. DOI:10.1111/cmi.12984 |
| [39] |
Arimoto KI, Funami K, Saeki Y, et al. Polyubiquitin conjugation to NEMO by triparite motif protein 23 (TRIM23) is critical in antiviral defense. Proc Natl Acad Sci USA, 2010, 107(36): 15856-15861. DOI:10.1073/pnas.1004621107 |
| [40] |
Liu J, Han CF, Xie B, et al. Rhbdd3 controls autoimmunity by suppressing the production of IL-6 by dendritic cells via K27-linked ubiquitination of the regulator NEMO. Nat Immunol, 2014, 15(7): 612-622. DOI:10.1038/ni.2898 |
| [41] |
Wang Q, Huang LY, Hong Z, et al. The E3 ubiquitin ligase RNF185 facilitates the cGAS-mediated innate immune response. PLoS Pathog, 2017, 13(3): e1006264. DOI:10.1371/journal.ppat.1006264 |
| [42] |
Wang Q, Liu X, Cui Y, et al. The E3 ubiquitin ligase AMFR and INSIG1 bridge the activation of TBK1 kinase by modifying the adaptor STING. Immunity, 2014, 41(6): 919-933. DOI:10.1016/j.immuni.2014.11.011 |
| [43] |
Cao ZF, Conway KL, Heath RJ, et al. Ubiquitin ligase TRIM62 regulates CARD9-mediated anti-fungal immunity and intestinal inflammation. Immunity, 2015, 43(4): 715-726. DOI:10.1016/j.immuni.2015.10.005 |
| [44] |
Cho JJ, Xu ZW, Parthasarathy U, et al. Hectd3 promotes pathogenic Th17 lineage through Stat3 activation and Malt1 signaling in neuroinflammation. Nat Commun, 2019, 10: 701. DOI:10.1038/s41467-019-08605-3 |
| [45] |
Ashida H, Kim M, Schmidt-Supprian M, et al. A bacterial E3 ubiquitin ligase IpaH9.8 targets NEMO/IKKgamma to dampen the host NF-kappaB-mediated inflammatory response. Nat Cell Biol, 2010, 12(1): 66-73. DOI:10.1038/ncb2006 |
| [46] |
Liu H, Zhang LL, Sun J, et al. Endoplasmic reticulum protein SCAP inhibits dengue virus NS2B3 protease by suppressing its K27-linked polyubiquitylation. J Virol, 2017, 91(9): e02234-16. DOI:10.1128/JVI.02234-16 |
| [47] |
Brown JS, Jackson SP. Ubiquitylation, neddylation and the DNA damage response. Open Biol, 2015, 5(4): 150018. DOI:10.1098/rsob.150018 |
| [48] |
Gatti M, Pinato S, Maiolica A, et al. RNF168 promotes noncanonical K27 ubiquitination to signal DNA damage. Cell Rep, 2015, 10(2): 226-238. DOI:10.1016/j.celrep.2014.12.021 |
| [49] |
Walden H, Muqit MMK. Ubiquitin and Parkinson's disease through the looking glass of genetics. Biochem J, 2017, 474(9): 1439-1451. DOI:10.1042/BCJ20160498 |
| [50] |
Zucchelli S, Codrich M, Marcuzzi F, et al. TRAF6 promotes atypical ubiquitination of mutant DJ-1 and alpha-synuclein and is localized to Lewy bodies in sporadic Parkinson's disease brains. Hum Mol Genet, 2010, 19(19): 3759-3770. DOI:10.1093/hmg/ddq290 |
| [51] |
Zucchelli S, Marcuzzi F, Codrich M, et al. Tumor necrosis factor receptor-associated factor 6 (TRAF6) associates with huntingtin protein and promotes its atypical ubiquitination to enhance aggregate formation. J Biol Chem, 2011, 286(28): 25108-25117. DOI:10.1074/jbc.M110.187591 |
| [52] |
Nucifora FC Jr, Nucifora LG, Ng CH, et al. Ubiqutination via K27 and K29 chains signals aggregation and neuronal protection of LRRK2 by WSB1. Nat Commun, 2016, 7: 11792. DOI:10.1038/ncomms11792 |
| [53] |
Cookson MR. The biochemistry of Parkinson's disease. Annu Rev Biochem, 2005, 74: 29-52. DOI:10.1146/annurev.biochem.74.082803.133400 |
| [54] |
Ashrafi G, Schwarz TL. The pathways of mitophagy for quality control and clearance of mitochondria. Cell Death Differ, 2013, 20(1): 31-42. DOI:10.1038/cdd.2012.81 |
| [55] |
Geisler S, Holmströ m KM, Skujat D, et al. PINK1/Parkin-mediated mitophagy is dependent on VDAC1 and p62/SQSTM1. Nat Cell Biol, 2010, 12(2): 119-131. DOI:10.1038/ncb2012 |
| [56] |
Birsa N, Norkett R, Wauer T, et al. Lysine 27 ubiquitination of the mitochondrial transport protein Miro is dependent on serine 65 of the Parkin ubiquitin ligase. J Biol Chem, 2014, 289(21): 14569-14582. DOI:10.1074/jbc.M114.563031 |
| [57] |
Hanahan D, Weinberg RA. The hallmarks of cancer. Cell, 2000, 100(1): 57-70. DOI:10.1016/S0092-8674(00)81683-9 |
| [58] |
Hanahan D, Weinberg RA. Hallmarks of cancer: the next generation. Cell, 2011, 144(5): 646-674. DOI:10.1016/j.cell.2011.02.013 |
| [59] |
Friedl P, Alexander S. Cancer invasion and the microenvironment: plasticity and reciprocity. Cell, 2011, 147(5): 992-1009. DOI:10.1016/j.cell.2011.11.016 |
| [60] |
El-Hachem N, Habel N, Naiken T, et al. Uncovering and deciphering the pro-invasive role of HACE1 in melanoma cells. Cell Death Differ, 2018, 25(11): 2010-2022. DOI:10.1038/s41418-018-0090-y |
| [61] |
Kong YJ, Wang ZH, Huang MB, et al. CUL7 promotes cancer cell survival through promoting Caspase-8 ubiquitination. Int J Cancer, 2019, 145(5): 1371-1381. DOI:10.1002/ijc.32239 |
| [62] |
Singh R, Karri D, Shen H, et al. TRAF4-mediated ubiquitination of NGF receptor TrkA regulates prostate cancer metastasis. J Clin Invest, 2018, 128(7): 3129-3143. DOI:10.1172/JCI96060 |
| [63] |
Xia T, Lévy L, Levillayer F, et al. The four and a half LIM-only protein 2 (FHL2) activates transforming growth factor β (TGF-β) signaling by regulating ubiquitination of the E3 ligase Arkadia. J Biol Chem, 2013, 288(3): 1785-1794. DOI:10.1074/jbc.M112.439760 |
| [64] |
Palicharla VR, Maddika S. HACE1 mediated K27 ubiquitin linkage leads to YB-1 protein secretion. Cell Signal, 2015, 27(12): 2355-2362. DOI:10.1016/j.cellsig.2015.09.001 |
| [65] |
Ikeda H, Kerppola TK. Lysosomal localization of ubiquitinated Jun requires multiple determinants in a lysine-27-linked polyubiquitin conjugate. Mol Biol Cell, 2008, 19(11): 4588-4601. DOI:10.1091/mbc.e08-05-0496 |
| [66] |
Xu WM, Liu LZ, Hornby D. c-IAP1 binds and processes PCSK9 protein: linking the c-IAP1 in a TNF-α pathway to PCSK9-mediated LDLR degradation pathway. Molecules, 2012, 17(10): 12086-12101. DOI:10.3390/molecules171012086 |
| [67] |
Gao Y, Li YC, Zhang CP, et al. Enhanced purification of ubiquitinated proteins by engineered tandem hybrid ubiquitin-binding domains (ThUBDs). Mol Cell Proteomics, 2016, 15(4): 1381-1396. DOI:10.1074/mcp.O115.051839 |
 2020, Vol. 36
2020, Vol. 36




