中国科学院微生物研究所、中国微生物学会主办
文章信息
- 彭稳, 何培民, 施定基, 贾睿
- Peng Wen, He Peimin, Shi Dingji, Jia Rui
- 橙色荧光蛋白的研究进展及其应用
- Advances in the research and applications of orange fluorescent protein
- 生物工程学报, 2020, 36(6): 1060-1068
- Chinese Journal of Biotechnology, 2020, 36(6): 1060-1068
- 10.13345/j.cjb.190465
-
文章历史
- Received: September 30, 2019
- Accepted: December 4, 2019
2. 中国科学院植物研究所,北京 100093
2. Institute of Botany, Chinese Academy of Sciences, Beijing 100093, China
细胞生物学的研究需要对活细胞内的各种代谢过程和细胞通路进行详细、准确的描述。各种荧光染料由于其毒性所以不能在活体细胞中很好地使用,而荧光蛋白(Fluorescent protein,FPs)可以在光谱成像中显示出明亮的荧光[1],在活细胞、深层组织和全身的成像容易[2],也就成为更好的替代品。随着荧光显微技术的进步,FPs可作为探针以跟踪细胞内分子间相互作用,追踪特定代谢产物的代谢途径,也可以结合到传感器中将细胞活动转化为生物信号[3-4]。
1962年,下村修等在维多利亚管状水母Aequorea victoria体内首次分离得到绿色FPs[5]。此后绿色FPs在生物学领域得到了广泛运用,但是其发射光谱仅仅局限于440–529 nm,细胞内成像背景较高,且不能解决皮下深层组织的成像问题[6]。1999年,Matz等从印度洋-太平洋地区的珊瑚虫(Discosoma sp.)中分离并改进得到红色FPs——DsRed[7],但由于其四聚体的特性限制了其在生物技术领域中的应用[8-9]。
近年来,蛋白质工程学已经成功地将天然荧光蛋白的激发光谱扩展到整个可见光波段甚至远红外波长,从而产生了一系列荧光蛋白[10]。当使用FPs时,通常需要同时成像多个光谱通道的图像以分析多种生物活动,或在定量研究中使用一个荧光团作为参照[11],所以丰富多样的荧光蛋白对生物学研究的推动作用显而易见。橙色荧光蛋白的出现使荧光蛋白家族的光谱范围进一步完善,它具有与常用的红色以及绿色荧光蛋白显著差异的发射波峰,使研究者在进行多荧光标记等实验时具有更多样的选择。同时它具有在显微镜下细胞自身荧光背景较暗、光散射较少、可用于活体组织成像、发射波长较长等优势。因此,现在更多的研究集中在橙色FPs的开发中。
1 橙色荧光蛋白的种类及其特点 1.1 常规橙色荧光蛋白目前报道的常规橙色FPs主要来源于两种亲本蛋白:Kusabira-Orange (KO)[12]、DsRed[13]。KO最初是从石质珊瑚Fungia concinna中分离出来的,通过在其N端引入10个氨基酸残基从而可为蛋白质提供明亮的橙色荧光[12],KO激发和发射峰分别为548 nm和561 nm [14]。但由于KO是四聚体结构,后来又通过引入另外22个氨基酸突变得到了一个单分子变体mKO,其保持了原有的亮度和荧光特性,最大发射峰略有变化(559 nm),分子量为28.1 kDa[14]。mKO被进一步改进,产生两个亮度更高的变体(mKO2[15]和mKOk[16])。
DsRed衍生物中橙色FPs最多,共有8种,Shaner等在DsRed的单分子变体mRFP1[17]基础上改进得到了mHoneydew、mBanana和mOrange[18]。mOrange的亮度比同为DsRed衍生物的mCherry[18]要高出3倍以上。虽然mOrange的亮度较高,但它对酸的敏感性也非常高,pKa值为6.5。mOrange的高消光系数和高量子产率使其成为潜在的荧光共振能量转移技术(Fluorescence resonance energy transfer,FRET)受体。随后,Shaner等又在mOrange的基础上,开发出mOrange2[19],其光稳定性是mOrange的25倍以上。此外,Strack等通过改造DsRed获得一种低细胞毒性的四聚体红色FPs——DsRed-Express 2[9, 20],并以此蛋白作为模板,在保留其低细胞毒性的基础上创造了橙色变体E2-Orange。
1.2 具有特殊表型的荧光蛋白此外,还有一些具有独特表型的橙色FPs,具有大斯托克位移(Large stoker shift,LSS)的橙色FPs有两种:LSSmOrange[21]以及亮度更高的CyOFP1[22],LSSmOrange是在mOrange的基础上突变得到的变体,CyOFP1是在mNeptune2的基础上定向突变得到,具有激发光为绿色以及高亮度的特点。除此之外还有具有从橙色到远红色荧光态的光转化特性的FPsPSmOrange和PSmOrange2[23]。
另外,在FPs发色团中还有一种被称为光敏剂的发色团,这种发色团被光照射后能够产生活性氧(ROS),进而灭活目的蛋白,即发色基团辅助光失活(Chromophore-assisted light inactivation,CALI)技术[29-30],对DNA、蛋白质、脂肪等造成损伤,影响细胞的代谢或增殖,诱导细胞死亡[31]。Sarkisyan等报道了一种红色FPsKillerRed的橙色突变体KillerOrange,当其被蓝光或绿光照射时,它会产生ROS从而杀伤细胞[32],所有荧光蛋白特性罗列在表 1中。
| Fluorescent protein | Excitation maximum (nm) | Excitation maximum (nm) | Extinction coefficient per chain (L/(mol·cm)) | Fluorescence quantum yield | Brightness of fully mature protein (% of EGFP) | Reference |
| EGFP* | 489 | 5 300 | 5 300 | 0.60 | 1.00 | [24] |
| KO | 548 | 561 | 109 750 | 0.45 | 1.55 | [12] |
| mKO | 548 | 559 | 51 600 | 0.60 | 0.97 | [12] |
| mKO2 | 551 | 565 | 63 800 | 0.57 | 1.14 | [15] |
| mKOK | 551 | 563 | 10 500 | 0.61 | 2.01 | [16] |
| DsRed* | 558 | 583 | 75 000 | 0.79 | 1.65 | [13] |
| mHoneydew | 504 | 562 | 17 000 | 0.12 | 0.06 | [18] |
| mBanana | 540 | 553 | 6 000 | 0.70 | 0.13 | [18] |
| mOrange | 548 | 562 | 71 000 | 0.70 | 1.54 | [18] |
| mOrange2 | 549 | 565 | 58 000 | 0.60 | 1.09 | [19] |
| LSSmOrange | 437 | 572 | 52 000 | 0.45 | 0.74 | [25] |
| CyOFP1 | 523 | 589 | 40 000 | 0.76 | 1.20 | [23] |
| PSmOrange | 548/636 | 565/662 | 113 300/32 700 | 0.51/0.28 | 1.82 | [26] |
| PSmOrange2 | 546/619 | 561/651 | 51 000/18 900 | 0.61/0.38 | 0.98 | [27] |
| DsRed-Express2* | 554 | 591 | 35 600 | 0.42 | 0.47 | [20] |
| E2-Orange | 540 | 561 | 36 500 | 0.54 | 0.62 | [28] |
| *EGFP, DsRed, DsRed-express2 are not orange FPs, only used as a control. | ||||||
大部分橙色荧光蛋白具有相似的β-桶状的结构。以mKO为例(图 1),11个β-折叠链组成β-筒的外周结构,筒两端分别被一些分子量较小的短α-螺旋覆盖,组成发色团的3个氨基酸残基与α-螺旋共价相连,位于圆筒中央螺旋中部。β-圆筒与短α-螺旋形成致密的结构域,使配体不能扩散进入,发色团被严格保护在筒内,从而形成稳定的结构,不易猝灭[33]。
2.2 发色团的结构通过研究,FPs发色团自成熟和光诱导化学的基本原理已经相对清楚[34]。FPs的光谱特性是由其发色团的结构决定的,橙色荧光蛋白的发色团由3个氨基酸残基组成,后两个较为保守,一般为酪氨酸和甘氨酸,第一位的氨基酸变化最大,也是此位氨基酸的侧链发生环化,形成特殊的三环发色团结构,从而扩展电子共轭体系为荧光蛋白提供橙色的荧光[18]。
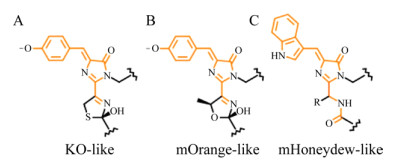
mKO和KO具有相同的发色团结构,发色团都由C-Y-G三肽组成(图 2A),Cys发生环化形成第3个环,扩展了显色团共轭体系[35]。DsRed的发色团由Q-Y-G组成,连接发色团的咪唑啉酮环和前一位氨基酸残基的酰基亚胺基团扩展了共轭体系从而导致了红色的荧光,DsRed中发色团的氨基酸残基的改变产生了3种橙色FPs:mHoney-dew (M-W-G)、mBanana (C-Y-G)和mOrange (T-Y-G)。在mOrange中,位于66位的Gln被Thr取代,从而形成色团中第3个杂环(恶唑环),导致橙色的光谱特性(图 2B)[7, 34]。在DsRed-Express2的橙色衍生物E2Orange的设计中,将相同的Q66T突变体引入DsRed-Express2中,从而产生了橙色的激发和发射峰。值得一提的是mHoneydew的发色团的结构很特别,与其他DsRed衍生物不同,它的发色团中的Tyr被取代为Trp (图 2C),但是这个特殊的结构并没有给它带来良好的荧光表现,它的荧光强度只有DsRed的3%,包括同为衍生物的mBanana,它们的特性都不是很良好,所以应用的实例也较少。
LSSmOrange、PSmOrange和PSmOrange2与亲本mOrange具有相同的显色团结构。它由对羟基苯基、咪唑啉酮和由T66侧链环化的2-羟基二氢恶唑环组成。在LSSmOrange中,大斯托克位移的产生源于分子内部的激发态质子转移[21],而包括I161D、M163L和W143M在内的关键突变对发色团周围环境造成显著影响从而为激发态质子转移的发生提供条件(图 3)。W143M置换使L165侧链向蛋白的内部转移,W143M和M163L均使发色团的结构趋向扁平,由这两个突变引起S146侧链构象的改变,从而使S146可以作为从发色团的对羟基苯环到D161的羧基之间激发态质子转移的中间传递体[36]。与LSSmOrange不同,CyOFP1的大斯托克位移源于K160与发色团的酚基团之间的氢键,激发态质子从发色团传递到K160的侧链从而完成激发态质子的转移[22]。
在PSmOrange和PSmOrange2中,Q64L突变对于其光转换特性至关重要。这种突变破坏了64位谷氨酰胺和95位精氨酸之间的氢键,Y99侧链羧基与I60主链羰基形成新的水介导的氢键。这两种变化导致的蛋白质可以被氧化剂或光刺激,从而转化为远红外的发光状态(图 4)。光转换伴随着65位Phe/Ile (PSmOrange/PSmOrange2)上Cα1-C键的断裂,使畸变的显色团恢复,并将2-羟基二氢恶唑氧OH-基团转化为C=O,使发色团共轭体系得到扩展。这是PSmOrange和PSmOrange2中大量(约90 nm)荧光红移的主要原因。PSmOrange2中A217S的取代使215位Glu向发色团靠近,并使其与发色团形成氢键,增加了发色团的极化和量子产率[36]。
KillerOrange以及mKillerOrange是KillerRed的突变体,三肽结构为Q-W-G,Trp替换KillerRed发色团中的Tyr,在这两种FPs中,发色团的第66位Trp与第159位Gln形成氢键,以维持一种不寻常的反式-顺式结构。这种顺反式构象与沿着β-桶轴从发色团延伸到筒体末端的充水通道是关键的结构特征,这两者为两种光敏剂提供了明亮的橙色荧光和光毒性[37]。
3 橙色荧光蛋白的应用 3.1 橙色荧光蛋白作为标记蛋白的应用橙色荧光蛋白所在的发射光波段,在某些特殊细胞中可以有效避开细胞自身的背景荧光。除此之外,橙色荧光在哺乳动物的深部组织成像优势巨大,因为相关组织对橙光及红光的通透性较好。某些变体自身的优异特性也使其成为标记蛋白的良好选择,比如大斯托克位移FPs在多荧光标记中的应用。
3.1.1 在微藻及植物中的应用蓝藻作为可以用于生物燃料和化学合成应用的模式生物越来越受到关注[38],蓝藻自身携带大量的光合色素,当对蓝藻进行荧光标记时,这些光合色素会与FPs竞争吸收激发光源,吸收FPs发射的荧光,并且放出自身荧光信号干扰检测,所以选择合适的标记蛋白对蓝藻的荧光标记至关重要。Ruffing等选择mOrange作为标记蛋白,探索其在Synechococcus sp. PCC7002中的标记性能,结果显示可以在光谱图上区分转基因型和野生型蓝藻。这为使用mOrange作为蓝藻生物中的标记蛋白提供参考和依据[39]。之前的研究也表明,橙色和红色FPs适合莱茵衣藻Chlamydomonas reinhardtii[40]的胞质表达。
除此之外,橙色FPs也在植物中作为标记蛋白来使用。Mann等将mOrange等6种FPs基因分别转入烟草和拟南芥中进行表达。结果显示橙色荧光蛋白可以用于植物转化[41]。在豆科植物中,过多的根瘤形成会抑制植物生长。植物激素脱落酸(ABA)可以抑制根瘤的形成,ljglu1基因是一种ABA反应基因。Osuki使用mOrange标记LjGlu1蛋白,从而观察其在根组织和结节中的定位[42],表明LjGlu1的表达是植物自身对根瘤菌感染作出的应激反应,并在根组织外发挥作用,从而抑制感染。发动蛋白和网格蛋白参与植物包膜囊泡的形成,Fujimoto等用GFP和mKO分别标记参与其中的发动蛋白以及网格蛋白,根据它们的聚集状况从而观察它们在植物内吞过程中的分子机制[43]。
3.1.2 在哺乳动物细胞中的应用Ⅱ型糖尿病是一种非胰岛素依赖型糖尿病,葡萄糖转运体4 (GLUT4)是将葡萄糖转运入细胞的主要蛋白之一,是治疗Ⅱ型糖尿病的靶点,其广泛分布于骨骼肌、心肌、脂肪组织、肾脏和大脑[44]。Zhao等通过构建融合表达mOrange和GLUT4的细胞株系,从而观察细胞内GLUT4动态变化及其与质膜融合状况[45]。在鸟类和哺乳动物的皮肤色素沉着过程中,黑色素在黑素细胞中合成,然后转移到邻近的角质形成细胞中,使含有黑色素的细胞覆盖全身。Murai等用EGFP和mOrange分别标记黑素细胞以及角质形成细胞,探索两种细胞间的相互作用[46],以了解细胞间相互作用机制以及体内皮肤色素沉着规律。
值得一提的是,CyOFP1的激发光为绿色,发射光为橙色,其开发者Chu等[22]利用这一特性,将其与绿色荧光蛋白分别标记到黑素瘤细胞的不同部位,从而通过单一的激发光源同时观察两个光通路的蛋白标记状况,这节省了布置双激光发射器的成本。
3.1.3 在病毒中的应用Etienne等将包括mOrange在内的多种FPs融合到单纯疱疹病毒1型(HSV-1)菌株的小衣壳蛋白(VP26)上,并且进入人黑色素瘤细胞进行扩增,从而探究病毒在体内的感染机制[47]。Chen等在竹花叶病毒(BaMV)感染宿主的过程中发现了一个上调的基因,该基因编码的蛋白包括一个环状结构与一个跨膜结构域,该蛋白可能具有泛素E3连接酶的功能,并将其命名为泛素E3连接酶(NbUbE3R1)。实验者将其与橙色FPs融合表达,进一步研究NbUbE3R1在BaMV积累中的功能作用[48]。
3.2 橙色荧光蛋白在FRET技术中的应用基于荧光寿命成像显微镜(FLIM)的荧光共振能量转移(FRET)测量正越来越多地用于探索生命系统中的分子构象和分子间相互作用。在适当位置的受体存在的情况下,可以根据供体荧光团的兴奋状态时间的缩短来判断其标记的蛋白质之间的分子构象。传统的青色FPs由于其耐光性差、低量子产量等缺点在荧光共振成像中的应用还达不到理想的效果[49],mOrange、mKO等由于其较高的消光系数和量子产率使其具有成为FRET受体的潜力,有望成为优秀的FRET。近几年来使用mOrange作为荧光受体的研究越来越多,为研究细胞内生物大分子相互作用以及探究生命过程提供有效工具。
Ke等通过将mOrange连接到哈氏弧菌荧光酶素α亚基的N端,从而使该融合蛋白获得了新的560 nm的发射峰。这为通过将荧光素酶与其他合适的FPs或发色团共价结合来改变细菌的生物发光颜色提供思路[50]。De等开发一种结合mOrange和荧光酶素的自发光融合蛋白(BRET3),新的融合蛋白在光强上提升了几倍,且能解决体内荧光成像等相关问题[51]。这两种应用都是将荧光酶素与FPs结合使用,利用荧光酶素本身利用生物底物发光的性质以及橙色FPs的高消光系数和高量子产率的优势进行创新的荧光标记手段,具有巨大的应用前景,可以为后人的实验设计提供新的思路。
植物依靠肌动蛋白细胞骨架来驱动基本的细胞活动,这对它们的正常生长和发育至关重要,包括与细胞分裂、细胞膨胀和膜重构等相关的生命活动[52]。Bayle等报道了在完整植物细胞中使用绿色FPs突变体TSapphire作为mOrange的荧光供体的应用。嵌合连接的TSapphire和mOrange之间发生大量的能量转移,导致TSapphire荧光寿命缩短。并且验证了该系统在不同的亚细胞中的通用性[49]。
视网膜鸟苷酰基环化酶激活蛋白(GCAPs)可以调节人类的视觉光反应。Peshenko等对整个GCAP1蛋白表面残基进行诱导突变,并用mOrange标记,在细胞基础上测试每个突变体与GFP标记的视网膜鸟苷酰基环化酶的体外结合以及激活能力,从而探索GCAPs与其靶酶相互作用的关键位点[53]。研究发现Orai1蛋白以及STIM1蛋白构建的Ca2+储存通道在Ca2+进入及信号传递中发挥重要作用[54]。Huang等用EGFP标记Orai1作为荧光供体,mOrange标记STIM1作为荧光受体在大鼠神经细胞中表达,探索两者之间的相互作用[55]。肌动蛋白细胞骨架参与多种生物过程,包括T细胞活化[56]。Larbret等将EGFP和mOrange分别融合到T细胞株的肌动蛋白上,当游离的G肌动蛋白互相连接组成F肌动蛋白时,靠近的EGFP和mOrange之间发生能量转移,从而观测活细胞中G肌动蛋白和F肌动蛋白之间的动态平衡[57]。Goedhart等构建mKO-mCherry FRET受体供体对,用于检测活细胞中NF-kB转录因子复合物亚族p65的同源二聚化,具有比CFP-YFP受体供体对更高的FRET效率[1]。表明在单个活细胞中,基于红色FPs的FRET方法更适合于检测蛋白质之间的相互作用。
4 总结与展望本文主要介绍了橙色FPs的研究和发展,以及其在蛋白标记、FRET、生物传感器等方面的运用。随着对FPs晶体结构研究的深入,人们大大提高了对于荧光发色团的认识。通过对一些优秀突变体的结构分析,我们了解了一些能够引起FPs性质改变的关键位点。结合目前现有的橙色FPs以及待发现的野生型橙色FPs,相信可以设计和研发出更多可应用的突变体。现有的常规橙色FPs如mOrange、mKO具有较高的消光性质和量子产率,在FRET中有巨大的应用前景。除此之外还有很多创新的应用,比如FPs与生物荧光酶素的融合表达也趋于流行,以及大斯托克位移的FPs如CyOFP1在多荧光标记中的应用。有理由相信,橙色FPs将会与众多FPs一起在未来的生物学研究中起到重要作用。
| [1] |
Okumoto S. Imaging approach for monitoring cellular metabolites and ions using genetically encoded biosensors. Curr Opin Biotechnol, 2010, 21(1): 45-54. DOI:10.1016/j.copbio.2010.01.009 |
| [2] |
Kremers GJ, Gilbert SG, Cranfill PJ, et al. Fluorescent proteins at a glance. J Cell Sci, 2011, 124(15): 2676. DOI:10.1242/jcs.095059 |
| [3] |
Zhang J, Campbell RE, Ting AY, et al. Creating new fluorescent probes for cell biology. Nat Rev Mol Cell Biol, 2002, 3(12): 906-918. DOI:10.1038/nrm976 |
| [4] |
van Roessel P, Brand AH. Imaging into the future: visualizing gene expression and protein interactions with fluorescent proteins. Nat Cell Biol, 2002, 4(1): E15-E20. DOI:10.1038/ncb0102-e15 |
| [5] |
Shimomura O, Johnson FH, Saiga Y. Extraction, purification and properties of aequorin, a bioluminescent protein from the luminous hydromedusan, Aequorea. J Cell Comp Physiol, 1962, 59(3): 223-239. DOI:10.1002/jcp.1030590302 |
| [6] |
Wang F, Yang HT, Wang ZF. Research progress on red fluorescent protein. Biotechnol Bull, 2017, 33(9): 32-47 (in Chinese). 王飞, 杨海涛, 王泽方. 红色荧光蛋白的研究进展. 生物技术通报, 2017, 33(9): 32-47. |
| [7] |
Matz MV. Erratum: fluorescent proteins from nonbioluminescent Anthozoa species. Nat Biotechnol, 1999, 17(12): 1227. DOI:10.1038/70787 |
| [8] |
Tao W, Evans BG, Yao J, et al. Enhanced green fluorescent protein is a nearly ideal long-term expression tracer for hematopoietic stem cells, whereas DsRed-Express fluorescent protein is not. Stem Cells, 2007, 25(3): 670-678. |
| [9] |
Strack RL, Strongin DE, Bhattacharyya D, et al. A noncytotoxic DsRed variant for whole-cell labeling. Nat Methods, 2008, 5(11): 955-957. DOI:10.1038/nmeth.1264 |
| [10] |
Ng HL, Lin MZ. Structure-guided wavelength tuning in far-red fluorescent proteins. Curr Opin Struct Biol, 2016, 39: 124-133. DOI:10.1016/j.sbi.2016.07.010 |
| [11] |
Depry C, Mehta S, Zhang J. Multiplexed visualization of dynamic signaling networks using genetically encoded fluorescent protein-based biosensors. Pflugers Archiv-Eur J Physiol, 2013, 465(3): 373-381. DOI:10.1007/s00424-012-1175-y |
| [12] |
Karasawa S, Araki T, Nagai T, et al. Cyan-emitting and orange-emitting fluorescent proteins as a donor/acceptor pair for fluorescence resonance energy transfer. Biochem J, 2004, 381(1): 307-312. |
| [13] |
Yarbrough D, Wachter RM, Kallio K, et al. Remington SJ. Refined crystal structure of DsRed, a red fluorescent protein from coral, at 2.0-Å resolution. Proc Natl Acad Sci USA, 2001, 98(2): 462-467. DOI:10.1073/pnas.98.2.462 |
| [14] |
Merzlyak EM, Goedhart J, Shcherbo D, et al. Bright monomeric red fluorescent protein with an extended fluorescence lifetime. Nat Methods, 2007, 4(7): 555-557. DOI:10.1038/nmeth1062 |
| [15] |
Sakaue-Sawano A, Kurokawa H, Morimura T, et al. Visualizing spatiotemporal dynamics of multicellular cell-cycle progression. Cell, 2008, 132(3): 487-498. DOI:10.1016/j.cell.2007.12.033 |
| [16] |
Tsutsui H, Karasawa S, Okamura Y, et al. Improving membrane voltage measurements using FRET with new fluorescent proteins. Nat Methods, 2008, 5(8): 683-685. DOI:10.1038/nmeth.1235 |
| [17] |
Campbell RE, Tour O, Palmer AE, et al. A monomeric red fluorescent protein. Proc Natl Acad Sci USA, 2002, 99(12): 7877-7882. DOI:10.1073/pnas.082243699 |
| [18] |
Shaner NC, Campbell RE, Steinbach PA, et al. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein. Nat Biotechnol, 2004, 22(12): 1567-1572. DOI:10.1038/nbt1037 |
| [19] |
Shaner NC, Lin MZ, McKeown MR, et al. Improving the photostability of bright monomeric orange and red fluorescent proteins. Nat Methods, 2008, 5(6): 545-551. DOI:10.1038/nmeth.1209 |
| [20] |
Strack RL, Hein B, Bhattacharyya D, et al. A rapidly maturing far-red derivative of dsred-express2 for whole-cell labeling. Biochemistry, 2009, 48(35): 8279-8281. DOI:10.1021/bi900870u |
| [21] |
Shcherbakova DM, Hink MA, Joosen L, et al. An orange fluorescent protein with a large stokes shift for single-excitation multicolor FCCS and FRET imaging. J Am Chem Soc, 2012, 134(18): 7913-7923. DOI:10.1021/ja3018972 |
| [22] |
Chu J, Oh YH, Sens A, et al. A bright cyan-excitable orange fluorescent protein facilitates dual-emission microscopy and enhances bioluminescence imaging in vivo. Nat Biotechnol, 2016, 34(7): 760-767. DOI:10.1038/nbt.3550 |
| [23] |
Subach OM, Patterson GH, Ting LM, et al. A photoswitchable orange-to-far-red fluorescent protein, PSmOrange. Nat Methods, 2011, 8(9): 771-777. DOI:10.1038/nmeth.1664 |
| [24] |
Patterson GH, Knobel SM, Sharif WD, et al. Use of the green fluorescent protein and its mutants in quantitative fluorescence microscopy. Biophys J, 1997, 73(5): 2782-2790. DOI:10.1016/S0006-3495(97)78307-3 |
| [25] |
Kennis JTM, van Stokkum IHM, Peterson DS, et al. Ultrafast proton shuttling in psammocora cyan fluorescent protein. J Phys Chem B, 2013, 117(38): 11134-11143. DOI:10.1021/jp401114e |
| [26] |
Griswold SL, Sajja KC, Jang CW, et al. Generation and characterization of iUBC-KikGR photoconvertible transgenic mice for live time lapse imaging during development. Genesis, 2011, 49(7): 591-598. DOI:10.1002/dvg.20718 |
| [27] |
Tsuboi T, Kitaguchi T, Karasawa S, et al. Age-dependent preferential dense-core vesicle exocytosis in neuroendocrine cells revealed by newly developed monomeric fluorescent timer protein. Mol Biol Cell, 2010, 21(1): 87-94. DOI:10.1091/mbc.e09-08-0722 |
| [28] |
Strack RL, Bhattacharyya D, Glick BS, et al. Noncytotoxic orange and red/green derivatives of DsRed-Express2 for whole-cell labeling. BMC Biotechnol, 2009, 9: 32. DOI:10.1186/1472-6750-9-32 |
| [29] |
Bulina ME, Chudakov DM, Britanova OV, et al. A genetically encoded photosensitizer. Nat Biotechnol, 2006, 24(1): 95-99. DOI:10.1038/nbt1175 |
| [30] |
Bulina ME, Lukyanov KA, Britanova OV, et al. Chromophore-assisted light inactivation (CALI) using the phototoxic fluorescent protein KillerRed. Nat Protocols, 2006, 1(2): 947-953. DOI:10.1038/nprot.2006.89 |
| [31] |
Rao PP, Wang X. The research progress of KillerRed in optogenetics. Chin J Cardiac Pac Electrophysiol, 2017, 31(4): 283-286 (in Chinese). 饶盼盼, 王晞. KillerRed在光遗传学中的研究进展. 中国心脏起搏与心电生理杂志, 2017, 31(4): 283-286. |
| [32] |
Sarkisyan KS, Zlobovskaya OA, Gorbachev DA, et al. KillerOrange, a genetically encoded photosensitizer activated by blue and green light. PLoS ONE, 2015, 10(12): e0145287. DOI:10.1371/journal.pone.0145287 |
| [33] |
Ausili A, Staiano M, Marabotti A, et al. Correlation between fluorescence and structure in the orange-emitting GFP-like protein, monomeric Kusabira Orange. J Photochem Photobiol B Biol, 2014, 138: 223-229. DOI:10.1016/j.jphotobiol.2014.04.028 |
| [34] |
Subach OM, Malashkevich VN, Zencheck WD, et al. Structural characterization of acylimine-containing blue and red chromophores in mTagBFP and TagRFP fluorescent proteins. Chem Biol, 2010, 17(4): 333-341. DOI:10.1016/j.chembiol.2010.03.005 |
| [35] |
Kikuchi A, Fukumura E, Karasawa S, et al. Structural characterization of a thiazoline-containing chromophore in an orange fluorescent protein, monomeric kusabira orange. Biochemistry, 2008, 47(44): 11573-11580. DOI:10.1021/bi800727v |
| [36] |
Pletnev S, Shcherbakova DM, Subach OM, et al. Orange fluorescent proteins: structural studies of LSSmOrange, PSmOrange and PSmOrange2. PLoS ONE, 2014, 9(6): e99136. DOI:10.1371/journal.pone.0099136 |
| [37] |
Pletneva NV, Pletnev VZ, Sarkisyan KS, et al. Crystal structure of phototoxic orange fluorescent proteins with a tryptophan-based chromophore. PLoS ONE, 2015, 10(12): e0145740. DOI:10.1371/journal.pone.0145740 |
| [38] |
Angermayr SA, Paszota M, Hellingwerf KJ. Engineering a cyanobacterial cell factory for production of lactic acid. Appl Environ Microbiol, 2012, 78(19): 7098-7106. DOI:10.1128/AEM.01587-12 |
| [39] |
Ruffing AM, Jensen TJ, Strickland LM. Genetic tools for advancement of Synechococcus sp. PCC 7002 as a cyanobacterial chassis. Microb Cell Factor, 2016, 15: 190. DOI:10.1186/s12934-016-0584-6 |
| [40] |
Rasala BA, Barrera DJ, Ng J, et al. Expanding the spectral palette of fluorescent proteins for the green microalga Chlamydomonas reinhardtii. Plant J, 2013, 74(4): 545-556. DOI:10.1111/tpj.12165 |
| [41] |
Mann DGJ, Abercrombie LL, Rudis MR, et al. Very bright orange fluorescent plants: endoplasmic reticulum targeting of orange fluorescent proteins as visual reporters in transgenic plants. BMC Biotechnol, 2012, 12: 17. DOI:10.1186/1472-6750-12-17 |
| [42] |
Osuki KI, Hashimoto S, Suzuki A, et al. Gene expression and localization of a β-1, 3-glucanase of Lotus japonicus. J Plant Res, 2016, 129(4): 749-758. DOI:10.1007/s10265-016-0811-6 |
| [43] |
Fujimoto M, Arimura SI, Ueda T, et al. Arabidopsis dynamin-related proteins DRP2B and DRP1A participate together in clathrin-coated vesicle formation during endocytosis. Proc Natl Acad Sci USA, 2010, 107(13): 6094-6099. DOI:10.1073/pnas.0913562107 |
| [44] |
Kotani K, Peroni OD, Minokoshi Y, et al. GLUT4 glucose transporter deficiency increases hepatic lipid production and peripheral lipid utilization. J Clin Invest, 2004, 114(11): 1666-1675. DOI:10.1172/JCI200421341 |
| [45] |
Zhao P, Ming Q, Xiong MR, et al. Dandelion chloroform extract promotes glucose uptake via the AMPK/GLUT4 pathway in L6 cells. Evid-Based Complement Alternat Med, 2018, 2018: 1709587. |
| [46] |
Murai H, Tadokoro R, Sakai KI, et al. In ovor gene manipulation of melanocytes and their adjacent keratinocytes during skin pigmentation of chicken embryos. Dev Growth Differ, 2015, 57(3): 232-241. DOI:10.1111/dgd.12201 |
| [47] |
Etienne L, Joshi P, Dingle L, et al. Visualization of herpes simplex virus type 1 virions using fluorescent colors. J Virol Methods, 2017, 241: 46-51. DOI:10.1016/j.jviromet.2016.12.012 |
| [48] |
Chen IH, Chang JE, Wu CY, et al. An E3 ubiquitin ligase from Nicotiana benthamiana targets the replicase of Bamboo mosaic virus and restricts its replication. Mol Plant Pathol, 2019, 20(5): 673-684. DOI:10.1111/mpp.12784 |
| [49] |
Bayle V, Nussaume L, Bhat RA. Combination of novel green fluorescent protein mutant TSapphire and DsRed variant mOrange to set up a versatile in planta FRET-FLIM assay. Plant Physiol, 2008, 148(1): 51-60. DOI:10.1104/pp.108.117358 |
| [50] |
Ke DC, Tu SC. Activities, kinetics and emission spectra of bacterial luciferase-fluorescent protein fusion enzymes. Photochem Photobiol, 2011, 87(6): 1346-1353. DOI:10.1111/j.1751-1097.2011.01001.x |
| [51] |
De A, Ray P, Loening AM, et al. BRET3: a red-shifted bioluminescence resonance energy transfer (BRET)-based integrated platform for imaging protein-protein interactions from single live cells and living animals. FASEB J, 2009, 23(8): 2702-2709. DOI:10.1096/fj.08-118919 |
| [52] |
Hussey PJ, Ketelaar T, Deeks MJ. Control of the actin cytoskeleton in plant cell growth. Ann Rev Plant Biol, 2006, 57: 109-125. DOI:10.1146/annurev.arplant.57.032905.105206 |
| [53] |
Peshenko IV, Olshevskaya EV, Lim S, et al. Identification of target binding site in photoreceptor Guanylyl Cyclase-Activating Protein 1 (GCAP1). J Biol Chem, 2014, 289(14): 10140-10154. DOI:10.1074/jbc.M113.540716 |
| [54] |
Liou J, Kim ML, Heo WD, et al. STIM is a Ca2+ sensor essential for Ca2+-store-depletion-triggered Ca2+ influx. Curr Biol, 2005, 15(13): 1235-1241. DOI:10.1016/j.cub.2005.05.055 |
| [55] |
Huang PC, Chiu TY, Wang LC, et al. Visualization of the orai1 homodimer and the functional coupling of orai1-STIM1 by live-cell fluorescence lifetime imaging. Microsc Microanal, 2010, 16(3): 313-326. DOI:10.1017/S1431927610000188 |
| [56] |
Samstag Y, Eibert SM, Klemke M, et al. Actin cytoskeletal dynamics in T lymphocyte activation and migration. J Leukocyte Biol, 2003, 73(1): 30-48. DOI:10.1189/jlb.0602272 |
| [57] |
Larbret F, Dubois N, Brau F, et al. Technical advance: Actin CytoFRET, a novel FRET flow cytometry method for detection of actin dynamics in resting and activated T cell. J Leukoc Biol, 2013, 94(3): 531-539. DOI:10.1189/jlb.0113022 |
| [58] |
Goedhart J, Vermeer JEM, Adjobo-Hermans MJW, et al. Sensitive detection of p65 homodimers using red-shifted and fluorescent protein-based FRET couples. PLoS ONE, 2007, 2(10): e1011. DOI:10.1371/journal.pone.0001011 |
 2020, Vol. 36
2020, Vol. 36