中国科学院微生物研究所、中国微生物学会主办
文章信息
- 王胜, 孟昆, 罗会颖, 姚斌, 涂涛
- Wang Sheng, Meng Kun, Luo Huiying, Yao Bin, Tu Tao
- 果胶甲酯酶的结构与功能研究进展
- Research progress in structure and function of pectin methylesterase
- 生物工程学报, 2020, 36(6): 1021-1030
- Chinese Journal of Biotechnology, 2020, 36(6): 1021-1030
- 10.13345/j.cjb.190418
-
文章历史
- Received: September 17, 2019
- Accepted: October 29, 2019
果胶物质存在于相邻细胞壁的胞间层中,起着将细胞粘在一起的作用,是植物细胞壁的主要成分之一。柑橘、柠檬、柚子等果皮中约含30%果胶,是果胶的最丰富来源。按果胶的组成可分为同质多糖和杂多糖两种类型:同质多糖果胶包括D-半乳聚糖、L-阿拉伯聚糖和D-半乳糖醛酸聚糖等;杂多糖果胶最常见,是由半乳糖醛酸、半乳糖和阿拉伯糖以不同比例组成,通常称为果胶酸。果胶具有复杂的结构,包含“平滑区域”和“毛状区域”两部分。平滑区域主要由α-1, 4糖苷键连接的D-聚半乳糖醛酸残基组成,其可在O6原子发生甲基化和在O2或O3原子发生乙酰化修饰,而毛状区域的特征在于含有1, 2-α-L-鼠李糖-1, 4-α-D-半乳糖醛酸二聚体的形式延伸,其中富含L-阿拉伯糖和D-半乳糖的侧链可被鼠李糖残基取代[1-2]。由于果胶的复杂性,需要一系列果胶酶协同降解,包括多聚半乳糖醛酸酶、果胶酸裂解酶、鼠李糖半乳糖醛酸酶、果胶甲酯酶、果胶乙酰酯酶等[3]。
果胶甲酯酶(Pectin methylesterase,PME;EC 3.1.1.11)能够水解甲酯基,释放甲醇从而降低果胶的甲酯化程度。PME在多种生物体中被发现,在高等植物中,大多数PME都存在于不同组织器官,如根、茎、叶、果实等,其对细胞壁组成和降解、细胞游离、花粉发育、种子萌发、根尖延伸、种子开裂、果实软化、抗病等方面具有重要作用[4-7],并且通常在一个物种中同时存在几种不同类型的PME。这些内源性的酶通常具有组织特异性,并且有助于植物发育和果实成熟期间细胞壁的变化;在微生物中,PME主要存在于真菌(如曲霉菌)、细菌(如欧文氏菌)。其次,某些植食性昆虫体内也相继发现了PME的存在[4, 8-10]。
PME是一种重要的果胶酶。目前在食品加工、茶饮料、造纸等生产工艺中已广泛应用PME来改善产品品质,如PME与多聚半乳糖醛酸酶一起使得果汁澄清的效果显著增强[11]。并且PME是许多植物病原体侵染植物的关键酶,对PME的研究也可以促进其对植物病原体侵染植物组织机理的理解,已成为植物学研究领域一个新的热点。但因为稳定性差、酶活低等特性,极大地限制了PME的广泛使用,所以对PME的功能改造也逐渐提上日程。而结构生物学研究是蛋白质功能定向改造强有力的支撑,通过对PME结构的研究来揭示酶结构与功能的关系成为目前研究的重点。本文在已报道PME的晶体结构基础上,对PME在结构生物学方面的研究进行综述。
1 果胶甲酯酶的总体结构PME属于碳水化合物酯酶第8家族[12] (Family 8 carbohydrate esterase,CE8s;http://afmb.cnrs-mrs.fr/CAZY)。目前,已有40种不同的CE8s被表征为PME蛋白,其从果胶内的甲酯化半乳糖醛酸中去除甲酯基,这也是细胞壁更新、果实成熟和致病等过程中的重要生物学事件[13]。
2000年Jenkins等[14]报道了欧文氏菌来源的PME (PemA;PDB ID:2NTB)晶体结构,这也是PME的第一个结构。随着对PME结构的深入研究,至今已经得到了7个不同来源的PME的晶体结构(表 1)。针对这7个不同来源的PME我们进行了多序列比对分析(图 1),尽管这7种不同来源PME氨基酸序列相似性较低(21%–62%),但它们三维结构相似,下面将进行具体介绍。

|
| 图 1 已报道结构的果胶甲酯酶多序列比对结果 Fig. 1 Multiple sequence alignment results of PME with known crystal structure. The sequences used in the multiple sequence alignment analysis are download from crystal structures of the PDB database |
| |
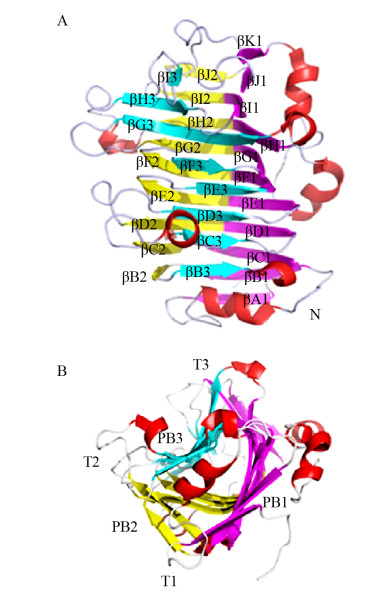
目前已报道的大多数酯酶采用α/β水解酶折叠结构并含有催化三联体Ser-His-Asp。但PME既没有共同的α/β水解酶折叠结构,也没有常见的催化三联体[14]。PME属于右手平行β-螺旋结构[19] (图 2A),对应每圈的β-折叠片分别定义为PB1、PB2和PB3。连接这3个β-折叠片的转角分别称为T1 (PB1-PB2)、T2 (PB2-PB3)和T3(PB3-PB1)[14, 18] (图 2B)。不同来源的PME其完整的β-螺旋圈数也不相同,其中番茄来源的PME (PDB ID:1XG2)只有7个完整的β-螺旋圈[17]。
|
| 图 2 胡萝卜PME的结构(PDB ID:1GQ8) Fig. 2 The crystal structure of PME from carrot (PDB ID: 1GQ8). (A) Front view. (B) Side view from N-terminal |
| |
以胡萝卜PME (PDB ID:1GQ8)为例,在整个结构中,PB1是最长的折叠片结构。结构中存在11股平行的PB1折叠片,其中第一条与最后一条大致垂直。第一个β折叠片之前的N末端存在7个游离的氨基酸残基,C末端也存在一段短的无序结构。但也有特殊情况存在,如水稻象鼻虫来源的PME (PDB ID:4PMH)的结构在整体的空间构象上与其他PME的结构保持一致,但是将其与其他来源的PME结构进行比对时,发现水稻象鼻虫来源的PME的C末端结构与其他来源PME的N末端结构能很好地拟合(图 3)。像这种蛋白质序列的循环排列是非常罕见的,但确实是自然发生的[20]。这种结构上的循环排列可能源于自然遗传或翻译后修饰导致的结构重排。从所有水稻象鼻虫PME蛋白的编码RNA序列来看,这种循环置换排列是起源于遗传的。研究人员又将其他来源的象鼻虫PME序列与水稻象鼻虫PME序列进行比对,发现这些蛋白质都具有延伸的N-末端区域和缩短的C-末端区域,表明该结构的循环置换属于昆虫家族特有的结构特征,因此也证明了蛋白质中存在结构循环排列现象[8]。

|
| 图 3 欧文氏菌PME (PDB ID:2NTB) (A)和水稻象鼻虫PME (PDB ID:4PMH) (B)的结构特点 Fig. 3 The structural features of Erwinia PME (PDB ID: 2NTB) (A) and rice weevil PME (PDB ID: 4PMH) (B) |
| |
T1转角区较短,其中胡萝卜来源的PME通常没有残基或只有1个残基连接PB1和PB2[18],而水稻象鼻虫来源的PME比其他来源的PME T1转角区较长。T2和T3转角区域通常包含较长的loop区,且蛋白分子表面的裂缝都由T2和T3区域这些较长的loop区组成。根据前面所述序列比对结果可知,来源于同一界的PME其序列一致性比较高,其在结构上的差异越小;来源于不同界的PME虽然在整体结构上都为右手平行β-螺旋结构且催化核心部分结构也较为保守,但是不同来源的PME在其结构上也存在很大差异,例如,β折叠片以及转角长度等,其中差异最大处在于连接β折叠片的转角区,由于其向外延伸长度各不相同,导致了转角处所形成的loop区的大小以及结构各异。例如将欧文氏菌来源的PME (PDB ID:2NTB)与胡萝卜来源的PME (PDB ID:1GQ8)进行结构比对(RMSD值为1.80 Å)发现,其最大的不同之处在于T2和T3 loop区链长度的不同,欧文氏菌来源的PME其T2与T3 loop区较长且结构更为复杂(图 4)。

|
| 图 4 欧文氏菌(2NTB)和胡萝卜(1GQ8)来源的PME基于序列以及结构对比分析结果(A:欧文氏菌(2NTB:橘色)和胡萝卜(1GQ8:浅蓝色)来源的PME序列比对结果,其中|为结构上相对应且残基一致,:为结构上相对应且残基相似,.为结构上相对应但二者残基不同. B:欧文氏菌(2NTB:橘色)和胡萝卜(1GQ8:浅蓝色)来源PME结构比对,其中灰色表示差异较大部分) Fig. 4 Results of sequence and structural comparison analysis of PME derived from Erwinia (2NTB) and carrot (1GQ8). (A) The sequence alignment of PME from Erwinia chrysanthemi (2NTB, up) and Daucus carota (1GQ, down), where | means structurally equivalent and identical residues, : means structurally equivalent and similar residues, . means structurally equivalent, but different residues. (B) The structure alignment of PME from bacteria (2NTB, orange) and plants (1GQ8, light blue), the gray color indicating the significance difference |
| |
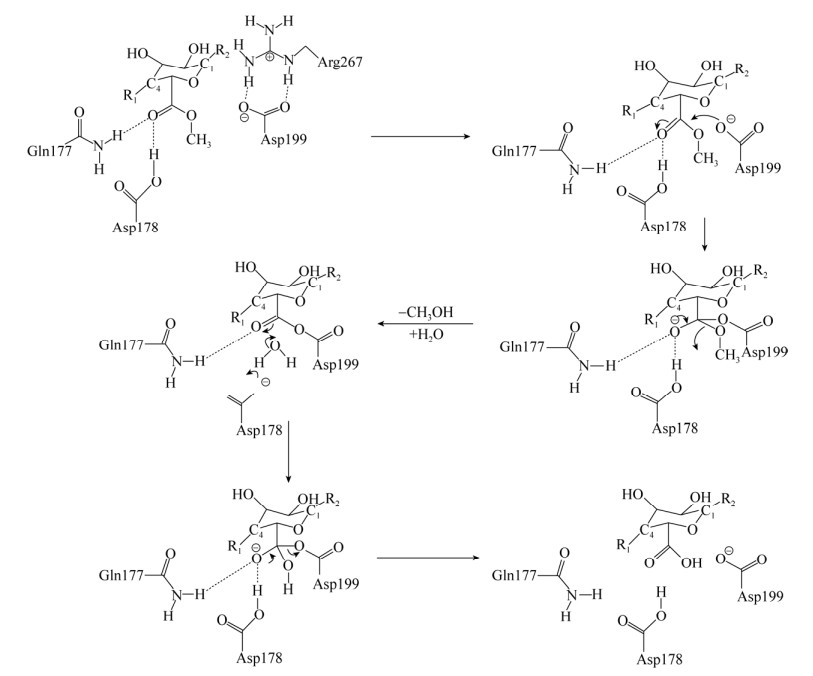
PME结构表面由较长的T2 loop区和T3 loop区形成裂缝,该裂缝的中心部分由几个芳香族氨基酸组成,这符合碳水化合物结合位点的特征[18]。Fries等[16]对欧文氏菌来源的PME (PDB ID:2NTB)与其底物共晶体的结构研究也进一步证实,六糖底物结合在酶表面的裂缝中。Teller等[8]分析了CE8s家族的PME的结构以及序列,总结了部分PME中的活性位点残基(表 2)。以欧文氏菌来源的PME为例,酶与底物复合物的结构表明以下残基参与催化反应:Asp199、Asp178和Gln177 (图 5)。
| PDB ID | Active site residues | ||
| 4PMH | Gln199 | Asp200 | Asp226 |
| 1GQ8 | Gln135 | Asp136 | Asp157 |
| 1QJV | Gln131 | Asp132 | Asp153 |
| 1XG2 | Gln177 | Asp178 | Asp199 |
| 3UW0 | Gln176 | Asp177 | Asp199 |
| 5C1C | Gln167 | Asp168 | Asp189 |
| 2NTB | Gln177 | Asp178 | Asp199 |

|
| 图 5 欧文氏菌PME (2NTB)活性位点相关残基 Fig. 5 The active sites of Erwinia PME (2NTB) |
| |
在之前的研究中,Markus等[15]对欧文氏菌来源的PME (PDB ID:2NTB)晶体结构进行研究时首先提出催化位点为保守的天冬氨酸(Asp178和Asp199)和精氨酸(Arg267)。但是经过进一步的研究发现第267位的精氨酸并不直接参与酶的催化[14]。后续研究提出了以下催化机制:残基Asp178通过与糖侧链上甲酯羰基的氧原子结合形成氢键,残基Asp199侧链上的羧基攻击糖侧链上的羰基碳原子形成四面体中间体,此时羰基氧原子上带有一个负电荷。该氧原子通过与Gln177和Asp178残基之间的相互作用来稳定自身的负电荷。由Asp178残基侧链上的羧基氢原子使离去基团质子化,形成酸酐中间体同时释放甲醇。随后由Asp178侧链去质子化后的羧基催化水分子水解酸酐中间体,并形成最终产物(图 6)。在该催化过程中,Asp199是直接攻击甲酯羰基碳的亲核试剂,Asp178是反应中的一般酸碱,而Gln177则通过形成氧阴离子孔来稳定过渡态,相邻的Gln153残基主要有助于底物结合,但是也有部分研究人员认为是由两个相邻的谷氨酰胺侧链形成的阴离子孔稳定带负电的四面体中间体[18]。与欧文氏菌来源的PME催化机理类似,植物来源的PME具有相同的催化机理。以胡萝卜来源的PME (PDB ID:1GQ8)为例,胡萝卜PME中Asp157、Asp136和Gln135分别起到亲核试剂、一般酸碱和稳定四面体中间体的作用。
人们对多糖的酶促转化提出了3种作用模式:(1)单链机制,酶与底物结合后,其链上所有连续的底物位点都被转化;(2)多链机制,每次反应完成后,酶-底物复合物解离,即每次反应仅转化单个底物残基;(3)多重攻击机制,对于每次形成的酶-底物复合物,酶催化平均数量有限的糖残基的转化[15]。针对这几种作用模式,部分研究中认为:植物和细菌来源的PME催化甲酯果胶产生多聚半乳糖醛酸链,对应提出单链和多链催化机制[21-23]。相反,真菌PME更加随机地行使催化活性,对应提出多重攻击机制[24-26]。但是在另一部分研究中提出了不同的见解:酸性微生物PME (日本曲霉、黑曲霉、油曲霉)催化甲酯基的随机裂解,属于多链催化机制。高等植物(番茄、橘子、苜蓿)和真菌(里氏木霉)来源的碱性PME水解产生游离羧基的嵌段,它们沿糖链线性催化果胶脱甲酯基,属于单链催化机制[27]。
在目前所有表征的结果中,PME持续催化的方向是从多聚半乳糖醛酸(High polygalacturonic acid,HG)链的非还原端朝向还原端。PME持续催化的能力很大程度上依赖于溶剂中的离子强度和pH等因素[16],由于不同来源PME其等电点不同,导致不同pH值缓冲溶液中PME的整体带电性质的不同,从而影响底物分子与酶活性口袋的结合。例如在Jean-Marc等[27]的研究中发现,苹果PME在不同的pH条件下由于酶与底物的作用方式的变化直接影响了其催化效果,在pH 7.0时具有典型的单链机理,但在pH 4.5时与单链和多链机理不同其属于多重攻击机制。同时Kent等[16]对黑曲霉来源的PME催化的持续性(Processive)特征进行了研究,结果表明酶和底物的高度带电性也会对酶与底物的结合产生影响。细菌和植物来源的PME通过静电吸引力使得带负电荷的底物持续保留在结合位点,从而有利于酶的持续性催化反应。相反,黑曲霉来源的PME对底物产生静电排斥作用,从而促进底物的解离和随机的非持续性酶促作用[16]。
3 碳水化合物结合方式以及底物特异性Fries等[15]对欧文氏菌来源的PME进行研究过程中,使用失活突变体D178A获得了PME与特异性甲基化的六聚半乳糖醛酸盐的晶体复合物。并使用了以下4种底物:底物Ⅰ-MMCCCM;底物Ⅱ- CCMMMC;底物Ⅲ-MCCCCM;底物Ⅳ-CMMMMC (寡糖序列以从非还原端到还原端的方式给出,其中M和C分别表示甲基化的聚半乳糖醛酸(Polygalacturonic acid,Gal A)和未甲基化的Gal A)对PME的底物特异性进行了研究。通过对这4种底物与酶结合方式的研究表征了酶的结合位点以及酶对不同甲基化底物的偏好性。底物Ⅰ和Ⅲ各自以相同的方式与PME结合,非还原末端位于亚位点-5位置,还原末端位于亚位点+1 (活性位点指定为亚位点+1)位置。底物Ⅱ和Ⅳ的结合模式不同于底物Ⅰ和Ⅲ的结合模式,底物Ⅱ与亚位点-2至+4位置结合,而底物Ⅳ占据亚位点-1至+5位置(表 3)。
| Substrate | Subsites | |||||||||
| -5 | -4 | -3 | -2 | -1 | +1 | +2 | +3 | +4 | +5 | |
| Preference | C | C | M | X | M | |||||
| Ⅰ | M | M | C | C | C | M | ||||
| Ⅱ | C | C | M | M | M | C | ||||
| Ⅲ | M | C | C | C | C | M | ||||
| Ⅳ | C | M | M | M | M | C | ||||
对晶体中底物-酶相互作用的分析表明,底物结合涉及的亚位点为-2、-1、+1、+2和+3,其中最重要的是亚位点-1和+1[15]。亚位点-1和-2总是被非甲基化的Gal A占据,这意味着该位点对糖链上羧基的强烈偏好。相反,在所有的复合物中发现催化位点结合的是甲基化的Gal A,表明催化位点对糖侧链上的甲酯基具有较强的偏好。在亚位点+2位置处,C6取代基面向溶剂,解除了酶对底物侧链的空间位阻;因此,甲基化或未甲基化的Gal A都可以结合在亚位点+2处。在亚位点+3位置含有疏水口袋,所以该亚位点更青睐于侧链含甲酯基的底物。虽然在亚位点+4、+5、-4和-5位置也分别结合着不同甲酯化的底物,但是这些位置氢键较少与底物的相互作用少,所以最佳的结合位点为亚位点-2至+3且其最佳底物为CCMXM (X表示甲基化或非甲基化底物皆可)。
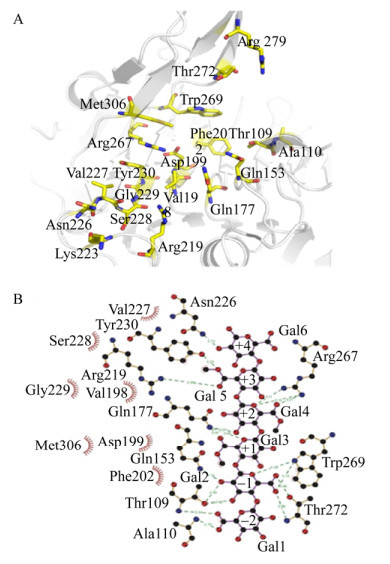
对PME同系物中底物结合涉及到的氨基酸保守程度进行统计分析,主要包括Thr109、Gln153、Gln177、Val198、Asp199、Phe202、Lys223、Val227、Ser228、Gly229、Arg267和Trp269。其中部分非保守性氨基酸也有助于底物的结合,如Ala110、Arg219、Asn226、Tyr230、Thr272、Arg279和Met306 (图 7)。其中与果胶分子中甲酯基和羧基结合的残基可能有助于稳定底物的构象。在亚位点-4和-5位置处没有氨基酸与果胶分子接触表明亚位点–3位置非还原端一侧底物侧链无论是甲酯基还是羧基都可以;另一方面,亚位点-3位置处的Arg279与底物的羧基结合,该带正电荷的残基与羧基的接触表明该位点优选侧链带羧基而不是甲酯基的底物。在亚位点-2位置处,Ala110的主链酰胺和另一些晶体复合物中的Thr109与底物羧基的两个氧形成氢键,与该位点对底物侧链为羧基的偏好性相关。同样,在亚位点-1处涉及的Trp269和Thr272 (主链酰胺和侧链羟基)与底物侧链羧基形成的氢键也揭示了此位点对羧基的偏好性。其次,甲酯基在活性位点的特异性结合可归因于由残基Phe202、Trp269和Met306产生的疏水环境。在亚位点+2处,底物侧链面向溶剂,因此亚位点+2不涉及底物特异性。Val198、Val227和Tyr230形成的疏水口袋,用于在亚点+3位置处结合酯的甲基官能团。亚点+4和+5处的Gal A的C6取代基不与蛋白质相互作用,因此预测亚基+4和+5不会对果胶底物的特定甲基化模式产生特异性。
|
| 图 7 PME活性口袋中涉及的与底物结合相关氨基酸残基分布以及与底物相互作用示意图(A:与各种底物结合相关的氨基酸残基分布情况;B:涉及的相互作用示意图)[15] Fig. 7 Schematic representation of the distribution of amino acid residues associated with substrate binding involved in the PME active pocket and the interaction of the enzyme with the substrate. (A) Distribution of amino acid residues associated with various substrate bindings. (B) Schematic diagram of interactions involved[15] |
| |
随着近几十年来对PME的不断深入研究,PME的结构与催化机理也逐渐被认识,但是目前仅仅对一种来源的PME与其底物复合物进行了晶体结构的表征,虽然其他科研人员也对PME与其底物的结合方式进行了一定的研究,但都是在此基础上对蛋白结构进行比对或者是利用分子对接技术等间接方法对酶的催化机制展开研究,具有一定的局限性。此外,目前已报道的PME晶体结构较少,对我们进一步了解PME催化机理以及底物特异性等方面也有一定的局限性。
其次,针对酶性能改造的蛋白质工程技术对于酶的商业化非常重要。候选酶的工业应用的一个瓶颈是低催化效率[28]。近几十年来研究人员已经开发了譬如从头设计、基于结构基础的蛋白质理性设计等多种方法来改善酶催化性能[29-31]。对酶的功能设计是基于对酶结构层面深入认知后展开的,并且理性设计也可以用于确定与酶催化相关的重要氨基酸残基等相关因素[32]。显然,针对这些基于结构生物学展开的研究工作,目前仅有的几个PME晶体结构不具广泛的代表性。
此外,除了对PME的催化性能进行理性设计以满足工业生产需求外,PME也是许多植物病原菌侵染植物的关键酶。通过对不同植物病原菌所产PME的催化机制研究,我们可以有效地针对其催化性质设计抑制剂,以减轻其对植物造成的危害;并且在现阶段的研究工作中,也已经有部分研究人员发现了PME的抑制蛋白[17],通过同时对PME以及其抑制蛋白作用方式的研究,进一步了解其催化特性以及起到抑制作用的关键因素,为理性设计抑制剂提供理论支持。
由此可见,继续推进对不同来源的PME结构研究,进一步揭示PME的结构特点以及催化机理与催化方式是十分重要的。目前报道的7种PME结构各不相同,并且已经报道不同来源的PME其催化方式也略有差异,这就更加要求我们加快研究的步伐。
| [1] |
Li YQ, Wang Y, Tu T, et al. Two acidic, thermophilic GH28 polygalacturonases from Talaromyces leycettanus JCM 12802 with application potentials for grape juice clarification. Food Chem, 2017, 237: 997-1003. DOI:10.1016/j.foodchem.2017.06.037 |
| [2] |
Tu T, Li YQ, Su XY, et al. Probing the role of cation-π interaction in the thermotolerance and catalytic performance of endo-polygalacturonases. Sci Rep, 2016, 6: 38413. DOI:10.1038/srep38413 |
| [3] |
Tu T, Meng K, Luo HY, et al. New insights into the role of T3 loop in determining catalytic efficiency of GH28 endo-polygalacturonases. PLoS ONE, 2015, 10(9): e135413. |
| [4] |
Ren CH, Kermode AR. An increase in pectin methyl esterase activity accompanies dormancy breakage and germination of yellow cedar seeds. Plant Physiol, 2000, 124(1): 231-242. DOI:10.1104/pp.124.1.231 |
| [5] |
Micheli F, Sundberg B, Goldberg R, et al. Radial distribution pattern of pectin methylesterases across the cambial region of hybrid aspen at activity and dormancy. Plant Physiol, 2000, 124(1): 191-200. DOI:10.1104/pp.124.1.191 |
| [6] |
Pilatzke-Wunderlich I, Nessler CL. Expression and activity of cell-wall-degrading enzymes in the latex of opium poppy, Papaver somniferum L. Plant Mol Biol, 2001, 45(5): 567-576. DOI:10.1023/A:1010624218855 |
| [7] |
Collmer A, Keen NT. The role of pectic enzymes in plant pathogenesis. Ann Rev Phytopathol, 1986, 24(1): 383-409. DOI:10.1146/annurev.py.24.090186.002123 |
| [8] |
Teller DC, Behnke CA, Pappan K, et al. The structure of rice weevil pectin methylesterase. Acta Crystallogr F Struct Biol Commun, 2014, 70(11): 1480-1484. DOI:10.1107/S2053230X14020433 |
| [9] |
Keeling CI, Henderson H, Li M, et al. Transcriptome and full-length cDNA resources for the mountain pine beetle, Dendroctonus ponderosae Hopkins, a major insect pest of pine forests. Insect Biochem Mol Biol, 2012, 42(8): 525-536. DOI:10.1016/j.ibmb.2012.03.010 |
| [10] |
Pauchet Y, Wilkinson P, Chauhan R, et al. Diversity of beetle genes encoding novel plant cell wall degrading enzymes. PLoS ONE, 2010, 5(12): e15635. DOI:10.1371/journal.pone.0015635 |
| [11] |
Tu T, Meng K, Bai YG, et al. High-yield production of a low-temperature-active polygalacturonase for papaya juice clarification. Food Chem, 2013, 141(3): 2974-2981. DOI:10.1016/j.foodchem.2013.05.132 |
| [12] |
Pan X, Tu T, Wang LW, et al. A novel low-temperature-active pectin methylesterase from Penicillium chrysogenum F46 with high efficiency in fruit firming. Food Chem, 2014, 162: 229-234. DOI:10.1016/j.foodchem.2014.04.069 |
| [13] |
Boraston AB, Abbott DW. Structure of a pectin methylesterase from Yersinia enterocolitica. Acta Crystallogr Sect F Struct Biol Cryst Commun, 2012, 68(2): 129-133. DOI:10.1107/S1744309111055400 |
| [14] |
Jenkins J, Mayans O, Smith D, et al. Three-dimensional structure of Erwinia chrysanthemi pectin methylesterase reveals a novel esterase active site. J Mol Biol, 2001, 305(4): 951-960. DOI:10.1006/jmbi.2000.4324 |
| [15] |
Fries M, Ihrig J, Brocklehurst K, et al. Molecular basis of the activity of the phytopathogen pectin methylesterase. EMBO J, 2007, 26(17): 3879-3887. DOI:10.1038/sj.emboj.7601816 |
| [16] |
Kent LM, Loo TS, Melton LD, et al. Structure and properties of a non-processive, salt-requiring, and acidophilic pectin methylesterase from Aspergillus niger provide insights into the key determinants of processivity control. J Biol Chem, 2016, 291(3): 1289-1306. DOI:10.1074/jbc.M115.673152 |
| [17] |
Di Matteo A, Giovane A, Raiola A, et al. Structural basis for the interaction between pectin methylesterase and a specific inhibitor protein. Plant Cell, 2005, 17(3): 849-858. DOI:10.1105/tpc.104.028886 |
| [18] |
Johansson K, El-Ahmad M, Friemann R, et al. Crystal structure of plant pectin methylesterase. FEBS Lett, 2002, 514(2/3): 243-249. |
| [19] |
Jenkins J, Pickersgill R. The architecture of parallel β-helices and related folds. Prog Biophys Mol Biol, 2001, 77(2): 111-175. DOI:10.1016/S0079-6107(01)00013-X |
| [20] |
Lindqvist Y, Schneider G. Circular permutations of natural protein sequences: structural evidence. Curr Opin Struct Biol, 1997, 7(3): 422-427. DOI:10.1016/S0959-440X(97)80061-9 |
| [21] |
Dongowski G, Bock W. On the effects of molecular-parameters of pectin-substrates on the activities of pectin-esterases from Aspergillus niger and from higher plants. Nahrung-Food, 1984, 28: 507-516. DOI:10.1002/food.19840280507 |
| [22] |
Kohn R, Dongowski G, Bock W. The distribution of free and esterified carboxyl groups within the pectin molecule after the influence of pectin esterase from Aspergillus niger and oranges. Nahrung, 1985, 29(1): 75-85. DOI:10.1002/food.19850290117 |
| [23] |
Grasdalen H, Andersen AK, Larsen B. NMR spectroscopy studies of the action pattern of tomato pectinesterase: Generation of block structure in pectin by a multiple-attack mechanism. Carbohyd Res, 1996, 289: 105-114. DOI:10.1016/0008-6215(96)00079-1 |
| [24] |
Limberg G, Körner R, Buchholt HC, et al. Analysis of different de-esterification mechanisms for pectin by enzymatic fingerprinting using endopectin lyase and endopolygalacturonase Ⅱ from A. niger. Carbohydr Res, 2000, 327(3): 293-307. DOI:10.1016/S0008-6215(00)00067-7 |
| [25] |
Van Alebeek GJM, Van Scherpenzeel K, Beldman G, et al. Partially esterified oligogalacturonides are the preferred substrates for pectin methylesterase of Aspergillus niger. Biochem J, 2003, 372(1): 211-218. |
| [26] |
Duvetter T, Fraeye I, Sila DN, et al. Mode of de-esterification of alkaline and acidic pectin methyl esterases at different pH conditions. J Agric Food Chem, 2006, 54(20): 7825-7831. DOI:10.1021/jf060013h |
| [27] |
Denès JM, Baron A, Renard CMGC, et al. Different action patterns for apple pectin methylesterase at pH 7.0 and 4.5. Carbohydr Res, 2000, 327(4): 385-393. DOI:10.1016/S0008-6215(00)00070-7 |
| [28] |
Choi JM, Han SS, Kim HS. Industrial applications of enzyme biocatalysis: Current status and future aspects. Biotechnol Adv, 2015, 33(7): 1443-1454. DOI:10.1016/j.biotechadv.2015.02.014 |
| [29] |
Korendovych IV, DeGrado WF. Catalytic efficiency of designed catalytic proteins. Curr Opin Struct Biol, 2014, 27: 113-121. DOI:10.1016/j.sbi.2014.06.006 |
| [30] |
Daczkowski CM, Pegan SD, Harvey SP. Engineering the organophosphorus acid anhydrolase enzyme for increased catalytic efficiency and broadened stereospecificity on Russian VX. Biochemistry, 2015, 54(41): 6423-6433. DOI:10.1021/acs.biochem.5b00624 |
| [31] |
Parashar D, Satyanarayana T. A chimeric α-amylase engineered from Bacillus acidicola and Geobacillus thermoleovorans with improved thermostability and catalytic efficiency. J Ind Microbiol Biot, 2016, 43(4): 473-484. DOI:10.1007/s10295-015-1721-7 |
| [32] |
Tu T, Li YQ, Luo HY, et al. A key residue for the substrate affinity enhancement of a thermophilic endo-polygalacturonase revealed by computational design. Appl Microbiol Biot, 2018, 102(10): 4457-4466. DOI:10.1007/s00253-018-8948-y |
 2020, Vol. 36
2020, Vol. 36