中国科学院微生物研究所、中国微生物学会主办
文章信息
- 李学龙, 刘军花, 刘茜阳, 于琳, 吴珊珊, 尹秀山
- Xuelong Li, Junhua Liu, Qianyang Liu, Lin Yu, Shanshan Wu, Xiushan Yin
- 一种基于荧光qPCR检测新型冠状病毒核酸的优化反应体系的建立及相关测试
- Optimization of a fluorescent qPCR detection for RNA of SARS-CoV-2
- 生物工程学报, 2020, 36(4): 732-739
- Chinese Journal of Biotechnology, 2020, 36(4): 732-739
- 10.13345/j.cjb.200088
-
文章历史
- Received: February 27, 2020
- Accepted: April 2, 2020
2. 拜澳泰克 (沈阳) 生物医学集团有限公司,辽宁 沈阳 110142;
3. 江苏谱录瑞医疗科技有限责任公司,江苏 徐州 221000;
4. 上海耐诺生物科技有限公司,上海 200000;
5. 拜澳泰克 (江西) 生物科技有限公司,江西 赣州 341000
2. BIOTECH (Shenyang) Biomedical Group Co., Ltd., Shenyang 110142, Liaoning, China;
3. Jiangsu Plumway Medical Technology Co., Ltd., Xuzhou 221000, Jiangsu, China;
4. Shanghai Nano Biotechnology co., Ltd., Shanghai 200000, China;
5. BIOTECH (Jiangxi) Biotechnology co., Ltd., Ganzhou 341000, Jiangxi, China
目前,由湖北省武汉市发现的可引发人患新型冠状病毒肺炎(Coronavirus disease,COVID-19)的病毒SARS-CoV-2[1-3],被鉴定为一种含单股正链RNA (ssRNA)的Beta型冠状病毒(Betacoronavirus),其核酸结构与SARS极其相似,主要包括有E (Envelope protein gene)、M (Membrane protein gene)、N (Nucleocapsid protein gene)、S (Spike protein gene)、ORF (Open reading frame)和RdRp (RNA-dependent RNA polymerase gene)[4](图 1),与蝙蝠和果子狸等野生动物体内所检测到的冠状病毒(Bat/Paguma larvata-SARS- related CoVs)具有极高的同源性[5-6]。该病毒主要借助飞沫方式在人群中进行传播感染。感染者通常会出现以高热为主的症状,但是在少数报道中显示在未有发病症状却有核酸检测阳性的病例,种种现象也提示该病毒潜在的传染途径及机制仍然存在不确定性。因此,对于这个突发的COVID-19疫情的防控,快速并精确地早期诊断是极其重要的一个环节。针对冠状病毒的诊断方法有很多,比如:胸部CT扫描[7]和分子诊断[8],普通电泳法[9]、Northern杂交[10]、RT-PCR (Reverse transcription-PCR)[11]、Real-time RT-PCR[12]和NASBA (Nucleic acid sequence-based amplification)[13]等方法。最近,虽然Corman等[14]的研究提及RT-PCR的方法用于检测SARS-CoV-2,但实验方法验证数据均依照SARS相关病毒分离株核酸检测所得,缺乏真实样本检测结果数据支撑。同样我们依照荧光定量PCR的原理建立起一套适用于初步筛查鉴定SARS-CoV-2病毒的新体系,并且采用取材于COVID-19确诊患者的病毒内容物进行体系测试,同时对未知临床样本进行检测。对比市场上的基因的单通道RT-PCR检测试剂盒,该方法的优势在于双通道法增加检测特异性,同时灵敏度高,扩增效率高,为临床医学诊断提供参考价值。

|
| 图 1 SARS-CoV-2基因序列组分图(源于WH-human_1 genome) Fig. 1 Sequence composition diagram of SARS-CoV-2 gene (originating from wh-human_1 genome) |
| |
PCR扩增仪(BIORAD-C1000);荧光光定量PCR仪(Bio-Rad CFX96 touch)和无核酸酶的PCR管等。
体外转录试剂盒(NEB);反转录试剂盒Superscript Ⅲ Reverse Transcription frist strand cDNA (Thermo公司);华大基因的2019-nCoV检测试剂盒,编号60202002;病毒核酸样本源于中国疾控中心。
引物设计与合成:以新型冠状病毒SARS- CoV-2基因组保守区N基因和ORF1a基因(WH- human_1 COVID-19患者分离的SARS-CoV-2序列见图 1)为靶点通过Primer Premier 5.0等软件进行设计特异性引物和TaqMan探针,在ORF1a基因探针的5′端携带FAM基团(蓝色荧光),以及3′端含BHQ1 (淬灭基团1),而在N基因的5′端携带HEX基团(绿色荧光),以及3′端含BHQ2 (淬灭基团2)。具体序列参见表 1。表中的序列均由TaKaRa公司合成。
| Primer number | Sequences (5′–3′) | Size (bp) | Targeted site |
| F1 | GTGARATGGTCATGTGTGGCGG | 100 | ORF1a gene |
| R1 | CARATGTTAAASACACTATTAGCATA | ||
| 1a-Probe | FAM*-CAGGTGGAACCTCATCAGGAGATGC-BHQ1* | ||
| N3_F | GGGAGCCTTGAATACACCAAAA | 72 | N gene |
| N3_R | TGTAGCACGATTGCAGCATTG | ||
| N3-Probe | HEX*-AYCACATTGGCACCCGCAATCCTG-BHQ2* | ||
| Note: FAM* and HEX* represent the addition of FAM and HEX fluorescence modifications at the 5′ end of the primer, respectively. | |||
实验样品处理流程包括SARS-CoV-2病毒核酸序列的获取、特异靶点捕获、荧光信号读取、实时定量检测等4个阶段。对于临床样本,我们将采集的鼻、咽拭子等经病毒RNA提取试剂盒提取纯化获得SARS-CoV-2病毒核酸RNA(该步骤至少需要在P3实验室进行,减少污染源传播),并在−40–−80 ℃条件下保存备用。
然后按照反应体系20 μL (如RNA取5μL (0.01 pg–1 µg),引物探针(10 µmol/L) 2 μL,反应液(SuperScriptTM Ⅲ RT/PlatinumTM Taq Mix和Reaction buffer)10 μL)加入PCR反应管,混匀并在低于2 000 r/min离心机中离心,再移至特定的荧光光定量PCR仪进行孵育反应。并设置激光通路为FAM (450–490 nm),HEX (515–535 nm),孵育参数分为两个连续阶段:50 ℃ 5 min,94 ℃ 2 min用于cDNA合成和94 ℃ 5 s,55 ℃ 10 s,循环35–40次左右完成PCR扩增步骤。反应结束后根据获取的荧光参数分析并解释数据。
1.3 反应体系的效率验证人工合成SARS-CoV-2病毒基因组序列,利用NEB体外转录试剂盒合成目的基因的RNA序列,经Qiagen纯化试剂盒纯化RNA后,使用Thermo的RNA荧光定量试剂盒进行RNA精准定量,然后梯度稀释后来验证试剂反应条件的效率。
精准定量后RNA按照以下比例用DEPC处理水稀释后进行PCR验证:A=1/10,B=1/50,C=1/100,D=1/500,E=1/1 000,F=1/5 000,G=1/10 000。
1.4 灵敏性试验通过疾控中心获得的阳性患者的核酸RNA,经过Thermo的RNA荧光定量仪精准定量浓度为9.5 ng/μL,用DEPC处理水稀释样本后进行qPCR验证,稀释比例如下:A=1/10,B=1/100,C=1/1 000,D=1/10 000,以确定最佳测试范围。
1.5 临床样品测试另从辽宁省疾控中心取得已诊断的218个样本(含25例阳性和193例阴性样本),我们按照本文建立的实验体系,在不定量的情况下,直接进行qPCR检测,验证该方法的临床适用性。
同时也参与测试了辽宁省疾控中心的44个样本,进一步临床验证该体系的可靠性。
1.6 特异性测试为了解决该实验体系能否排除其他病毒的干扰问题,我们选择了两种较为常见的病毒,如甲流病毒、猪流感病毒,通过测试来验证该体系的特异性能。
1.7 差异性分析利用上述方法建立的试剂混合液体系与目前市售SARS-CoV-2检测试剂盒(如BGI kit)在同样的条件下进行差异性分析。
2 结果与分析 2.1 反应体系的效率验证将荧光定量后的SARS-CoV-2病毒RNA (130 ng/μL)在7个不同稀释浓度下进行荧光PCR检测,结果见图 2所示。当稀释比例为1/10,反应循环数(Ct) < 10可达到预期阈值。当稀释比例逐渐增大,反应循环数也不断增加,呈线性正相关。但当稀释比例增加为1/10 000时,反应循环数(Ct)需20个就达到预期。由此可以说明该方法所检测浓度的下限可调至1/10 000。
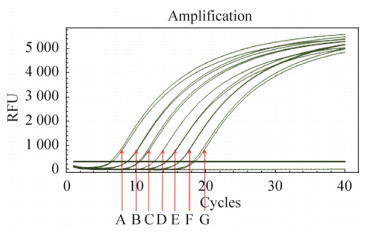
|
| 图 2 反应体系效率验证图 Fig. 2 The image of efficiency verification of reaction system. A–G represents four different dilution ratios, respectively: A=1/10, B=1/50, C=1/100, D=1/500, E=1/ 1 000, F=1/5 000, and G=1/10 000. |
| |
根据上述已建立的检测体系,对来源于一名确诊病例的阳性COVID-19核酸进行荧光PCR检测(图 3),当浓度以10倍的稀释梯度逐渐从1/10降低至1/10 000时,其RFU结果达到阈值时的循环数(Ct)均小于35,因此,此方法符合实际条件下的荧光定量PCR扩增情况,表明该方法具有一定的可靠性。
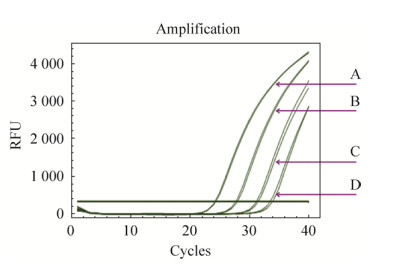
|
| 图 3 灵敏性测试结果 Fig. 3 The results of sensitivity test. A–D represents four different dilution ratios, respectively: A=1/10, B= 1/100, C=1/1 000, and D=1/10 000. |
| |
同时在阳性对照初始值设为9–10 ng/μL的基础条件下,浓度稀释比例达到10 000倍,仍有循环数(Ct)低于40的结果呈现,由此可知,此方法对于阳性病毒的最低检测值可达到1/10 000,该方法具有良好的敏感性。
2.3 临床样品测试在辽宁省疾控中心得到的262例经诊断的核酸样本中,其中46例患者被确诊为阳性患者,216例患者被确诊为阴性患者。按照国家卫健委颁布的新冠肺炎诊疗指南第6、7版,将这些样本划分为两组,分别是COVID-19患者组和COVID-19非患者组,进行本实验体系适用性验证,在测试结果中显示的Ct值均与疾控中心的诊断结果基本一致,非患者组的检测结果均一致,而患者组中45例检测呈阳性,其中一份漏检,符合度为97.83%。因此,本实验体系符合且适用于初步筛查病毒感染(表 2)。
| Patient ID | COVID-19 diagnosis sample from CDC | The testing results in our systems | |
| Infected groups | Non-infected group | Coincidence rate (%) | |
| 001–050 | 5 | 45 | 100 |
| 051–100 | 7 | 43 | 100 |
| 101–150 | 5 | 45 | 100 |
| 151–200 | 5 | 45 | 100 |
| 201–250 | 19 | 31 | 94.74 (18+/19+) |
| 251–262 | 5 | 7 | 100 |
| Total | 46 | 216 | 97.83 (45+/46+) |
| Note: Coincidence rate (%) refers to the data obtained by comparing the number of positive samples tested in this experimental system with the known clinical positive samples. | |||
现选择其中两个COVID-19阳性样本,在不定量的情况下,按照阴性样本、COVID-19#1、COVID-19#2和阳性样本的顺序上样,并对每个样本设置2个复孔,结果如图 4显示,阳性对照循环数(Ct)为22–24之间,COVID-19#1号患者的循环数(Ct)为28–30之间,COVID-19#2患者的循环数(Ct)为32–34之间。因此,说明本实验中所提供的新体系可以初步用于疑似病例检测SARS-CoV-2病原感染程度。但由于本次实验没有进行定量操作,所以无法判断1号患者的感染程度大于2号患者的感染程度。
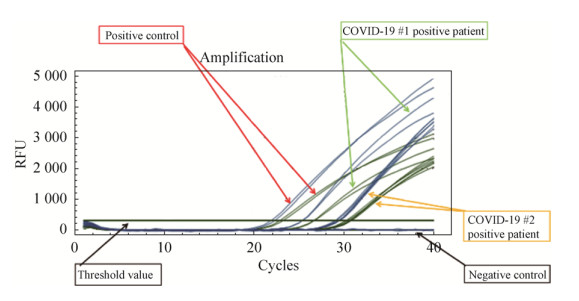
|
| 图 4 临床样品检测图谱 Fig. 4 The diagram of clinical sample test. |
| |
实验中为了验证该体系的特异性,我们进行了两种甲流病毒和猪流感病毒对SARS-CoV-2病毒的干扰性测试实验,具体的测试结果见图 5。图中显示的甲流病毒(Influenza A virus)和猪流感病毒(Swine influenza)的扩增曲线图近乎为一条平行于横坐标轴的直线,而SARS-CoV-2的检测曲线迅速增长,这说明本实验体系可以排除甲流和猪流感病毒等干扰。
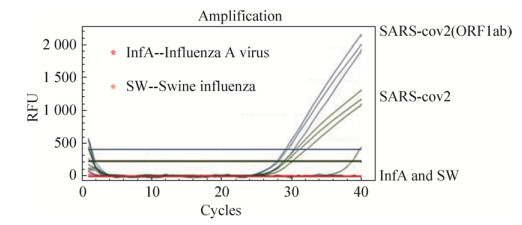
|
| 图 5 特异性测试结果图 Fig. 5 Results of specificity test. |
| |
为了验证本实验所采用的方法与上市的试剂盒的差异性(图 6A为上市试剂盒检测结果,图 6B为本实验方法输出的结果)。在同等的上样量情况下,产生的结果一致,并且本实验体系检测循环数(Ct)为22,而BGI试剂盒测试循环数(Ct)为24。由此说明该检测体系的灵敏度稍高于BGI试剂盒。同时,实验中采用双通道检测不仅增加检测特异性,而且降低假阳/阴性率,可初步用于检测SARS-CoV-2病毒的感染情况。

|
| 图 6 对比目前上市试剂盒结果分析图 Fig. 6 The analysis diagram of the method adopted in this experiment comparing to current market kit about COVID-19. (A) The result of BGI kit test. (B) The method provided in this experiment. The four lines refer that the two channels of the two samples respectively show the amplification curves, such as blue referring to FAM-ORF1a/b gene, and green referring to the HEX-N gene in the B diagram. |
| |
对于在湖北武汉地区突发的SARS-CoV-2病毒而引起的严重肺部炎症,目前尚无特异性治疗药物,而且关于该病毒的入侵机制也不甚清楚。虽然最近在一些COVID-19相关研究取得重大进展,比如Wrapp等[15]直接揭示SARS-CoV-2病毒的Cryo-EM结构,随后,Yan[16]等报道了结合SARS-CoV-2 Spike糖蛋白后的人肺泡细胞的ACE2 (Angiotensin-converting enzyme 2)的Cryo-EM结构,表明SARS-CoV-2与SARS-CoV具有相似感染人肺部细胞的机制。同时,最近有几篇文章同样报道了人细胞表面受体ACE2和SARS-CoV-2的Spike糖蛋白之间的传递媒介,并提出一些阻断剂,如TMPRSS2抑制剂[17]以及SARS-CoV S鼠多克隆抗体(交叉中和性抗体)[18],进一步深入了解了病毒的入侵机制,为COVID-19疫苗的研制提供了理论基础。但是,在一些地区出现数例核酸检测阳性的患者中,却没有出现普遍的高热等发病症状。鉴于该病毒传播能力较强,易潜伏,且可引起同流感相似的症状,因此,除了依赖于高热等体征变化来判别感染,在病毒的高发区实施全面筛查是非常有必要的。在本试验中,利用荧光定量实时PCR技术建立的优化体系,直接将患者RNA提取物进行病毒检测,省略多步扩增的过程,且在初始定量为10 ng/μL条件下,检测量的下限可调整至1/10 000 (即0.001 ng/μL),提高了病毒检测灵敏度。在与目前上市的COVID-19检测试剂盒的测试项目中,该实验体系采用双通道检测体系可明显减少非特异性结果的出现,同时本体系的实验操作时间(45–50 min)远低于目前上市的试剂盒操作时间(通常为90 min)。经过一系列的临床样本的测试流程,该方法可以用于早期初步的筛查COVID-19,或与临床CT诊断联用可直接且更精确地确诊SARS-CoV-2病毒感染情况。目前,基于此方法现已开发了一款基于荧光定量检测SARS-CoV-2的试剂盒产品(编号:BIO2020-02)。整个流程操作简便,一个反应1 h以内可完成操作,大大提升了检测效率,至少解决此次突发事件的某些地区出现的试剂短缺等重大问题。综上所述,这项研究的结果提示我们优化后的RT-PCR检测方法可用于快速且准确筛查SARS-CoV-2病毒感染情况。
| [1] |
Wu F, Zhao S, Yu B, et al. A new coronavirus associated with human respiratory disease in China. Nature, 2020, 579(7798): 265-269. DOI:10.1038/s41586-020-2008-3 |
| [2] |
Hui DS, Azhar EI, Madani TA, et al. The continuing 2019-nCoV epidemic threat of novel coronaviruses to global health - The latest 2019 novel coronavirus outbreak in Wuhan, China. Int J Infect Dis, 2020, 91: 264-266. |
| [3] |
Coronaviridae Study Group of the International Committee on Taxonomy of Viruses. The species Severe acute respiratory syndrome-related coronavirus: classifying 2019-nCoV and naming it SARS-CoV-2. Nat Microbiol, 2020. DOI:10.1038/s41564-020-0695-z |
| [4] |
Chan JFW, Kok KH, Zhu Z, et al. Genomic characterization of the 2019 novel human-pathogenic coronavirus isolated from a patient with atypical pneumonia after visiting Wuhan. Emerg Microb Infect, 2020, 9(1): 221-236. |
| [5] |
Chen Y, Liu QY, Guo DY. Emerging coronaviruses: Genome structure, replication, and pathogenesis. J Med Virol, 2020, 92(4): 418-423. |
| [6] |
Xu XT, Chen P, Wang JF, et al. Evolution of the novel coronavirus from the ongoing Wuhan outbreak and modeling of its spike protein for risk of human transmission. Sci China Life Sci, 2020, 63(3): 457-460. DOI:10.1007/s11427-020-1637-5 |
| [7] |
Lei JQ, Li JF, Li X, et al. CT imaging of the 2019 novel coronavirus (2019-nCoV) pneumonia. Radiology, 2020, 200236. DOI:10.1148/radiol.2020200236 |
| [8] |
Chu DKW, Pan Y, Cheng SMS, et al. Molecular diagnosis of a novel coronavirus (2019-nCoV) causing an outbreak of pneumonia [published online ahead of print, 2020 Jan 31]. Clin Chem, 2020: hvaa029, doi: 10.1093/clinchem/hvaa029.
|
| [9] |
刘军花, 李学龙, 刘茜阳, 等.一种基于琼脂糖凝胶电泳法检测SARS-CoV-2的新方法[EB/OL].微生物学杂志: 1-5. [2020-04-07] http://kns.cnki.net/ kcms/detail/21.1186.q.20200302.0849.002.html.
|
| [10] |
Gildow FE, Reavy B, Mayo MA, et al. Aphid acquisition and cellular transport of Potato leafroll virus-like particles lacking P5 readthrough protein. Hytopathology, 2000, 90(10): 1153-1161. |
| [11] |
Shirato K, Nao N, Katano H, et al. Development of genetic diagnostic methods for novel coronavirus 2019 (nCoV-2019) in Japan. Jpn J Infect Dis, 2020. DOI:10.7883/yoken.JJID.2020.061 |
| [12] |
Poon LLM, Chan KH, Wong OK, et al. Early diagnosis of SARS coronavirus infection by real time RT-PCR. J Clin Virol, 2003, 28(3): 233-238. |
| [13] |
Keightley MC, Sillekens P, Schippers W, et al. Real-time NASBA detection of SARS-associated coronavirus and comparison with real-time reverse transcription-PCR. J Med Virol, 2005, 77(4): 602-608. |
| [14] |
Corman VM, Landt O, Kaiser M, et al. Detection of 2019 novel coronavirus (2019-nCoV) by real-time RT-PCR. Euro Surveill, 2020, 25(3). DOI:10.2807/1560-7917.ES.2020.25.3.2000045 |
| [15] |
Wrapp D, Wang NS, Corbett KS, et al. Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation. Science, 2020, 367(6483): 1260-1263. DOI:10.1126/science.abb2507 |
| [16] |
Yan R, Zhang Y, Li Y, et al. Structural basis for the recognition of SARS-CoV-2 by full-length human ACE2. Science, 2020, 367(6485): 1444-1448. DOI:10.1126/science.abb2762 |
| [17] |
Hoffmann M, Kleine-Weber H, Schroeder S, et al. SARS-CoV-2 cell entry depends on ACE2 and TMPRSS2 and is blocked by a clinically proven protease inhibitor. cell, 2020, S0092-8674(20): 30229-4. DOI:10.1016/j.cell.2020.02.052 |
| [18] |
Walls AC, Park YJ, Tortorici MA, et al. Structure, function, and antigenicity of the SARS-CoV-2 spike glycoprotein. Cell, 2020, S0092-8674(20): 30262-2. DOI:10.1016/j.cell.2020.02.05 |
 2020, Vol. 36
2020, Vol. 36




