中国科学院微生物研究所、中国微生物学会主办
文章信息
- 高建芳, 柯丽萍, 孙玉强
- Gao Jianfang, Ke Liping, Sun Yuqiang
- 基于花青素代谢培育蓝色花卉的研究进展
- Progress in the genetic modification of blue flowers based on anthocyanin metabolism
- 生物工程学报, ${metaVo.year}, 36(4): 678-692
- Chinese Journal of Biotechnology, ${metaVo.year}, 36(4): 678-692
- 10.13345/j.cjb.190335
-
文章历史
- Received: July 25, 2019
- Accepted: October 11, 2019
- Published: October 30, 2019
五彩缤纷的花朵,是大自然进化的杰作,更是人工选择和培育的结果。亿万年前,裸子植物主要依靠风力传粉,后来进化的被子植物有了花被,分化出五颜六色的花萼和花冠,有些还生有蜜腺,分泌汁液或散发香味,吸引昆虫或鸟类为其授粉[1-2]。此外,花色的形成能有效防御害虫和紫外线照射,对植物在自然界的生存和繁衍起重要作用。从观赏的角度来看,花色是观赏植物最重要的性状之一,是花卉育种者关注的焦点和选择的目标性状。然而,自然界中蓝色系花卉的数量极少,仅占所有花卉的15%-20%,并且只在极少数高度进化的被子植物家族中存在[3],所以在这种情况下,用传统的育种方法培育蓝色花朵非常困难。时至今日,玫瑰、菊花、百合和康乃馨等常见的观赏性花卉都无法利用传统的育种方法创造出蓝色的花朵,因为它们的基因组中不具有产生蓝色花系的基因,利用杂交、回交育种等方法难以实现蓝色花卉的培育[4-5]。
飞燕草色素是植物花青素的一种,早在1988年,黄胤怡等对18种蓝色花系植物花瓣的色素组成进行分析,发现飞燕草色素是蓝色花卉中最常见、最主要的花色素[6]。植物呈现蓝色,在一定程度上还与金属离子、花青素所在液泡中的pH值、花青素苷修饰和结构的多样性及环境因子等因素有关,但最重要的是飞燕草色素在花瓣细胞中的存在。在花青素合成代谢通路中,类黄酮3’, 5’羟基化酶(Flavonoid-3’, 5’-hydoxylase,F3’5’H)是产生蓝色花色素的关键酶,因此编码该酶的F3’5’H基因也被称为蓝色基因。另外,花青素的含量对花色的改变也有重要作用,Ohno等对15个不同颜色的大丽花品种进行色彩、黄酮类化合物成分和花青素含量测定分析,结果发现不同颜色的大丽花中含有的黄酮类化合物成分不同。进一步研究发现深紫色、紫色和粉红色品种的黄酮组成没有差异,花青素含量却依次降低,而在象牙白品种中未检测到花青素[7]。还有研究报道了在合成飞燕草色素苷的基础上进行后续的修饰才能得到蓝色色素[8]。
近年来,利用基因工程培育新型花卉的成功报道日益增多,定向改造花卉颜色的技术也发展迅速,这样就使得不同蓝色花系品种的培育成为可能。利用基因工程,可从以下几个途径来改良花卉的蓝色性状。一种是定量定性地控制花卉内源类黄酮色素的组成和含量,可通过RNAi、反义RNA技术和基因编辑等技术抑制与内源花青素合成相关的关键基因,大量积累无色底物,使花色变浅甚至无色,现在的基因编辑技术可以精确编辑相关基因,能更高效彻底降低本底色素关键酶基因的表达量。其次是积累花卉非天然色素,通过导入编码类黄酮3’, 5’-羟化酶的F3’5’H基因,使花卉累积飞燕草色素及衍生物,花卉颜色变为紫色或蓝色[9]。再者,通过调控花青素合成途径中转录因子的表达改变花色等性状[10-11],这也是改良花色的一条重要途径。此外,合成生物学可以系统设计分子模块,对植物体内的基础元件进行人工组装,构建出复杂的类黄酮色素合成、转运和沉积通路,改变花青素类代谢通路或代谢调控,使花色的改良更加可控。
文中将介绍植物花色素合成的主要途径,并对蓝色花形成的结构基因和调节基因进行详细的剖析,阐述花青素苷的结构修饰对蓝色花形成的影响,并且根据基因工程培育蓝色花的成功案例,简要归纳了不同基因工程改良策略,为蓝色花卉的培育提供理论参考,并为其他植物的色泽改良或花青素累积提供技术支持,如彩色棉蓝色纤维的培育等。棉花是一种不可替代的经济作物,彩色棉纤维具有天然色彩,在纺织过程中无需染色,可减少废水的排放和加工成本,并且避免化学染料对人体的伤害。但是天然彩色棉颜色类型单一,生产上只有绿色和棕色两种色系,这严重限制了彩棉行业的发展,因此,创造不同色泽的彩色棉具有重大意义。
1 植物花青素的结构与合成途径花青素是广泛存在于自然界中的一种植物水溶性色素,是一类重要的黄酮类化合物[12]。花青素是糖基化的多酚类化合物,在植物花朵、种子、果实等组织中,呈现出橙色、红色、紫色和蓝色等不同颜色[13]。自然界中天然存在的花青素有250多种,已经确定的有20种,在植物中常见的有6种[14],分别为天竺葵色素(Pelargonidin,Pg)、矢车菊色素(Cyanidin,Cy)、飞燕草色素(Delphindin,Dp)、芍药色素(Peonidin,Pn)、锦葵色素(Malvidin,Mv)和矮牵牛色素(Petunidin,Pt)[15-16]。
1.1 花青素的结构花青素的基本结构为α-苯基苯丙吡喃阳离子,具有一个基本的C6(A环)-C3(C环)-C6(B环)碳骨架结构[17]。由于环上不同位置发生的甲基化和羟基化等修饰,主要为B环上R1和R2位置的基团不同,从而形成不同的花青素类型并呈现出不同的颜色(表 1)[18]。花青素的颜色随着花青素B环上羟基的数量变化,在B环中有3’, 4’, 5’-OH三个取代基,飞燕草色素是通过B环3’和5’位置的羟化形成的,但多数缺乏蓝色基调的花都没有飞燕草色素。而红色和洋红色的花含有天竺葵基或矢车菊基的花青素,含有4’-一羟基取代基或3’, 4’-二羟基取代基[19]。
| Basic structure of anthocyanins | Anthocyanidin | R1 | R2 |
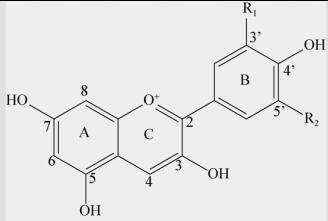 |
Pelargonidin (Pg) | H | H |
| Cyanidin (Cy) | OH | H | |
| Delphindin (Dp) | OH | OH | |
| Peonidin (Pn) | OCH3 | H | |
| Petunidin (Pt) | OH | OCH3 | |
| Malvidin (Mv) | OCH3 | OCH3 |
花青素的生物合成途径是研究较为成熟的苯丙氨酸类化合物途径(图 1),具体的代谢途径始于一个由苯丙氨酸经过一系列反应合成的4-香豆酰辅酶A (4-coumaroyl-CoA)分子与脂肪酸代谢产生的3个丙烯辅酶A分子(Malonyl-CoA)的缩合,形成黄色的查尔酮[20],该反应由查尔酮合成酶(Chalcone synthase,CHS)催化。然后该途径继续由黄烷酮-3-羟化酶(Flavanone-3- hydroxylase,F3H),类黄酮-3’-羟化酶(Flavanone-3’-hydroxylase,F3’H)和类黄酮-3’, 5’-羟化酶(Flavanone-3’5’- hydroxylase,F3’5’H)催化反应,分别形成不同的无色类黄酮类产物,再经二氢黄酮醇-4-还原酶(Dihydroflavonol 4 reductase,DFR)的作用分别形成无色天竺葵素、无色矢车菊素和无色飞燕草色素[21-22],形成的无色花青素再经无色花青素双加氧酶/花青素合成酶(Leucoanthocyanidin dioxygenase/ Anthocyanidinsynthase,LDOX/ANS)的作用形成有色的花色素。其中矢车菊色素和飞燕草色素在甲基转移酶(O-methyltransferase, OMT)作用下,由矢车菊色素在3’位置甲基化形成芍药色素,由飞燕草色素在5’位置甲基化形成矮牵牛色素,在3’和5’位置甲基化形成锦葵色素[13],最后花青素在类黄酮-3-O-糖苷转移酶(UDP glucose- flavonoid-3-O-glycosyltransferase,UFGT)的作用下与一个或多个葡萄糖、鼠李糖、半乳糖、木糖和阿拉伯糖等形成糖苷键,最终转化为稳定的花色素苷[23-24]。
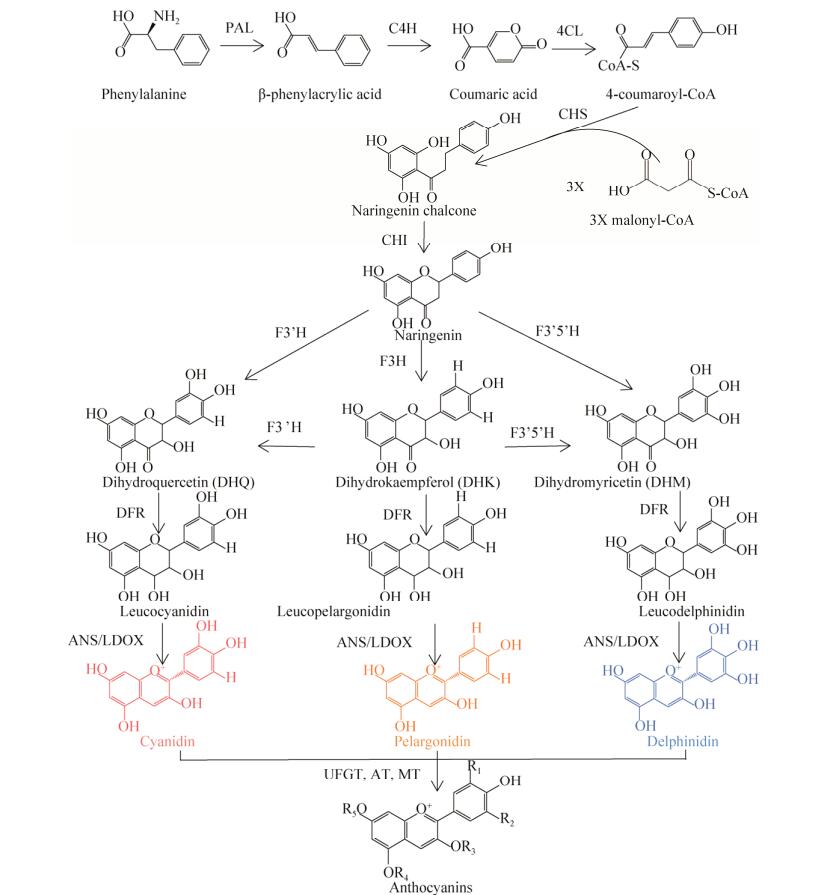
|
| 图 1 类黄酮生物合成途径及类黄酮化合物在花卉中的积累[13, 20] Fig. 1 Biosynthesis pathway of flavonoids and accumulation of flavonoid compounds in flowers[13, 20]. PAL: Phenylalanine ammonialyase; C4H: Cinnamate 4-hydroxylase; 4CL: 4-coumarate CoAligase; CHS: Chalconesynthase; CHI: Chalconeisomerase; F3H: Flavanone-3-hydroxylase; F3'H: Flavanone-3'-hydroxylas; F3'5'H: Flavanone 3'5'-hydroxylase; DFR: Dihydroflavonol 4 reductase; LDOX/ANS: Leucoanthocyanidin dioxygenase/ Anthocyanidinsynthase; UFGT: Flavonoid-3-O-glycosyltransferase. |
| |
花青素苷的合成主要在内质网表面进行,随后需要有效地向液泡中转运并贮藏,这一过程的异常也会影响植物的着色[25]。花青素苷的液泡转运方式主要有3种:一是在谷胱甘肽转移酶(Glutathione S-transferase,GST)的协助下经液泡膜上的多药耐药抗性相关蛋白(Multidrug resistance associated protein,MRP)类转运蛋白识别后将其跨膜转运至液泡;二是由液泡膜上的多药和有毒化合物排出家族(Multidrug and toxic compound extrusion,MATE)类转运蛋白将其跨膜转运到液泡中;三是由囊泡包裹通过膜融合的方式进入液泡。这几种转运机制可能同时进行,具体的作用机制尚不清楚[26-27]。花青素的转运过程及作用机理非常复杂,金属离子的络合作用、液泡的pH值、温度、光照等环境因子也会影响花青素最终的呈色,但起决定性作用的是代谢途径中的结构基因和调控因子及其互作[8, 28-29]。
1.2.2 飞燕草色素合成相关的结构基因在各种植物中,花青素合成途径中的结构基因直接影响合成花青素的种类,编码催化花青素合成过程中的各种酶,包括大多数植物中的保守基因群(PAL、C4H、4CL、CHS、CHI、F3H等)和一些植物中编码合成不同花青素单体合成的基因群(F3’H、F3’5’H、DFR、ANS和UFGT等),其中与飞燕草色素的合成和积累相关的基因有F3’H、F3’5’H和DFR[29]。在棉花中,天然彩色棉纤维的形成主要是由不同的花青素及其衍生物在纤维腔中累积而呈现不同色泽。Xiao等从棕色棉中克隆了GhCHI、GhF3H、GhDFR、GhANS和GhANR等5个结构基因,这些类黄酮合成途径上的关键基因在棕色棉纤维中的表达量明显高于在白色棉中的表达量[30]。Feng等研究表明GhCHS、GhC4H、GhF3’H和GhF3’5’H四个类黄酮合成途径上的关键基因,在棕色棉纤维中的表达量明显高于在白色棉中的表达量,HPLC分析表明柑橘素(Naringenin)、槲皮素(Quercetin)、莰非醇(Kaempferol)和杨梅素(Myricetin)四种黄酮类化合物在棕色纤维中的积累量明显高于在白色棉纤维中的积累量[31]。Feng等通过对提取、分离棕色棉纤维色素物质的NMR鉴定,发现棕色棉色素类物质为原花青素,且构成原花青素的主要结构单元是表棓儿茶素,同时有少量的表儿茶素结构单元,由于棕色棉纤维发育过程中,早期棕色棉的纤维是白色的,直到纤维发育到脱水时期,纤维才逐渐转变为棕色,推断这与原花青素的氧化聚合有关[32]。根据我们的研究,在彩色棉纤维花青素的合成代谢通路中,F3’H、F3’5’H和DFR三个基因与矢车菊素和飞燕草色素的积累有密切的关系。
在花青素生物合成途径中,F3’H和F3’5’H基因分别编码合成偏红色色调的矢车菊色素和偏蓝色色调的飞燕草色素,是决定花青素种类的关键基因。F3’H和F3’5’H均属于细胞色素P450单加氧酶[4, 33-34],分别催化类黄酮B环3’-位和3’5’-位发生羟基化反应,二者竞争底物二氢莰非醇(Dihydrokaempferol,DHK),决定类黄酮的最终产物,是确定形成花色的重要步骤和关键酶[35]。F3’H和F3’5’H的表达丰度是影响花青素合成的重要因素,F3’5’H高丰度表达,F3’H低丰度表达,能有效合成以飞燕草色素为主的蓝色系花。因在缺乏F3’5’H基因的植物中,飞燕草色素的合成受阻而不能形成蓝色花系,所以F3’5’H又被称为“蓝色基因”[36]。
DFR是合成后期表达的一个关键基因,F3’H和F3’5’H基因发挥作用后形成不同分支途径,DFR在辅因子NADPH的作用下将第4位羰基还原为羟基,催化二氢槲皮醇(Dihydroquercetin,DHQ)、二氢莰非醇(Dihydrokaempferol,DHK)和二氢杨梅黄酮(Dihydromyricetin,DHM)分别生成无色矢车菊素、无色天竺葵色素和无色飞燕草色素[37]。有研究显示,DFR对不同底物具有选择催化性[38],如在矮牵牛中,DFR首先催化DHM生成无色飞燕草色素,其次是DHQ,但并不能催化DHK,因此矮牵牛不能生成天竺葵色素,而将外源的玉米DFR基因导入矮牵牛,便可以催化DHK生成天竺葵色素[39]。
因此,F3’H和F3’5’H活性的强弱和DFR对底物的选择性等因素共同导致了花青素组成的改变,目前基因工程创制蓝色花系的过程基本都是围绕这3个酶的编码基因进行[40]。
1.2.3 花青素合成途径转录水平的调控植物花青素的合成与转录调控因子有重要关系。目前已经报道参与花青素合成转录水平调控的调节基因至少有3类,分别是MYB基因家族、编码碱性螺旋-环-螺旋(bHLH)的基因家族(也被称为MYC基因家族)和WD40基因家族。这三大家族基因形成协同调控系统,与结构基因的启动子结合来调控花青素的合成代谢,还影响许多双子叶植物的花色素沉积[41-42]。
MYB转录因子包含保守的DNA结合区域(MYB结构域),根据MYB结构域的重复数不同可分成3种:含1个结构域的R3-MYB、含2个结构域的R2R3-MYB和含3个结构域的R1R2R3-MYB[43-44],其中起主要作用的是R2R3-MYB[45]。在一些双子叶植物中,如拟南芥的R2R3-MYB转录因子可以独立调控花青素生物代谢过程中早期基因的表达,不需要其他辅因子的参与,在后期则需要由MYB-bHLH-WD40转录因子组成的三元复合体共同调控起作用。而在玉米中花青素的整个合成途径中,3类转录因子均有不同程度的参与[46]。葡萄中的R2R3-MYB调控因子会诱导F3’5’H等结构基因表达量上调,使果皮中的花青素大量合成[47]。此外,将拟南芥MYB家族的PAP1基因转入烟草中并超量表达,发现烟草的转基因植株在叶片、茎和花瓣上出现了不同程度的紫红色。在月季中,R2R3-MYB调控因子可激活多个结构基因的表达,使得叶片和花瓣的花青素沉积[48],MYB蛋白是调控发育、代谢和应答的调控网络中的关键因子[49]。
在植物中,bHLH蛋白是第二大转录因子类,在调节次生代谢、细胞分化和形态建成、根中的铁吸收和植物色素介导的光信号等过程中发挥着非常重要的作用,一般与R2R3-MYB结合起作用[50-51]。
WD40重复蛋白的结构高度保守,是以约40个色氨酸-天冬氨酸(W-D)的氨基酸重复结构WD基元,一般含有4-16个串联重复的基元[52-53]。这类蛋白缺少DNA结合域,并不能直接识别基因的启动子,而是其他蛋白互作的纽带,将苦荞麦的FtWD40基因转入烟草中,花瓣的颜色明显加深,且DFR和ANS的表达量上调[54]。
研究表明,3类转录因子还可以结合组成一个MYB-bHLH-WD40三元复合体(MBW复合物)共同调控黄酮类化合物的生物合成途径,MBW复合物主要用于调控晚期类黄酮生物合成基因,对结构基因的激活能力比单独的转录因子作用强[46, 55]。WD40蛋白通过与R2R3-MYB相互作用的bHLH转录因子结合成稳定MBW的复合物,而MBW复合物组成成分的多样化会导致类黄酮的合成通路调控模式发生细微的变化。因此,MBW复合物的准确组装也会影响黄酮类色素的产生、生物合成途径基因的激活以及靶基因表达的强度[56-57]。在构成MBW复合物的3种蛋白中,R2R3-MYB被认为直接与花青素生物合成相关的DNA靶点结合[58-59]。这些转录因子基因超表达可以增加花青素合成的含量,在此基础上间接地促进飞燕草色素的合成,为基因工程和模块化分子育种培育蓝色花奠定基础[60-61]。
1.2.4 花青素合成途径转录后水平的调控转录后调控是在mRNA基础上加工、翻译等过程中的调控,包括蛋白质泛素化、MicroRNA (miRNA)以及小干扰siRNA (Small interfering RNAs)介导的转录后基因沉默。在拟南芥中发现,重要光形态建成因子COP1 (Constitutively photomorphogenic 1,COP1)和SPA (Suppressor of phyA)通过泛素/26S蛋白酶体系统(Ubiquitin/ 26S proteasome system,UPS)靶向结合多个花青素苷合成的关键活性因子如PAP1、PAP2、HY5及其同源物HYH抑制光诱导的花青素积累[62-63]。
呈发夹结构的单链RNA前体经过Dicer酶加工后生成miRNA后,与靶基因RNA序列互补将其切割或抑制其翻译来发挥转录后水平的调控作用[64]。Yang等利用miR828过表达载体转化拟南芥,表明miR828/TAS4-siR81(-)能通过抑制花青素正调控因子PAP1、PAP2、MYB113的表达进而负调控花青素的积累[65]。在拟南芥中控制莲座丛与茎的连接部位花青素积累的是基因SPL9 (Squamosa promoter binding protein-like 9),SPL9通过降低MYB-bHLH-WD40复合物稳定性,抑制花青素合成积累,miR156能明显抑制SPL9表达,激活合成花青素关键酶基因以及转录因子家族基因的表达,使花青素合成增加[66-67]。
与miRNA不同,siRNA来源于双链RNA前体,通过互补配对降解靶标转录本[64, 68]。Morita等在双色牵牛花中两个不同的颜色区域检测到CHS-A的前体RNA,但在白色区域中却没有CHS-A的成熟RNA,却存在含有21个核苷酸的siRNA,可能是由于siRNA介导抑制了CHS-A的表达导致了双色牵牛花的出现[69]。
1.2.5 花青素苷的分子修饰植物花青素苷B环的羟基水平越高结构越不稳定[70],需要经过后续的分子修饰来使其结构更加稳定,分子修饰的作用因修饰的种类、数量和位置而异。糖基化能提高花青素的稳定性和水溶性,糖基转移酶通常将糖基转移到花青素分子的C3位羟基上,有些糖基也会转移到其他位置,如C5、C7位的羟基上。C3位的糖基化提高了花青素的稳定性,双糖苷元的稳定性强于单糖苷元,还会将颜色稍微转向红色,而C5的糖基化会降低色素的强度[20, 70]。糖基化结束后才能形成显色的花青素苷,花青素苷合成后的分子修饰增加了结构和颜色的多样性及稳定性。
甲基化修饰通常发生于花青素苷的C3’和C5’位羟基上,由于甲基化替代了羟基化,羟基化的数量减少,使得最后的颜色偏红[71]。
酰基化修饰使花青素苷的稳定性和水溶性更强,避免在中性或酸性溶液中褪色[72],包括芳香族酰基化和脂肪族酰基化,其中芳香族酰基化修饰还能改变花青素的吸收波长,使其颜色向蓝色转变,且分子间的堆积力使蓝色更加稳定[71, 73-74]。
2 蓝色花的分子改良途径与蓝色相关的基因表达、转录因子的调控作用和花青素的分子修饰等因素都与蓝色花的形成有关,结合这些影响因素,通过基因操纵的基因工程创制蓝色花主要有以下几种策略:其一是定量定性的控制花瓣中的内源性黄酮类色素,其二是积累体内非天然的飞燕草色素[9],其三是利用转录因子基因调控来改良花色[75],再者利用合成生物学技术重组生命体内元件来改变花青素合成通路,这几种策略均能有效改良花色[76] (表 2)。
| Genetic engineering strategy | Gene source | Experimental plant | Produced color | References |
| Endogenous F3’5’H | Petunia hybrida | Petunia hybrida (Light pink) |
Reddish violet | [77] |
| F3’5’H | Vinca major | Petunia hybrida | Nearly black flower | [78] |
| F3’5’H, Introduced | Petunia hybrida | Petunia hybrida (Pink) |
Reddish-purple, variegated chimeric color |
[79] |
| CHS, Introduced | Petunia hybrida | Petunia hybrida (Light pink) |
White, purple and white | [80] |
| CHS, RNAi | Gentiana scabra | Gentiana sp (Blue) | Pale blue to white | [81] |
| ANS, RNAi | Gentiana scabra | Gentiana sp Blue) | Pale blue | [81] |
| F3’5’H, RNAi | Gentiana scabra | Gentiana sp (Blue) | Lilac to pale blue | [82] |
| ANS, RNAi | Petunia hybrida | Petunia hybrid (Blue) |
White to pale blue | [83] |
| CHS, Antisense | Gentiana scabra | Gentiana sp (Blue) | White | [83] |
| ANS, Antisense | Petunia hybrida | Petunia hybrida (Blue) |
Pale blue | [83] |
| F3’5’H, Over expression | Viola sp. | Rosa hybrid (Red to pale cyanic) |
Pale blue | [84] |
| F3’H, RNAi & F3’5’H, Introduced |
Viola sp. | Chrysanthemum | Blue | [85] |
| F3'5'H, A3'5'GT Introduced | Campanula latifolia, Clitoria ternatea |
Chrysanthemum (pink) | Blue | [86] |
| F3’5’H, Over expression | Phalaenopsis amabilis | Lilium brownii (pink) | Lavender | [87-88] |
控制内源性黄酮类色素的策略最直接的方法是减少内源花色素的含量,通过抑制或敲除编码这些关键酶的基因,控制关键酶的合成,如抑制花青素代谢途径中的CHS、F3’H、F3’5’H、DFR、ANS等基因来减少花青素的累积,产生白色或浅色花。这种策略可以通过RNAi技术、反义RNA技术及基因编辑技术等方法来实现。此外,过表达负转录调控因子也可以抑制花瓣中花青素的形成[89-90]。在产生浅色花的基础上再导入外源基因,合成蓝色飞燕草色素单体来改良蓝色花卉,在控制内源基因表达的过程中会产生不同的颜色,如白蓝相间,这对于丰富花卉的颜色具有重要意义。
2.1.1 RNAi技术RNAi技术是通过外源性或细胞内源性的双链RNA介导的转录后基因沉默技术干涉目标基因的表达。沉默后,目标基因的表达量一般会降低,进而影响编码相应酶的含量。Nishihara等利用RNAi技术对龙胆的同源基因CHS、ANS、F3’5’H、DFR和A3’5’GT分别进行基因沉默,结果发现蓝色花变成不同的浅色花[81-82]。
2.1.2 反义RNA技术反义RNA技术可将目的基因的反义链插入表达载体中,导入植物体内后,反向DNA转录的RNA与内源的互补mRNA结合,降解或阻止其作为模板合成蛋白质,影响关键酶或转录因子的合成从而改变花色。vander Krol等首次运用这个技术将CHS基因的反义链导入矮牵牛中,抑制了花青素苷前体的合成,使得花色变浅[91]。Nakamuraet等运用反义RNA技术抑制蓝色龙胆中CHS基因的表达,得到了白色的龙胆,又用同样的方法降低蓝色矮牵牛中ANS基因的表达量,最终花瓣颜色转变为淡蓝色。由于抑制上游基因会导致类黄酮类物质的缺失,转基因植物在抵御紫外线、逆境以及信号转导等方面的能力大大降低,不利于植物生长发育等,因此利用该方法的研究重点转向抑制花青素代谢通路中的下游基因[80]。
2.1.3 基因编辑技术CRISPR (Clustered regularly interspaced short palindromic repeats)技术问世以来一直是生物界的焦点,发展至今的CRISPR/Cas9系统已成功用于多种生物的定向编辑和诱变,增加反向遗传学研究手段,特别是对于具有成熟转化系统和已知高质量基因组序列的植物[92-93]。Watanabe等利用CRISPR/Cas9系统对日本牵牛花花青素合成途径中的DFR-B基因进行双等位基因突变[94]。在牵牛花中,DFR-A、DFR-B和DFR-C是一个排列整齐的三基因家族,3个基因均有表达,但DFR-B基因为主要基因,是负责茎和花色素沉积的显色基因,该基因编辑的结果显示茎和花的颜色均变浅且花青素含量减少,后代符合遗传规律。该研究首次利用CRISPR/Cas9系统实现了高等植物花色变化的调控。
2.2 引入外源基因合成非天然类黄酮色素有些植物中缺少合成飞燕草色素的关键基因F3’5’H和DFR,因此通过导入植物本身不含有的外源结构基因使其表达产生飞燕草色素是一种有效获得蓝色花系的途径。在此基础上,将影响飞燕草色素合成的相关基因过量表达,增加飞燕草色素及衍生物的含量。选择合适来源的F3’5’H基因对合成途径有重要影响,将三色堇和牵牛花的F3’5’H基因导入康乃馨、玫瑰和菊花导致飞燕草色素的积累,检测到三色堇F3’5’H基因比牵牛花的F3’5’H基因产生飞燕草素的效果更好[4, 85],而且表达载体和启动子与导入基因的有效组合也会显著影响最终的显色结果[95]。
培育蓝色花卉,通常将控制内源性黄酮类色素与导入外源基因合成飞燕草色素结合起来。内源性F3’H与引入外源基因合成的F3’5’H竞争底物,为了使花瓣颜色进一步向紫色或蓝色靠近,为了更好地提高飞燕草素含量,先沉默F3’H基因,降低F3’H的含量,在此基础上过量表达F3’5’H基因,这样可使飞燕草色素及衍生物有效的积累[85]。
2.3 花青素合成调控相关转录因子的基因工程花青素合成途径中转录因子的改变可能会导致植物众多性状的改变,包括花瓣颜色。Butelli等在番茄中导入了金鱼草的Rose1和Del基因(分别编码MYB和bHLH类),发现果实中的花青素含量增加,并且出现了紫色[96]。Han等将玉米中编码bHLH类转录因子的Lc基因和编码MYB类转录因子的Pl基因分别或共同转入匍匐剪股颖中,结果发现转基因植株在整个植株出现了不同程度的紫色[75]。Ohno等在对大丽花的研究中发现,siRNA介导的CHS基因转录后基因沉默会导致大丽花呈现纯白色[97]。
2.4 利用合成生物学创造蓝色花卉合成生物学是在自然界中已有生命系统的基础上,重新设计和构建生命体系,优化自然生命[98]。已有研究证明了利用合成生物学技术可成功合成黄酮类化合物,通过在大肠杆菌或者酵母中表达一些关键酶基因,添加外源的前体物质香豆酸、苯丙氨酸、肉桂酸等,可将一些常见的底物转变为柚皮素[99]和无色花青素[100]等次级代谢产物[101]。Ankanahalli等利用微生物非核糖体肽合成酶(Nonribosomal peptide synthetase,NRPS)合成了蓝色的色素苷,生成蓝色玫瑰[76]。每个非核糖体肽都是由特定NRPS产生的,NRPS含一组特定的结构形成一个新的生命系统,在连续的多步酶促过程中合成肽,参与花青素生物合成。
3 总结与展望目前,蓝色花系形成分子机理的研究比较深入,也获得了很多跨越式的进展,利用基因工程培育出许多自然界中没有的蓝色花卉,像蓝色玫瑰、康乃馨、菊花等植物已经商业化,而且不同基因工程策略结合起来,有利于增加花卉色泽的丰富度,如产生白蓝相间的花。
然而,进一步利用基因工程改良花色仍有很大的困难。首先,对影响飞燕草色素合成的转录因子所知甚少,外源基因转入受体植物体内,往往在一些转录因子的影响下,并未达到预期的效果。目前,调控花青素代谢通路的转录因子所知甚少,应该着重研究更多与花青素代谢有关的转录调控因子。其次,还要关注转录因子调节下游靶基因和信号通路,转录因子被激酶磷酸化修饰等方面,进一步了解转录调控的机制,完善花青素代谢通路中的转录调控网络。第二,关于花青素苷的甲基化、酰基化等修饰研究的很少,但却是影响蓝色花形成的一大因素。针对这一问题,要先对花青素苷的分子修饰进行进一步的研究,如在花青素代谢通路中,甲基化会替代羟基化,降低甲基化的修饰会反馈调控羟基化修饰,促进天竺葵色素、矢车菊色素和飞燕草色素的合成,结合其他方法累积飞燕草色素。第三,物种特异性导致了基因工程育种的进展缓慢,且结果存在着不稳定性,不同的植物之间存在很大的差异,同一种植物转基因后出现的结果也不相同。今后可加强对不同植物或同一植物不同时空花青素代谢的特异性和共性分析,并找出其共性规律和导致物种特异性的原因,利用好物种特异性来丰富花卉性状的多样性。第四,能高效合成飞燕草色素的基因资源很少,今后的研究应该倾向挖掘更多能形成蓝色花的基因,同时也不能忽视野生蓝色花卉的基因资源,在此基础上通过人工育种建立基因资源库很有必要。
此外,蓝色花形成调控网络的研究为其他植物提供了理论基础和技术支持,包括纺织纤维材料彩色棉,彩色棉花青素合成代谢通路中包含了合成飞燕草色素的基因。目前对棉花花青素的研究也比较缺乏,而将蓝色和彩色棉纤维色泽改良联系起来,研究蓝色纤维的彩色棉花尚未开展,因此研制彩棉“蓝色妖姬”具有重要的研究意义和社会生产价值。利用分子生物学和基因工程的技术手段构建RNAi载体对F3’H基因进行沉默使其表达量降低。还可以构建RISPR/Cas9载体,这样可以避免沉默靶基因的同源序列或冗余序列的问题,定点敲除F3’H基因。同时构建超表达载体超量表达F3’5’H基因使二氢莰非醇生成二氢杨梅黄酮,达到累积飞燕草色素的目的,得到转基因植株后,筛选飞燕草色素大量累积的棉花品种。然而,蓝色彩棉纤维的研制也依然存在着很大的挑战,天然彩色棉纤维色素形成的生化和分子机制尚不清楚,调控彩棉纤维色素累积基因的表达和调控途径错综复杂,累积色素的物质组成与沉积机理也不明晰,飞燕草色素还要经过复杂修饰过程和转录因子的调控等。
| [1] |
Chittka L, Menzel R. The evolutionary adaptation of flower colours and the insect pollinators' colour vision. J Comp Physiol A, 1992, 171(2): 171-181. |
| [2] |
Rausher MD. The evolution of flavonoids and their genes//Grotewold E, Ed. The Science of Flavonoids. New York: Springer, 2006, 175-211. |
| [3] |
Mackenzie JMD. Fluctuations in the numbers of British Tetraonids. J Anim Ecol, 1952, 21(1): 128-153. DOI:10.2307/1914 |
| [4] |
Tanaka Y. Flower colour and cytochromes P450. Phytochem Rev, 2006, 5(2/3): 283-291. |
| [5] |
Tanaka Y, Brugliera F. Metabolic engineering of flower color pathways using cytochromes P450//Yamazaki H, Ed. Fifty Years of Cytochrome P450 Research. Tokyo: Springer, 2014, 207-229. |
| [6] |
Huang YY, Shen MS, Li P, et al. Gene engineering of blue color flower. Plant Physiol Commun, 2002, 38(2): 203-206 (in Chinese). 黄胤怡, 沈明山, 李鹏, 等. 蓝色花的基因工程. 植物生理学报, 2002, 38(2): 203-206. |
| [7] |
Ohno S, Deguchi A, Hosokawa M, et al. A basic helix-loop-helix transcription factor DvIVS determines flower color intensity in cyanic dahlia cultivars. Planta, 2013, 238(2): 331-343. DOI:10.1007/s00425-013-1897-x |
| [8] |
Noda N. Recent advances in the research and development of blue flowers. Breed Sci, 2018, 68(1): 79-87. DOI:10.1270/jsbbs.17132 |
| [9] |
Nishihara M, Nakatsuka T. Genetic engineering of flavonoid pigments to modify flower color in floricultural plants. Biotechnol Lett, 2011, 33(3): 433-441. DOI:10.1007/s10529-010-0461-z |
| [10] |
Cronk QCB. Plant evolution and development in a post-genomic context. Nat Rev Genet, 2001, 2(8): 607-619. DOI:10.1038/35084556 |
| [11] |
Schwinn K, Venail J, Shang YJ, et al. A small family of MYB-Regulatory genes controls floral pigmentation intensity and patterning in the genus Antirrhinum. Plant Cell, 2006, 18(4): 831-851. DOI:10.1105/tpc.105.039255 |
| [12] |
Tanaka Y, Sasaki N, Ohmiya A. Biosynthesis of plant pigments: anthocyanins, betalains and carotenoids. Plant J, 2008, 54(4): 733-749. DOI:10.1111/j.1365-313X.2008.03447.x |
| [13] |
Liu Y, Tikunov Y, Schouten RE, et al. Anthocyanin biosynthesis and degradation mechanisms in Solanaceous vegetables: a review. Front Chem, 2018, 6: 52. DOI:10.3389/fchem.2018.00052 |
| [14] |
Smeriglio A, Barreca D, Bellocco E, et al. Chemistry, pharmacology and health benefits of anthocyanins. Phytother Res, 2016, 30(8): 1265-1286. DOI:10.1002/ptr.5642 |
| [15] |
Zhao CL, Chen ZJ, Bai XS, et al. Structure-activity relationships of anthocyanidin glycosylation. Mol Divers, 2014, 18(3): 687-700. DOI:10.1007/s11030-014-9520-z |
| [16] |
Kong JM, Chia LS, Goh NK, et al. Analysis and biological activities of anthocyanins. Phytochemistry, 2003, 64(5): 923-933. DOI:10.1016/S0031-9422(03)00438-2 |
| [17] |
De Brito ES, De Araújo MC, Alves RE, et al. Anthocyanins present in selected tropical fruits: Acerola, Jambolúo, Jussara, and Guajiru. J Agric Food Chem, 2007, 55(23): 9389-9394. DOI:10.1021/jf0715020 |
| [18] |
Dai SL, Hong Y. Molecular breeding for flower colors modification on ornamental plants based on the mechanism of anthocyanins biosynthesis and coloration. Sci Agric Sin, 2016, 49(3): 529-542 (in Chinese). 戴思兰, 洪艳. 基于花青素苷合成和呈色机理的观赏植物花色改良分子育种. 中国农业科学, 2016, 49(3): 529-542. DOI:10.3864/j.issn.0578-1752.2016.03.011 |
| [19] |
Jaakola L. New insights into the regulation of anthocyanin biosynthesis in fruits. Trends Plant Sci, 2013, 18(9): 477-483. DOI:10.1016/j.tplants.2013.06.003 |
| [20] |
Zhang Y, Butelli E, Martin C. Engineering anthocyanin biosynthesis in plants. Curr Opin Plant Biol, 2014, 19: 81-90. DOI:10.1016/j.pbi.2014.05.011 |
| [21] |
Devic M, Guilleminot J, Debeaujon I, et al. The BANYULS gene encodes a DFR-like protein and is a marker of early seed coat development. Plant J, 1999, 19(4): 387-398. DOI:10.1046/j.1365-313X.1999.00529.x |
| [22] |
Huang H, Hu K, Han KT, et al. Flower colour modification of chrysanthemum by suppression of F3'H and overexpression of the exogenousSenecio cruentus F3'5'H gene. PLoS ONE, 2013, 8(11): e74395. DOI:10.1371/journal.pone.0074395 |
| [23] |
Hou ZH, Wang SP, Wei SD, et al. Anthocyanin biosynthesis and regulation in plants. Guihaia, 2017, 37(12): 1603-1613 (in Chinese). 侯泽豪, 王书平, 魏淑东, 等. 植物花青素生物合成与调控的研究进展. 广西植物, 2017, 37(12): 1603-1613. DOI:10.11931/guihaia.gxzw201705002 |
| [24] |
Fu HH, Xin PY, Xu YL, et al. Bioinformatics analysis of UFGT gene from several economic plants. Genomics Appl Biol, 2011, 30(1): 92-102 (in Chinese). 付海辉, 辛培尧, 许玉兰, 等. 几种经济植物UFGT基因的生物信息学分析. 基因组学与应用生物学, 2011, 30(1): 92-102. DOI:10.3969/gab.030.000092 |
| [25] |
Zhao J, Dixon RA. The 'ins' and 'outs' of flavonoid transport. Trends Plant Sci, 2010, 15(2): 72-80. DOI:10.1016/j.tplants.2009.11.006 |
| [26] |
Gomez C, Conejero G, Torregrosa L, et al. In vivo grapevine anthocyanin transport involves vesicle-mediated trafficking and the contribution of anthoMATE transporters and GST. Plant J, 2011, 67(6): 960-970. DOI:10.1111/j.1365-313X.2011.04648.x |
| [27] |
Wang L, Dai SL, Jin XH, et al. Advances in plant anthocyanin transport mechanism. Chin J Biotech, 2014, 30(6): 848-863 (in Chinese). 王璐, 戴思兰, 金雪花, 等. 植物花青素苷转运机制的研究进展. 生物工程学报, 2014, 30(6): 848-863. |
| [28] |
Shoji K, Momonoi K, Tsuji T. Alternative expression of vacuolar iron transporter and ferritin genes leads to blue/purple coloration of flowers in tulip cv. 'Murasakizuisho'. Plant Cell Physiol, 2010, 51(2): 215-224. DOI:10.1093/pcp/pcp181 |
| [29] |
Zhuang WB, Liu TY, Shu XC, et al. The molecular regulation mechanism of anthocyanin biosynthesis and coloration in plants. Plant Physiol J, 2018, 54(11): 1630-1644 (in Chinese). 庄维兵, 刘天宇, 束晓春, 等. 植物体内花青素苷生物合成及呈色的分子调控机制. 植物生理学报, 2018, 54(11): 1630-1644. |
| [30] |
Xiao YH, Zhang ZS, Yin MH, et al. Cotton flavonoid structural genes related to the pigmentation in brown fibers. Biochem Biophys Res Commun, 2007, 358(1): 73-78. DOI:10.1016/j.bbrc.2007.04.084 |
| [31] |
Feng HJ, Tian XH, Liu YC, et al. Analysis of flavonoids and the flavonoid structural genes in brown fiber of upland cotton. PLoS ONE, 2013, 8(3): e58820. DOI:10.1371/journal.pone.0058820 |
| [32] |
Feng HJ, Li YJ, Wang SF, et al. Molecular analysis of proanthocyanidins related to pigmentation in brown cotton fibre (Gossypium hirsutum L.). J Exp Bot, 2014, 65(20): 5759-5769. DOI:10.1093/jxb/eru286 |
| [33] |
Han YP, Vimolmangkang S, Soria-Guerra RE, et al. Ectopic expression of apple F3'H genes contributes to anthocyanin accumulation in the Arabidopsis tt7 mutant grown under nitrogen stress. Plant Physiol, 2010, 153(2): 806-820. DOI:10.1104/pp.109.152801 |
| [34] |
Zhang SH, Li CQ, Guo HM, et al. Effects of Eupatorium adenophorum flavonoid 3'-hydroxylase over-expression on POD and PAL activity in transgenic tobacco. J Agric Sci Technol, 2009, 11(3): 98-101 (in Chinese). 张松焕, 李春奇, 郭惠明, 等. 过量表达紫茎泽兰类黄酮3'-羟化酶基因对转基因烟草POD、PAL活性的影响. 中国农业科技导报, 2009, 11(3): 98-101. DOI:10.3969/j.issn.1008-0864.2009.03.018 |
| [35] |
Wang YS, Xu YJ, Gao LP, et al. Functional analysis of flavonoid 3′, 5′-hydroxylase from tea plant (Camellia sinensis): critical role in the accumulation of catechins. BMC Plant Biol, 2014, 14: 347. DOI:10.1186/s12870-014-0347-7 |
| [36] |
Zhou TS, Yu YB, Xiao B, et al. Engineering of a flavonoid 3'-hydroxylase from tea plant (Camellia sinensis) for biosynthesis of B-3', 4'-dihydroxylated flavones. Acta Microbiol Sin, 2017, 57(3): 447-458 (in Chinese). 周天山, 余有本, 肖斌, 等. 操纵茶树类黄酮3'-羟基化酶生物合成B环-3', 4'-二羟基黄酮类化合物. 微生物学报, 2017, 57(3): 447-458. |
| [37] |
Lim SH, You MK, Kim DH, et al. RNAi-mediated suppression of dihydroflavonol 4-reductase in tobacco allows fine-tuning of flower color and flux through the flavonoid biosynthetic pathway. Plant Physiol Biochem, 2016, 109: 482-490. DOI:10.1016/j.plaphy.2016.10.028 |
| [38] |
Yoshida K, Iwasaka R, Shimada N, et al. Transcriptional control of the dihydroflavonol 4-reductase multigene family in Lotus japonicus. J Plant Res, 2010, 123(6): 801-805. DOI:10.1007/s10265-010-0325-6 |
| [39] |
Aharoni A, De Vos CH, Wein M, et al. The strawberry FaMYB1 transcription factor suppresses anthocyanin and flavonol accumulation in transgenic tobacco. Plant J, 2001, 28(3): 319-332. DOI:10.1046/j.1365-313X.2001.01154.x |
| [40] |
Fan RH, Huang ML. Progress in regulation of anthocyanins. Chin J Cell Biol, 2013, 35(5): 741-746 (in Chinese). 樊荣辉, 黄敏玲. 花青素苷调控研究进展. 中国细胞生物学学报, 2013, 35(5): 741-746. |
| [41] |
Sagawa JM, Stanley LE, LaFountain AM, et al. An R2R3-MYB transcription factor regulates carotenoid pigmentation in Mimulus lewisii flowers. New Phytol, 2016, 209(3): 1049-1057. DOI:10.1111/nph.13647 |
| [42] |
Yamagishi M, Shimoyamada Y, Nakatsuka T, et al. Two R2R3-MYB genes, homologs of petunia AN2, regulate anthocyanin biosyntheses in flower tepals, tepal spots and leaves of Asiatic hybrid lily. Plant Cell Physiol, 2010, 51(3): 463-474. DOI:10.1093/pcp/pcq011 |
| [43] |
Dubos C, Stracke R, Grotewold E, et al. MYB transcription factors in Arabidopsis. Trends Plant Sci, 2010, 15(10): 573-581. DOI:10.1016/j.tplants.2010.06.005 |
| [44] |
He XJ, Zhao XC, Gao LP, et al. Isolation and characterization of key genes that promote flavonoid accumulation in purple-leaf tea (Camellia sinensis L.). Sci Rep, 2018, 8(1): 130. DOI:10.1038/s41598-017-18133-z |
| [45] |
Buer CS, Imin N, Djordjevic MA. Flavonoids: new roles for old molecules. J Integr Plant Biol, 2010, 52(1): 98-111. DOI:10.1111/j.1744-7909.2010.00905.x |
| [46] |
Petroni K, Tonelli C. Recent advances on the regulation of anthocyanin synthesis in reproductive organs. Plant Sci, 2011, 181(3): 219-229. DOI:10.1016/j.plantsci.2011.05.009 |
| [47] |
Liu Y, Zheng TC, Dai LJ, et al. Construction of plant expression vector and genetic transformation analysis of Arabidopsis thaliana AtPAP1 gene in Nicotiana tabacum. Plant Physiol Commun, 2017, 53(7): 1199-1207 (in Chinese). 刘轶, 郑唐春, 代丽娟, 等. 拟南芥AtPAP1基因植物表达载体构建及在烟草中遗传转化分析. 植物生理学报, 2017, 53(7): 1199-1207. |
| [48] |
Boss PK, Davies C. Molecular biology of anthocyanin accumulation in Grape berries//Roubelakis-Angelakis KA, Ed. Grapevine Molecular Physiology & Biotechnology. Dordrecht: Springer, 2009, 263-292. |
| [49] |
Takahashi R, Yamagishi N, Yoshikawa N. A MYB transcription factor controls flower color in soybean. J Hered, 2013, 104(1): 149-153. DOI:10.1093/jhered/ess081 |
| [50] |
Lloyd A, Brockman A, Aguirre L, et al. Advances in the MYB-bHLH-WD repeat (MBW) pigment regulatory model: addition of a WRKY factor and co-option of an anthocyanin MYB for betalain regulation. Plant Cell Physiol, 2017, 58(9): 1431-1441. DOI:10.1093/pcp/pcx075 |
| [51] |
Feller A, Machemer K, Braun EL, et al. Evolutionary and comparative analysis of MYB and bHLH plant transcription factors. Plant J, 2011, 66(1): 94-116. DOI:10.1111/j.1365-313X.2010.04459.x |
| [52] |
Park KI, Hoshino A. A WD40-repeat protein controls proanthocyanidin and phytomelanin pigmentation in the seed coats of the Japanese morning glory. J Plant Physiol, 2012, 169(5): 523-528. DOI:10.1016/j.jplph.2011.12.001 |
| [53] |
Liu YJ, Hou H, Jiang XL, et al. A WD40 repeat protein from Camellia sinensis regulates anthocyanin and proanthocyanidin accumulation through the formation of MYB-bHLH-WD40 ternary complexes. Int J Mol Sci, 2018, 19(6): 1686. DOI:10.3390/ijms19061686 |
| [54] |
Yao PF, Zhao HX, Luo XP, et al. Fagopyrum tataricum FtWD40 functions as a positive regulator of anthocyanin biosynthesis in transgenic tobacco. J Plant Growth Regul, 2017, 36(3): 755-765. DOI:10.1007/s00344-017-9678-6 |
| [55] |
Schaart JG, Dubos C, De La Fuente IR, et al. Identification and characterization of MYB-bHLH-WD40 regulatory complexes controlling proanthocyanidin biosynthesis in strawberry (Fragaria×ananassa) fruits. New Phytol, 2013, 197(2): 454-467. DOI:10.1111/nph.12017 |
| [56] |
Xu WJ, Dubos C, Lepiniec L. Transcriptional control of flavonoid biosynthesis by MYB-bHLH-WDR complexes. Trends Plant Sci, 2015, 20(3): 176-185. DOI:10.1016/j.tplants.2014.12.001 |
| [57] |
Davies KM, Albert NW, Schwinn KE. From landing lights to mimicry: the molecular regulation of flower colouration and mechanisms for pigmentation patterning. Funct Plant Biol, 2012, 39(8): 619-638. DOI:10.1071/FP12195 |
| [58] |
Hichri I, Barrieu F, Bogs J, et al. Recent advances in the transcriptional regulation of the flavonoid biosynthetic pathway. J Exp Bot, 2011, 62(8): 2465-2483. DOI:10.1093/jxb/erq442 |
| [59] |
Fournier-Level A, Hugueney P, Verriès C, et al. Genetic mechanisms underlying the methylation level of anthocyanins in grape (vitis vinifera L.). BMC Plant Biol, 2011, 11: 179. DOI:10.1186/1471-2229-11-179 |
| [60] |
Ben-Simhon Z, Judeinstein S, Nadler-Hassar T, et al. A pomegranate (Punica granatum L.) WD40-repeat gene is a functional homologue of Arabidopsis TTG1 and is involved in the regulation of anthocyanin biosynthesis during pomegranate fruit development. Planta, 2011, 234(5): 865-881. DOI:10.1007/s00425-011-1438-4 |
| [61] |
Paolocci F, Robbins MP, Passeri V, et al. The strawberry transcription factor FaMYB1 inhibits the biosynthesis of proanthocyanidins in Lotus corniculatus leaves. J Exp Bot, 2011, 62(3): 1189-1200. DOI:10.1093/jxb/erq344 |
| [62] |
Li YY, Mao K, Zhao C, et al. MdCOP1 ubiquitin E3 ligases interact with MdMYB1 to regulate light-induced anthocyanin biosynthesis and red fruit coloration in apple. Plant Physiol, 2012, 160(2): 1011-1022. DOI:10.1104/pp.112.199703 |
| [63] |
Shin DH, Choi M, Kim K, et al. HY5 regulates anthocyanin biosynthesis by inducing the transcriptional activation of the MYB75/PAP1 transcription factor in Arabidopsis. FEBS Lett, 2013, 587(10): 1543-1547. DOI:10.1016/j.febslet.2013.03.037 |
| [64] |
Molnár A, Schwach F, Studholme DJ, et al. miRNAs control gene expression in the single-cell alga Chlamydomonas reinhardtii. Nature, 2007, 447(7148): 1126-1129. DOI:10.1038/nature05903 |
| [65] |
Yang FX, Cai J, Yang Y, et al. Overexpression of microRNA828 reduces anthocyanin accumulation in Arabidopsis. Plant Cell, Tissue Organ Cult, 2013, 15(2): 159-167. |
| [66] |
Eriksson M, Moseley JL, Tottey S, et al. Genetic dissection of nutritional copper signaling in Chlamydomonas distinguishes regulatory and target genes. Genetics, 2004, 168(2): 795-807. DOI:10.1534/genetics.104.030460 |
| [67] |
Gou JY, Felippes FF, Liu CJ, et al. Negative regulation of anthocyanin biosynthesis in Arabidopsis by a miR156-targeted SPL transcription factor. Plant Cell, 2011, 23(4): 1512-1522. DOI:10.1105/tpc.111.084525 |
| [68] |
Brodersen P, Voinnet O. The diversity of RNA silencing pathways in plants. Trends Genet, 2006, 22(5): 268-280. DOI:10.1016/j.tig.2006.03.003 |
| [69] |
Morita Y, Saito R, Ban Y, et al. Tandemly arranged Chalcone synthase A genes contribute to the spatially regulated expression of siRNA and the natural bicolor floral phenotype in Petunia hybrida. Plant J, 2012, 70(5): 739-749. DOI:10.1111/j.1365-313X.2012.04908.x |
| [70] |
Woodward G, Kroon P, Cassidy A, et al. Anthocyanin stability and recovery: implications for the analysis of clinical and experimental samples. J Agric Food Chem, 2009, 57(12): 5271-5278. DOI:10.1021/jf900602b |
| [71] |
Xie Y, Sun Y, Huang JR. Anthocyanin modification in Arabidopsis. Plant Physiol Commun, 2013, 49(2): 101-110 (in Chinese). 谢烨, 孙毅, 黄继荣. 拟南芥中花青素的修饰. 植物生理学报, 2013, 49(2): 101-110. |
| [72] |
Luo J, Nishiyama Y, Fuell C, et al. Convergent evolution in the BAHD family of acyl transferases: identification and characterization of anthocyanin acyl transferases from Arabidopsis thaliana. Plant J, 2007, 50(4): 678-695. DOI:10.1111/j.1365-313X.2007.03079.x |
| [73] |
Quattrocchio F, Baudry A, Lepiniec L, et al. The regulation of flavonoid biosynthesis//Grotewold E, Ed. The Science of Flavonoids. New York: Springe, 2006, 97-122. |
| [74] |
Yoshida K, Mori M, Kondo T. Blue flower color development by anthocyanins: from chemical structure to cell physiology. Nat Prod Rep, 2009, 26(7): 884-915. DOI:10.1039/b800165k |
| [75] |
Han YJ, Kim YM, Lee JY, et al. Production of purple-colored creeping bentgrass using Maize transcription factor genes Pl and Lc through Agrobacterium-mediated transformation. Plant Cell Rep, 2009, 28(3): 397-406. DOI:10.1007/s00299-008-0648-0 |
| [76] |
Nanjaraj Urs AN, Hu YL, Li PW, et al. Cloning and expression of a nonribosomal peptide synthetase to generate blue Rose. ACS Synth Biol, 2019, 8(8): 1698-1704. DOI:10.1021/acssynbio.8b00187 |
| [77] |
Holton TA, Brugliera F, Lester DR, et al. Cloning and expression of cytochrome P450 genes controlling flower colour. Nature, 1993, 366(6452): 276-279. DOI:10.1038/366276a0 |
| [78] |
Mori S, Kobayashi H, Hoshi Y, et al. Heterologous expression of the flavonoid 3′, 5′-hydroxylase gene of Vinca major alters flower color in transgenic Petunia hybrida. Plant Cell Rep, 2004, 22(6): 415-421. DOI:10.1007/s00299-003-0709-3 |
| [79] |
Shimada Y, Ohbayashi M, Nakano-Shimada R, et al. Genetic engineering of the anthocyanin biosynthetic pathway with flavonoid-3′, 5′-hydroxylase: specific switching of the pathway in petunia. Plant Cell Rep, 2001, 20(5): 456-462. DOI:10.1007/s002990100319 |
| [80] |
Shao L, Li Y, Yang MZ, et al. Gene expression of Chalcone synthase a (CHSA) in flower colour alterations and male sterility in transgenic Petunia. Acta Bota Sin, 1996, 38(7): 517-524 (in Chinese). 邵莉, 李毅, 杨美珠, 等. 查尔酮合酶基因对转基因植物花色和育性的影响. 植物学报, 1996, 38(7): 517-524. |
| [81] |
Nakatsuka T, Mishiba KI, Abe Y, et al. Flower color modification of gentian plants by RNAi-mediated gene silencing. Plant Biotechnol, 2008, 25(1): 61-68. DOI:10.5511/plantbiotechnology.25.61 |
| [82] |
Nishihara M, Nakatsuka T, Hosokawa K, et al. Dominant inheritance of white-flowered and herbicide-resistant traits in transgenic gentian plants. Plant Biotechnol, 2006, 23(1): 25-31. DOI:10.5511/plantbiotechnology.23.25 |
| [83] |
Nakamura N, Fukuchi-Mizutani M, Miyazaki K, et al. RNAi suppression of the anthocyanidin synthase gene in Torenia hybrida yields white flowers with higher frequency and better stability than antisense and sense suppression. Plant Biotechnol, 2006, 23(1): 13-17. |
| [84] |
Katsumoto Y, Fukuchi-Mizutani M, Fukui Y, et al. Engineering of the rose flavonoid biosynthetic pathway successfully generated blue-hued flowers accumulating delphinidin. Plant Cell Physiol, 2007, 48(11): 1589-1600. DOI:10.1093/pcp/pcm131 |
| [85] |
Brugliera F, Tao GQ, Tems U, et al. Violet/blue chrysanthemums—metabolic engineering of the anthocyanin biosynthetic pathway results in novel petal colors. Plant Cell Physiol, 2013, 54(10): 1696-1710. DOI:10.1093/pcp/pct110 |
| [86] |
Noda N, Yoshioka S, Kishimoto S, et al. Generation of blue Chrysanthemums by anthocyanin B-ring hydroxylation and glucosylation and its coloration mechanism. Sci Adv, 2017, 3(7): e1602785. DOI:10.1126/sciadv.1602785 |
| [87] |
Qi YY, Lou Q, Quan YH, et al. Flower-specific expression of the Phalaenopsis flavonoid 3′, 5′-hydoxylase modifies flower color pigmentation in Petunia and Lilium. Plant Cell, Tissue Organ Cult, 2013, 115(2): 263-273. DOI:10.1007/s11240-013-0359-2 |
| [88] |
Qi YY, Du LJ, Quan YH, et al. Agrobacterium-mediated transformation of embryogenic cell suspension cultures and plant regeneration in Lilium tenuifolium oriental×trumpet 'Robina'. Acta Physiol Plant, 2014, 36(8): 2047-2057. DOI:10.1007/s11738-014-1582-0 |
| [89] |
Nakatsuka T, Saito M, Yamada E, et al. Isolation and characterization of GtMYBP3 and GtMYBP4, orthologues of R2R3-MYB transcription factors that regulate early flavonoid biosynthesis, in gentian flowers. J Exp Bot, 2012, 63(18): 6505-6517. DOI:10.1093/jxb/ers306 |
| [90] |
Nakatsuka T, Yamada E, Saito M, et al. Heterologous expression of gentian MYB1R transcription factors suppresses anthocyanin pigmentation in tobacco flowers. Plant Cell Rep, 2013, 32(12): 1925-1937. DOI:10.1007/s00299-013-1504-4 |
| [91] |
Van Der Krol AR, Lenting PE, Veenstra J, et al. An anti-sense chalcone synthase gene in transgenic plants inhibits flower pigmentation. Nature, 1988, 333(6176): 866-869. DOI:10.1038/333866a0 |
| [92] |
Jiang WZ, Zhou HB, Bi HH, et al. Demonstration of CRISPR/Cas9/sgRNA-mediated targeted gene modification in Arabidopsis, tobacco, sorghum and rice. Nucleic Acids Res, 2013, 41(20): e188. DOI:10.1093/nar/gkt780 |
| [93] |
Puchta H. Applying CRISPR/Cas for genome engineering in plants: the best is yet to come. Curr Opin Plant Biol, 2017, 36: 1-8. |
| [94] |
Watanabe K, Kobayashi A, Endo M, et al. CRISPR/Cas9-mediated mutagenesis of the dihydroflavonol-4-reductase-B (DFR-B) locus in the Japanese morning glory Ipomoea (Pharbitis) nil. Sci Rep, 2017, 7(1): 10028. DOI:10.1038/s41598-017-10715-1 |
| [95] |
Noda N, Aida R, Kishimoto S, et al. Genetic engineering of novel bluer-colored chrysanthemums produced by accumulation of delphinidin-based anthocyanins. Plant Cell Physiol, 2013, 54(10): 1684-1695. DOI:10.1093/pcp/pct111 |
| [96] |
Butelli E, Titta L, Giorgio M, et al. Enrichment of tomato fruit with health-promoting anthocyanins by expression of select transcription factors. Nat Biotechnol, 2008, 26(11): 1301-1308. DOI:10.1038/nbt.1506 |
| [97] |
Ohno S, Hosokawa M, Kojima M, et al. Simultaneous post-transcriptional gene silencing of two different chalcone synthase genes resulting in pure white flowers in the octoploid dahlia. Planta, 2011, 234(5): 945-958. DOI:10.1007/s00425-011-1456-2 |
| [98] |
Wang Y. New materia medica project: synthetic biology based bioactive metabolites research in medicinal plant. Chin J Biotech, 2017, 33(3): 478-485 (in Chinese). 王勇. 新本草计划——基于合成生物学的药用植物活性代谢物研究. 生物工程学报, 2017, 33(3): 478-485. |
| [99] |
Santos CNS, Koffas M, Stephanopoulos G. Optimization of a heterologous pathway for the production of flavonoids from glucose. Metab Eng, 2011, 13(4): 392-400. DOI:10.1016/j.ymben.2011.02.002 |
| [100] |
Chemler JA, Fowler ZL, McHugh KP, et al. Improving NADPH availability for natural product biosynthesis in Escherichia coli by metabolic engineering. Metab Eng, 2010, 12(2): 96-104. DOI:10.1016/j.ymben.2009.07.003 |
| [101] |
Zou LQ, Wang CX, Kuang XJ, et al. Advance in flavonoids biosynthetic pathway and synthetic biology. China J Chin Meter Med, 2016, 41(22): 4124-4128 (in Chinese). 邹丽秋, 王彩霞, 匡雪君, 等. 黄酮类化合物合成途径及合成生物学研究进展. 中国中药杂志, 2016, 41(22): 4124-4128. |
 ${metaVo.year}, Vol. 36
${metaVo.year}, Vol. 36




