中国科学院微生物研究所、中国微生物学会主办
文章信息
- 侯艳红, 龚桂芝, 彭祝春, 董倩倩, 罗艾, 洪棋斌
- Hou Yanhong, Gong Guizhi, Peng Zhuchun, Dong Qianqian, Luo Ai, Hong Qibin
- MIME——有丝分裂替代减数分裂及其在作物无融合生殖中的应用
- MIME—Mitosis instead of meiosis and its application in crop apomixis
- 生物工程学报, 2020, 36(4): 612-621
- Chinese Journal of Biotechnology, 2020, 36(4): 612-621
- 10.13345/j.cjb.190225
-
文章历史
- Received: May 30, 2019
- Accepted: July 29, 2019
- Published: August 26, 2019
有丝分裂(Mitosis)是基因组一次复制后,浓缩的染色体在纺锤丝的牵引下平均分配到两个子代细胞,每个子代细胞具有与母细胞完全一样的遗传物质,它保证了细胞增殖过程中遗传的稳定。减数分裂(Meiosis)则是基因组一次复制后,连续两次分裂,第一次染色体倍数减半,第二次姊妹染色单体等分,最后形成4个子代细胞,每个子代细胞仅具有母细胞一半的遗传物质,由于同源染色体间的交换重组,子代细胞的遗传物质产生差异,其融合后代导致生殖的遗传多样性[1]。有丝分裂和减数分裂是在发生时机和功能上分隔的两个过程,然而许多植物能通过调控减数分裂配子的形成过程而产生未减数配子,使其减数分裂过程类似于有丝分裂,MIME (Mitosis instead of Meiosis)是人为构建第一次染色单体等分,第二次减数分裂跳过的无融合减数分裂突变体[2],构建MIME突变体的生物工程技术,已成为研究工作者创制无融合生殖的有力“工具”。
无融合生殖因产生基因型与母本完全一致的种子,所以在固定杂种优势、提高育种效率、降低制种成本方面有着巨大潜力[3-4],被誉为杂交育种界的“无性生殖革命”[5]。根据胚的来源途径可将无融合生殖分为两种类型,一种是由未减数胚囊形成无融合胚的配子体无融合生殖,另一种是由胚珠内珠心细胞直接形成一个或者多个胚的孢子体无融合生殖,也称为珠心胚现象[6]。国外对无融合生殖的研究主要集中在配子体无融合生殖上,研究工作者曾对天然存在配子体无融合生殖现象的禾本植物[7-10]进行研究,筛选出与无融合生殖相关的一段基因重组抑制区,并试图通过杂交将该区域引进其各自的近缘栽培品种中[11],但都没有成功。孢子体无融合生殖的研究主要以柑橘类植物为模式植物,但由于采用不同的杂交材料得到不同的杂交分离结果,因此对于珠心胚的遗传控制位点一直存在争议,有单基因、单主基因加微效多基因以及双基因等多种调控理论假说[12-14]。我们团队长期跟踪无融合生殖,以柑橘对无融合生殖进行研究,希望将其无融合生殖机制或机理拓展到重要粮油作物的杂种优势固定,提出了柑橘类植物中无融合生殖的双基因调控假说,该假说认为柑橘无融合生殖可能受位于细胞核的两对互补的显性基因调控,且其中一对基因存在显性纯合致死效应,该假说能解释已有的大部分柑橘杂交分离数据[14]。通过遗传分离群体分析和无融合生殖突变材料的RNA-seq分析,我们定位了一些QTL位点和候选基因,并且在柑橘和烟草中开展了部分候选基因的转基因验证工作。
有关无融合生殖的遗传机制研究仍未取得决定性进展[15-16],人工创制无融合生殖则成为突破无融合生殖壁垒的另一种有效方法[17]。目前人工创制无融合生殖主要从配子体无融合生殖入手,创制需满足3个要素,第一是植物体中减数分裂的缺失与改变,即无融合减数分裂;第二是卵细胞不受精的独立发育,即孤雌生殖;第三是胚乳的正常启动发育[18],其中无融合减数分裂是构建无融合生殖的先决条件。MIME作为一种人造无融合减数分裂突变体,产生与母本基因型完全一致的功能性配子,成功解决了自然发生的未减数配子发生频率低且育性不稳定的问题,将MIME与孤雌生殖相结合即可获得无融合生殖的种子,成功创制无融合生殖。MIME是打开人工创制无融合生殖大门的钥匙,对MIME的基因及发生机制的研究,有利于将MIME扩大到更多栽培作物中,对农业杂交育种有着重要意义。文中对MIME的发生及其在作物无融合生殖中的应用以及MIME应用中遇到的问题进行讨论综述,并对MIME在作物中的扩展和对无融合研究的推动进行了展望。
1 MIME的产生自然界中存在产生未减数配子的自发突变体,根据减数分裂突变发生的时期,产生未减数配子的减数分裂突变被分为“第一次分裂重组” (FDR, the first division restitution)和“第二次分裂重组” (SDR, the second division restitution)[19]。拟南芥Arabidopsis thaliana的parallel-spindle1 (ps1, At1g34355)和jason (jas, At1g06660)编码维持花粉母细胞第二次减数分裂过程中的纺锤体构象的功能蛋白,它们的缺失突变导致二倍体雄性配子的形成,属于SDR突变,但由于它们的精子细胞中存在同源重组的基因标记,因此并不是真正的克隆配子[20-22]。第一次观察到真正的无融合减数分裂是在拟南芥DYAD突变体中,DYAD编码一种进入减数分裂所必需的核缠绕蛋白,缺失该基因导致减数分裂转化为有丝分裂,从而产生未减数分裂的配子[23-24]。但DYAD突变体只产生0.2%的可育未减数雌性配子,远低于育种应用所需频率。直到d’Erfurth等[2]将拟南芥中影响关键减数分裂过程的3个基因突变相结合,人为构建了减数分裂被有丝分裂取代的MIME突变体。与DYAD相比,MIME产生了更高频率(雄配子100%和雌配子85%)的未减数配子,且MIME植株育性不受影响,此后又有报道在水稻Oryza sativa L.中成功构建MIME[25]。
MIME的形成是对减数分裂区别有丝分裂的3个关键点同时破坏,即把同源染色体之间配对和重组、第一次减数分裂时姐妹染色单体不分离、一次DNA复制后发生两次分裂3个点同时突变,使减数分裂过程类似于有丝分裂,产生的配子保留了母本所有的遗传信息,从而完成无融合生殖三要素中最关键的无融合减数分裂过程。值得注意的是,许多基因在细胞分裂周期中发挥着至关重要的作用,一旦缺失可能引起有丝分裂的终止或育性的严重损害,因此选择合适的基因构建MIME突变体则显得尤为重要。拟南芥是最早成功构建MIME的物种,且是植物染色体行为研究的模式植物,因此本文主要以构建拟南芥MIME突变的spo11-1、rec8和osd1三基因突变为主,分析在MIME产生过程中染色体的行为变化,参见图 1。
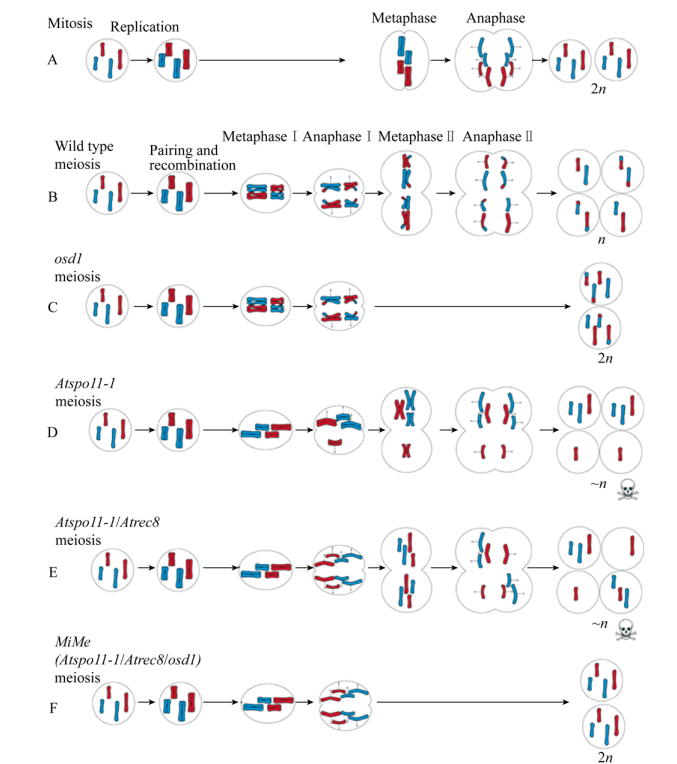
|
| 图 1 MIME产生过程染色体变化示意图(d’Erfurth等[2]) Fig. 1 Schematic diagram of chromosomal changes produced by MIME production process (d'Erfurth et al[2]). (A) wild type mitosis. Chromosomal replication and sister chromatid separation result in diploid daughter cells that are genetically identical to maternal cells. (B) Wild type meiosis. DNA replicates once and divides twice. homologous chromosome recombination and isolation in Meiosis Ⅰ. Sister chromatids equal distribution in meiosis Ⅱ, haploid gametes carrying recombinant genetic information was obtained. (C) Atosd1 mutant meiosis. The meiosis Ⅱ was skipped and a diploid gamete carrying recombinant genetic information was obtained. (D) Atspo11-1 mutant meiosis. chromosome homologous recombination was inhibited, following unequal distribution in meiosis Ⅰ. Sister chromatids separation in meiosis Ⅱ and sterile gametes was obtained. (E) Atspo11-1/Atrec8 double mutant meiosis. chromosome homologous recombination was inhibited with sister chromatids were separated advanced in meiosis Ⅰ, thus undergoes a mitotic-like division instead of a normal first meiotic division. Unbalanced distribution of chromatids in meiosis Ⅱ, and sterile gametes were obtained. (F) Atspo11-1/Atrec8/Atosd1 triple mutant meiosis. chromosome homologous recombination was inhibited and sister chromatids were separated advanced in meiosis Ⅰ, thus undergoes a mitotic-like division instead of a normal first meiotic division. The triple mutant skips the meiosis Ⅱ and obtains fertile gametes that are genetically identical to the maternal cells. |
| |
酿酒酵母Saccharomyces cerevisiae H. Spo11蛋白是古菌DNA拓扑异构酶Via亚基(topo Via)的真核同源物,介导DNA双链断裂,从而启动减数分裂重组[26]。Grelon等[27]在拟南芥中发现Spo11蛋白的3个同源基因Atspo11-1、Atspo11-2和Atspo11-3,遗传学证据表明Atspo11-1是酿酒酵母S. cerevisiae spo11的真正的同源体,也是减数分裂重组所必需的基因。在拟南芥中构建MIME时,选择突变Atspo11-1基因来阻断第一次减数分裂时同源染色体重组互换,为产生与母本基因型一致的配子打下基础。此外,参与DNA双链重组修复过程的基因众多,Atspo11-2、prd1、prd 2、prd 3/pair 1、dfo和mtopvib等都有相关功能的研究报道[28-29],Mieulet等[25]通过实验证实prd1、prd2、prd3 (pair1)突变能够替代Atspo11-1在拟南芥中阻断同源重组而产生MIME突变体。水稻Ospair1是减数分裂重组启动中必需的基因[30],它的缺失突变能在水稻中重现拟南芥spo11-1突变现象,是在构建水稻MIME表型时阻断同源重组的首选。
1.2 rec8突变致使姐妹染色单体提前分离第一次减数分裂成功的决定条件之一是同源染色体配对,并成功向细胞的两极移动分离。这一过程的完成需要第一次减数分裂中期的姐妹染色单体定向到同极,且在第二次减数分裂后期之前保持着丝粒的凝聚力。减数分裂凝集亚基Rec8是保持着丝粒凝聚力的关键分子之一,Rec8亚基的缺失会导致着丝粒凝聚力下降,姐妹染色单体提前分离[31]。在拟南芥中突变Atrec8基因,致使姐妹染色体提前分离且不平衡分配到两极,破坏了第一次减数分裂过程,使其与有丝分裂相接近,而破坏第一次减数分裂正是构建MIME表型中非常重要的一环。Atscc3是目前已知的另一控制姐妹染色单体单极定向所必需的基因,除在减数分裂中起作用以外,还是有丝分裂正常发生所必需的基因[32],它的突变会导致有丝分裂畸形而影响后代的正常生长发育。因此,目前暂没有发现能替代Atrec8在MIME中发挥相同功能的基因。
1.3 osd1突变导致第二次减数分裂的跳跃相较于上述两种产生FDR的基因功能鉴定,导致SDR突变的基因osd1发现得更晚一些,osd1突变会导致第二次减数分裂过程跳过。d’Erfurth等[2]使用基因表达谱分析工具在拟南芥中筛选出与已知的减数分裂基因的表达模式相似的候选基因Atosd1,对该基因的功能研究发现,osd1纯合突变体无任何体细胞发育缺陷,雌雄配子育性也与野生型相当,但在突变体自交后代中,会产生大量的四倍体和少量的三倍体植株,无二倍体植株。当突变花粉用于野生型植物受精时,其后代均为三倍体,突变胚珠与野生型花粉受精时,分离得到12%的二倍体和88%的三倍体植株,这些结果表明osd1基因的缺失能产生高频率的二倍体配子。对减数分裂过程中染色体行为观察发现,osd1突变体是由于第二次减数分裂过程的跳跃而产生了二倍体配子。osd1基因突变在不损害配子育性的基础上,导致第二次减数分裂过程跳过,使得配子倍性增加,是构建MIME表型中起决定性作用的一环。
同样是d’Erfurth团队利用编码减数分裂必需周期蛋白的tam1基因突变代替osd1突变[33],成功在拟南芥中构建了MIME-2表型,但由于tam1并不是完全的显性突变[34],MIME-2植物在产生功能性二倍体配子的同时也会产生少量的单倍体和非整倍体配子,且MIME-2植株的育性水平也低于野生型或tam1单突变型,因此在拟南芥中,osd1是比tam1构建MIME突变体更好的选择。在拟南芥中有一个osd1的旁系同源基因uvi4[35-36],osd1和uvi4均参与细胞周期调控,但uvi4是维持有丝分裂状态所必需的,因此不能用于构建MIME突变型植株。Mieulet等[25]利用同源性分析在水稻中找到与拟南芥osd1基因同源的基因Os02g37850,发现该基因的插入突变会在水稻中重现拟南芥osd1突变,导致第二次减数分裂的跳跃,因此将Os02g37850基因命名为Ososd1,并结合水稻中的Ospair1和Osrec8成功在水稻中建立MIME突变体。
1.4 基因间的相互作用在先前的研究报道中,拟南芥spo11-1/rec8双突变体的第一次减数分裂被一个类似于有丝分裂的过程所取代,随后是染色体不平衡分配的第二次分裂,导致配子严重不育[32]。但将阻止第二次减数分裂的osd1突变与破坏第一次减数分裂的spo11-1/rec8相结合,osd1基因突变抑制了spo11-1/rec8双突变体的染色体不平衡分配,使spo11-1/rec8/osd1三突变体中产生了减数分裂被有丝分裂取代且遗传信息与母本完全一致的可育无融合配子。
在水稻中也有相似的发现,Mieulet等[25]在对水稻Ospair1、Osrec8和Ososd1单突变体筛选杂交获得三重突变体的过程中发现,Osrec8单突变体在减数分裂中期出现纠缠的DNA团,随后出现染色体断裂和不平衡分离。但在Ospair1/Osrec8双突变体中,染色体断裂和不平衡分配的现象却消失了,说明Ospair1能够消除Osrec8的染色体断裂且改变了染色体分配模式,这两个基因之间具有一定的修正互补作用,但这种双突变体产生的仍然是严重不育的配子,而Ososd1突变能弥补Ospair1/Osrec8双突变体产生不育配子的特点,在水稻中产生高频率的可育无融合配子。
2 MIME在作物无融合生殖中的应用由于MIME产生的克隆配子参与正常的自受精会产生倍性加倍的后代,因此要获得正常的无融合生殖种子,还必须防止染色体加倍。启动MIME配子发育成为单倍体植株,是MIME引入作物杂交育种的关键。
Marimuthu等[37]通过将拟南芥cenh3-1染色体消除突变体系GEM和拟南芥MIME系进行正反交,筛选出包括二倍体、三倍体和非整倍体的F1代无性系,在二倍体种子后代当中,分别有98%和42%的二倍体植株保留MIME株系的杂合性,证明通过MIME获得无融合种子是可行的。
中国水稻研究所王春等[38]利用CRISPR/Cas9基因编辑技术在水稻春优84中敲除pair1、rec8、osd1三个内源基因构成MIME的基础上,还对编码花粉特异性磷脂酶的matrilineal (mtl)基因进行了敲除,该基因能诱导玉米和水稻单倍体的发生[39-42],最终的四重突变体植株产生了大量的四倍体与二倍体种子,二倍体克隆种子获得率为6.2%。该四重突变体在营养生长阶段表现正常,但和诱导父系基因组消除的mtl基因单突变植株相似,它的育性显著下降。虽然存在育性下降的缺陷,但他们的研究是在不引入杂交授粉的基础上,将MIME与水稻无融合生殖相结合的成功案例,是MIME应用中的一项重大突破。
同样是在水稻当中,Khanday等[43]将能诱导珍珠粟Pennisetum americarum (L.) Leeke.孤雌生殖的baby-boom-like (bbm)基因[44]与水稻MIME相结合,成功获得了无融合水稻种子。Anderson等[45]曾对水稻合子转录组研究发现,bbm1在受精后的受精卵中特异表达,表明bbm1在受精卵胚胎发生过程中具有潜在的作用,Khanday等[43]对水稻的bbm家族基因跟进研究发现,bbm1、bbm2、bbm3基因三重突变体植株的胚胎发育异常,更重要的是,在这个三重突变体里引入bbm1基因,又能重启胚胎发育,同时超表达bbm1基因能引起水稻的孤雌生殖。这些结果证实bbm1是启动胚胎发育的“开关”,也正是MIME应用研究中所需的孤雌生殖“开关”。随后研究人员将这一开关引入MIME植株,在bbm1超表达株系中敲除pair1、rec8、osd1基因,获得两株S-Apo (MIME+bbm1ee)转基因植株,从中获得频率分别为11%和29%的无融合生殖种子后代,但与野生型相比,S-Apo植株的育性也存在显著下降。
3 MIME技术在当前应用中存在的问题 3.1 MIME的应用扩展目前只在拟南芥和水稻中成功获得MIME植株,如何将其扩大到更多的物种是当前MIME应用中所面临的问题。减数分裂基因在真核生物中通常是比较保守的,因此利用模式植物拟南芥相应的同源基因推广到其他物种当中是比较可行的办法,但在不同物种之间也存在减数分裂机制不同的情况。比如大多数单子叶植物在每次减数分裂之后都会发生胞质分裂,而双子叶植物通常在第二次减数分裂时发生胞质分裂[46],这暗示着在不同植物之间可能存在不同的分裂机制。此外,控制细胞分裂周期的基因有可能在植物中存在多个拷贝,比如osd1在大麦、油菜中是单一基因,而在玉米Zea mays L.、高粱Sorghum bicolor和狗尾草Setaria italica中却表现为串联重复[32],因此在构建MIME表型时需要使用RNAi或基因组编辑技术进行定点突变,增加了一定的操作难度。这些相关的证据表明植物基因组具有高度复杂性,因此将MIME应用扩大时不能完全依赖于同源基因的功能识别,还需要在特定感兴趣的作物上进行表型验证。
3.2 孤雌生殖MIME只完成了产生无融合配子的过程,要达到以种子进行无性繁殖的目的,还需要通过无融合配子的孤雌生殖或与能诱导基因组消除的着丝粒特异组蛋白CENH3突变系杂交产生自主发育胚胎。找到调控胚胎发育的关键基因,诱导植物体产生孤雌生殖,无疑是比引入杂交更为理想的获得无融合种子的方式,但目前对孤雌生殖机制的研究一直未取得较大进展。bbm和mtl是当前少数明确能够控制孤雌生殖的两个基因,在玉米和水稻中都有促进单性繁殖的报道[44, 47-48],但bbm和mtl都会导致突变体植株生育力严重下降,这在产业应用上是致命的缺陷。此外,系统进化树分析显示,mtl基因在单子叶的谷类作物中高度保守,但未在双子叶植物中找到同源基因[40],因此,mtl孤雌生殖诱导技术不具有广泛应用的基础。并且这两种孤雌生殖诱导单倍体后代产生的频率都比较低,极大地限制了克隆繁殖的效率。
CENH3系诱导基因组消除技术是当前在拟南芥中运用比较成熟的单倍体诱导技术[49-50],但除能在玉米中诱导较低频率的单倍体种子外[51],在其他物种中成功应用的研究仍少见报道,且因其需依赖授粉过程才能产生克隆种子后代的特点,不利于无融合种子的产生与商业推广。因此改进孤雌生殖技术将会是无融合育种研究中的一个重要方向。
3.3 胚乳发育MIME产生无融合配子,孤雌生殖使无融合配子启动胚胎发育,除此以外,还需要胚乳的启动发育以保证种子的形成。在bbm1诱导MIME植株产生无融合种子的水稻体系中,导入bbm1基因胚胎虽然会启动发育,但由于没有受精形成胚乳,胚胎发育提前终止[43],因此它们的二倍体无融合种子仍然是通过自花授粉所获得。与孤雌生殖启动胚胎发育类似,自然界也存在许多特殊的形成胚乳的情况,比如在山柳菊Hieracium以及龙须草Eulaliopsis binatat (Rotz) C.E Hubb.中发现,其胚乳能够由极核不经受精作用自主分裂发生[52],若将该胚乳自主发生机制与当前的无融合分裂与孤雌生殖技术相结合,将能完成彻底不经受精即产生无融合种子的无性革命。但目前对无融合生殖的胚乳发育机制还知之甚少,对无融合生殖植物中胚乳发育分子机制的研究还有很长的路要走。
4 总结与展望MIME的产生是无融合生殖研究中的一项重大突破,将它产生的功能性无融合配子与孤雌生殖技术相结合,能在有性植物中进行无性克隆繁殖,对杂交育种具有重要意义。运用MIME已成功在拟南芥和水稻中获得无融合二倍体种子,是基因工程创制无融合生殖的重要里程碑事件。植物中存在许多与生殖发育相关的关键基因,目前我们所了解到的仅是其中一小部分,可能还存在很多能引起减数分裂抑制而不影响营养生长的基因,对这些基因进行探索将会是MIME未来研究的方向。
玉米是当前的单倍体诱导技术研究得最多且应用也最为广泛的作物[53-55],将其成熟的单倍体诱导技术与MIME相结合,有望对玉米杂交育种产生巨大推动作用。虽然目前还没有在玉米中成功构建MIME的研究报道,但玉米中多个减数分裂突变体的功能变化暗示MIME的构建是可行的。玉米中含有与拟南芥rec8同源的基因afd1,该基因突变同样使姐妹染色单体提前分离[56]。此外,玉米中的spo11-1同源基因也被成功地克隆和鉴定[57],但玉米的osd1基因有多个拷贝,暂未鉴定出与控制细胞周期相关的主效基因[58],而玉米的elongate1基因突变体通过跳过第二次减数分裂,产生功能性二倍体胚囊[59],表现出与水稻、拟南芥osd1基因突变相似的变化。对玉米中的afd1、spo11-1、elongate1三个基因同时突变构建MIME突变体,对于玉米的无融合生殖构建具有重要意义,不过能否在玉米中重现MIME还有待实验验证。
有学者曾提出,无融合生殖的发生过程是有性通路时空表达改变的结果,这一过程会招募有性生殖相关基因的表达[11]。通过人为调控这些基因,能在非自然无融合物种中将有性途径转换为无融合途径,MIME的构建正是对有性生殖关键基因的修改,再结合孤雌生殖将有性生殖的种子变成了无融合生殖种子,是对无融合生殖是叠加在有性途径上这一观点的印证。下一步对产生MIME的基因之间的调控网络进行研究,以MIME作为突破口将能给无融合生殖发生的分子机理探究提供依据。
与MIME创制的配子体无融合生殖相比,以柑橘为代表的孢子体无融合生殖具有明显不同的发生和遗传控制机制,其研究无论从材料获得、研究基础和遗传控制过程等考量均相对更简单,柑橘无融合生殖基因的利用不需要考虑复杂的胚乳发生机制等问题,而且更贴近水稻、油菜等重要二倍体粮油作物。若能将孢子体无融合生殖模式导入作物,将对无法采用MIME构建无融合生殖的物种提供新的补充途径。
| [1] |
Wang YX, Copenhaver GP. Meiotic recombination: Mixing it up in plants. Annu Rev Plant Biol, 2018, 69: 577-609. DOI:10.1146/annurev-arplant-042817-040431 |
| [2] |
d'Erfurth I, Jolivet S, Froger N, et al. Turning meiosis into mitosis. PLoS Biol, 2009, 7(6): e1000124. DOI:10.1371/journal.pbio.1000124 |
| [3] |
Ozias-akins P. Apomixis: developmental characteristics and genetics. Crit Rev Plant Sci, 2006, 25(2): 199-214. DOI:10.1080/07352680600563926 |
| [4] |
Spillane C, Steimer A, Grossniklaus U. Apomixis in agriculture: the quest for clonal seeds. Sex Plant Reprod, 2001, 14(4): 179-187. DOI:10.1007/s00497-001-0117-1 |
| [5] |
Calzada JPV, Crane CF, Stelly DM. Apomixis: the asexual revolution. Science, 1996, 274(5291): 1322-1323. DOI:10.1126/science.274.5291.1322 |
| [6] |
Koltunow AM. Apomixis: embryo sacs and embryos formed without meiosis or fertilization in ovules. Plant cell, 1993, 5(10): 1425-1437. DOI:10.2307/3869793 |
| [7] |
Bantin J, Matzk F, Dresselhaus T. Tripsacum dactyloides (Poaceae): a natural model system to study parthenogenesis. Sex Plant Reprod, 2001, 14(4): 219-226. DOI:10.1007/s00497-001-0119-z |
| [8] |
Roche D, Chen ZB, Hanna WW, et al. Non-Mendelian transmission of an apospory-specific genomic region in a reciprocal cross between sexual pearl millet (Pennisetum glaucum) and an apomictic F1 (P. glaucum×P. squamulatum). Sex Plant Reprod, 2001, 13(4): 217-223. DOI:10.1007/s004970000060 |
| [9] |
Ebina M, Nakagawa H, Yamamoto T, et al. Co-segregation of AFLP and RAPD markers to apospory in Guineagrass (Panicum maximum Jacq.). Grassland Sci, 2005, 51(1): 71-78. DOI:10.1111/j.1744-697X.2005.00011.x |
| [10] |
Miles JW, Escandón ML. Further evidence on the inheritance ofreproductive mode in Brachiaria. Can J Plant Sci, 1997, 77(1): 105-107. DOI:10.4141/P95-187 |
| [11] |
Barcaccia G, Albertini E. Apomixis in plant reproduction: a novel perspective on an old dilemma. Plant Reprod, 2013, 26(3): 159-179. DOI:10.1007/s00497-013-0222-y |
| [12] |
Parlevliet JE, Cameron JW. Evidence on the inheritance of nucellar embryony in citrus. Proc Am Soc Hort Sci, 1959, 74: 252-260. |
| [13] |
Iwamasa M, Ueno I, Nishiura M. Inheritance of nucellar embryony in Citrus. Bull Hort Res Sta Japan, 1967, 7: 1-10. |
| [14] |
Hong QB, Xiang SQ, Chen KL, et al. Two complementary dominant genes controlling apomixis in genus Citrus and Poncirus. Acta Genet Sin, 2001, 28(11): 1062-1067 (in Chinese). 洪棋斌, 向素琼, 陈克玲, 等. 两对互补的显性基因控制着柑桔属和枳属的无融合生殖. 遗传学报, 2001, 28(11): 1062-1067. |
| [15] |
Hand ML, Koltunow AMG. The genetic control of apomixis: asexual seed formation. Genetics, 2014, 197(2): 441-450. |
| [16] |
Ozias-Akins P, van Dijk PJ. Mendelian genetics of apomixis in plants. Annu Rev Genet, 2007, 41: 509-537. DOI:10.1146/annurev.genet.40.110405.090511 |
| [17] |
Spillane C, Curtis MD, Grossniklaus U. Apomixis technology development-virgin births in farmers' fields?. Nat Biotechnol, 2004, 22(6): 687-691. DOI:10.1038/nbt976 |
| [18] |
Koltunow AM, Grossniklaus U. Apomixis: a developmental perspective. Annu Rev Plant Biol, 2003, 54: 547-574. DOI:10.1146/annurev.arplant.54.110901.160842 |
| [19] |
Bretagnolle F, Thompson JD. Gametes with the somatic chromosome number: mechanisms of their formation and role in the evolution of autopolyploid plants. New Phytol, 1995, 129(1): 1-22. DOI:10.1111/j.1469-8137.1995.tb03005.x |
| [20] |
d'Erfurth I, Jolivet S, Froger N, et al. Mutations in AtPS1 (Arabidopsis thaliana Parallel Spindle 1) lead to the production of diploid pollen grains. PLoS Genet, 2008, 4(11): e1000274. DOI:10.1371/journal.pgen.1000274 |
| [21] |
De Storme N, Geelen D. The Arabidopsis mutant jason produces unreduced first division restitution male gametes through a parallel/fused spindle mechanism in meiosis Ⅱ. Plant Physiol, 2011, 155(3): 1403-1415. DOI:10.1104/pp.110.170415 |
| [22] |
Erilova A, Brownfield L, Exner V, et al. Imprinting of the polycomb group gene MEDEA serves as a ploidy sensor in Arabidopsis. PLoS Genet, 2009, 5(9): e1000663. DOI:10.1371/journal.pgen.1000663 |
| [23] |
Boateng KA, Yang XH, Dong FQ, et al. SWI1 is required for meiotic chromosome remodeling events. Mol Plant, 2008, 1(4): 620-633. DOI:10.1093/mp/ssn030 |
| [24] |
Mercier R, Vezon D, Bullier E, et al. SWITCH1 (SWI1): a novel protein required for the establishment of sister chromatid cohesion and for bivalent formation at meiosis. Genes Dev, 2001, 15(14): 1859-1871. DOI:10.1101/gad.203201 |
| [25] |
Mieulet D, Jolivet S, Rivard M, et al. Turning rice meiosis into mitosis. Cell Res, 2016, 26(11): 1242-1254. DOI:10.1038/cr.2016.117 |
| [26] |
Keeney S, Giroux CN, Kleckner N. Meiosis-specific DNA double-strand breaks are catalyzed by spo11, a member of a widely conserved protein family. Cell, 1997, 88(3): 375-384. DOI:10.1016/S0092-8674(00)81876-0 |
| [27] |
Grelon M, Vezon D, Gendrot G, et al. AtSPO11-1 is necessary for efficient meiotic recombination in plants. EMBO J, 2001, 20(3): 589-600. DOI:10.1093/emboj/20.3.589 |
| [28] |
de Muyt A, Vezon D, Gendrot G, et al. AtPRD1 is required for meiotic double strand break formation in Arabidopsis thaliana. EMBO J, 2007, 26(18): 4126-4137. DOI:10.1038/sj.emboj.7601815 |
| [29] |
de Muyt A, Pereira L, Vezon D, et al. A high throughput genetic screen identifies new early meiotic recombination functions in Arabidopsis thaliana. PLoS Genet, 2009, 5(9): e1000654. DOI:10.1371/journal.pgen.1000654 |
| [30] |
Nonomura KI, Nakano M, Fukuda T, et al. The novel gene Homologous pairing aberration in rice meiosis1 of rice encodes a putative coiled-coil protein required for homologous chromosome pairing in meiosis. Plant Cell, 2004, 16(4): 1008-1020. DOI:10.1105/tpc.020701 |
| [31] |
Bannister LA, Reinholdt LG, Munroe RJ, et al. Positional cloning and characterization of mouse mei8, a disrupted allele of the meiotic cohesin Rec8. Genesis, 2004, 40(3): 184-194. DOI:10.1002/gene.20085 |
| [32] |
Chelysheva L, Diallo S, Vezon D, et al. AtREC8 and AtSCC3 are essential to the monopolar orientation of the kinetochores during meiosis. J Cell Sci, 2005, 118(20): 4621-4632. DOI:10.1242/jcs.02583 |
| [33] |
d'Erfurth I, Cromer L, Jolivet S, et al. The CYCLIN-A CYCA1;2/TAM is required for the meiosis I to meiosis Ⅱ transition and cooperates with OSD1 for the prophase to first meiotic division transition. PLoS Genet, 2010, 6(6): e1000989. |
| [34] |
Cifuentes M, Jolivet S, Cromer L, et al. TDM1 regulation determines the number of meiotic divisions. PLoS Genet, 2016, 12(2): e1005856. DOI:10.1371/journal.pgen.1005856 |
| [35] |
Hase Y, Trung KH, Matsunaga T, et al. A mutation in the uvi4 gene promotes progression of endo-reduplication and confers increased tolerance towards ultraviolet B light. Plant J, 2006, 46(2): 317-326. DOI:10.1111/j.1365-313X.2006.02696.x |
| [36] |
Lloyd AH, Ranoux M, Vautrin S, et al. Meiotic gene evolution: can you teach a new dog new tricks?. Mol Biol Evol, 2014, 31(7): 1724-1727. DOI:10.1093/molbev/msu119 |
| [37] |
Marimuthu MPA, Jolivet S, Ravi M, et al. Synthetic clonal reproduction through seeds. Science, 2011, 331(6019): 876. DOI:10.1126/science.1199682 |
| [38] |
Wang C, Liu Q, Shen Y, et al. Clonal seeds from hybrid rice by simultaneous genome engineering of meiosis and fertilization genes. Nat Biotechnol, 2019, 37(3): 283-286. |
| [39] |
Liu CX, Li X, Meng DX, et al. A 4-bp Insertion at ZmPLA1 encoding a putative phospholipase a generates haploid induction in maize. Mol Plant, 2017, 10(3): 520-522. DOI:10.1016/j.molp.2017.01.011 |
| [40] |
Yao L, Zhang Y, Liu CX, et al. OsMATL mutation induces haploid seed formation in indica rice. Nat Plants, 2018, 4(8): 530-533. DOI:10.1038/s41477-018-0193-y |
| [41] |
Li X, Meng DX, Chen SJ, et al. Single nucleus sequencing reveals spermatid chromosome fragmentation as a possible cause of maize haploid induction. Nat Commun, 2017, 8(1): 991. |
| [42] |
Gilles LM, Khaled A, Laffaire JB, et al. Loss of pollen-specific phospholipase NOT LIKE DAD triggers gynogenesis in maize. EMBO J, 2017, 36(6): 707-717. DOI:10.15252/embj.201796603 |
| [43] |
Khanday I, Skinner D, Yang B, et al. A male-expressed rice embryogenic trigger redirected for asexual propagation through seeds. Nature, 2019, 565(7737): 91-95. DOI:10.1038/s41586-018-0785-8 |
| [44] |
Conner JA, Mookkan M, Huo HQ, et al. A parthenogenesis gene of apomict origin elicits embryo formation from unfertilized eggs in a sexual plant. Proc Natl Acad Sci USA, 2015, 112(36): 11205-11210. DOI:10.1073/pnas.1505856112 |
| [45] |
Anderson SN, Johnson CS, Chesnut J, et al. The zygotic transition is initiated in unicellular plant zygotes with asymmetric activation of parental genomes. Dev Cell, 2017, 43(3): 349-358. |
| [46] |
Hamant O, Ma H, Cande WZ. Genetics of meiotic prophase I in plants. Annu Rev Plant Biol, 2006, 57: 267-302. DOI:10.1146/annurev.arplant.57.032905.105255 |
| [47] |
Conner JA, Podio M, Ozias-Akins P. Haploid embryo production in rice and maize induced by PsASGR-BBML transgenes. Plant Reprod, 2017, 30(1): 41-52. DOI:10.1007/s00497-017-0298-x |
| [48] |
Kelliher T, Starr D, Richbourg L, et al. MATRILINEAL, a sperm-specific phospholipase, triggers maize haploid induction. Nature, 2017, 542(7639): 105-109. DOI:10.1038/nature20827 |
| [49] |
Karimi-Ashtiyani R, Ishii T, Niessen M, et al. Point mutation impairs centromeric CENH3 loading and induces haploid plants. Proc Natl Acad Sci USA, 2015, 112(36): 11211-11216. DOI:10.1073/pnas.1504333112 |
| [50] |
Kuppu S, Tan EH, Nguyen H, et al. Point mutations in centromeric histone induce post-zygotic incompatibility and uniparental inheritance. PLoS Genet, 2015, 11(9): e1005494. DOI:10.1371/journal.pgen.1005494 |
| [51] |
Kelliher T, Starr D, Wang WL, et al. Maternal haploids are preferentially induced by CENH3-tailswap transgenic complementation in maize. Front Plant Sci, 2016, 7: 414. |
| [52] |
Yao JL. Study on apomixis mechanism and evaluation of resources in Eulaliopsis binata[D]. Wuhan: Huazhong Agricultural University, 2005 (in Chinese). 姚家玲.龙须草无融合生殖机理研究及资源评价[D].武汉: 华中农业大学, 2005. http://cdmd.cnki.com.cn/Article/CDMD-10504-2006190226.htm |
| [53] |
Kermicle JL. Androgenesis conditioned by a mutation in maize. Science, 1969, 166(3911): 1422-1424. |
| [54] |
Eder J, Chalyk S. In vivo haploid induction in maize. Theor Appl Genet, 2002, 104(4): 703-708. DOI:10.1007/s00122-001-0773-4 |
| [55] |
Zhang ZL, Qiu FZ, Liu YZ, et al. Chromosome elimination and in vivo haploid production induced by Stock 6-derived inducer line in maize (Zea mays L.). Plant Cell Rep, 2008, 27(12): 1851-1860. DOI:10.1007/s00299-008-0601-2 |
| [56] |
Golubovskaya IN, Hamant O, Timofejeva L, et al. Alleles of afd1 dissect REC8 functions during meiotic prophase I. J Cell Sci, 2006, 119(16): 3306-3315. DOI:10.1242/jcs.03054 |
| [57] |
Ronceret A, Vielle-Calzada JP. Meiosis, unreduced gametes, and parthenogenesis: implications for engineering clonal seed formation in crops. Plant Reprod, 2015, 28(2): 91-102. DOI:10.1007/s00497-015-0262-6 |
| [58] |
Hu X, Cheng X, Jiang H, et al. Genome-wide analysis of cyclins in maize (Zea mays). Genet Mol Res, 2010, 9(3): 1490-1503. DOI:10.4238/vol9-3gmr861 |
| [59] |
Barrell PJ, Grossniklaus U. Confocal microscopy of whole ovules for analysis of reproductive development: the elongate1 mutant affects meiosis Ⅱ. Plant J, 2005, 43(2): 309-320. DOI:10.1111/j.1365-313X.2005.02456.x |
 2020, Vol. 36
2020, Vol. 36




