中国科学院微生物研究所、中国微生物学会主办
文章信息
- 陈才, 陈伟, 郑尧, 顾浩, 王伟, 王宵燕, 高波, 宋成义
- Chen Cai, Chen Wei, Zheng Yao, Gu Hao, Wang Wei, Wang Xiaoyan, Gao Bo, Song Chengyi
- 转座子引起的猪ktn1基因结构变异及其与生产性能的关联分析
- Structural variations caused by transposons in porcine ktn1 gene and their association with production traits
- 生物工程学报, 2020, 36(2): 267-275
- Chinese Journal of Biotechnology, 2020, 36(2): 267-275
- 10.13345/j.cjb.190179
-
文章历史
- Received: May 9, 2019
- Accepted: July 10, 2019
在猪基因组中,转座子是基因组的主要组成成分,占猪基因组的40.72%,其中反转录转座子是主要成分,占转座子的91.49%[1]。研究表明,转座子是基因组大小的重要决定因素,基因组的大小与其转座子的含量成正相关[2]。同时转座子可移动的特性使其成为基因组结构变异的重要贡献者,并在多个水平上影响宿主基因的活性。已有超过100例反转录转座子介导的插入导致了人类遗传疾病的报道[3]。同时在动物上已经观察到许多由转座子插入引起的表型变化,例如短散在核元件(Short interspersed nuclear elements,SINE)插入引起狗的体型变化和毛色变化[4-6],以及内源性反转录病毒(Endogenous retroviruses transposon,ERV)插入引起鸡的蛋壳颜色变化[7]。在猪中也观察到两例因L1插入引起的性状变化[8-9]。
驱动连接蛋白基因(kinectin 1,ktn1)产物是生物进化过程中保留下来的内质网膜上的一种保守性膜受体蛋白。猪(Sus scrofa) ktn1基因(GeneID: 100152397)位于Chr1: 184 881 555–184 990 936 (+) (https://www.ncbi.nlm.nih.gov/gene/100152397),全长109 382 bp。ktn1于1992年从鸡胚脑的cDNA文库中发现[10],其后有研究表明该基因与鸡胫骨软骨病有关[11]。同时,在人的疾病研究中,通过转录组测序鉴定了ktn1在皮肤鳞状细胞癌(cSCC)中发挥重要作用,并可作为治疗干预的新靶点[12]。
目前对猪ktn1基因的研究较少,而猪的ktn1基因及其侧翼序列中转座子及结构变异(Structural variations,SV)的分布情况还未见报道。因此全面解析ktn1基因中存在的结构变异,揭示转座子对其结构变异的贡献非常必要,其研究结果将会对进一步理解转座子对功能基因的影响提供参考。
1 材料与方法 1.1 实验动物本研究所用动物样品为大白猪(Large White pig)耳样62个,来自安徽省某种猪育种公司(安徽省);长白猪(Landrace pig)抗凝血样30个和杜洛克(Duroc pig)抗凝血样30个,来自徐州六马种猪科技有限公司(江苏省);梅山猪(Meishan pig)耳样30个和二花脸(Erhualian pig)耳样41个,来自苏州苏太企业有限公司(江苏省);巴马香猪(Bama miniature pig)耳样20个,来自巴马原种香猪农牧实业有限公司(广西省);苏姜猪(Sujiang pig)耳样184个,来自姜曲海种猪场(江苏省);藏猪(Tibetan pig)耳样36个,来自甘孜市畜牧所采集自四川省康定市地区(四川省)。野猪(Wild boar)肉样3头,采购自安徽地区。
1.2 基因组提取使用试剂盒TaKaRa的MiniBEST Universal Genomic DNA Extraction Kit Ver.5.0 (TaKaRa,大连,中国)从血液或耳组织中提取基因组DNA。提取的基因组DNA通过紫外分光光度计和琼脂糖凝胶电泳进行DNA浓度和质量检测,然后置于−20 ℃保存备用。
1.3 获取ktn1基因和侧翼序列从NCBI数据库(https://www.ncbi.nlm.nih.gov/gene/?term=100152397)中定位ktn1基因的位置,向其5′侧翼区和3′侧翼区分别延伸5 kb和3 kb作为参考序列。利用参考序列与NCBI的WGS数据库中的基因组序列数据进行比对,获取到另外13个基因组序列中ktn1基因及侧翼区的序列,其中部分基因组中的序列由于数据库中基因组测序数据拼接长度不够,需要手动拼接。
1.4 猪ktn1基因在不同物种间的保守性分析为了分析ktn1 (基因和侧翼区)序列的保守性,将猪ktn1 (基因和侧翼区)序列定位于ENSEMBL数据库(http://www.ensembl.org/index.html)中的猪参考基因组上,同时进行区域比对(Region Comparison),获取牛、绵羊、马、人、小鼠、狗参考基因组中相对应的序列,然后使用mVISTA (http://genome.lbl.gov/vista/index.shtml)进行保守分析。
1.5 转座子注解通过使用RepeatMasker (version:4.0.7,-cutoff 250,-nolow)结合本实验室构建的猪转座子数据库[1]对杜洛克参考基因组的ktn1 (基因和侧翼区)序列进行转座子注释,仅保留比对得分超过1 000且标记长度超过100 bp的位点进行后续分析。
1.6 ktn1基因多序列比对及结构突变分析将14条ktn1 (基因和侧翼区)序列使用ClustalX (version:2.0)软件进行多序列比对以鉴定结构突变。我们定义2–10 bp的变异为小型结构变异,且仅当有3个及以上品种基因组数据中出现一致变异才被认定为确定存在的变异,少于3个品种发生的结构变异被认为可能由测序或拼接不准造成的不确定变异,未被统计。大于50 bp的变异被定为大结构变异,且仅当2个及以上品种基因组数据中出现时才被统计,但由测序不确定(如超过500 bp的gap或超过100 bp的N)造成的大结构变异未被统计。同时将大结构变异与1.5中转座子注释位点进行相互对应,超过60%长度的结构变异对应转座子标记位点的被认定为是由转座子引起的结构变异。
1.7 ktn1中SINE转座子插入多态检测选择一个ktn1中年轻SINE转座子插入位点两侧的序列使用Oligo7设计常规PCR引物,引物名称及序列信息见表 1。同时选取杜洛克猪、长白猪、大白猪、野猪、梅山猪、藏猪、二花脸猪、巴马香猪8个品种,每个品种3个个体进行插入多态性检测。
| Primer | Primer sequences (5'–3') | Length of product (bp) |
| ktn1-STIP-F | CATGGTGTTTGTCAGGAGGA | 676/381 |
| ktn1-STIP-R | GGAAAGAAAGTGCAATCCAGA |
对1.7中SINE插入位点在不同品种的群体中(大白猪62头、长白猪48头、杜洛克44头、苏姜猪184头、巴马香猪36头)插入多态性及其频率进行检测。计算基因型频率、等位基因频率。对基因型分布进行Hardy-Weiberg平衡的卡方适合性检验(chi-square test):
| ${\chi ^2} = \sum {\frac{{{{(O - E)}^2}}}{E}} $ |
O (Observed)代表每个基因型的观测数目,E (Expected)代表每一个基因型在哈代-温伯格平衡定律成立的假定下的期望数目。同时在苏姜猪中与相关生长繁殖性状进行关联分析,使用SPSS通过Duncan测验进行差异显著性分析(P < 0.05)。
2 结果与分析 2.1 获取到14条ktn1基因的序列经过与NCBI的WGS数据库中不同品种的猪基因组测序数据比对,获取到14个测序基因组中ktn1 (基因及上游5 kb和下游3 kb侧翼区)的序列,ktn1序列的来源及在各基因组中的长度见表 2。从不同品种的测序基因组中获取到的ktn1 (基因及侧翼区)长度存在一定的差异,八眉猪(Bamei pig)、长白猪(Landrace pig)和大白猪(Large White pig)中获取的序列长度与杜洛克猪(Duroc pig)参考序列长度相近(117 382 bp),在杂交猪(Cross-bred pig)、哥根廷猪(Gottingen pig)、金华猪(Jinhua pig)和荣昌猪(Rongchang pig)中,序列长度略小于参考序列,但在五指山猪(Wuzhishan pig)和巴克夏猪(Berkshire pig)的基因组中获取的序列明显短于参考序列,分别只有115 708 bp和114 968 bp,提示在ktn1基因或其侧翼区中存在结构变异。
| Breed | ktn1 gene and its flanks | |
| Length (bp) | Candidate | |
| Duroc | 117 382 | Sscrofa11.1 chr1:184876555–184993936 |
| Bamei | 117 090 | LUXV01064060.1|2073403–2190492 |
| Berkshire | 114 968 | LUXW01084868.1|678706–793673 |
| Cross-bred | 116 678 | NPJO01000001.1|97934762–98051439 |
| Landrace | 117 513 | LUXT01022056.1|93089–210601 |
| LargeWhite | 117 310 | LUXX01064257.1|446207–563516 |
| Wuzhishan | 115 708 | AJKK01162022.1|7850–46400; AJKK01162023.1; AJKK01162024.1; AJKK01162025.1; AJKK01162026.1; AJKK01162027.1|1–5135 |
| Gottingen | 116 317 | LIDP01000001.1|205407929–205524245 |
| Jinhua | 116 753 | LUXY01023594.1|677735–794487 |
| Rongchang | 116 594 | LUXR01025216.1|677232–793825 |
| Meishan | 117 274 | LUXQ01132148.1:44173–111886; LUXQ01092367.1:31135–80699 |
| Tibetan | 121 664 | AORO02060094.1:1107224–1228894 |
| Hampshire | 117 432 | LUXS01014866.1:36154–153585 |
| Pietrain | 117 378 | LUXU01042406.1:30907–148284 |
根据NCBI对猪ktn1基因的注解,ktn1基因的长度为109 382 bp,共有54个外显子,但前6个外显子只是起始位置不同,结束位置相同,而最后4个外显子的起始位置相同,终止位置不同。这些外显子通过不同的拼接组合形成15个转录本(图 1)。
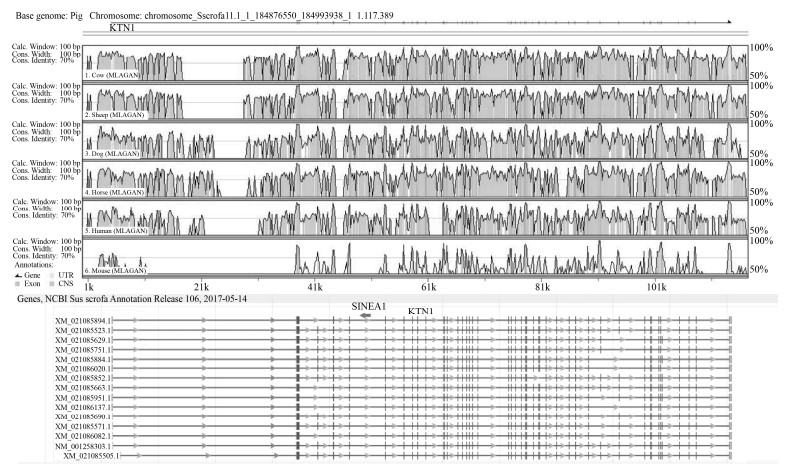
|
| 图 1 ktn1基因保守性分析及转录本示意图 Fig. 1 Schematic diagram of the ktn1 gene conservation analysis and its transcripts. The upper part is the result of conservative analysis. The light blue area represents the UTR area of the porcine ktn1, and the light purple represents the CDS area of the porcine ktn1. The following is a schematic diagram of 15 transcripts of the porcine ktn1 gene, green squares represent exons and thin lines represent introns. Red arrow represents the SINEA1 retrotransposon. |
| |
通过mVISTA将猪ktn1 (基因和侧翼)与牛、绵羊、马、狗、人、小鼠中相应区域的序列进行保守性分析。结果如图 1所示,猪的ktn1基因与牛、绵羊、狗和马的保守性较高,其次是人,与小鼠的保守性最差。但在上述7个物种中外显子区尤其CDS区(浅紫色)均非常保守,而第一内含子区保守性相对较弱。
2.3 杜洛克参考基因组中转座子对ktn1基因的贡献由于杜洛克参考基因组中ktn1基因序列最为完整,因此以杜洛克中ktn1基因的转座子注解信息进行统计分析,在ktn1基因及其侧翼区中鉴定到77个转座子标记位点,其中76个为反转录转座子(98.70%),其中SINE最多,有54个位点,最年轻的家族SINEA有49个位点。对标记长度进行统计分析发现,117 382 bp的ktn1 (基因和侧翼)序列中共有26 364 bp (22.46%)被转座子标记,其中最多是SINE (13 638/11.62%),其次是LINE (11 328/9.65%),其中最年轻SINE家族SINEA标记了12 588 bp占10.72%。ktn1基因中转座子数量及序列贡献信息见表 3。
| TE type | TE site | Percentage of TE site (%) | TE length (bp) | Percentage of TE (%) |
| DNA | 1 | 1.30 | 334 | 0.28 |
| hAT_Charlie | 1 | 1.30 | 334 | 0.28 |
| LINE | 19 | 24.68 | 11 328 | 9.65 |
| L1A | 8 | 10.39 | 4 463 | 3.80 |
| L1B | 2 | 2.60 | 1 110 | 0.95 |
| L1D | 1 | 1.30 | 294 | 0.25 |
| L1other | 6 | 7.79 | 3 604 | 3.07 |
| L2 | 2 | 2.60 | 1 857 | 1.58 |
| LTR | 3 | 3.90 | 1 064 | 0.91 |
| ERV1 | 1 | 1.30 | 508 | 0.43 |
| ERVⅢ | 2 | 2.60 | 556 | 0.47 |
| SINE | 54 | 70.13 | 13 638 | 11.62 |
| SINEA | 49 | 63.64 | 12 588 | 10.72 |
| SINEB | 5 | 6.49 | 1 050 | 0.89 |
| 总计 | 77 | 100.00 | 26 364 | 22.46 |
将14条ktn1 (基因和侧翼区)序列进行多序列比对,统计结构突变位点。外显子区未发现结构变异。在5′侧翼区和3′侧翼区各找到1个小结构突变,在内含子区找到7个小结构变异。而在ktn1基因的内含子区,只发现4个超过50 bp的大结构变异。具体数据及统计条件见表 4,其中较大的4个结构突变全部对应转座子注释位点。
| No. | Breed or Strain | Mutation type | In reference sequence(Duroc) | Sequence or TE corresponding to structural variation site | Gene structure | Insertion orientation | |
| Begin | End | ||||||
| 1 | Tibetan, Wuzhishan, Jinhua, Meishan, Rongchang, Cross-bred | Deletion | 755 | 756 | TT | 5′Flank | Sense |
| 2 | Bamei, Landrace, Hampshire, Berkshire, Pietrain, Tibetan, Wuzhishan, Jinhua, Meishan, Rongchang | Deletion | 23 150 | 23 151 | CA | Intron | Sense |
| 3 | Tibetan, Wuzhishan, Jinhua, Meishan, Rongchang | Insertion | 46 898 | 46 899 | CT | Intron | Sense |
| 4 | Bamei, Tibetan, Wuzhishan, Jinhua, Meishan, Rongchang, Cross-bred, Goettingen | Deletion | 54 931 | 54 936 | CTGTGG | Intron | Sense |
| 5 | Tibetan, Wuzhishan, Jinhua, Meishan, Rongchang | Insertion | 80 024 | 80 025 | AGAA | Intron | Sense |
| 6 | Tibetan, Wuzhishan, Jinhua, Meishan, Rongchang, Cross-bred | Deletion | 87 485 | 87 487 | GAG | Intron | Sense |
| 7 | Bamei, Tibetan, Wuzhishan, Jinhua, Meishan, Rongchang | Insertion | 88 134 | 88 135 | AGCCCTAG | Intron | Sense |
| 8 | Tibetan, Wuzhishan, Jinhua, Meishan, Rongchang | Deletion | 90 199 | 90 201 | CAG | Intron | Sense |
| 9 | Landrace, Hampshire, Berkshire, Pietrain, Tibetan, Wuzhishan, Jinhua, Meishan, Rongchang | Insertion | 116 920 | 116 921 | CT | 3′Flank | Sense |
| A | Tibetan, Wuzhishan, Meishan | Insertion | 16 032 | 16 033 | ERV16/LTR | Intron | Antisense |
| B | Bamei, Tibetan, Wuzhishan, Jinhua, Meishan, Rongchang, Cross-bred | Deletion | 50 974 | 51 268 | SINEA1/SINE | Intron | Sense |
| C | Tibetan, Wuzhishan | Deletion | 65 417 | 65 541 | SINEA1/SINE | Intron | Antisense |
| D | Tibetan, Wuzhishan, Jinhua, Meishan, Rongchang, Cross-bred | Deletion | 68 188 | 68 474 | L1D1/LINE | Intron | Sense |
在8个不同猪品种(杜洛克、长白猪、大白猪、野猪、梅山猪、藏猪、二花脸、巴马香猪)的24个个体中对ktn1基因上的一个SINEA1转座子插入位点(表 4中的B位点)的多态性进行检测,该位点在8个品种的24个个体中呈现出良好的多态现象,且检测结果清晰易判断(图 2)。此

|
| 图 2 ktn1-STIP位点在8个品种中多态检测结果 Fig. 2 Polymorphic detection results of the ktn1-STIP loci in 8 porcine breeds. |
| |
位点在ktn1基因上的位置以红色箭头标注在图 1中,其插入方向与ktn1基因相反,位于ktn1基因的内含子区域(图 1),未影响ktn1基因的编码区。由电泳图(图 2)可以看出,该分子标记在猪群体中有3种基因型,SINE插入纯合基因型(SINE+/+),为单条大带676 bp,SINE无插入纯合基因型(SINE-/-),为单条小带381 bp,SINE插入杂合基因型(SINE+/-),为两条带676 bp和381 bp。
2.6 ktn1-STIP与断奶窝重呈显著性相关将ktn1-STIP在62个大白个体、48个长白猪个体、44个杜洛克个体、36个巴马香猪个体和184个苏姜猪个体中进行多态检测。由图 3可以看出,该位点的SINE在本研究群体中均呈现出插入多态现象。对插入基因型进行统计,结果见表 5,大白猪和长白猪的基因型SINE+/+的频率高于杜洛克猪、苏姜猪和巴马猪。而杜洛克猪和巴马香猪中SINE–/–的个体明显多于另外两种基因型。
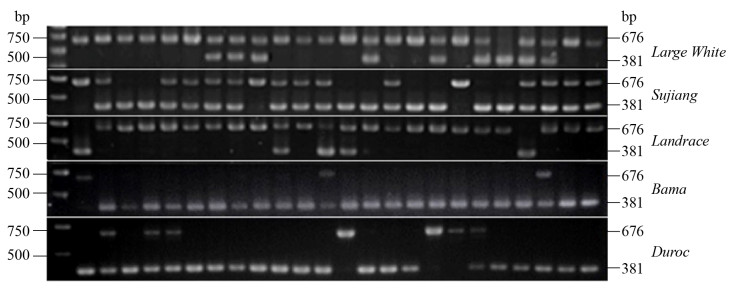
|
| 图 3 ktn1-STIP位点大白猪、苏姜猪、长白猪、巴马香猪和杜洛克猪群体中多态性检测代表电泳图。 Fig. 3 Representative electrophoresis figure of ktn1-STIP polymorphism in Large White, Sujiang, Landrace, Bama and Duroc pigs. |
| |
| Pig breed | Number | Genotype frequency (%) | Allele frequency (%) | Hardy-Weinberg/χ2 | ||||
| SINE+/+ | SINE+/- | SINE-/- | SINE+ | SINE- | ||||
| LargeWhite | 48 | 91.30 | 6.52 | 2.17 | 94.57 | 5.43 | 6.15 | |
| Landrace | 48 | 77.08 | 16.67 | 6.25 | 85.42 | 14.58 | 5.26 | |
| Duroc | 44 | 9.09 | 22.73 | 68.18 | 20.46 | 79.54 | 4.00 | |
| Sujiang | 184 | 11.41 | 59.78 | 28.80 | 41.30 | 58.70 | 9.98 | |
| Bama | 36 | 5.56 | 16.67 | 77.78 | 13.90 | 86.10 | 3.31 | |
| Note: χ0.052(df=1)=3.84, χ0.012(df=1)=6.63. | ||||||||
选取ktn1-STIP在苏姜猪猪群体中(184)进行多态性检测,并将每个个体的生长繁殖性状与ktn1-STIP位点的多态性进行关联分析,结果显示,在苏姜猪中断奶窝重与ktn1基因中是否有SINE插入显著相关(P < 0.05),无插入个体(SINE-/-)断奶窝重((64.20±10.6) kg)比纯合有插入个体(SINE+/+) ((74.14±9.0) kg)和杂合有插入个体(SINE+/-) ((69.71±7.7) kg)轻。产活仔数、断奶仔猪数和初生窝重与ktn1基因中ktn1-STIP位点是否有SINE插入无显著相关性(表 6)。
| Genotype | Number | Number of born alive | Number of weaned piglets | Primary litter weight | Waning litter weight |
| SINE+/+ | 25 | 10.45±1.6a | 10.60±1.0a | 14.25±1.5a | 74.14±9.0a |
| SINE+/- | 106 | 10.64±2.0a | 9.59±2.1a | 13.96±2.6a | 69.71±7.7a |
| SINE-/- | 53 | 10.65±2.0a | 9.50±1.6a | 14.11±2.3a | 64.20±10.6b |
| Note: identical letters in the same column indicates no significant difference; different letters indicate significant differences (P < 0.05). | |||||
尽管SINE仅占猪基因组的8%左右,但它们广泛分布在整个基因组中[13-14],已经有研究证明一些SINE插入与猪生长、繁殖和胴体性状显著相关[15-16]。SINE可以在多个水平上影响基因活性或功能(基因结构、转录和翻译等)。在某些情况下SINE可作为顺式调控元件参与基因表达调控,如多腺苷酸化和选择性剪接的过程[17-18]。此外,SINE RNA还可以作为反式调控因子调节基因的转录和翻译[17, 19]。本研究通过对ktn1 (基因和侧翼区)序列进行转座子注释,发现仅在杜洛克参考基因组中的ktn1基因及其侧翼区中就含有77个转座子片段,绝大部分为SINE类反转录转座子,有49个插入,这说明,转座子对ktn1的结构和序列组成具有重要的影响,值得进一步探究。进一步对14个不同猪品种/品系的基因组中的ktn1 (基因和侧翼区)序列进行比对,发现了9个小结构变异及4个大结构变异,且大结构变异全部由转座子插入引起,其中2个为SINEA1插入引起。这也进一步验证了之前的研究,反转录转座子是基因组的主要组成成分,是基因变异的重要来源[1, 20-22]。
年轻转座子在不同品种中,存在丰富的插入多态性[1],因此通过使用不同品种和个体样本对每个转座子位点引起的结构变异进行多态检测,将会全面揭示转座子对猪ktn1基因结构突变的影响。本研究中,我们在ktn1基因的内含子区检测到2个SINEA1插入引起的结构变异,对其中一个进行插入多态性检测,结果表明,此位点在多个品种中存在插入多态性(图 2),且在大白猪、长白猪中SINEA1的插入频率(SINE+)高于无插入频率(SINE-),而杜洛克、苏姜猪和巴马香猪中呈现相反的现象。并且引入猪种大白猪、长白猪和杜洛克猪和培育猪种苏姜猪偏离哈代温伯格平衡,巴马香猪等中国地方猪种符合哈代温伯格平衡(表 5),说明被检测的巴马香猪群体符合群体基因遗传平衡,而杜洛克、大白猪、长白猪是经过人工选育的西方商品猪,培育猪种苏姜猪的亲本为杜洛克、姜曲海和枫泾,在世代选育过程中造成群体偏离遗传平衡。
仔猪在哺乳期体重的增加依赖于自身生长发育潜力和母猪的哺乳能力及营养、环境因素等。目前研究表明SINE转座子对基因具有多种调控功能。将184个苏姜猪个体的生长繁殖性状与SINE-ktn1位点的转座子插入多态性进行关联分析,显示SINE转座子的插入可以显著增加仔猪断奶窝重,提示猪ktn1基因中SINE的插入可能影响了ktn1基因的表达,通过一定信号通路,进而影响仔猪生长发育,或者通过影响母猪泌乳生理机能,间接影响仔猪生长发育,其具体分子机制值得深入探究。
4 结论本研究通过对14条ktn1 (基因及其侧翼区)序列的多序列比对和转座子注释,全面解析转座子对ktn1的影响。最终在参考基因组的ktn1基因及其侧翼区中鉴定到77个转座子片段,其中绝大部分(98.70%)为SINE类反转录转座子,与基因组中大多数转座子的分布情况一致。并鉴定到9个小结构变异和4个由转座子引起的大结构变异,证明了转座子是基因变异的重要来源。进一步检测了其中一个SINEA1转座子的插入多态性,并在苏姜猪群体中与相关性状进行关联分析,结果表明,无插入个体(SINE-/-)的断奶窝重较纯合插入个体(SINE+/+)和杂合插入个体(SINE+/-)轻(P < 0.05)。本研究表明基于转座子插入多态研发分子标记具有可行性,同时此分子标记在分子辅助育种中具有很强的应用潜力。
| [1] |
Chen C, Wang W, Wang XY, et al. Retrotransposons evolution and impact on lncRNA and protein coding genes in pigs. Mobile DNA, 2019, 10: 19. |
| [2] |
G ao, B, Shen D, Xue SL, et al. The contribution of transposable elements to size variations between four teleost genomes. Mobile DNA, 2016, 7: 4. |
| [3] |
Hancks DC, Kazazian HH JR. Roles for retrotransposon insertions in human disease. Mobile DNA, 2016, 7: 9. |
| [4] |
Gray MM, Sutter NB, Ostrander EA, et al. The IGF1 small dog haplotype is derived from Middle Eastern grey wolves. BMC Biol, 2010, 8: 16. |
| [5] |
Clark LA, Wahl JM, Rees CA, et al. Retrotransposon insertion in SILV is responsible for merle patterning of the domestic dog. Proc Natl Acad Sci USA, 2006, 103(5): 1376-1381. |
| [6] |
Murphy SC, Evans JM, Tsai KL, et al. Length variations within the Merle retrotransposon of canine PMEL:Correlating genotype with phenotype. Mobile DNA, 2018, 9: 26. DOI:10.1186/s13100-018-0131-6 |
| [7] |
Wang ZP, Qu LJ, Yao JF, et al. An EAV-HP insertion in 5′ flanking region of SLCO1B3 causes blue eggshell in the chicken. PLoS Genet, 2013, 9(1): e1003183. |
| [8] |
Giuffra E, Törnsten A, Marklund S, et al. A large duplication associated with dominant white color in pigs originated by homologous recombination between LINE elements flanking KIT. Mamm Genome, 2002, 13(10): 569-577. |
| [9] |
Sironen A, Uimari P, Iso-Touru T, et al. L1 insertion within SPEF2 gene is associated with increased litter size in the Finnish Yorkshire population. J Anim Breed Genet, 2012, 129(2): 92-97. DOI:10.1111/j.1439-0388.2011.00977.x |
| [10] |
Toyoshima I, Yu H, Steuer ER, et al. Kinectin, a major kinesin-binding protein on ER. J Cell Biol, 1992, 118(5): 1121-1131. |
| [11] |
Tian WX, Li JK, Qin P, et al.Differentially expressed genes in broiler tibia cartilage by gene chip//Chinese Association of Animal Science and Veterinary Medicine 2010 Annual Conference.Chang Chun, 2010(in Chinese). 田文霞, 李家奎, 覃平, 等.基因芯片筛选肉鸡胫骨软骨发育不良差异表达基因//中国畜牧兽医学会2010学术年会暨第二届中国兽医临床大会论文集.长春: 中国畜牧兽医学会, 2010. |
| [12] |
Zhang Y, Gao L, Ma SD, et al. MALAT1-KTN1-EGFR regulatory axis promotes the development of cutaneous squamous cell carcinoma. Cell Death Differ, 2019, 25: 1. |
| [13] |
Groenen MAM, Archibald AL, Uenishi H, et al. Analyses of pig genomes provide insight into porcine demography and evolution. Nature, 2012, 491(7424): 393-398. |
| [14] |
Zhang XY, Wang DX, Han Y, et al. Altered imprinted gene expression and methylation patterns in mid-gestation aborted cloned porcine fetuses and placentas. J Assist Reprod Genet, 2014, 31(11): 1511-1517. |
| [15] |
Han SH, Shin KY, Lee SS, et al. Porcine SPP1 gene polymorphism association with phenotypic traits in the Landrace × Jeju (Korea) black pig F2 population. Mol Biol Rep, 2012, 39(7): 7705-7709. DOI:10.1007/s11033-012-1606-z |
| [16] |
Magotra A, Naskar S, Das B, et al. A comparative study of SINE insertion together with a mutation in the first intron of follicle stimulating hormone beta gene in indigenous pigs of India. Mol Biol Rep, 2015, 42(2): 465-470. |
| [17] |
Kramerov DA, Vassetzky NS. Origin and evolution of SINEs in eukaryotic genomes. Heredity, 2011, 107(6): 487-495. |
| [18] |
Lunyak VV, Atallah M. Genomic relationship between SINE retrotransposons, Pol Ⅲ-Pol Ⅱ transcription, and chromatin organization:the journey from junk to jewel. Biochem Cell Biol, 2011, 89(5): 495-504. DOI:10.1139/o11-046 |
| [19] |
Longo MS, Brown JD, Zhang C, et al. Identification of a recently active mammalian SINE derived from ribosomal RNA. Genome Biol Evol, 2015, 7(3): 775-788. |
| [20] |
Callinan PA, Wang JX, Herke SW, et al. Alu retrotransposition-mediated deletion. J Mol Biol, 2005, 348(4): 791-800. |
| [21] |
Han KD, Sen SK, Wang JX, et al. Genomic rearrangements by LINE-1 insertion-mediated deletion in the human and chimpanzee lineages. Nucleic Acids Res, 2005, 33(13): 4040-4052. DOI:10.1093/nar/gki718 |
| [22] |
Salem AH, Kilroy GE, Watkins WS, et al. Recently integrated Alu elements and human genomic diversity. Mol Biol Evol, 2003, 20(8): 1349-1361. |
 2020, Vol. 36
2020, Vol. 36




